基于人工染色體的DNA信息存儲前沿進展
楊洋,樊春海,2
(1上海交通大學醫(yī)學院附屬仁濟醫(yī)院,分子醫(yī)學研究院,上海 200127;2上海交通大學化學化工學院,上海 200240)
近二十年來人類在信息技術方面取得的進步,尤其是互聯(lián)網和移動互聯(lián)技術的發(fā)展,帶來了前所未有的數(shù)據(jù)爆炸和信息存儲危機。在科研與社會服務層面,更高分辨率的天文觀測、醫(yī)學成像以及交通監(jiān)控正在不斷產生大量的圖像視頻類數(shù)據(jù)。社交網絡則是另一個重要場景,除了互動信息之外,個人化的照片與視頻創(chuàng)造和傳播呈現(xiàn)加速趨勢,預計到2030年全球網民比例將從2017年的51%增長至近乎全覆蓋,屆時數(shù)據(jù)產生的速度也將翻倍。全球數(shù)字化的這一發(fā)展趨勢使得數(shù)據(jù)量快速增長,據(jù)國際數(shù)據(jù)公司統(tǒng)計,2018年人類產生的數(shù)據(jù)總量達到了33 ZB(1 ZB≈109TB),而到2025年這一數(shù)字將增長為驚人的175 ZB。面對快速增長的海量數(shù)據(jù),基于磁、光、電等的傳統(tǒng)介質的存儲技術面臨功耗、體積以及使用壽命等限制,而DNA存儲提供了應對數(shù)據(jù)存儲發(fā)展挑戰(zhàn)的新契機。
DNA因其極高的信息密度和非凡的穩(wěn)定性成為存儲系統(tǒng)的有力候選。首先,DNA的信息密度非常大,自然界中大部分生物的全部生命信息都存儲在DNA中,以人類為例,人體大約可以產生40萬種蛋白質,而它們需要在不同的發(fā)育階段,以不同的數(shù)量和速度在不同的細胞中被表達、使用和代謝。所有這些蛋白及其程序控制相關的信息都被存儲于僅僅23對染色體上。一個細胞中染色體所折疊的DNA全部拉直并連接雖然可以長達3 m,但其重量卻僅有10-11g。一方面理論上,1 g DNA可以存儲455 EB(1 EB≈106TB)的數(shù)據(jù)量,據(jù)此計算1億部高清電影如果存儲于DNA中,這些DNA只需要占據(jù)一塊橡皮的大小,而利用2T的硬盤來存儲的話則需要10萬個硬盤。另一方面,DNA存儲數(shù)據(jù)具有極高的穩(wěn)定性,不但動植物化石中保存的DNA可以歷經千年保持可讀性,提純的DNA經過濃縮與干燥,可在惰性氣體保護下保存至少百年的時間。而如果把攜載外部信息的DNA借由微生物進行保存,其拷貝數(shù)可以以指數(shù)形式大量擴增并代代相傳,相比承載與維持服務器機組工作所需的巨大機房和空調系統(tǒng),利用微生物攜載DNA用于信息存儲也是極其綠色節(jié)能的選擇。因此,越來越多的國家已經開始將基于DNA的數(shù)據(jù)存儲列為戰(zhàn)略層面的發(fā)展方向,例如2021年1月,美國半導體產業(yè)協(xié)會(SIA)發(fā)布的《半導體10年計劃》,將DNA數(shù)據(jù)存儲列為未來海量數(shù)據(jù)存儲的重要選項。我國科技部也早在2018年即開展了基于DNA的信息存儲相關項目部署。在2021年3月份通過的國家“十四五”規(guī)劃綱要的第九章中更明確提出“推動生物技術和信息技術融合創(chuàng)新”的目標,為大力發(fā)展合成生物學及DNA信息存儲技術提供了政策引導。本文評述作者長期從事核酸分析、納米技術以及DNA編碼與計算相關的交叉學科研究并取得了顯著成果,將在下文中簡要綜述DNA信息存儲的歷史發(fā)展,對近期基于酵母的人工染色體構建與DNA信息存儲的進展工作稍作點評。
從1960年代蘇聯(lián)物理學家Mikhail Samiolvich Neiman首次提出關于DNA作為信息存儲物質的設想[1],到1986年麻省理工學院的研究員Joe Davis將12個字母的詞組轉換為28個堿基對的DNA序列并插入大腸桿菌(E.coli)細胞中[2],DNA作為存儲材料的潛力早已為人所知。但存儲數(shù)據(jù)量和檢索讀出技術距離實用還有很遠的距離。自1980年代以來不斷進步的DNA固相合成技術快速發(fā)展,為大量數(shù)據(jù)的寫入提供了基礎,與此同時DNA測序技術的迭代升級使得信息的高效讀取成為可能。近年來飛速發(fā)展的新一代測序技術(next generation sequencing,NGS)[3]提供了同時平行測序百萬條短DNA序列的平臺,利用NGS技術,一個人的基因組可以在一天內測序拼接完成,而傳統(tǒng)的桑格爾(Sanger)測序法[4]在一臺測序儀上完成這一工作則需要十年的時間。伴隨著整個分子生物學的發(fā)展,我們終于可以編寫、存儲、檢索和讀取大量的DNA序列。同時,以DNA為媒介存儲信息的工作也不斷涌現(xiàn),存儲的數(shù)據(jù)量和數(shù)據(jù)類型都不斷增加。1999年,紐約大學Risca等[5]利用69個堿基對成功編碼和檢索了含有22個字母、數(shù)字和字符的消息。2012年,哈佛大學Church課題組在《科學》雜志發(fā)表論文,詳細介紹了如何使用DNA來存儲一本含由53 426個詞的書[6]。在存儲算法研究方面,簡單的二進制向四進制的轉換并不能最大程度地利用DNA的存儲能力,隨機產生的特殊的序列組成(例如連續(xù)的G序列或C序列)有可能給合成與測序帶來錯誤概率的積累,因檢索與糾錯需求帶來的信息冗余又會使得存儲密度大打折扣。因此,信息的編碼算法起到舉足輕重的作用。2017年Yaniv Erlich和Dina Zielinski開發(fā)了一種新型的“噴泉“碼算法,可以將凈信息密度提高到1.57 bit/bp,將DNA的實際容量提高到86%[7]。美國微軟公司(Microsoft)在DNA存儲領域一直推進技術革新,2016年他們與華盛頓大學合作發(fā)表的一篇有關DNA數(shù)據(jù)存儲前景的文章描述了如何利用合成的DNA編寫和檢索三幅圖像[8];在2019年,他們進一步開發(fā)了一套全自動的DNA存儲與讀取設備[9];同年,他們又利用納米孔技術實現(xiàn)了1.67 MB的信息讀取[10]。從這一領域快速增長的文章(2018年、2019年每年在PubMed上統(tǒng)計DNA data/information storage的相關文章超過1000篇)和專利數(shù)量(WIPO關于DNA數(shù)據(jù)存儲的國際專利申請超過1 700余件)可以判斷,國際上關于DNA信息存儲的競爭在未來十幾年中還將持續(xù)白熱化。
2021年2月12日,天津大學元英進教授團隊帶領的跨學科團隊于National Science Review上在線發(fā)表了以“An artificial chromosome for data storage”為題的研究論文(天津大學微電子學院青年教師陳為剛副教授、化工學院博士研究生韓明哲以及助理研究員周見庭為論文共同第一作者)。該工作中,研究者從頭編碼設計合成了一條長度為254 886 bp,專用于數(shù)據(jù)存儲的酵母人工染色體,存儲了兩張圖片及一段視頻,編碼覆蓋率超過95%,并實現(xiàn)了數(shù)據(jù)的穩(wěn)定復制與快速可靠讀出(圖1)。
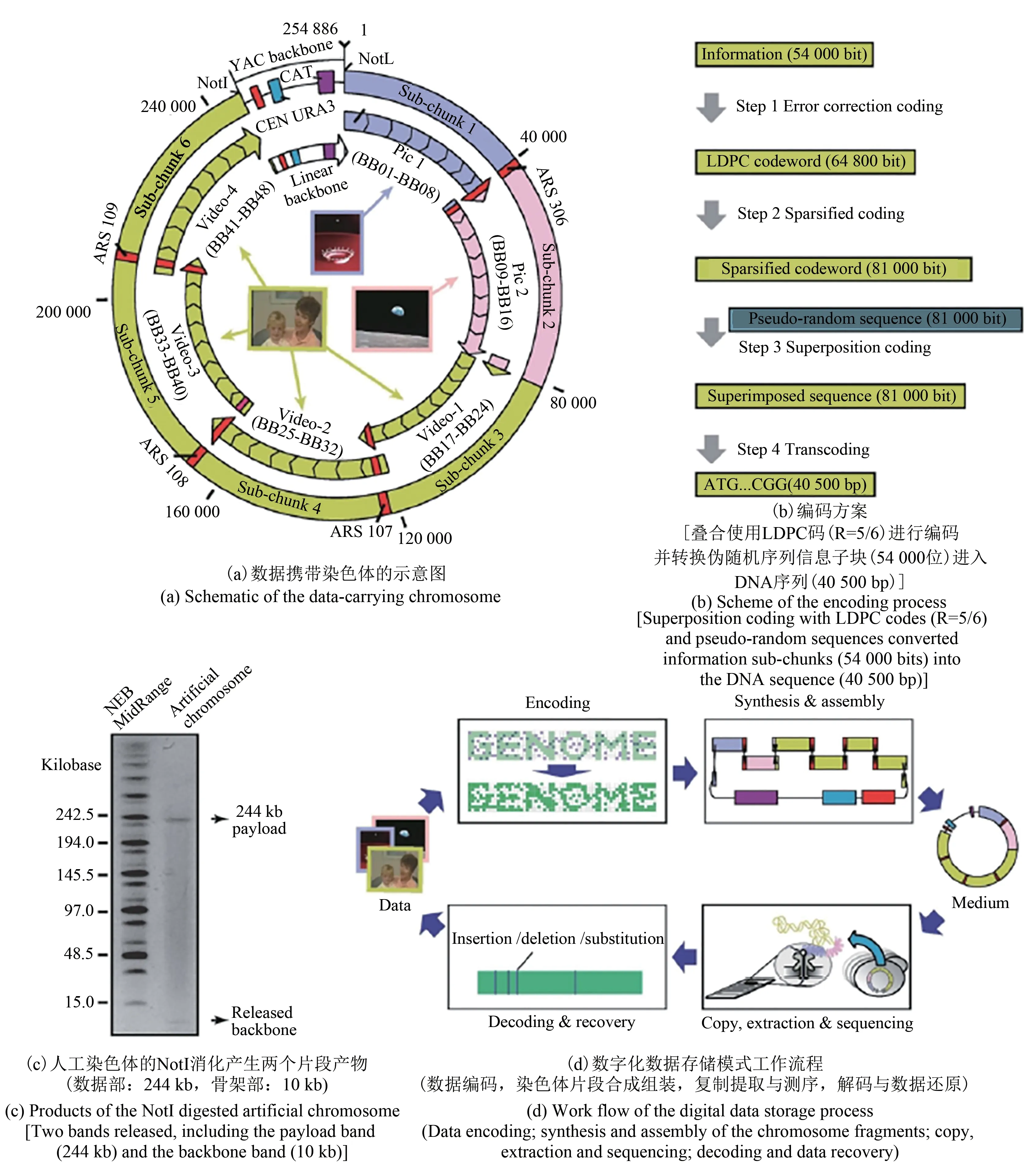
圖1 設計與組裝攜載信息的人工染色體Fig.1 Design and assembly of the artificial chromosome that carriesdigital information
在存儲環(huán)節(jié),一方面該研究借助疊加偽隨機序列應對三代測序的插入/刪除(insertion/deletion)錯誤,采用現(xiàn)代通信中常用的低密度奇偶校驗(low-density parity-check,LDPC)碼糾正替代錯誤,實現(xiàn)了在高達10%錯誤率時的數(shù)據(jù)可靠恢復。另一方面,該染色體設計中,插入一定數(shù)量的酵母自主復制序列(autonomously replicating sequence,ARS),提升了染色體的穩(wěn)定性,保障了其高效組裝和穩(wěn)定復制(>100代復制仍可讀出)。該存儲模式中,數(shù)據(jù)邏輯密度(包含載體)為1.19 bit/bp,與目前文獻中指標最高的四進制編碼DNA噴泉方案相當[8]。
在數(shù)據(jù)讀取環(huán)節(jié),該工作利用三代納米孔測序技術在大約10 min時間內獲得足夠的原始讀段后,結合自主設計的生物信息學與糾錯譯碼混合流程,便可實現(xiàn)數(shù)據(jù)可靠恢復,所需測序覆蓋度僅為16.8×。相比純粹利用合成DNA存儲再利用聚合酶鏈反應(PCR)技術進行備份的傳統(tǒng)做法,利用酵母菌存儲信息可以實現(xiàn)一次寫入,多次讀出,體現(xiàn)了極好的低成本與便攜性優(yōu)勢。
這一最新研究成果為DNA存儲技術提供了新穎的角度與方案,可以期待的是,通過進一步降低合成成本和構建多條人工染色體,人們能夠在酵母菌中存儲更多數(shù)據(jù)。隨著合成生物學領域的更多技術進步,利用DNA和生命系統(tǒng)存儲與利用信息將會成為大勢所趨,然而相比于以硅基硬盤為基礎的電子化信息存儲,核酸/微生物硬盤的廣泛應用還有賴于存儲密度的進一步提高,合成組裝操作的進一步簡化,存、檢、讀方案的全面整合以及全流程的自動化集成。以DNA存儲為核心的上述全鏈條的技術研發(fā)有望引領多學科的交互發(fā)展與共同進步。

