橡膠樹‘熱研7-33-97’死皮相關研究的qRT-PCR內參基因篩選
盧亞莉 張世鑫 楊署光 田維敏 史敏晶
摘 ?要:在橡膠樹死皮以及死皮康復相關研究中,為獲得可靠的基因表達結果,篩選合適的內參基因顯得尤為重要。本研究以‘熱研7-33-97健康樹、三級死皮樹和死皮康復樹為試驗材料,收集其中的膠乳,采用qRT-PCR技術,結合GeNorm、NormFinder和BestKeeper三種內參篩選軟件,綜合分析了20個候選內參基因在不同膠乳樣品中的表達情況。結果表明:eif2、ACT7a、UBC2b在健康樹中較穩定;UBC3、eifAb、eifAa、ADF4、UBC4、UBC2b、eif2、RH2b、ACT7b在三級死皮樹中較穩定;UBC2b、eif2和ROC3在三級死皮恢復樹中較穩定。選擇膠乳代謝相關基因HbDXS1、HbHMGS2和HbNIN3對候選的內參基因進行驗證,篩選出本研究中適合的一組穩定可靠的內參基因是eif2和UBC2b,其中ejf2是最佳內參基因,18S不適合作為本研究的內參基因。
關鍵詞:橡膠樹;死皮相關;內參基因;qRT-PCR
中圖分類號:S794.1 ? ? ?文獻標識碼:A
Abstract: In order to obtain reliable gene expression results for TPD-related study of rubber tree, the selection of suita-ble reference genes is very important. In the present study, using the latex from the healthy rubber tree, the third grade tapping panel dryness (TPD) tree and the TPD-recovering tree of ‘Reyan7-33-97 as the experimental materials, the expression stability of 20 candidate reference genes were evaluated by using quantitative real-time PCR (qTR-PCR) and?softwares of geNorm, NormFinder and BestKeeper. The results showed that, reference genes eif2、ACT7a and UBC2bwere the top three stable genes in the healthy tree; UBC3、eifAb、eifAa、ADF4、UBC4、UBC2b、eif2、RH2b and ACT7b?were all stable genes in the third grade TPD tree; UBC2b、eif2 and ROC3 were stable genes for the TPD-recovering?rubber tree. Based on the different stable reference genes, relative expressions of HbDXS1, HbHMGS2 and HbNIN3 genes were analyzed respectively. The results showed that eif2 and UBC2b were a couple of stable reference genes for TPD-related study; the eif2 was the most stable internal reference gene; 18S gene was not suitable as reference gene for qTR-PCR in this study.
Keywords: Hevea brasiliensis Muell. Arg.; TPD-related; reference genes; qTR-PCR
DOI: 10.3969/j.issn.1000-2561.2021.05.012
橡膠樹死皮病,即割面干涸病(tapping panel dryness,TPD),是在橡膠樹的割線上出現局部或全部不排膠的癥狀,在世界各國的植膠園中普遍發生,嚴重影響天然橡膠產量,縮短橡膠樹的經濟壽命,給橡膠產業帶來嚴重危害。我國橡膠樹死皮病發生情況尤其嚴重,成為當前天然橡膠產業的主要限制因子之一[1],因此,探索死皮病的發生機理并尋求解決辦法已成為當務之急。利用分子生物學手段,研究橡膠樹死皮病發生和康復的分子機理已成為近年來的研究熱點,其中,對差異表達基因的篩選則是進行基因功能研究和進行分子調控的基礎。
基因表達分析的手段眾多,單個RNA的穩態水平可以用不依賴PCR的方法如Northern blotting[2]和原位雜交[3]等來衡量,但這些技術耗時長,RNA用量大,實驗難度較大,并且重現性較差。實時熒光定量PCR(qRT-PCR)的技術由于具有特異性強、靈敏度高、精確度高、重現性好、通量較高、動態范圍較寬、無需后PCR處理[4-7]等優點而成為目前最受歡迎的mRNAs檢測方法。但qRT-PCR的準確性受到樣品間起始RNA的質量和數量、cDNA合成效率以及PCR擴增效率等諸多因素的影響[8],控制和減少誤差的一個較好的方法就是選擇合適的內參基因。理想的內參基因的表達水平應該在不同的生理與環境條件下穩定不變[9],但已有研究表明,生物體內并沒有真正的、完全穩定的內參基因。目前,在植物qRT-PCR分析中普遍使用的內參基因有Actin、18S等,但已有研究發現一種植物中的某個較為理想的內參基因并不一定適合在其他植物中使用。因此,為獲得準確的基因表達數據,每進行一項研究實驗,篩選出試驗條件下特異性的內參基因對獲得準確的基因表達結果至關重要[6, 10]。一般用于植物和動物內參基因分析的常用軟件是GeNorm、NormFinder和BestKeeper[11-12],借用這些軟件,篩選研究所需的合適的內參基因是可以實現的。
大量研究表明,常用的內參基因18S rRNA、HbActin在橡膠樹中不具有普適性,不同的實驗系統最好需要單獨篩選內參基因。目前,關于橡膠樹內參基因的報道主要是針對乳管分化、膠乳再生、排膠、不同組織、不同激素處理等展開,橡膠樹死皮發生,尤其是死皮康復研究中,缺乏相應的內參基因的篩選。為準確研究死皮以及康復這一過程中相關基因的表達,對該過程中的內參基因進行分析,篩選合適的內參基因顯得尤為重要。本研究選取18S rRNA(簡稱18S)、ACT7a、ACT7b、ADF、ADF4、CYP2、eifAa、eifAb、eif2、eif3、UBC2a、UBC2b、UBC1、UBC3、UBC4、ROC3、PTP、RH2a、RH2b、FP共20個基因作為健康橡膠樹、死皮樹和施用死皮康復營養劑后的死皮恢復植株相關研究中的候選內參基因,通過qRT-PCR技術,并借助GeNorm、NormFinder和BestKeeper軟件對20個內參基因在‘熱研7-33-97不同樣品中的表達穩定性進行評價,以期篩選出合適的內參基因,為橡膠樹死皮以及康復相關研究中的基因表達分析奠定基礎。
1 ?材料與方法
1.1 ?材料
巴西橡膠樹(Hevea brasiliensis Muell. Arg.)無性系‘熱研7-33-97定植于中國熱帶農業科學院海南儋州兩院試驗場。選取長勢大致相同的‘熱研7-33-97的健康割膠樹、三級死皮樹和施用死皮康復營養劑后的死皮恢復樹作為試驗對象,每種類型橡膠樹至少3株重復。采用3 d一刀,1/2 S的割膠制度,按照以下方法分別采集不同的膠乳樣品:(1)健康樹距離地面1 m處連續割膠5刀,收集每一刀的膠乳,分別編號為0(CK)、1、2、3、4刀;(2)三級死皮樹在正常割膠的情況下,針刺收集不排膠部位、點狀排膠部位、連續排膠部位水囊皮中的膠乳;移液器收集點狀排膠部位和連續排膠部位割線上的膠乳;(3)死皮后經施用康復營養劑恢復的橡膠樹,距離地面1 m處連續割膠5刀,收集每一刀的膠乳,分別編號為0(CK)、1、2、3、4刀。收集的膠乳直接加入到裂解液SL中,混勻后,冰上保存,帶回實驗室后立即提取總RNA,于–80 ℃貯存備用。
1.2 ?方法
1.2.1 ?膠乳總RNA提取與cDNA合成 ?橡膠樹的膠乳總RNA的提取根據RNAprep Pure Plant Kit多糖多酚植物總RNA提取試劑盒(天根生化科技北京有限公司)操作說明進行。通過瓊脂糖凝膠電泳和紫外分光光度計檢測提取的總RNA的完整度和純度。按照RevertAidTM First Strand cDNA Synthesis Kit(Thermo Fisher Scientific Inc.,USA)試劑盒說明進行cDNA的反轉錄合成,合成產物于–20 ℃冰箱保存備用。
1.2.2 ?內參基因的表達分析 ?根據已報道的橡膠樹內參基因,選擇CYP2、18S、ROC3、FP、PTP、ACT7a、ACT7b、ADF、ADF4、RH2a、RH2b、eifAa、eifAb、eif2、eif3、UBC1、UBC2a、UBC2b、UBC3和UBC4共20個基因作為候選的內參基因,采用Real-time PCR方法,分析這些基因在不同健康狀況下橡膠樹的不同部位和不同刀次收集膠乳中的表達情況。所選內參基因的PCR引物來源于本研究團隊。
1.2.3 基因qRT-PCR分析 ?以cDNA為模板,分別對候選內參基因進行實時熒光定量PCR分析,反應平臺為CFX384 Real-Time PCR Detection (Bio-rad, USA)。反應體系為10 L體系,反應程序為:95 ℃預變性5 min;95 ℃變性10 s,60 ℃退火20 s,72 ℃延伸10 s,共40個循環。反應使用的試劑為2x Transstart Tip Green qPCR Super Mix。每個PCR反應進行3次技術重復。利用Bio-rad CFX manager 3.0軟件,采用2–ΔΔCT算法分析基因的相對表達量。
1.3 ?數據處理
采用Gern Norm和Norm Finder軟件對內參基因穩定性進行分析;Original 8.0軟件繪制內參基因穩定性圖;SPSS 19.0以及Excel 2007進行統計學分析;AI軟件進行圖形組合。以篩選出來的內參基因為參照,對所選的目的基因HbDXS1、HbHMGS2和HbNIN3的表達水平進行定量分析,驗證內參基因的有效性。
2 ?結果與分析
2.1 ?巴西橡膠樹膠乳總RNA質量檢測
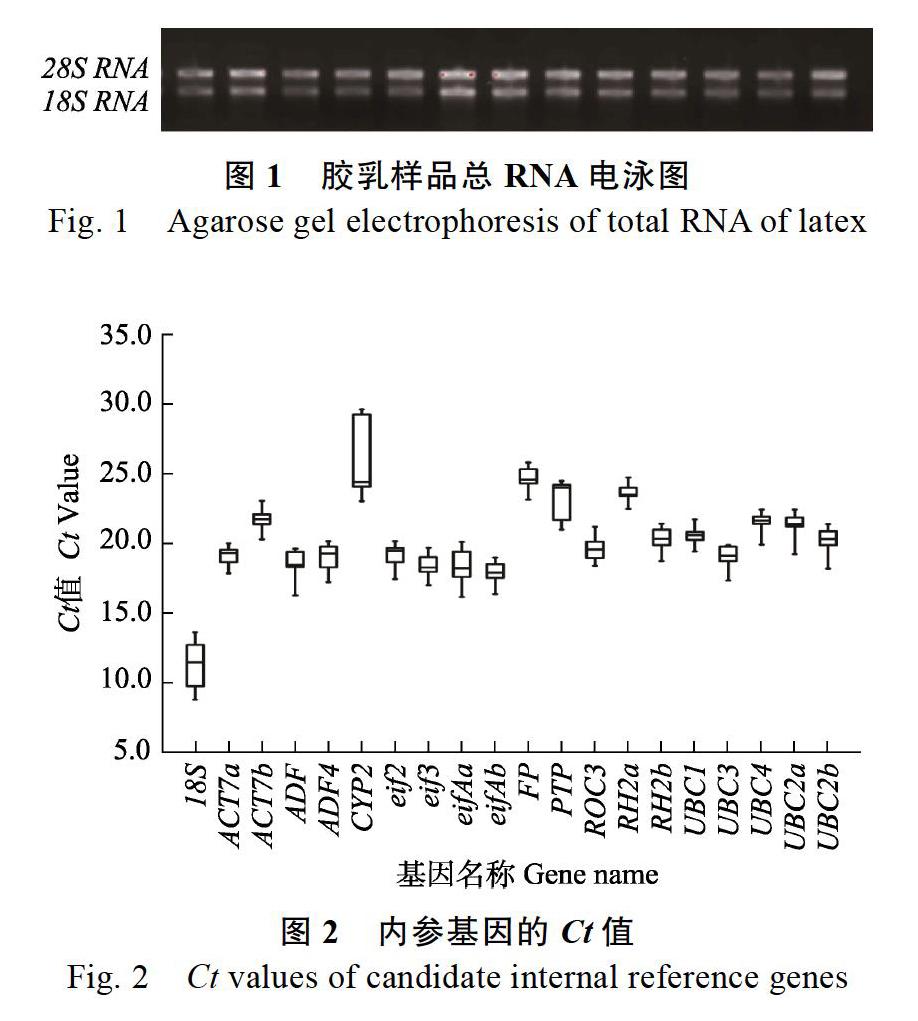
在qRT-PCR基因表達分析中,總RNA的質量直接影響后續實驗結果。提取的膠乳總RNA經瓊脂糖凝膠電泳檢測顯示,28S rRNA和18S rRNA條帶清晰完整(圖1),無降解現象,亮度比值約為2∶1。NanoDrop 2000(Thermo Fisher Scientific Inc., USA)測定OD260/OD280比值在1.8~2.1之間,出現單個峰值,表明RNA完整性好,純度較高,可用于后續研究。
2.2 ?內參基因Ct值分析
基因的Ct值大小與其表達量成反相關,Ct值越小,表達量越高,反之亦然[13]。對20個候選內參基因在不同膠乳樣品中得到的Ct值進行分析發現,這些基因的Ct值在8~32之間,范圍較廣,表明不同基因表達的豐度存在較大的差異。其中18S的Ct值最小,表明其基因的表達量最高;CYP2的Ct值最高,表明其表達量最低;其余基因的Ct值介于15~25之間,分布較集中,表達豐度居中(圖2)。
2.3 ?候選內參基因在不同樣品中表達穩定性分析與評價
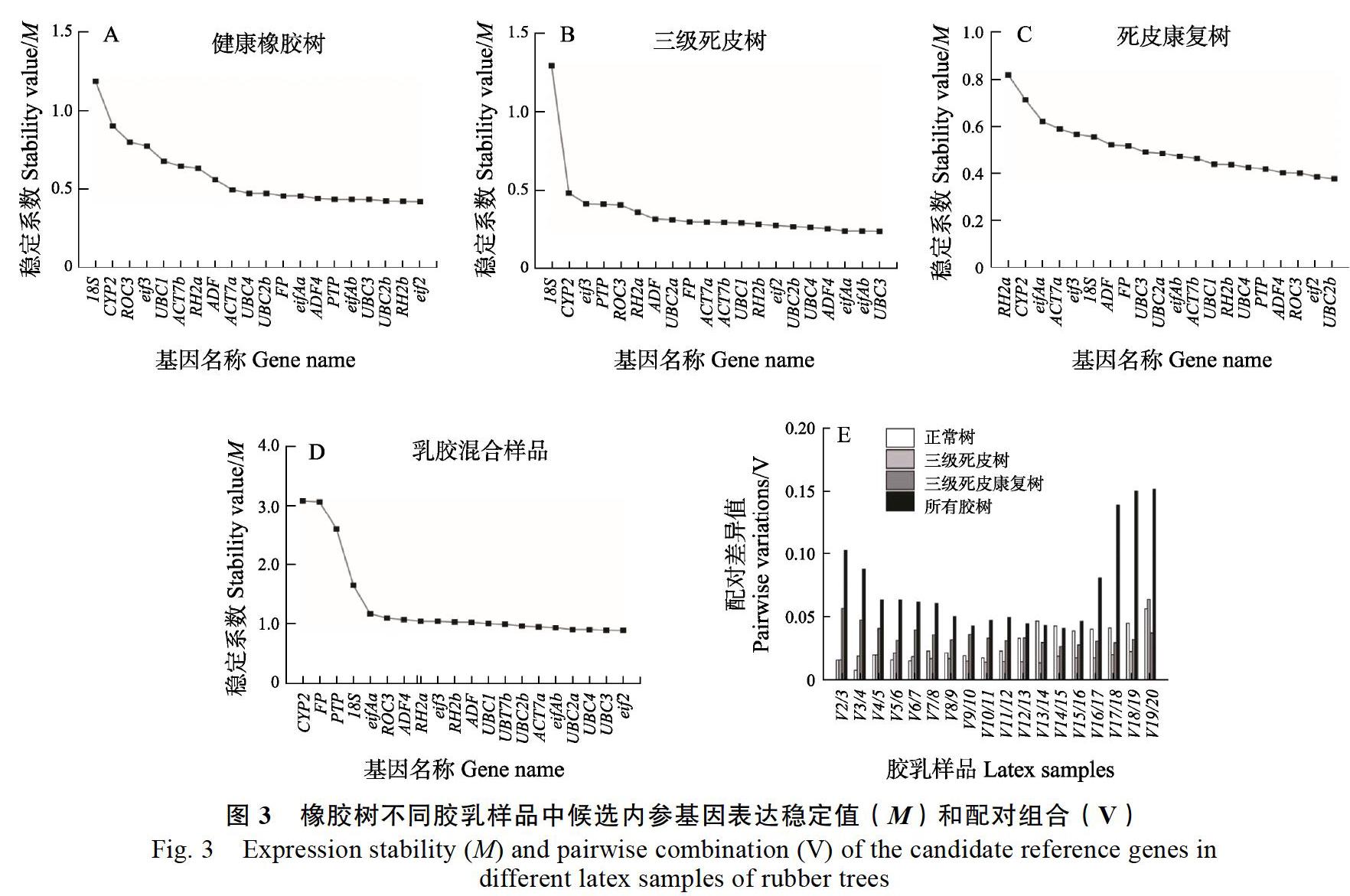
2.3.1 ?GeNorm分析 ?利用GeNorm軟件對候選內參基因的相對表達量進行統計分析,通過計算基因的表達穩定值(M)評價基因的表達穩定性,基因的M值越小,表達越穩定[14]。根據內參基因的穩定性從低到高進行排序作折線圖,除混合的膠乳樣品外,其他樣品中大部分基因的M值低于0.5,有較好的穩定性(圖3)。
在健康樹膠乳樣品中,穩定性由高到低排名前10的基因分別是eif2、RH2b、UBC2a、UBC3、eifAb、PTP、ADF4、eifAa、FP和UBC2b,并且其M值差異不大,均低于0.5。其中eif2、RH2b和UBC2a最穩定,M值約為0.4。最不穩定的3個內參基因為18S、CYP2、ROC3,M值高于0.75(圖3A)。
在三級死皮樹不同膠乳樣品中,穩定性由高到低排名前10的基因分別是UBC3、eifAb、eifAa、ADF4、UBC4、UBC2b、eif2、RH2b、UBC1、ACT7b,且M值差異不明顯,均低于0.4,都可以作為合適的內參基因。其中,UBC3、eifAb和eifAa最穩定,M值約為0.2。最不穩定的內參基因為18S,M值高于1.0;其次為CYP2、eif3,但M值也在0.5以內(圖3B)。
在死皮康復樹膠乳樣品中,穩定性由高到低排名前10位的基因分別是UBC2b、eif2、ROC3、ADF4、PTP、UBC4、RH2b、UBC1、ACT7b、eifAb,且M值均低于0.5。其中,UBC2b、eif2和 ROC3最穩定,M值約為0.4。最不穩定的3個內參基因為RH2a、CYP2、eifAa,M值均高于0.6(圖3C)。
在所有膠乳混合的樣品中,有4個基因的M值大于1.5,其他基因的M值也在1.0偏上,總體來看,表達穩定性略差。穩定性排名前10的基因分別是eif2、UBC3、UBC4、UBC2a、eifAb、ACT7a、UBC2b、ACT7b、UBC1、ADF。其中,eif2、UBC3和UBC4最穩定,M值約為0.9。最不穩定的3個內參基因為CYP2、FP和 PTP,M值高達2.5以上(圖3D)。之所以在這個組合中M值比較大,可能與膠乳樣品是來自狀態完全不同、差異明顯的膠乳樣品混合有關。
綜上所述,根據M值的高低,在全部樣品中均有良好穩定性的5個內參基因有eif2、UBC2b、RH2b、UBC3、UBC4,穩定性一直都較差的有18S和CYP2兩個基因。
GeNorm軟件還可以通過成對的變異值(Vn/Vn+1)來確定標準化所需的參考基因的最佳數量[8],閾值上限是0.15,以確定所需內參的最佳數目。在此研究中,成對的變異值在0.15左右(圖3E),利用GeNorm軟件對配對組合(Vn/Vn+1)分析發現,在橡膠樹的不同膠乳樣品中選出2個內參基因就可以進行標準化,這與之前一些物種的研究相似[15-16]。
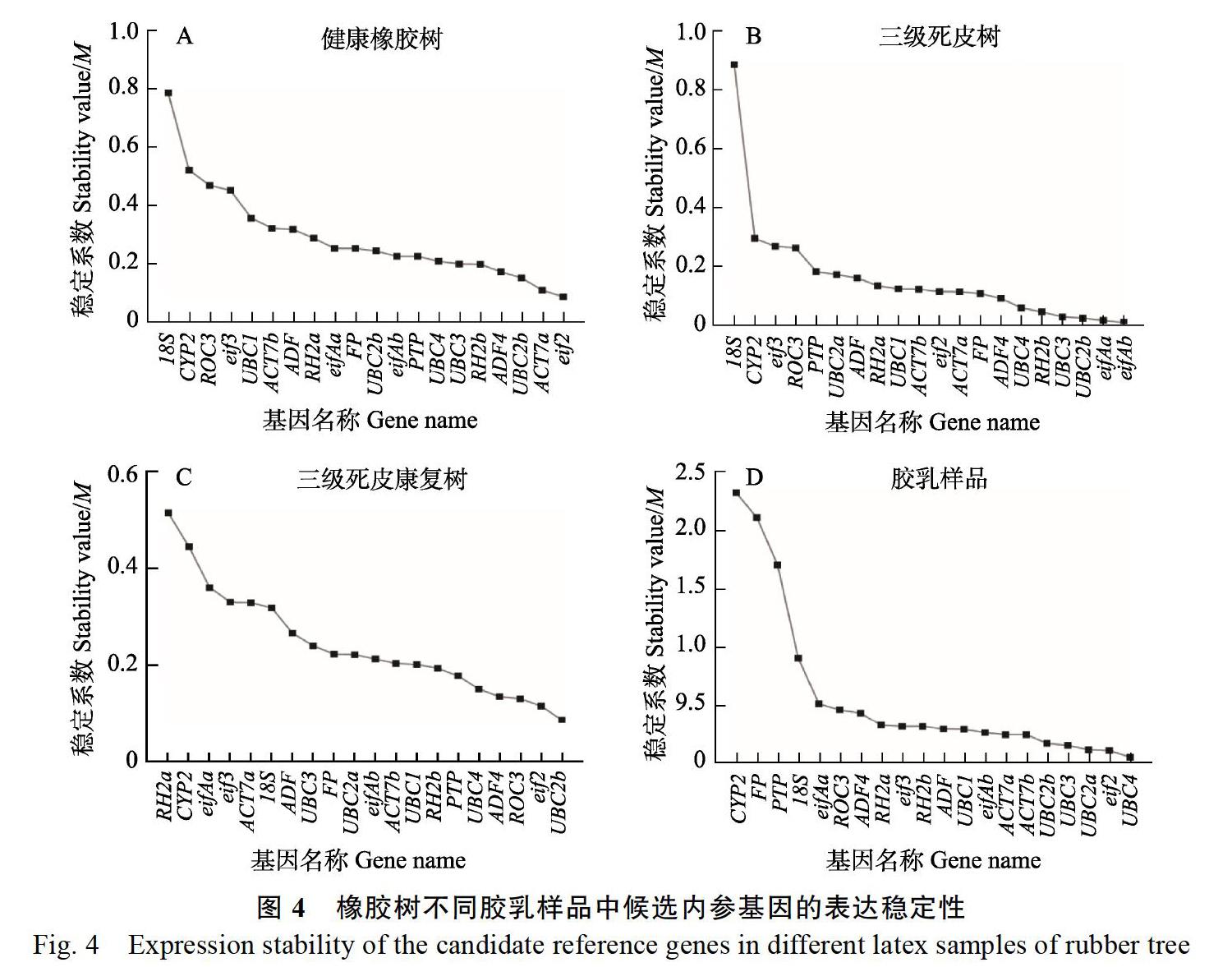
2.3.2 ?NormFinder分析 ?采用NormFinder軟件分析穩定值(SV),其數值大小與穩定值呈負相關[17],即數值越小穩定性越高,進一步驗證geNorm分析的結果。
結果表明,在健康樹膠乳樣品中,穩定性由高到低排名前10的基因分別是eif2、ACT7a、UBC2a、ADF4、RH2b、UBC3、UBC4、PTP、eifAb、UBC2b(圖4A),其中有8個基因與geNorm評價的前10位基因相同;在三級死皮樹膠乳樣品中,排名前10位的基因分別是eifAb、eifAa、UBC2b、UBC3、RH2b、UBC4、ADF4、FP、ACT7a、eif2(圖4B),其中也有9個基因與geNorm評價的前10位基因相同;在死皮康復膠乳樣品中,排名前10位的基因分別是UBC2b、eif2、ROC3、ADF4、UBC4、PTP、RH2b、UBC1、ACT7b、eifAb,與geNorm評價的前10位基因排序一致(圖4C);在所有膠乳的混合樣品中,排名前10位的基因分別是UBC4、eif2、UBC2a、UBC3、UBC2b、ACT7b、ACT7a、eifAb、UBC1、ADF,與geNorm評價的前10位基因相同(圖4D)。綜合來看,NormFinder和geNorm分析的實驗結果中,得出eif2、UBC2b、UBC3、UBC4、RH2b穩定性好,而18S和CYP2表達穩定性較差的結果是一致的。
2.3.3 ?BestKeeper分析 ?BestKeeper軟件可用于對候選內參基因在各樣品中的平均Ct值進行分析[18]。一般穩定的內參基因在BestKeeper中具有較高的決定系數(coefficient of determination, R2)。由結果可知(表1),健康橡膠樹膠乳的穩定性排名前10的基因依次是ACT7a、UBC2b、PTP、UBC3、eif2、FP、RH2b、RH2a、UBC2a、eifAb;三級死皮樹膠乳則是RH2b、UBC2b、FP、UBC1、eifAa、ACT7a、eif2、UBC3、ACT7b、eifAb;死皮康復橡膠樹膠乳則是FP、ROC3、UBC2b、eifAb、ADF、18S、eif2、RH2a、ACT7a、RH2b;在混合膠乳中則是:eif2、eifAa、ADF、ADF4、UBC3、eifAb、UBC2a、ACT7a、eif3、UBC4。
GeNorm、NormFinder和BestKeeper三個軟件的評估結果既有一致性,也有差異性。GeNormh和NormFinder的分析結果高度一致,而與BestKeeper的一致性略差。綜合3種結果,eif2和UBC2b兩個基因的穩定性表現最好,是合適的內參基因選擇。
2.4 ?內參基因穩定性驗證
根據軟件分析結果,選擇各樣品中3個較穩定的基因和穩定性較差的18S基因,分別對HbDXS1、HbHMGS2和HbNIN3共3個膠乳代謝相關基因的相對表達量進行分析。結果表明,健康樹不同割膠刀次之間,HbDXS1、HbHMGS2和HbNIN3三個基因在以eif2、UBC2b、UBC2a為內參時的表達規律基本一致,而以18S為內參時則基本表現出相反的變化趨勢(圖5A)。
三級死皮樹不同死皮程度部位采集的膠乳樣品中,分別以穩定的UBC3、eifAb和UBC2b為內參分析時,HbDXS1、HbHMGS2和HbNIN3三個目的基因的表達變化規律基本一致,并且相對健康樹而言,表達量整體水平上降低。以18S為內參進行分析時,3個目的基因的表達規律明顯不同于以3個穩定性好的內參基因為參照的結果,甚至出現近似于相反的表達規律(圖5B)。
死皮康復樹中,分別以穩定性好的eif2、UBC2b和ROC3為內參基因分析時,HbDXS1、HbHMGS2和HbNIN3基因的表達規律基本一致,其中尤以eif2和UBC2b為內參時表達規律一致性更好;當以18S為內參進行分析時,基因的表達量和表達規律都與前3個內參有較大的區別(圖5C)。
3 ?討論
為獲取準確的基因表達數據,內參基因的選擇顯得尤為重要。合適的、穩定的內參基因才能客觀、真實地反映目標基因的相對表達量。已有研究表明,內參基因不具有通用性。不同的物種、不同的組織、不同的發育階段,甚至不同的處理條件下都可能需要不同的內參基因,不經篩選而直接選用常見的內參基因可能使分析結果的精確性降低甚至導致得出錯誤的結論。目前,篩選內參基因常用的方法主要有Genorm、NormFinder、BestKeeper[8,17-18]。Manoli等[19]利用這些方法研究了玉米在不同實驗條件下、不同組織中12個內參基因的差異,篩選得到在玉米中肌動蛋白(ACT)的穩定性較好,而18S的穩定性差。早期在橡膠樹qRT-PCR分析中常采用18S作為內參基因,后來該基因也被發現并不適合作為橡膠樹所有的基因研究中的內參。Li等[4]認為在橡膠樹不同組織中RH2b和UBC2b穩定性較好,在割膠處理中eifAb穩定性較好。何斌[20]對20個內參基因進行分析,認為適合膠乳再生研究的內參基因是UBC4、ADF、UBC2a、eif2、ADF4。Long等[21]在連續割膠實驗中評估了22個候選基因,認為穩定性好的前5個參考基因分別為UBC2a、UBC2b、UBC4、UBC3和ADF。Chao等[22]針對不同排膠時間篩選出合適的膠乳內參基因是UBC2a和UBC2b。最近,吳紹華等[23]研究TSA誘導乳管分化過程中篩選的最佳內參基因是UBC3。由此可見,橡膠樹有關分子機理的研究中,也沒有一個普適的內參基因。針對不同的實驗系統,有必要篩選相應的最適內參基因。死皮相關尤其是包含死皮康復樹方面的qRT-PCR內參基因篩選的研究尚未見報道,為研究死皮發生以及死皮康復的分子機理,尋找合適的內參基因顯得尤為重要。
本研究是針對橡膠樹死皮相關研究展開的內參基因篩選,通過3個計算軟件比較了20個候選內參基因的表達穩定性。結合基因穩定性驗證,綜合分析認為eif2、ACT7a、UBC2b在健康樹中是最為穩定的3個內參基因;UBC3、eifAb、eifAa、ADF4、UBC4、UBC2b、eif2、RH2b、ACT7b在三級死皮樹不同部位中都表現出較好的穩定性;ROC3、eif2和UBC2b在三級死皮恢復樹中是較穩定的基因。可見,在3種類型的橡膠樹中都有良好的穩定性表現的內參基因是eif2和UBC2b,尤其其中的eif2一直都有良好的穩定性,即使在所有膠乳混合這樣的來源差異比較大的樣品中也是最穩定的。因此,在死皮相關研究中,認為eif2和UBC2b是最佳的一組內參基因,而其中的ejf2是最佳的內參基因。18S表達豐度在20個候選基因中最高,但穩定性最差,基因驗證也表明不適合作為死皮相關研究的內參基因。
本研究中利用了Genorm、NormFinder、BestKeeper三種方法來分析候選基因的穩定性,但其中Genorm和NormFinder的分析結果一致性更高,與最終分析的結果更吻合,是較適合用來篩選內參基因的軟件。
參考文獻
[1] 何 ?晶, 馮成天, 郭秀麗, 等. 高濃度乙烯利刺激誘導橡膠樹死皮發生過程中的膠乳生理研究[J]. 西北林學院學報, 2018, 33(2): 123-128.
[2] Alwine J C, Kemp D J, Stark G R. Method for detection of specific RNAs in agarose gels by transfer to diazobenzy-loxymethyl-paper and hybridization with DNA probes[J]. Proceedings of the National Academy of Sciences of the USA, 1977, 74(12): 5350-5354.
[3] Kupper H, Seib L O, Sivaguru M, et al. A method for cellu-lar localization of gene expression via quantitative in situ hybridization in plants[J]. Plant Journal, 2007, 50(1) : 159-175.
[4] Li H P, Qin Y X, Xiao X H, et al. Screening of valid refer-ence genes for real-time RT-PCR data normalization in He-vea brasiliensis, and expression validation of a sucrose transporter gene HbSUT3[J]. Plant Science, 2011, 181(2): 132-139.
[5] 袁 ?偉, 萬紅建, 楊悅儉. 植物實時熒光定量PCR內參基因的特點及選擇[J]. 植物學報, 2012, 47(4): 427-436
[6] Ginzinger D G. Gene quantification using real-time quantitative PCR: an emerging technology hits the mainstream[J]. Experimental Hematology, 2002, 30(6) : 503-512.
[7] Garson J A, Grant P R, Ayliffe U, et al. Real-time PCR quantitation of hepatitis B virus DNA using automated sam-ple preparation and murine cytomegalovirus internal con-trol[J]. Journal of Virological Methods, 2005, 126(1): 207-213.
[8] Vandesompele J, Preter K D, Pattyn F, et al. Accurate nor-malization of real-time quantitative RT-PCR data by geome-tric averaging of multiple internal control genes[J]. Genome Biology, 2002, 3(7): 467-470.
[9] 黃雪玲, 馮 ?浩, 康振生. 小麥條銹菌實時熒光定量PCR分析中內參基因的選擇[J]. 農業生物技術學報, 2012, 20(2) : 181-187.
[10] Radoni? A, Thulke S, Mackay I M, et al. Guideline to refer-ence gene selection for quantitative real-time PCR[J]. Bio-chemical and Biophysical Research Communications, 2004, 313(4): 856-862.
[11] 姜 ?婷, 蘇 ?喬, 安利佳. 多重脅迫下玉米實時定量PCR內參基因的篩選與驗證[J]. 植物生理學報, 2015, 51(9): 1457-1464.
[12] Chen K, Fessehaie A, Arora R. Selection of reference genes for normalizing gene expression during seed priming and germination using qPCR in Zea mays, and Spinacia olera-cea[J]. Plant Molecular Biology Reporter, 2012, 30(2): 478-487.
[13] 聞志彬, 張明理. 松葉豬毛菜干旱脅迫下實時定量PCR內參基因的篩選[J]. 植物生理學報, 2015, 51(11): 2031-2038.
[14] 黃瓊林, 梁凌玲, 何 ?瑞, 等. 青天葵實時熒光定量PCR內參基因的選擇[J]. 中草藥, 2013, 44(14): 1979-1983.
[15] Reddy D S, Bhatnagar-Mathur P, Cindhuri K S, et al. Evalu-ation and validation of reference genes for normalization of quantitative real-time PCR based gene expression studies in peanut[J]. PLoS One, 2013, 8(10): e78555.
[16] Taylor C M, Jost R, Erskine W, et al. Identifying stable reference genes for qRT-PCR normalization in gene expres-sion studies of narrow-leafed Lupin (Lupinus angustifolius L.)[J]. PLoS One, 2016, 11(2): e0148300.
[17] Andersen C L, Jensen J L, Orntoft T F. Normalization of real-time quantitative reverse transcription-PCR data: a model-based variance estimation approach to identify genes suited for normalization, applied to bladder and colon cancer data sets[J]. Cancer Research, 2004, 64(15): 5245-5250.
[18] Pfaffl M W, Tichopad A, Prgomet C, et al. Determination of stable housekeeping genes, differentially regulated target genes and sample integrity: Best Keeper-Excel-based tool using pair-wise correlations[J]. Biotechnology Letters, 2004, 26(6): 509-515.
[19] Manoli A, Sturaro A, Trevisan S, et al. Evaluation of candi-date reference genes for qPCR in maize[J]. Journal of Plant Physiology, 2012, 169(8): 807-815.
[20] 何 ?斌. 橡膠樹膠乳再生過程中糖代謝相關基因的表達與膠乳生理生化參數分析[D]. 海口: 海南大學, 2015.
[21] Long X, He B, Gao X, et al. Validation of reference genes for quantitative real-time PCR during latex regeneration in rubber tree[J]. Gene, 2015, 563(2): 190-195.
[22] Chao J, Yang S, Chen Y, et al. Evaluation of reference genes for quantitative real-time PCR analysis of the gene expression in laticifers on the basis of latex flow in rubber tree (Hevea brasiliensis Muell. Arg.)[J]. Frontiers in Plant Science, 2016, 7: 1149.
[23] 吳紹華, 張世鑫, 楊署光, 等. TSA誘導橡膠樹次生乳管分化過程qRT-PCR內參基因的篩選[J]. 熱帶作物學報, 2020, 41(3): 504-512.
責任編輯:白 ?凈

