蘋果Dof轉錄因子生物信息學及其表達分析
王新亮 彭玲 王健 賈晶晶 唐立平

摘要:?為了解蘋果Dof轉錄因子家族的生物學信息與功能,利用蘋果基因組GDDH13 v1.1檢索及RNA-seq轉錄本重構找到51個Dof 基因,并通過 Pfam 和SMART檢測確認,進一步對這些Dof基因進行全面分析。結果表明,除MD07G1265700外,其他50個Dof蛋白均含有一個明顯的Dof結構域,且有一個CX2CX21CX2C基序。這些Dof基因編碼氨基酸數為163~523,相對分子質量為18 210~55 800,等電點為5.01~10.30,多數Dof成員定位于細胞核中, 少數定位于葉綠體或線粒體中。組織特異表達顯示多數Dof 基因在營養器官中的表達量高于生殖器官,而MD01G1084700、MD07G1153300、MD08G1040100、MD15G1034500基因在未成熟的果肉中表達量最高。鹽堿脅迫下,除MD05G1023800基因沒有檢測到表達外,其他Dof基因的表達均受鹽堿脅迫影響,只是響應強度和時間有差異,基因表達顯著上調的有7個,顯著下調的有15個。該研究結果為進一步揭示蘋果Dof轉錄因子生物功能奠定了理論基礎。
關鍵詞:?蘋果;Dof基因;轉錄因子;表達分析;生物信息學
中圖分類號:?S661.1??文獻標識碼:?A??文章編號:?1000-4440(2021)02-0480-13
Abstract:?In order to understand the biological information and function of apple Dof transcription factor family, 51 Dof genes were found by searching apple genome GDDH13 v1.1 and RNA-seq transcripts reconstruction, and they were confirmed as members of Dof transcription factor family by Pfam and SMART detection. The Dof genes were generally analyzed furtherly. The results showed that, besides MD07G1265700, the other 50 Dof proteins all contained an obvious Dof domain and a CX2CX21CX2C motif. The number of amino acids encoded by Dof gene ranged from 163 to 523, and the relative molecular weight ranged from 18 210 to 55 800, while the isoelectric point ranged from 5.01 to 10.30. Most of the Dof proteins were located in the nucleus, and a few of them were located in the chloroplast or mitochondria. The results of tissue-specific expression showed that the expression level of most of the Dof genes expressed in vegetative organs were higher than in reproductive organs, while the expression levels of MD01G1084700, MD07G1153300, MD08G1040100 and MD15G1034500 were the highest in immature fruit flesh. Under saline-alkali stress, except for MD05G1023800 gene, the expression of other Dof genes were affected by saline-alkali stress, but the intensity and time of response were different. There were seven Dof genes significantly up-regulated and 15 Dof genes significantly down regulated. The research results lay a theoretical foundation for further study on the biological function of Dof transcription factor in apple.
Key words:?apple;Dof gene;transcription factor;expression analysis;bioinformatics
蘋果(Malus pumila Mill.)是薔薇科中重要的果樹之一,與其他果樹相比,具有種植面積大、貨架期長等特點,蘋果在中國的種植面積和產量均為世界首位。土壤鹽堿、干旱、倒春寒等非生物脅迫制約著蘋果產業的發展[1]。
Dof (DNA binding with one finger)蛋白是一類植物特有的轉錄因子[2],其N端含有一個由50~52個氨基酸組成的高度保守的鋅指結構——Dof結構域,此結構域中含有1個高度保守的CX2CX21CX2C基序。Dof蛋白既能與特定的DNA序列結合調節基因表達,也能與某些蛋白質相互作用,參與植物生長發育及非生物脅迫響應的調控[3-5]。Dof蛋白識別的核心序列是5′-AAAG-3′或5′-CTTT-3′[6];其C端氨基酸序列變異性較大,是Dof蛋白的轉錄調控結構域[7]。在單子葉植物和雙子葉植物均發現了許多Dof蛋白。目前,在小麥、大豆、水稻和擬南芥的基因組中,分別有31、78、30 和 36個Dof基因被發現[8-10]。植物中存在如此多的Dof基因說明其參與植物多種復雜的生理功能控制。
本研究利用蘋果基因組,通過Dof結構域序列檢索和RNA-seq轉錄本重構,篩選并鑒定出51 個蘋果Dof基因家族成員,全面分析了它們編碼的Dof轉錄因子的Dof結構域、保守基序、亞細胞定位、系統進化樹及其特異性表達。以期為進一步研究Dof基因在調控蘋果生長發育及非生物脅迫響應等過程中的作用奠定基礎。
1?材料與方法
1.1?試驗材料與處理
平邑甜茶種子用0.2% KMnO4浸泡30 min,然后用流水沖洗干凈,與洗凈的細沙混勻置于4 ℃層積60 d。種子露白后播種在裝有種植土(細砂、蛭石、土的體積比為3∶2∶1 )的育苗盤中,并置于濱州學院黃河三角洲生態環境重點實驗室苗圃中露天管理,每隔5 d澆1次 1/2 Hoagland營養液(pH=6.8±0.2)。當平邑甜茶苗長到6片真葉時,用加入了100 mmol/L NaCl、50 mmol/L Na2SO4及50 mmol/L NaHCO3的1/2 Hoagland營養液(pH=8)處理,在第0 d、1 d、3 d、6 d分別采集葉片和新根(每個樣品3次重復)存放在液氮中備用。
1.2?RNA 提取和轉錄組測序
樣品總RNA用TRIzol試劑盒提取,利用磁珠富集mRNA,然后利用打斷Buffer將獲得的RNA片段化,使用隨機N6引物進行反轉錄合成,再合成cDNA第二條鏈。通過特異引物進行PCR擴增。熱變性使PCR產物形成單鏈,再利用橋式引物將單鏈DNA環化最終得到單鏈環狀DNA文庫。使用BGISEQ-500平臺測序,所得原始數據經純化去除低質量、接頭及未知堿基N含量過高的read后得到可靠的高質量序列,使用HISAT將所得序列比對到參考基因組序列(Malus x domestica GDDH13 Whole Genome v1.1,https://www.rosaceae.org/species/malus/malus_x_domestica/genome_GDDH13_v1.1)[11]。
1.3?GDDH13Dof轉錄因子篩選與鑒定
利用Dof結構域序列,在蘋果基因組GDDH13 v1.1 mRNA進行BLAST序列比對并搜索“Dof”關鍵詞篩選蘋果Dof候選基因。利用StringTie (http://ccb.jhu.edu/software/stringtie) 對平邑甜茶轉錄組測序結果進行轉錄本重構,然后用Cuffmerge (http://cole-trapnell-lab.github.io/cufflinks) 將所有樣品的重構信息整合在一起,再使用Cuffcompare (http://cole-trapnell-lab.github.io/cufflinks) 將整合后的轉錄本與參考注釋信息比較,挑選出新轉錄本,然后使用CPC2 (http://cpc2.cbi.pku.edu.cn/)[12]預測新轉錄本編碼蛋白質的潛力,然后將具有蛋白質編碼潛力的新轉錄本加入參考基因中,最后通過Pfam (http://pfam.xfam.org)[13] 和SMART (http://www.smart.embl-heidelberg.de/)對鑒定出的Dof蛋白進行確認。通過ExPASy網站 (https://web.expasy.org/protparam/)對蘋果Dof蛋白的理化性質進行分析[14]。
1.4?Dof蛋白系統進化樹、保守域和保守基序分析
從擬南芥信息數據庫(TAIR https://www.arabidopsis.org/ )下載獲得擬南芥的Dof轉錄因子的氨基酸序列。利用MAFFT version 7 (https://mafft.cbrc.jp/alignment/server/)比對 Dof結構域[15]。通過MAFFT online service和Evolview v3 (https://www.evolgenius.info//evolview/#mytrees//)進行系統進化樹構建[15-16],用Average linkage (UPGMA)法構建進化樹,參數均為默認值。利用GSDS2 (http://gsds.cbi.pku.edu.cn/)繪制基因結構圖[17]。利用MEME 5.1.1版本 (http://memesuite.org/tools/meme)分析Dof蛋白保守基序(數目設為15,寬度為6~50 aa)[18]。Dof蛋白亞細胞定位用在線軟件WoLF PSORT (https://wolfpsort.hgc.jp/) 預測。
1.5?Dof表達分析與功能注釋
從AppleMDO (http://bioinformatics.cau.edu.cn/AppleMDO/)網站的RNA-seq數據下載Dof 基因在7個不同組織(莖尖、葉片、莖、花、未成熟果肉、成熟果肉、成熟果皮)中的表達數據[19],利用Excel繪制表達熱圖。利用AppleMDO數據庫的GO analysis功能以GDDH13 v1.1 homology with Arabidopsis為參考進行GO分析。
2?結果與分析
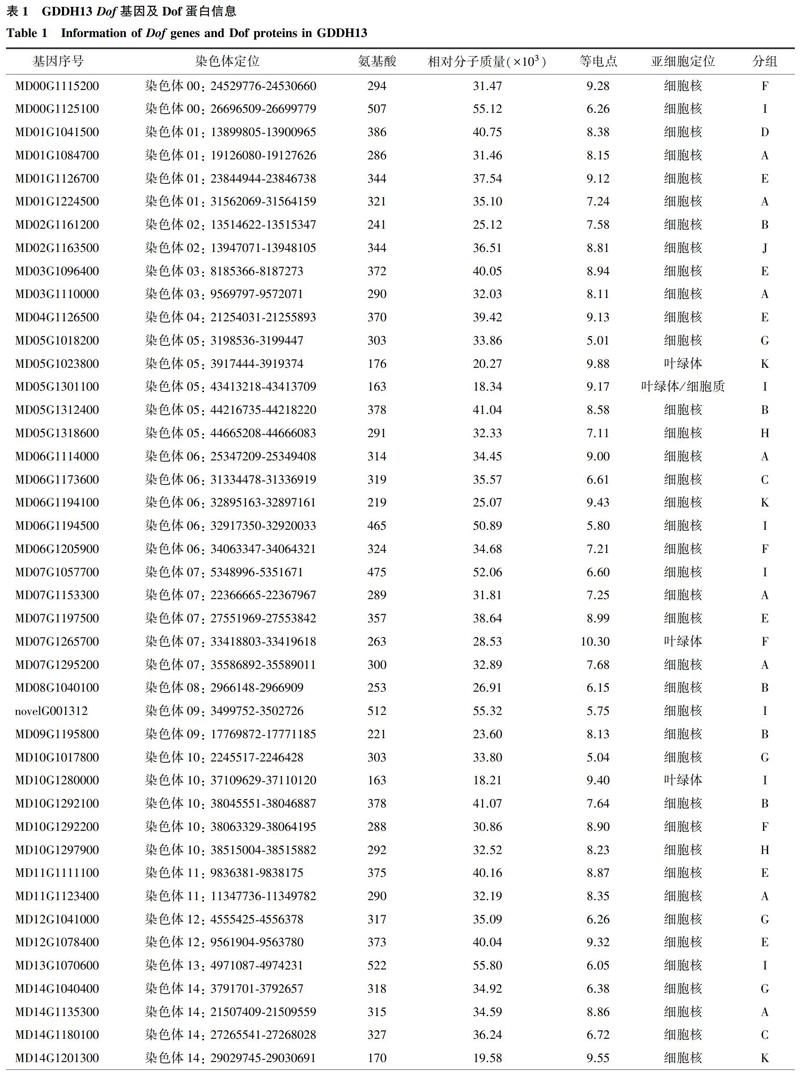
2.1?Dof基因的篩選與鑒定
利用Dof結構域序列,在GDR數據庫蘋果基因組(Malus x domestica GDDH13 Whole Genome v1.1)[11]進行BLAST序列比對,篩選Dof 候選基因。并對平邑甜茶轉錄組測序結果進行轉錄本重構,挑選出新轉錄本,然后使用CPC2預測新轉錄本的蛋白質編碼潛力,將新轉錄本中具有蛋白質編碼潛力的添加到參考基因中。然后通過Pfam和SMART在線工具對篩選出的Dof蛋白進行確認,共篩選鑒定出51個Dof家族成員。但是通過MAFFT version 7對這些Dof轉錄因子的Dof結構域進行比對,發現只有50個基因具有典型的Dof結構域(圖1),由圖1可以看出,有50個Dof轉錄因子均含有1個明顯的高度保守的Dof結構域,且結構域內均含有CX2CX21CX2C基序構成的單鋅指結構。而MD07G1265700雖然通過Pfam和SMART在線工具被鑒定為Dof家族成員,但是通過序列對比發現其并不存在典型的CX2CX21CX2C單鋅指結構。對Dof基因的定位,編碼蛋白質的相對分子質量、等電點等理化性質進行分析(表1)。這些 Dof 基因在蘋果各個染色體上均有分布,它們編碼的蛋白質的長度為163~523 aa;相對分子質量為18 210~55 800;等電點為5.01~10.30。亞細胞定位預測結果表明,除MD05G1023800、MD07G1265700、MD10G1280000可能定位于葉綠體中,MD05G1301100 可能定位于葉綠體或/和細胞核中,MD15G1275300可能定位于線粒體中,其他Dof蛋白均主要定位于細胞核中。
2.2?Dof家族蛋白系統進化樹
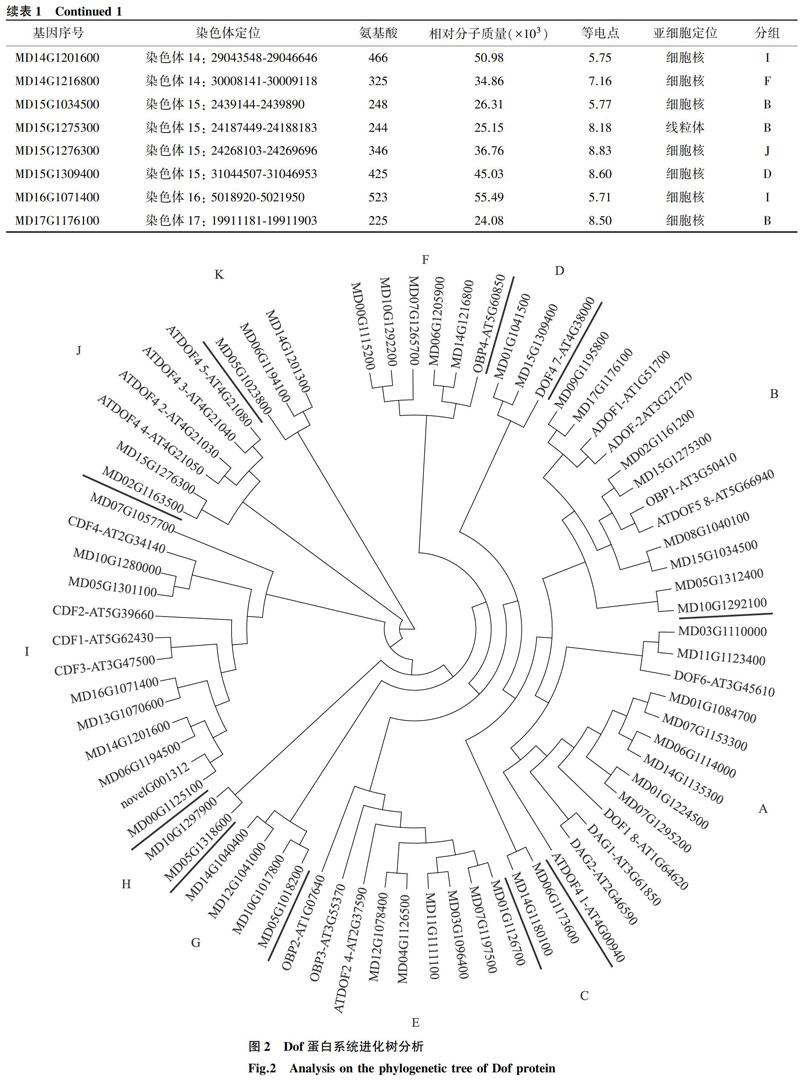
利用鑒定的Dof蛋白與擬南芥的Dof蛋白氨基酸序列構建了一個系統進化樹(圖2)。 Dof蛋白分成11組。在這51個Dof蛋白中,8個分到A組,8個分到B組,2個分到C組,2個分到D組,6個分到E組,5個分到F組,4個分到G組,2個分到H組,9個分到I組,2個分到J組,3個分到K組。
2.3?Dof基因結構分析
利用GDDH13 v1.1基因組注釋文件gene_models_20170612.gff3.gz繪制了Dof基因結構圖(圖3)。圖3顯示,進化樹分析中親緣關系近的基因,它們的基因結構也相似。但是成員較多的A、B和I組中,基因結構差異較大。
2.4?Dof蛋白保守motif分析
利用MEME在線軟件對Dof蛋白氨基酸序列進行motif分析,可以看到Dof蛋白含有15個高度保守的motif,雖然它們的數目和位置差異較大,但是根據進化樹分析,親緣關系近的Dof蛋白的motif數量和序列相似度較高(圖4)。其中50個Dof蛋白均含有1個保守的CX2CX21CX2C基序(含有motif 1或motif 13),而MD07G1265700 既不含有motif 1,也不含有motif 13。
2.5?Dof基因組織特異性表達
利用AppleMDO數據庫的RNA-seq數據,分析了Dof基因在莖尖、葉片、莖、花、未成熟果肉、成熟果肉和成熟果皮中的表達(圖5)。除MD12G1041000基因和MD14G1040400基因沒在任何組織中檢測到表達外,其他Dof 基因均至少在一個組織中檢測到表達;這些Dof基因在莖、葉片、莖尖等營養器官(組織)中的表達量大多高于在花、果肉、果皮等生殖器官(組織)中的表達量。而MD01G1084700、MD07G1153300、MD08G1040100、MD15G1034500基因在未成熟的果肉中表達量最高,在成熟果肉中均未被檢測到。
2.6?平邑甜茶Dof基因在鹽堿脅迫下的表達分析
近年來,越來越多的研究結果表明Dof基因參與植物對干旱、鹽堿等非生物脅迫響應的調控。本研究利用RNA-seq技術檢測了平邑甜茶在鹽堿脅迫0 d、1 d、3 d、6 d后葉片和根中Dof基因的表達情況(圖6)。在平邑甜茶中,除MD05G1023800基因沒有檢測到表達外,其他Dof基因均檢測到1次及以上的表達,而且大多數Dof基因在根中的表達量顯著高于葉片。與對照(0 d)相比,在鹽堿脅迫下平邑甜茶根系中表達顯著上調的Dof基因有MD13G1070600、MD00G1125100、novelG001312、MD16G1071400、MD05G1318600、MD10G1297900、MD05G1018200基因。與對照(0 d)相比,表達顯著下調的基因有MD15G1309400、MD02G1161200、MD14G1040400、MD01G1041500、MD14G1216800、MD07G1265700、MD15G1034500、MD15G1275300、MD06G1194100、MD06G1173600、MD06G1205900、MD05G1312400、MD14G1180100、MD07G1057700、MD05G1301100基因。
2.7?功能注釋
利用AppleMDO數據庫的GO analysis功能對已有的50個Dof基因進行了GO功能分析,結果顯示,共涉及到40個GO條目 (圖7),其中細胞組成 (CC) 9個、分子功能(MF) 3個、生物過程(BP) 28個。除分子功能中蛋白質結合和生物過程中細胞組分組織、對非生物脅迫反應、對刺激反應等9個條目參與的基因較少,其余GO條目均有50個Dof基因參與。
3?結論與討論
Dof轉錄因子在植物調節生長發育與響應非生物脅迫中起著重要作用[20-22]。目前,Dof基因的報道主要集中在草本植物中[8-10],在木本植物中報道較少。本研究對GDDH13 v1.1基因組預測基因進行了較全面的檢索,從已有基因中共檢索并鑒定了50個Dof轉錄因子基因。另外通過對平邑甜茶轉錄組測序結果進行轉錄本重構,挑選出新轉錄本,然后使用CPC2對新轉錄本進行蛋白質編碼潛力預測,然后通過Pfam和SMART在線工具鑒定出1個新的Dof基因,并命名為novelG001312。其中MD07G1265700雖然通過Pfam和SMART在線工具被鑒定為Dof轉錄因子家族成員,但是通過氨基酸序列比較和motif 分析發現其并不存在典型的CX2CX21CX2C單鋅指結構,說明MD07G1265700可能不屬于Dof轉錄因子家族。
有研究結果表明:不同植物Dof蛋白的理論等電點基本處于5.41~6.97,且一般堿性氨基酸個數多于酸性氨基酸[23]。但本研究中蘋果Dof蛋白的等電點主要集中在5.01~10.30,多為堿性氨基酸,與小桐子和大豆Dof蛋白的等電點研究結果相似[24-25],說明不同植物間Dof家族成員的等電點差異較大[26]。
系統進化樹能為預測同源基因的功能提供一些依據。DAG1 (AtDof3.7) 能通過抑制GA3ox1基因,減少赤霉素含量,從而抑制種子萌發;而DAG2(AtDof2.5)能促進種子萌發[27-28],MD01G1084700、MD01G1224500、MD06G1114000、MD07G1153300、MD07G1295200、MD14G1135300與DAG1、DAG2位于A組的一個分支上,說明它們也可能參與種子的萌發調控。擬南芥CDF1、CDF2和CDF3通過抑制CONSTANS基因轉錄來控制開花期[29-30],I組中MD00G1125100、MD06G1194500、MD13G1070600、MD14G1201600、MD16G1071400、novelG001312與上述3個轉錄因子較近,它們也可能參與蘋果花期調控。B組中,MD09G1195800、MD17G1176100和ADOF1在一個分支,ADOF1在胼胝質組織中表達量較高,ADOF1可能影響酚類化合物的合成[31]。因此,MD09G1195800和MD17G1176100也可能與該功能有關。在氧化脅迫和水楊酸誘導下,OBP1能調節防御基因表達,OBP1還與細胞分裂周期有關[32],與其在同一分支上的MD02G1161200、MD08G1040100、MD15G1034500、MD15G1275300也可能具有類似的功能。
進化樹分析中,位于同一分支的基因親緣關系近,而親緣關系越近[33-36],Dof基因的結構及其編碼的氨基酸序列的motif數量和相似度越高。轉錄因子家族含有的motif 在相同的組中是高度保守的[37]。通過motif 分析,51個Dof蛋白含有15個高度保守的 motif,其中motif 1和motif 13含有CX2CX21CX2C單鋅指結構。除MD07G1265700不含有motif 1和motif 13,MD14G1201300、MD06G1194100、MD05G1023800各含有1個motif 13,其他Dof蛋白各含有1個motif 1。表明在相同組中含有相同保守基序的Dof蛋白也許具有相似的功能。
基因表達模式能為其功能預測提供一定的參考[37-38]。OBP2在擬南芥的根和葉片中表達量最高,在莖和花中表達量低于根和葉[39],OBP2可以調控擬南芥硫代葡萄糖苷的生物合成[40]。同在E組的MD04G1126500、MD12G1078400、MD03G1096400、MD07G1197500、MD11G1111100、MD01G1126700基因也是多數在根、葉中表達量較高。GO分析結果表明,它們都參與生物合成和代謝的調控,因此推斷這些基因可能參與蘋果次生代謝的調控。OBP4在擬南芥各個組織器官中均有表達,并參與細胞周期和細胞擴增的負調控[22]。同在F組的MD00G1115200、MD10G1292200、MD07G1265700、MD06G1205900、MD14G1216800基因也在各個組織均表達,且多數在花中的表達量最高。說明它們可能主要參與花中細胞周期的調控。MD01G1084700、MD07G1197500、MD08G1040100和MD15G1034500基因在未成熟的果肉中表達量最高,在成熟果肉中均未被檢測到,它們可能與果實的生長發育有關。
本研究中,除MD05G1023800基因沒有檢測到表達外,其他Dof基因的表達均對鹽堿處理有響應,只是響應強度和時間有差異。在鹽堿脅迫下,顯著上調的Dof基因有7個,顯著下調的有15個。總之,蘋果大部分Dof 家族基因可能在鹽堿脅迫響應中起重要的調控作用,但它們的功能還需要進一步驗證。
參考文獻:
[1]?張婷婷,康?慧,付璐璐,等. 蘋果MdCYP707A家族基因表達分析和MdCYP707A1的功能鑒定[J]. 園藝學報, 2019, 46(8): 1429-1444.
[2]?UMEMURA Y, ISHIDUKA T, YAMAMOTO R, et al. The Dof domain, a zinc finger DNA-binding domain conserved only in higher plants, truly functions as a Cys2/Cys2 Zn finger domain[J]. The Plant Journal, 2004, 37 (5): 741-749.
[3]?MARZABAL P, GAS E, FONTANET P, et al. The maize Dof protein PBF activates transcription of γ-zein during maize seed development[J]. Plant Molecular Biology, 2008, 67 (5): 441-454.
[4]?TOKUNAGA S, SANDA S, URAGUCHI Y, et al. Overexpression of the DOF-Type transcription factor enhances lipid synthesis in Chlorella vulgaris[J]. Applied Biochemistry and Biotechnology, 2019, 189(1): 116-128.
[5]?KURAI T, WAKAYAMA M, ABIKO T, et al. Introduction of the ZmDof1 gene into rice enhances carbon and nitrogen assimilation under low-nitrogen conditions[J]. Plant Biotechnology Journal, 2011, 9 (8): 826-837.
[6]?李?婭,丁文杰,江海燕,等. Dof基因家族調節植物生長發育功能的研究進展[J]. 西北植物學報, 2018, 38(9): 1758-1766.
[7]?YANAGISAWA S. The Dof family of plant transcription factors[J]. Trends in Plant Science, 2002, 7(12): 555-560.
[8]?SHAW L M, MCINTYRE C L, GRESSHOFF P M, et al. Members of the Dof transcription factor family in Triticum aestivum are associated with light-mediated gene regulation[J]. Functional & Integrative Genomics, 2009, 9(4): 485-498.
[9]?GUO Y, QIU L J. Genome-wide analysis of the Dof transcription factor gene family reveals soybean-specific duplicable and functional characteristics[J]. PLoS One, 2013, 8(9): e76809.
[10]LIJAVETZKY D, CARBONERO P, VICENTE-CARBAJOSA J. Genome-wide comparative phylogenetic analysis of the rice and Arabidopsis Dof gene families[J]. BMC Evolutionary Biology, 2003, 3(1): 17.
[11]JUNG S, LEE T, CHENG C H, et al. 15 years of GDR: New data and functionality in the Genome Database for Rosaceae[J]. Nucleic Acids Research, 2019, 47(D1): D1137-D1145.
[12]KANG Y J, YANG D C, KONG L, et al. CPC2: a fast and accurate coding potential calculator based on sequence intrinsic features[J]. Nucleic Acids Research, 2017, 45(W1): W12-W16.
[13]EL-GEBALI S, MISTRY J, BATEMAN A, et al. The Pfam protein families database in 2019[J]. Nucleic Acids Research, 2019, 47(D1): D427-D432.
[14]GASTEIGER E, HOOGLAND C, GATTIKER A, et al. Protein identification and analysis tools on the ExPASy server[M]New Jersey: Humana Press, 2005: 571-607.
[15]KATOH K, ROZEWICKI J, YAMADA K D. MAFFT online service: multiple sequence alignment, interactive sequence choice and visualization[J]. Briefings in Bioinformatics, 2019, 20(4): 1160-1166.
[16]SUBRAMANIAN B, GAO S, LERCHER M J, et al. Evolview v3: a webserver for visualization, annotation, and management of phylogenetic trees[J], Nucleic Acids Research, 2019, 47(W1): W270-W275.
[17]HU B, JIN P, GUO A Y, et al. GSDS 2.0: an upgraded gene feature visualization server[J]. Bioinformatics, 2015, 31(8): 1296-1297.
[18]BAILEY T L , ELKAN C. Fitting a mixture model by expectation maximization to discover motifs in biopolymers[C]// ALTMAN R, BRUTLAG D, KARP P, et al. Proceedings of the Second International Conference on Intelligent Systems for Molecular Biology. Menlo Park, California: AAAI Press,1994: 28-36.
[19]DA L L, LIU Y, YANG J T, et al. AppleMDO: a multi-dimensional omics database for apple co-expression networks and chromatin states[J]. Frontiers in Plant Science, 2019(10): 1333.
[20]EWAS M, KHAMES E, ZIAF K, et al. The tomato DOF daily fluctuations 1, TDDF1 acts as flowering accelerator and protector against various stresses[J]. Scientific Reports, 2017(7): 10299.
[21]CORRALES A R, CARRILLO L, LASIERRA P, et al. Multifaceted role of cycling DOF factor 3 (CDF3) in the regulation of flowering time and abiotic stress responses in Arabidopsis[J]. Plant Cell and Environment, 2017, 40 (5): 748-764.
[22]XU P P, CHEN H Y, YING L, et al. At DOF5.4/OBP4, a DOF transcription factor gene that negatively regulates cell cycle progression and cell expansion in Arabidopsis thaliana[J]. Scientific Reports, 2016(6): 27705.
[23]李?輝,黃?蔚,劉志薇,等. 茶樹兩個Dof 轉錄因子的分離及其在溫度脅迫中的響應分析[J]. 茶葉科學, 2016, 36(3): 312-322.
[24]王海波,唐利洲. 基于基因組鑒定小桐子Dof 轉錄因子家族及其表達分析[J]. 分子植物育種, 2018, 16(3): 764-771.
[25]劉?蓓,邱?爽,何佳琦,等. 8個大豆Dof轉錄因子的生物信息學分析及干旱誘導表達[J]. 大豆科學, 2020, 39(3): 377-383.
[26]程冬梅,鄧志勇,郭靄光. 小麥高分子量麥谷蛋白亞基等電點的特性分析[J]. 西北植物學報, 2006, 26(3): 532-536.
[27]GABRIELE S, RIZZA A, MARTONE J, et al. The Dof protein DAG1 mediates PIL5 activity on seed germination by negatively regulating GA biosynthetic gene AtGA3ox1[J]. The Plant Journal, 2010, 61(2): 312-323.
[28]GUALBERTI G, PAPI M, BELLUCCI L, et al. Mutations in the Dof zinc finger genes DAG2 and DAG1 influence with opposite effects the germination of Arabidopsis seeds[J]. The Plant Cell, 2002, 14(6): 1253-1263.
[29]SONG Y H, SMITH R W, TO B J, et al. FKF1 conveys crucial timing information for CONSTANS stabilization in the photoperiodic flowering[J]. Science, 2012, 336(6084): 1045-1049.
[30]FORNARA F, PANIGRAHI K C S, GISSOT L, et al. Arabidopsis DOF transcription factors act redundantly to reduce CONSTANS expression and are essential for a photoperiodic flowering response[J]. Developmental Cell, 2009, 17(1): 75-86.
[31]TSUJIMOTO-INUI Y, NAITO Y, SAKURAI N, et al. Functional genomics of the Dof transcription factor family genes in suspension-cultured cells of Arabidopsis thaliana[J]. Plant Biotechnology, 2009(26): 15-28.
[32]SKIRYCZ A, RADZIEJWOSKI A, BUSCH W, et al. The DOF transcription factor OBP1 is involved in cell cycle regulation in Arabidopsis thaliana[J]. The Plant Journal , 2008,56(5): 779-792.
[33]裴徐梨,荊贊革,徐?境,等. 青花菜BoDof5.3基因的克隆及漬水脅迫表達特征分析[J].江蘇農業學報,2020,36(6):1498-1502.
[34]韓利紅,劉?潮,張維維,等. 鐵皮石斛熱激轉錄因子(Hsf)基因家族鑒定及生物信息學分析[J]. 南方農業學報,2019,50(4):677-684.
[35]龐文玉,王安,楊寶誼,等. 大白菜ENT基因家族的鑒定與生物信息學分析[J].江蘇農業科學,2019, 47(12):52-57.
[36]楊冬靜,孫厚俊,謝逸萍,等. 甘薯等8種植物JAZ1基因的生物信息學分析[J].江蘇農業學報,2019,35(5):1021-1027.
[37]唐躍輝,包欣欣,王?健,等. 小桐子Dof基因家族生物信息學與表達分析[J]. 江蘇農業學報, 2019, 35(1): 15-25.
[38]SADDEE A A, MALVANKAR M R, KUMAR K. Selection of reference genes for quantitative real-time PCR analysis in halophytic plant Rhizophora apiculata[J]. The Journal of Life and Environmental Sciences, 2018, 6(8): e5226.
[39]KANG H G, SINGH K B. Characterization of salicylic acid-responsive, Arabidopsis Dof domain proteins: overexpression of OBP3 leads to growth defects[J]. The Plant Journal, 2000,21(4): 329-339.
[40]SKIRYCZ A, REICHELT M, BUROW M, et al. DOF transcription factor AtDof1.1 (OBP2) is part of a regulatory network controlling glucosinolate biosynthesis in Arabidopsis[J]. The Plant Journal, 2006,47(1): 10-24.
(責任編輯:陳海霞)

