利用單片段代換系鑒定巴西陸稻IAPAR9中的粒型基因
張 波 裴瑞琴 楊維豐 朱海濤 劉桂富 張桂權(quán) 王少奎
利用單片段代換系鑒定巴西陸稻IAPAR9中的粒型基因
張 波 裴瑞琴 楊維豐 朱海濤 劉桂富 張桂權(quán) 王少奎*
廣東省植物分子育種重點(diǎn)實(shí)驗(yàn)室/ 亞熱帶農(nóng)業(yè)生物資源保護(hù)與利用國(guó)家重點(diǎn)實(shí)驗(yàn)室/ 華南農(nóng)業(yè)大學(xué), 廣東廣州 510642
水稻粒型是由多基因控制的復(fù)雜數(shù)量性狀, 是稻米產(chǎn)量和品質(zhì)的重要影響因子, 水稻粒型基因的定位與遺傳研究有助于稻米產(chǎn)量與品質(zhì)的協(xié)同改良。本研究利用巴西陸稻IAPAR9為供體、以華南地區(qū)高產(chǎn)秈稻品種華粳秈74為受體, 構(gòu)建的153份水稻單片段代換系材料, 連續(xù)兩季通過(guò)單因素方差分析和鄧肯氏多重比較, 結(jié)合代換片段重疊群作圖, 定位了13個(gè)控制水稻粒型及粒重的QTL。這13個(gè)位點(diǎn)分別分布于水稻1、2、4、5、6、7、9和11號(hào)染色體上, 包括9個(gè)控制谷粒長(zhǎng)的QTL、1個(gè)控制谷粒寬的QTL和3個(gè)控制千粒重的QTL。其中,-、-和為新鑒定的QTL位點(diǎn)。新的粒型QTL定位將為進(jìn)一步的基因克隆與粒型遺傳調(diào)控網(wǎng)絡(luò)解析提供依據(jù)和線索, 也可為稻米產(chǎn)量與品質(zhì)協(xié)同改良提供新的種質(zhì)資源。
水稻; 單片段代換系; 粒型; QTL定位
水稻是世界上最重要的糧食作物之一, 有超過(guò)半數(shù)的人口以稻米為食。隨著現(xiàn)代生活水平逐步提高, 人們對(duì)稻米品質(zhì)的要求也日益增高, 培育高產(chǎn)、優(yōu)質(zhì)的水稻品種已成為水稻育種重要目標(biāo)。水稻粒型直接決定稻米外觀品質(zhì), 同時(shí)也是影響稻谷千粒重的重要因素, 因此, 粒型是水稻高產(chǎn)優(yōu)質(zhì)育種的重要靶標(biāo)性狀, 粒型的遺傳分析與分子機(jī)制研究備受關(guān)注。
水稻粒型是受多基因控制的復(fù)雜農(nóng)藝性狀。伴隨著近代遺傳學(xué)與基因組學(xué)的不斷發(fā)展, 通過(guò)構(gòu)建F2、重組自交系(recombinant inbreed line, RIL)等初級(jí)作圖群體以及染色體片段代換系(chromosome segment substitute line, CSSL)等次級(jí)作圖群體, 大量控制水稻粒型的QTL (quantitative trait locus)陸續(xù)被定位[1-8]; 近年來(lái), 全基因組關(guān)聯(lián)分析(genome-wide association study, GWAS)、突變位點(diǎn)圖譜(MutMap)等技術(shù)更是加快了水稻粒型QTL定位的速度與效率[9-12]。截至目前, 已經(jīng)有超過(guò)600個(gè)粒型相關(guān)的QTL被鑒定, 涵蓋粒寬、粒長(zhǎng)、粒厚、長(zhǎng)寬比和粒重等, 被鑒定到的QTL遍布于水稻12條染色體[1,13]。但目前已被克隆的水稻粒型基因數(shù)量還較少, 其中, 利用自然變異材料克隆到的粒型基因, 僅有[14]、[15]、[16]、[17]、/[18-20]、[21-22]、[23]、[24]、[25]、[26]/[27]、[28]、[29]和[30]等十余個(gè)。
已克隆的粒型基因數(shù)量遠(yuǎn)遠(yuǎn)低于已定位的QTL數(shù)量, 這與定位群體的復(fù)雜性密切相關(guān)。傳統(tǒng)定位中用到的F2、RIL等群體背景較為復(fù)雜, 后代往往持續(xù)分離, 對(duì)基因的精細(xì)定位造成一定的干擾。近年來(lái), GWAS技術(shù)被廣泛用于QTL定位, 它能夠?qū)⒛繕?biāo)QTL定位在某一區(qū)間, 但由于實(shí)驗(yàn)材料的復(fù)雜多樣, 且缺乏必要的遺傳材料對(duì)基因進(jìn)行功能解析, 獲得的優(yōu)良基因也難以直接運(yùn)用到水稻育種實(shí)踐中。水稻單片段代換系(single segment substitution lines, SSSL)材料經(jīng)過(guò)多代回交和分子標(biāo)記輔助選擇培育而成, 通常只攜帶一段來(lái)自供體親本的染色體片段, 遺傳背景與受體親本高度相似, 是鑒定復(fù)雜農(nóng)藝性狀QTL, 解析目的基因功能的理想材料[31]。經(jīng)過(guò)長(zhǎng)期積累, 本團(tuán)隊(duì)已構(gòu)建供體親本2360份, 包括亞洲栽培稻、非洲栽培稻以及AA基因組野生稻的SSSL材料[31], 但尚未針對(duì)單一親本為供體的SSSL材料系統(tǒng)考察粒型相關(guān)QTL。巴西陸稻IAPAR9谷粒較大, 千粒重較高, 本研究利用以其為供體的153份SSSL材料鑒定粒型QTL, 并對(duì)鑒定到的QTL進(jìn)行初步定位和遺傳分析。
1 材料與方法
1.1 試驗(yàn)材料
試驗(yàn)材料為華南地區(qū)高產(chǎn)秈稻品種華粳秈74 (HJX74)、巴西陸稻IAPAR9和153份以華粳秈74為受體親本、IAPAR9為供體親本的水稻單片段代換系材料。水稻材料由本團(tuán)隊(duì)水稻種質(zhì)資源庫(kù)低溫保存。2份親本材料及153份單片段代換系材料分別于2018年晚季、2019年早季在廣州市華南農(nóng)業(yè)大學(xué)啟林北試驗(yàn)農(nóng)場(chǎng)種植。每個(gè)材料3個(gè)重復(fù), 每個(gè)重復(fù)1個(gè)雙壟(20株), 按照10 cm × 15 cm單株插秧, 常規(guī)管理。
1.2 材料的鑒定
插秧7 d后, 按照單株取水稻幼嫩葉片, 利用TPS法[32]提取基因組DNA。以合格的DNA樣品作為模板, 使用SSR標(biāo)記進(jìn)行PCR擴(kuò)增, 結(jié)合6%聚丙烯酰胺變性凝膠電泳進(jìn)行檢測(cè)。帶型與HJX74帶型一致的標(biāo)記為“1”, 與IAPAR9帶型一致的標(biāo)記為“3”, 據(jù)此判定代換片段的位置與長(zhǎng)度, 并選取攜帶純合代換片段的材料用于表型分析。
1.3 材料表型考察
種子完熟后, 按單株收種, 充分晾干后, 考察粒長(zhǎng)、粒寬和千粒重。粒長(zhǎng)和粒寬的考察: 按照不同材料編號(hào)隨機(jī)挑選發(fā)育健康、大小均一的種子100粒, 利用彩色平臺(tái)式掃描儀(MICROTER MRS- 9600TFU2L)掃描記錄; 采用種子外觀分析軟件(萬(wàn)深SC-E型)進(jìn)行圖像處理, 讀取粒長(zhǎng)和粒寬等數(shù)據(jù)。千粒重考察方法為: 充分晾干谷粒, 隨機(jī)取出500粒飽滿健康的種子進(jìn)行稱重, 重復(fù)3次(每次重量之差小于平均值的3%), 折算成千粒重。
1.4 數(shù)據(jù)分析與QTL定位
實(shí)驗(yàn)數(shù)據(jù)采用SPSS 23.0以及Microsoft Excel 2016進(jìn)行統(tǒng)計(jì)與分析; 圖表繪制采用MapChart、Origin 2018和Photoshop CS進(jìn)行。利用單因素方差分析(ANOVA)和鄧肯氏(Duncan’s)多重比較分析單片段代換系材料和HJX74目的性狀的差異顯著性。在0.01水平下進(jìn)行多重比較, 與HJX74在同一組別的SSSL材料不攜帶粒型相關(guān)QTL, 而與HJX74不在同一組別的SSSL材料則攜帶粒型QTL; 在2個(gè)測(cè)試季節(jié)均被檢測(cè)到表型變異的SSSL被認(rèn)為攜帶穩(wěn)定遺傳的QTL, 僅在一個(gè)季節(jié)檢測(cè)到的表型變化不記為QTL。采用重疊群作圖法, 對(duì)代換片段存在重疊區(qū)間的粒型QTL進(jìn)行初步定位[33]。加性效應(yīng)計(jì)算方法為HJX74的表型值與攜帶粒型基因的SSSL的表型值的差值的一半[34]。
2 結(jié)果與分析
2.1 IAPAR9的粒型與攜帶的粒型基因
HJX74是本團(tuán)隊(duì)育成的高產(chǎn)品種, 籽粒大小適中。IAPAR9由巴西農(nóng)業(yè)科學(xué)院稻豆研究中心于1970年育成, 1992年由中國(guó)農(nóng)業(yè)科學(xué)院檢疫、試種并陸續(xù)在江西、貴州和北京等地審定。該品種谷粒長(zhǎng)為(9.65 ± 0.05) cm, 較HJX74增長(zhǎng)約17.60%, 粒寬增加約4.20%, 千粒重也比HJX74重約10.50%, 相關(guān)性狀差異均達(dá)到極顯著水平(圖1)。以二者為親本構(gòu)建定位群體, 鑒定水稻粒型QTL切實(shí)可行。
a, b: HJX74和IAPAR9的粒型, 標(biāo)尺為5 mm; c: HJX74和IAPAR9的粒長(zhǎng); d: HJX74和IAPAR9的粒寬; e: HJX74和IAPAR9的粒重。**表示HJX74的粒長(zhǎng)、粒寬和千粒重與IAPAR9之間的差異達(dá)到極顯著水平(< 0.01),檢驗(yàn)。
a, b: the grain shape of HJX74 and IAPAR9, Bar: 5 mm; c: the grain length of HJX74 and IAPAR9; d: the grain width of HJX74 and IAPAR9; e: the grain weight of HJX74 and IAPAR9. ** means significant difference in grain length, grain width and grain weight between HJX74 and IAPAR9 by Student’s-test (< 0.01).
利用水稻自然變異已克隆了[14]、[16]等十余個(gè)粒型基因。本研究利用本團(tuán)隊(duì)重測(cè)序數(shù)據(jù)結(jié)合Sanger測(cè)序, 系統(tǒng)分析了IAPAR9和HJX74中相關(guān)粒型基因的功能位點(diǎn)。結(jié)果表明, IAPAR9和HJX74二者中的[14]、[24][25][28]和[30]等基因在功能位點(diǎn)上序列一致。而[35][16]、[17][21-22]、[23]、[26]和[29]等粒型基因在二者中存在功能位點(diǎn)上的序列差異, 上述基因位點(diǎn)在IAPAR9中均為功能變異類(lèi)型。針對(duì)存在功能變異的位點(diǎn), 開(kāi)發(fā)了功能基因緊密連鎖標(biāo)記(引物序列見(jiàn)附表1), 相關(guān)標(biāo)記可明確區(qū)分HJX74和IAPAR9中粒型基因的不同等位變異(圖2)。盡管IAPAR9中存在[16]、[21-22]和[26]等多個(gè)粒型基因功能變異, 但粒型是極為復(fù)雜的數(shù)量性狀位點(diǎn), 繼續(xù)解析造成IAPAR9和HJX74粒型差異的其他基因位點(diǎn)將為系統(tǒng)理解水稻粒型的遺傳調(diào)控網(wǎng)絡(luò)提供更多證據(jù)。
2.2 SSSL材料的代換片段
本團(tuán)隊(duì)經(jīng)過(guò)長(zhǎng)期積累, 已經(jīng)建成了供體來(lái)源豐富的水稻單片段代換系材料文庫(kù), 其中以IAPAR9為供體的材料總計(jì)153份。材料中所攜帶的來(lái)自IAPAR9的代換片段分布于水稻12條染色體上, 覆蓋了約52.99%的水稻染色體, 每條染色體上的代換片段總長(zhǎng)度變幅在3.45~123.65 cM之間, 9號(hào)染色體覆蓋率最高, 12號(hào)染色體覆蓋最低(圖3和附表2)。每個(gè)材料中攜帶的代換片段長(zhǎng)度大小不一, 代換片段平均長(zhǎng)度為20.95 cM; 每條染色體上的代換片段數(shù)量不同, 位于1號(hào)染色體的代換片段最多, 達(dá)到30個(gè), 而12號(hào)染色體上的代換片段最少, 僅有1個(gè)(附表2)。
白色線段指示相關(guān)引物經(jīng)PCR擴(kuò)增獲得的目的條帶, 其余帶紋為引物非特異性擴(kuò)增條帶。
The white lines indicate the target bands obtained by PCR, and the other bands are non-specific amplifications.
遺傳距離(單位: cM)見(jiàn)左側(cè)標(biāo)尺; 染色體上黑色部分代表該區(qū)域可以被來(lái)自IAPAR9的代換片段所覆蓋; 藍(lán)色字體突出顯示相關(guān)位點(diǎn)上已克隆的水稻粒型基因。
The genetic distance (cM) is shown as rulers on the left margin. Filled and open bars represent chromosomal segments homozygous for IAPAR9 and HJX74 alleles, respectively. The cloned genes are shown in blue font.
2.3 親本與群體的粒長(zhǎng)、粒寬和千粒重
在2018年晚季和2019年早季, 連續(xù)兩季的粒型分析表明, 153份SSSL材料的粒長(zhǎng)、粒寬和千粒重性狀變幅很大, 粒寬變幅在2.23~2.87 mm之間, 粒長(zhǎng)為7.53~9.04 mm, 千粒重變化區(qū)間為14.00~25.44 g。粒長(zhǎng)、粒寬和千粒重均變現(xiàn)為連續(xù)變異, 接近正態(tài)分布; 其中, 粒寬和千粒重在2個(gè)種植季節(jié)均存在超親分離; 粒長(zhǎng)在2019年早季也觀測(cè)到超親分離現(xiàn)象(圖4)。
2.4 QTL定位分析
利用153份SSSL對(duì)水稻的粒長(zhǎng)、粒寬和千粒重進(jìn)行QTL檢測(cè), 共檢測(cè)到13個(gè)QTL。分別分布在水稻1、2、4、5、6、7、9和11號(hào)染色體上(圖5和表1), 其中包括9個(gè)粒長(zhǎng)QTL、1個(gè)粒寬QTL和3個(gè)控制千粒重的QTL。
控制水稻粒長(zhǎng)的9個(gè)QTL, 分布于水稻1、2、4、5、6、7、9和11號(hào)染色體, 2018晚季對(duì)粒長(zhǎng)的貢獻(xiàn)率在2.12%~4.55%, 2019年早季為1.96%~5.32%。其中, 有6個(gè)QTL加性效應(yīng)為負(fù), 其他3個(gè)QTL加性效應(yīng)為正。本研究在水稻2號(hào)染色體上檢測(cè)到1個(gè)控制粒寬的QTL, 其加性效應(yīng)為正值, 顯著增加水稻粒寬, 連續(xù)兩季對(duì)粒型的貢獻(xiàn)率均在3.00%以上。檢測(cè)到的3個(gè)千粒重QTL中, 有2個(gè)位于水稻1號(hào)染色體, 1個(gè)位于4號(hào)染色體, 其中位于1號(hào)染色體長(zhǎng)臂端的-加性效應(yīng)為正, 與控制粒長(zhǎng)的-共定位;-與-共定位, 對(duì)表型貢獻(xiàn)率均表現(xiàn)為負(fù)效應(yīng) (表1)。該研究獲得的攜帶優(yōu)異粒型QTL的SSSL材料有望直接運(yùn)用于本團(tuán)隊(duì)搭建的水稻設(shè)計(jì)育種平臺(tái), 也有助于克隆并解析新的粒型基因, 為闡明水稻粒型的遺傳調(diào)控網(wǎng)絡(luò)提供材料基礎(chǔ)和理論依據(jù)。
3 討論
本研究利用水稻SSSL材料定位了9個(gè)控制水稻粒長(zhǎng)、1個(gè)控制水稻粒寬和3個(gè)影響千粒重的QTL。研究中用到的IAPAR9為耐旱性良好的粳型水稻, 我國(guó)已利用該材料育成多個(gè)審定品種, 挖掘和研究其中的粒型QTL/基因, 有助于我們深入了解粒型等位基因在水稻秈粳亞種間的遺傳特點(diǎn), 也將直接為水稻外觀品質(zhì)改良提供新的種質(zhì)和基因資源。
a~c:2018晚季親本和SSSL的粒長(zhǎng)、粒寬和千粒重的頻次分布; d~f:2019早季親本和SSSL的粒長(zhǎng)、粒寬和千粒重的頻次分布。
a–c: the frequency distribution of grain length, width, and 1000-grain weight in SSSLs and the parents in later season of 2018; d–f: the frequency distribution of grain length, width, and 1000-grain weight in SSSLs and the parents in early season of 2019.
遺傳距離見(jiàn)左側(cè)標(biāo)尺; 染色體右側(cè)的黑色矩形代表QTL及其定位區(qū)間。
The genetic distance is shown as rulers on the left margin. Black bars on the right of each chromosome are the location intervals of QTLs for grain size with their names on the right.
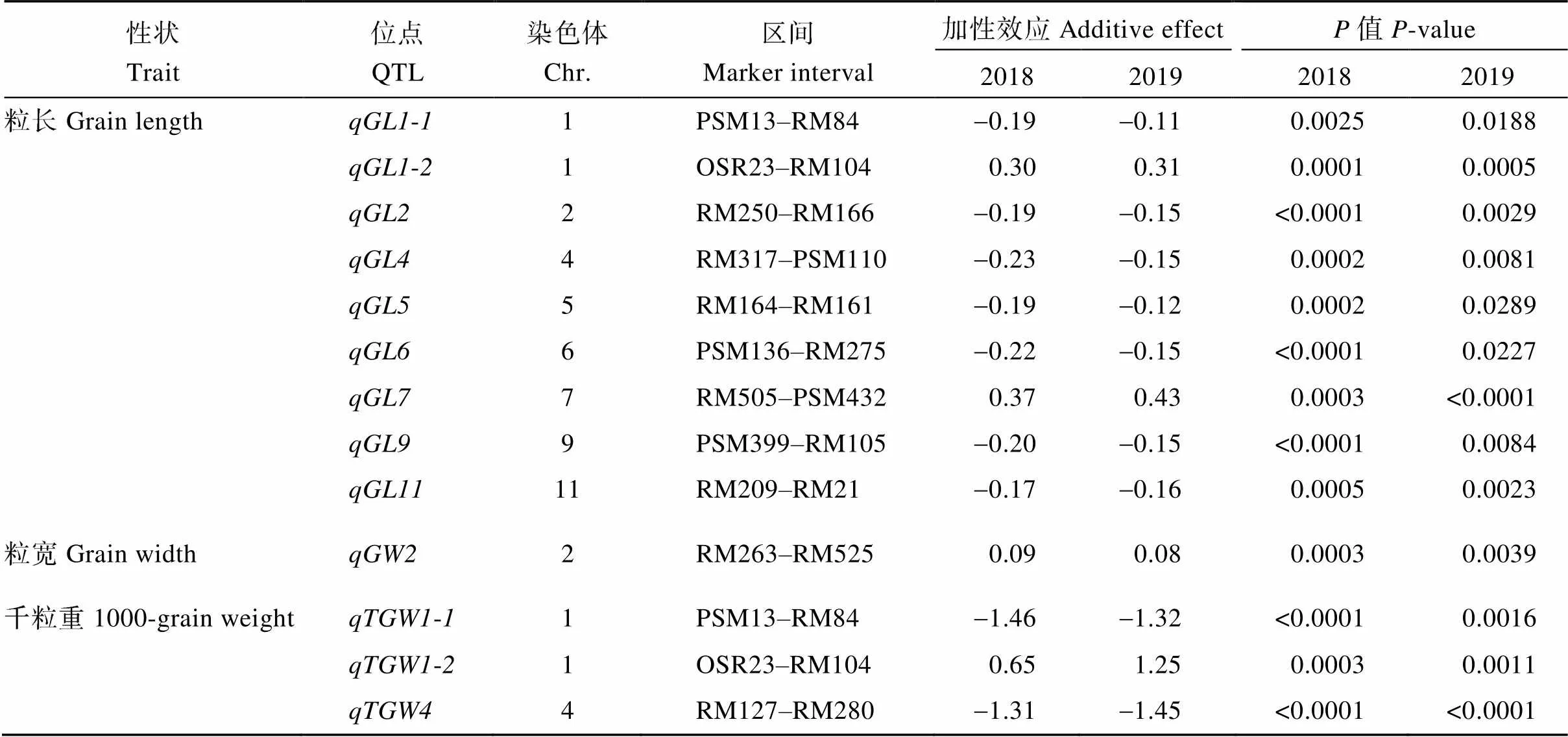
表1 利用SSSL定位到的粒型QTL及其加性效應(yīng)
本研究定位的多個(gè)QTL與前人已有的定位區(qū)間重疊, 部分QTL所定位的區(qū)間內(nèi)已有控制水稻粒型的主效基因被克隆。定位于1號(hào)染色體上的-和-與前人定位到的[36]位于同一區(qū)間內(nèi), 可能為相同的QTL。定位于2號(hào)染色體上的與已有的研究中定位的[37]、[38]和-[39]等存在定位區(qū)間的重疊, 該區(qū)間里包含影響粒型的[40],編碼轉(zhuǎn)錄激活因子, 通過(guò)影響調(diào)控細(xì)胞增殖和伸長(zhǎng)基因的表達(dá), 調(diào)控水稻籽粒大小。本研究定位到的與-[2]-[41]等QTL所在區(qū)間重疊, 該區(qū)間存在一個(gè)已克隆的、負(fù)向調(diào)控水稻粒寬和粒重的[42], 該基因的編碼蛋白與調(diào)控細(xì)胞周期的KRP1蛋白存在互作, 影響細(xì)胞增殖和細(xì)胞生長(zhǎng), 但我們定位到的主要控制水稻粒長(zhǎng), 二者效應(yīng)不同, 也有可能是一個(gè)控制水稻粒長(zhǎng)的新位點(diǎn)。在我們定位的位于4號(hào)染色體上的所在區(qū)間, 已有[2]、-[43]以及[41]等QTL報(bào)道; 位于4號(hào)染色體上主要控制千粒重的則與已克隆的[44]位于同一區(qū)間內(nèi),調(diào)控水稻籽粒大小, 進(jìn)而影響水稻的千粒重與單株產(chǎn)量, 二者遺傳效應(yīng)相似, 極有可能為同一基因。5號(hào)染色體上的與鄒德堂等[37]定位的有重疊部分, 該區(qū)間涵蓋一個(gè)編碼轉(zhuǎn)錄因子的,是RPBF和GBSSI的直接調(diào)控因子, 能夠負(fù)調(diào)控水稻籽粒大小[40], 可能是的候選基因。定位于6號(hào)染色體, 其所在區(qū)間內(nèi)有已被克隆的、調(diào)控水稻粒長(zhǎng)和粒重的[24], 但I(xiàn)APAR9的與HJX74并無(wú)功能位點(diǎn)上的序列變異, 這暗示了該區(qū)間內(nèi)存在一個(gè)尚未被發(fā)現(xiàn)的水稻粒型新位點(diǎn)。[26]/[27]是位于水稻7號(hào)染色體, 控制粒長(zhǎng)和粒寬的主效QTL, 序列分析表明, IAPAR9攜帶功能變異位點(diǎn), 本研究定位的極有可能與[26]/[27]為同一位點(diǎn)。定位于9號(hào)染色體上的與已報(bào)道的[2]所在區(qū)間重疊。位于4號(hào)染色體長(zhǎng)臂末端, 該位點(diǎn)沒(méi)有發(fā)現(xiàn)粒型、粒寬的變化, 千粒重的變化可能由于谷粒厚度或灌漿飽滿程度的影響造成的。-、-和等3個(gè)QTL所在區(qū)間尚未有利用作圖群體定位到粒型QTL的報(bào)道, 其中,-和-共定位于水稻1號(hào)染色體長(zhǎng)臂末端, 該位點(diǎn)可能存在一個(gè)協(xié)同增加水稻粒長(zhǎng)和千粒重的正效應(yīng)因子, 有望為稻米產(chǎn)量與品質(zhì)協(xié)同改良提供新的種質(zhì)資源。上述發(fā)現(xiàn)豐富了我們對(duì)水稻粒型遺傳調(diào)控的認(rèn)識(shí)。
IAPAR9攜帶[16]、[35]、[17]、[21-22]、[23]和[26]等多個(gè)粒型基因功能變異, 但是由于本研究所利用的SSSL材料中的代換片段未能完全覆蓋水稻的整個(gè)染色體組, 特別是[16]、[35]、[21,22]、[23]和[29]所在區(qū)間均未被代換片段覆蓋(圖3), 上述基因位點(diǎn)并未在本研究中被檢測(cè)出來(lái); 而7號(hào)染色體上具有覆蓋粒型主效基因[26]/[27]所在區(qū)間的代換片段, 在該區(qū)間鑒定到控制粒長(zhǎng)的,在2個(gè)檢測(cè)季節(jié)中均是具有最高貢獻(xiàn)率的粒長(zhǎng)QTL。綜上, 只要具有足夠的代換片段覆蓋度, 利用SSSL材料可以全面解析控制相關(guān)農(nóng)藝性狀的QTL。另外, SSSL也是研究復(fù)雜性狀QTL的理想材料, 與受體親本相比, SSSL只攜帶一個(gè)來(lái)自供體的染色體片段, 其遺傳背景與受體親本高度相似, 是受體親本的天然近等基因系, 能夠極大限度地排除遺傳背景干擾[31], 這將為后續(xù)基因的克隆與功能解析提供扎實(shí)的材料基礎(chǔ)。而且, 本團(tuán)隊(duì)已經(jīng)建成HJX74背景下, 供體涵蓋稻屬AA基因組7個(gè)種的SSSL文庫(kù), 全部代換片段大約覆蓋水稻基因組29倍[30], 為功能基因的等位分析提供了充分的材料保障。基于SSSL材料建立的設(shè)計(jì)育種體系[45-46], 則進(jìn)一步促進(jìn)了發(fā)掘到的優(yōu)良基因資源在水稻設(shè)計(jì)育種中的應(yīng)用。
4 結(jié)論
本研究利用153份以巴西陸稻IAPAR9為供體親本秈稻品種HJX74為受體親本構(gòu)建的SSSL材料, 在2018年晚季和2019年早季鑒定水稻粒長(zhǎng)、粒寬和千粒重QTL, 本研究共鑒定到13個(gè)在2個(gè)不同季節(jié)穩(wěn)定遺傳的QTL, 包括9個(gè)粒長(zhǎng)QTL、1個(gè)粒寬QTL和3個(gè)千粒重QTL。其中, 僅和所在的定位區(qū)間有已克隆的粒型相關(guān)基因被報(bào)道。本研究結(jié)果為新粒型基因進(jìn)一步精細(xì)定位、克隆和功能解析奠定了良好的基礎(chǔ), 獲得的優(yōu)異種質(zhì)資源將有助于高產(chǎn)優(yōu)質(zhì)水稻設(shè)計(jì)育種。
[1] 楊維豐, 詹鵬麟, 林少俊, 茍亞軍, 張桂權(quán), 王少奎. 水稻粒形的遺傳研究進(jìn)展. 華南農(nóng)業(yè)大學(xué)學(xué)報(bào), 2019, 40(5): 203–210. Yang W F, Zhan P L, Lin S J, Gou Y J, Zhang G Q, Wang S K. Research progress of grain shape genetics in rice., 2019, 40(5): 203–210 (in Chinese with English abstract).
[2] 侯俊, 周浩, 高冠軍, 李平波, 何予卿, 張慶路. 利用F2和F3群體定位水稻粒形和千粒重QTL. 分子植物育種, 2019, 17: 3938–3944. Hou J, Zhou H, Gao G J, Li P B, He Y Q, Zhang Q L. Mapping QTL for rice grain shape and 1000-grain weight using F2and F3populations., 2019, 17: 3938–3944 (in Chinese with English abstract).
[3] 李永洪, 李傳旭, 劉成元, 何珊, 向箭宇, 謝戎. 利用崗46 B/A 232 RILs群體定位秈稻粒形和粒重的QTL. 分子植物育種, 2018, 16: 3956–3966. Li Y H, Li C X, Liu C Y, He S, Xiang J Y, Xie R. Location of the QTL of grain shape and grain weight ofrice by Gang 46B/A232RILs., 2018, 16: 3956–3966 (in Chinese with English abstract).
[4] 方先文, 張?jiān)戚x, 肖西林, 張所兵, 林靜, 汪迎節(jié). 基于重組自交系群體的水稻粒形QTL定位. 江蘇農(nóng)業(yè)學(xué)報(bào), 2017, 33: 241–247. Fang X W, Zhang Y H, Xiao X L, Zhang S B, Lin J, Wang Y J. Mapping of QTLs for grain shape using recombinant inbred lines in rice (L.)., 2017, 33: 241–247 (in Chinese with English abstract).
[5] 李生強(qiáng), 崔國(guó)昆, 關(guān)成冉, 王俊, 梁國(guó)華. 基于水稻單片段代換系的粒形QTL定位. 中國(guó)水稻科學(xué), 2011, 25: 163–168. Li S Q, Cui G K, Guan C R, Wang J, Liang G H. QTL detection for grain shape using chromosome single segment substitution lines., 2011, 25: 163–168 (in Chinese with English abstract).
[6] 葉少平, 張啟軍, 李杰勤, 趙兵, 李平. 用(培矮64s/Nipponbare) F2群體對(duì)水稻產(chǎn)量構(gòu)成性狀的QTL定位分析. 作物學(xué)報(bào), 2005, 31: 1620–1627. Ye S P, Zhang Q J, Li J Q, Zhao B, Li P. QTL mapping for yield component traits using (Pei’ai 64s/Nipponbare) F2population., 2005, 31: 1620–1627 (in Chinese with English abstract).
[7] 劉冠明, 李文濤, 曾瑞珍, 張澤民, 張桂權(quán). 水稻單片段代換系代換片段的QTL鑒定. 遺傳學(xué)報(bào), 2004, 31: 1395–1400. Liu G M, Li W T, Zeng R Z, Zhang Z M, Zhang G Q. Identification of QTLs on substituted segments in single segment substitution lines of rice., 2004, 31: 1395–1400 (in Chinese with English abstract).
[8] 張麗霞, 劉丕慶, 劉學(xué)義. 染色體單片段代換系的構(gòu)建及應(yīng)用于QTL精細(xì)定位. 分子植物育種, 2004, 2: 743–746. Zhang L X, Liu P Q, Liu X Y. Construction of chromosome single segment substitution lines and QTL fine mapping., 2004, 2: 743–746 (in Chinese with English abstract).
[9] Visscher P M, Brown M A, McCarthy M I, Yang J. Five years of GWAS discovery., 2012, 90: 7–24.
[10] Visscher P M, Wray N R, Zhang Q, Sklar P, McCarthy M I, Brown M A, Yang J. 10 years of GWAS discovery: biology, function, and translation., 2017, 101: 5–22.
[11] Abe A, Kosugi S, Yoshida K, Yoshida K, Natsume S, Takagi H, Kanzaki H, Matsumura H, Yoshida K, Mitsuoka C, Tamiru M, Innan H, Cano L, Kamoun S, Terauchi R. Genome sequencing reveals agronomically important loci in rice using MutMap., 2012, 30: 174–179.
[12] Lyn Y, Wang Y X, Noushin J, Hu H T, Chen P, Shang L G, Lin H Y, Dong G J, Hu J, Gao Z Y, Qiao Q, Zhang Y, Guo L B. Genome-wide association analysis and allelic mining of grain shape-related traits in rice., 2019, 26: 384–392.
[13] 涂坦, 付洪. 水稻主要粒型基因及其遺傳調(diào)控的研究進(jìn)展. 山地農(nóng)業(yè)生物學(xué)報(bào), 2016, 35(5): 58–65. Tu T, Fu H. Research progress in major genes controlling grain shape of rice and their genetic regulation., 2016, 35(5): 58–65 (in Chinese with English abstract).
[14] Song X J, Huan W, Shi M, Zhu M Z, Lin X. A QTL for rice grain width and weight encodes a previously unknown RING-type E3 ubiquitin ligase., 2007, 39: 623–630.
[15] Hu J, Wang Y, Fang Y, Zeng L J, Xu J, Yu P H, Shi Z Y, Pan J, Zhang D, Kang S J, Zhu L, Dong G J, Guo L B, Zeng D L, Zhang G H, Xie L H, Xiong G S, Li J Y, Qian Q. A rare allele ofenhances grain size and grain yield in rice., 2015, 8: 1455–1465.
[16] Fan C C, Xing Y Z, Mao H L, Lu T T, Han B, Xu C G, Li X H, Zhang Q F., a major QTL for grain length and weight and minor QTL for grain width and thickness in rice, encodes a putative transmembrane protein., 2006, 112: 1164–1171.
[17] Qi P, Lin Y S, Song X J, Shen J B, Huang W, Shan J X, Zhu M Z, Jiang L W, Gao J P, Lin H X. The novel quantitative trait locuscontrols rice grain size and yield by regulating Cyclin-T1;3., 2012, 22: 1666–1680.
[18] Xia D, Zhou H, Liu R J, Dan W H, Li P B, Wu B, Chen J X, Wang L Q, Gao G J, Zhang Q L, He Y Q., a novel QTL encoding a GSK3/SHAGGY-like Kinase, epistatically interacts withto Produce extra-long grains in rice., 2018, 11: 754–756.
[19] Hu Z J, Lu S J, Wang M J, He H H, Sun L, Wang H R, Liu X H, Jiang L, Sun J L, Xin X Y, Kong W, Chu C C, Xue H W, Yang J S, Luo X J, Liu J X. A novel QTLencodes the GSK3/SHAGGY-like kinase OsGSK5/OsSK41 that interacts with OsARF4 to negatively regulate grain size and weight in rice., 2018, 11: 736–749.
[20] Ying Z J, Ma M, Bai C, Huang X H, Liu J L, Fan Y Y, Song X J., a major QTL that negatively modulates grain length and weight in rice., 2018, 11: 750–753.
[21] Liu J F, Chen J, Zheng X M, Wu F Q, Lin Q B, Heng Y Q, Tian P, Cheng Z J, Yu X W, Zhou K N, Zhang X, Guo X P, Wang J L, Wang H Y, Wan J M.acts in the brassinosteroid signaling pathway to regulate grain width and weight in rice., 2017, 3: 17043.
[22] Weng J F, Gu S H, Wan X Y, Gao H, Guo T, Su N, Lei C L, Zhang X, Cheng Z J, Guo X P, Wang J L, Jiang L, Zhai H Q, Wan J M. Isolation and initial characterization of, a major QTL associated with rice grain width and weight., 2008, 18: 1199–1209.
[23] Li Y B, Fan C C, Xing Y Z, Jiang Y H, Luo L J, Sun L, Shao D, Xu C J, Li X H, Xiao J H, He Y Q, Zhang Q F. Natural variation inplays an important role in regulating grain size and yield in rice., 2011, 43: 1266–1269.
[24] Ishimaru K, Hirotsu N, Madoka Y, Murakami N, Hara N, Onodera H, Kashiwagi T, Ujiie K, Shimizu B, Onishi A, Miyagawa H, Katoh E. Loss of function of the IAA-glucose hydrolase geneenhances rice grain weight and increases yield., 2013, 45: 707–711.
[25] Shi C L, Dong N Q, Guo T, Ye W W, Shan J X, Lin H X. A quantitative trait locuscontrols rice grain size and yield through the gibberellin pathway., 2020, 103: 1174–1188.
[26] Wang S K, Li S, Liu Q, Wu K, Zhang J Q, Wang S S, Wang Y, Chen X B, Zhang Y, Gao C X, Wang F, Huang H X, Fu X D. The-regulatory module determines grain shape and simultaneously improves rice yield and grain quality., 2015, 47: 949–954.
[27] Wang Y X, Xiong G S, Hu J, Jiang L, Yu H, Xu J, Fang Y X, Zeng L J, Xu E B, Xu J, Ye W J, Meng X B, Liu R F, Chen H Q, Jing Y H, Wang Y H, Zhu X D, Li J Y, Qian Q. Copy number variation at thelocus contributes to grain size diversity in rice., 2015, 47: 944–948.
[28] Si L Z, Chen J Y, Huang X H, Gang H, Luo J H, Hou Q Q, Zhou T Y, Lu T T, Zhu J J, Shang-Guan Y Y, Chen E W, Gong C X, Zhao Q, Jing Y F, Zhao Y, Li Y, Cui L L, Fan D L, Lu Y Q, Weng Q J, Wang Y C, Zhan Q L, Liu K Y, Wei X H, An K, An G, Han B.controls grain size in cultivated rice., 2016, 48: 447–456.
[29] Wang S K, Wu K, Yuan Q B, Liu X Y, Liu Z B, Lin X Y, Zeng R Z, Zhu H T, Dong G J, Qian Q, Zhang G Q, Fu X D. Control of grain size, shape and quality byin rice., 2012, 44: 950–954.
[30] Zhao D S, Li Q F, Zhang C Q, Zhang C, Yang Q Q, Pan L X, Ren X Y, Lu J, Gu M H, Liu Q Q.acts as a transcriptional activator to regulate rice grain shape and appearance quality., 2018, 9:1240.
[31] 張桂權(quán). 基于SSSL文庫(kù)的水稻設(shè)計(jì)育種平臺(tái). 遺傳, 2019, 41: 754–760. Zhang G Q. The platform of breeding by design based on the SSSL library in rice., 2019, 41: 754–760 (in Chinese with English abstract).
[32] Andrews P B, Bishop M, Issar S, Nesmith D, Pfenning F, Xi H W. TPS: a theorem-proving system for classical type theory., 1996, 16: 321–353.
[33] Paterson A H, DeVerna J W, Lanini B, Tanksley S D. Fine mapping of quantitative trait loci using selected overlapping recombinant chromosomes, in an interspecies cross of tomato., 1990, 124: 735–742.
[34] Mackay T F C. Epistasis and quantitative traits: using model organisms to study gene–gene interactions., 2014, 15: 22.
[35] Liu Q, Han R X, Wu K, Zhang J Q, Ye Y F, Wang S S, Chen J F, Pan Y J, Li Q, Xu X P, Zhou J W, Tao D Y, Wu Y J, Fu X D. G-protein βγ subunits determine grain size through interaction with MADS-domain transcription factors in rice., 2018, 9: 852.
[36] 王軍, 周勇, 楊杰, 朱金燕, 范方軍, 李文奇, 梁國(guó)華, 仲維功. 利用染色體單片段代換系定位水稻粒重QTL. 華北農(nóng)學(xué)報(bào), 2013, 28(6): 11–17. Wang J, Zhou Y, Yang J, Zhu J Y, Fan F J, Li W Q, Liang G H, Zhong W G. Mapping of QTLs for grain weight using chromosome single segment substitution lines in rice.-, 2013, 28(6): 11–17 (in Chinese with English abstract).
[37] 鄒德堂, 劉忠良, 趙宏偉, 孫健, 鄭洪亮, 陳賓賓, 劉博文. 利用F2:3和BC2F2群體定位水稻粒型和粒重QTL. 東北農(nóng)業(yè)大學(xué)學(xué)報(bào), 2014, 45(9): 9–17. Zou D T, Liu Z L, Zhao H W, Sun J, Zheng H L, Chen B B, Liu B W. Mapping QTLs for grain shape and weight using F2:3and BC2F2population in rice., 2014, 45(9): 9–17 (in Chinese with English abstract).
[38] 張亞?wèn)|, 張穎慧, 董少玲, 陳濤, 趙慶勇, 朱鎮(zhèn), 周麗慧, 姚姝, 趙凌, 于新, 王才林. 特大粒水稻材料粒型性狀的QTL檢測(cè). 中國(guó)水稻科學(xué), 2013, 27: 122–128. Zhang Y D, Zhang Y H, Dong S L, Chen T, Zhao Q Y, Zhu Z, Zhou L H, Yao S, Zhao L, Yu X, Wang C L. Identification of QTL for rice grain traits based on an extra-large grain material., 2013, 27: 122–128 (in Chinese with English abstract).
[39] 蔣鈺東, 羅俊濤, 況浩池, 楊揚(yáng), 陳光珍, 何興材, 付均, 鄭軍, 曾正明. 利用兩個(gè)秈稻雜交F2定位水稻粒型、粒重QTLs. 分子植物育種, 2015, 13: 1689–1694. Jiang Y D, Luo J T, Kuang H C, Yang Y, Chen G Z, He X C, Fu J, Zheng J, Zeng Z M. Identification of QTLs for grain shape and weight using F2generation of tworice varieties., 2015, 13: 1689–1694 (in Chinese with English abstract).
[40] Yu X Q, X S S, Xu Q K, Cui Y J, Gong M, Zeng D L, Zhang Q, Shen L, Jiao G A, Gao Z Y, Hu J, Zhang G H, Zhu L, Guo L B, Ren D Y, Qian Q.encodes OsMADS6 and determines palea identity and affects rice grain yield and quality., 2020, 63: 228–238.
[41] 王小雷, 李煒星, 曾博虹, 孫曉棠, 歐陽(yáng)林娟, 陳小榮, 賀浩華, 朱昌蘭. 基于染色體片段置換系對(duì)水稻粒形及千粒重QTL檢測(cè)與穩(wěn)定性分析. 作物學(xué)報(bào), 2020, 46: 1517–1525. Wang X L, Li W X, Zeng B H, Sun X T, Ou-Yang L J, Chen X R, He H H, Zhu C L. QTL detection and stability analysis of rice grain shape and thousand-grain weight based on chromosome segment substitution lines., 2020, 46: 1517–1525 (in Chinese with English abstract).
[42] Ruan B P, Shang L G, Zhang B, Hu J, Wang Y X, Lin H, Zhang A P, Liu C L, Peng Y L, Zhu L, Ren D Y, Shen L, Dong G J, Zhang G H, Zeng D L, Guo L B, Qian Q, Gao Z Y. Natural variation in the promoter ofdetermines grain width and weight in rice., 2020, 227: 629–640.
[43] Lu B Y, Yang C Y, Xie K, Zhang L, Wu T, Li L F, Liu X, Jiang L, Wan J M. Quantitative trait loci for grain-quality traits across a rice F2population and backcross inbred lines., 2013, 192: 25–35.
[44] Wu W G, Liu X Y, Wang M H, Meyer R S, Luo X J, Ndjiondjop M N, Tan L B, Zhang J W, Wu J Z, Cai H W, Sun C Q, Wang C Q, Wang X K, Wing R A, Zhu Z F. A single-nucleotide polymorphism causes smaller grain size and loss of seed shattering during African rice domestication., 2017, 3: 17064.
[45] Dai Z J, Lu Q, Luan X, Ou-Yang L, Guo J, Liang J Y, Zhu H T, Wang W J, Wang S K, Zeng R Z, Liu Z Q, Zhang Z M, Zhu X Y, Zhang G Q. Development of a platform for breeding by design of CMS restorer lines based on an SSSL library in rice (L.)., 2016, 66: 768–775.
[46] Luan X, Dai Z J, Yang W F, Tan Q Y, Lu Q, Guo J, Zhu H T, Liu G F, Wang S K, Zhang G Q. Breeding by design of CMS lines on the platform of SSSL library in rice., 2019, 39: 126.
附表1 引物名稱及序列
Table S1 Primers and the sequence of the primers
附表2 單片段代換系代換片段分布
Table S2 The distribution of the substitution segment in the SSSLs
TL: total lengths of the substituted segments; CL: the coverage length of the substituted segments; CP (%): coverage percent. It means the ratio of the contig length of the substitution segments to the length of the same chromosome.
Mapping and identification QTLs controlling grain size in rice (L.) by using single segment substitution lines derived from IAPAR9
ZHANG Bo, PEI Rui-Qing, YANG Wei-Feng, ZHU Hai-Tao, LIU Gui-Fu, ZHANG Gui-Quan, and WANG Shao-Kui*
Guangdong Provincial Key Laboratory of Plant Molecular Breeding / State Key Laboratory for Conservation and Utilization of Subtropical Agro-Bioresources / South China Agricultural University, Guangzhou 510642, Guangdong, China
Rice grain size is a complex quantitative trait controlled by multiple genes. Grain size is an important factor affecting rice yield and quality. The mapping and genetic analysis of genes controlling rice grain size are essential for the concurrent improvement of rice yield and quality. Here 13 QTLs for grain size were detected using 153 rice single-segment substitution lines in rice, which were derived from HJX74 as the receptor parent, and IAPAR9 as the donor parent. One-way ANOVA and Duncan’s multiple comparison were employed to detect the genetic bases of rice grain size in two consecutive years. Based on the substitution mapping using overlapped substitution-fragment in the SSSLs, a total of 13 grain size-related QTLs were detected on chromosomes 1, 2, 4, 5, 6, 7, 9, and 11, including nine QTLs controlling grain length, one QTL controlling grain width, and three QTLs controlling 1000-grain weight. Furthermore,-,-, andwere novel identified QTLs. This study provided new basis for cloning and functional analysis of genes regulating grain size.
rice; single segment substitution line; grain size; QTL mapping
10.3724/SP.J.1006.2021.02056
本研究由國(guó)家重點(diǎn)研發(fā)計(jì)劃項(xiàng)目(2016YFD0100406), 廣東省基礎(chǔ)與應(yīng)用基礎(chǔ)研究重大項(xiàng)目(2019B030302006)和廣東省特支計(jì)劃項(xiàng)目(2016TX03N224)資助。
This study was supported by the National Key Research and Development Program of China (2016YFD0100406), the Key Projects of Basic Research andAppliedBasic Research of Guangdong Province (2019B030302006), and the Special Support Program of Guangdong Province for High-level Talents (2016TX03N224).
王少奎, E-mail: shaokuiwang@scau.edu.cn
E-mail: zhangboscau@163.com
2020-08-17;
2021-01-13;
2021-02-25.
URL: https://kns.cnki.net/kcms/detail/11.1809.S.20210225.1147.006.html

