中國荷斯坦牛肢蹄結構和乳房形態的全基因組關聯分析
王添禎,高 雪,宋文芹,姚大為,陳麗麗,陳成彬*,馬 毅*
(1. 天津市畜牧獸醫研究所,天津 300381;2. 南開大學 生命科學院,天津 300071;3. 中國農業科學院 北京畜牧獸醫研究所,北京 100193)
伴隨全基因組關聯分析(Genome-wide Association Study, GWAS)在人類[1]和家畜[2]中的應用,許多與復雜疾病和數量性狀相關的候選基因和因果變異位點被鑒定。該方法不僅有助于解析復雜表型的分子遺傳機制,同時有助于促進基因組選擇的應用及加快遺傳改良速度[3-4]。中國荷斯坦牛是我國主要奶牛品種,目前不少研究報道對中國荷斯坦牛的產奶量[5]、乳脂等[6]進行了GWAS分析,確定了多個候選基因以及數量性狀位點(Quantitative trait locus, QTLs)。然而,其他重要的經濟及健康性狀尚研究不足。
奶牛體型性狀包含肢蹄結構(包括蹄角度、肢蹄評分以及整體的肢蹄結構評分)和乳房形態(包括乳房深度、后乳頭位置、乳頭長度、后乳房寬度、前乳房附著、后乳房高度以及整體的乳房形態評分)。具體的評分標準參照美國2018年荷斯坦牛的評分標準。肢蹄病是常見的疾病,可以造成生產性能下降;乳房形態(乳房長、寬、基部圍等)則與產奶量呈中等以上正相關。在過去幾十年里,研究者主要關注產奶性狀,利用基因組選擇(Genetic Selection,GS)、調整牛場管理模式以及改良飼料配方等多種策略,使奶牛的產奶量實現快速增長。當前,奶牛育種更加關注平衡育種,體型性狀作為奶牛的重要性狀,影響奶牛的健康以及產奶量等重要經濟指標,因此體型性狀也逐漸被重視。然而體型性狀的遺傳基礎研究國內外報道仍然較少。本研究旨在進行中國荷斯坦牛肢蹄結構與乳房形態兩個性狀的全基因組關聯分析,確定顯著的SNP候選位點,為進一步開展奶牛分子育種提供參考。
1 材料與方法
1.1 樣本基因型和質量控制
選取天津地區奶牛場中2014年出生的300頭中國荷斯坦母牛(使用8個父系凍精,從PCA的結果中也可以看出,樣本大致可分為3個家系)進行血液樣本采集,并使用TiangenTIANamp Blood基因組提取試劑盒提取全基因組DNA,采用GeneSeek Genomic Profiler Bovine 50 K SNP chip芯片對所有個體進行基因分型,SNP均勻分布在牛基因組上,平均標記密度為59 kb。
用PLINK v1.9[7]軟件進行基因型數據的質量控制,包括個體水平和SNPs水平兩個方面:(1)刪除檢出率低于94%的個體數據;刪除所有基因型數據缺失超過10%的個體;去除缺失基因型超過1%、最小等位基因頻率小于5%和哈代-溫伯格平衡檢驗P值小于1.0×10-7的SNP數據。經過質量控制后,共有274個樣本的40 501個高質量SNPs用于GWAS分析,占總SNPs的84.7%,其所在牛染色體上的分布情況如表1所示。
1.2 預期傳遞力
根據美國農業部奶牛育種委員會(The Council of Dairy Cattle Breeding, CDCB, https://www.uscdcb.com/)2018年更新的標準方法計算得到預期傳遞力(Predicted transfer Ability, PTA)作為表型[8],對體型性狀中的肢蹄結構、乳房形態性狀進行全基因組關聯分析。

表1 質控后SNPs的分布和相鄰SNPs的平均距離
1.3 統計分析
使用PLINK (v1.9)進行群體主成分分析(Principal Components Aalysis,PCA),對膨脹系數(λ)進行評估。根據PCA結果對群體進行校正,避免因群體分層而導致較高的假陽性[9]。采用EMMAX[10]軟件中基于混合線性模型[11]的單標記回歸方法進行全基因組關聯分析,建立模型如下:
y=Xβ+Zυ+e
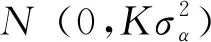
1.4 關聯分析的顯著性檢驗
采用Bonferroni檢驗校正。單次檢驗的顯著性水平為:染色體水平顯著(1/N)、全基因組水平顯著(0.05/N),其中N為質控后可用SNPs數為40 501。即染色體水平顯著閾值為(1/40 501=2.47×10-5),全基因組水平顯著(0.05/40 501=1.23×10-6)。
1.5 群體分層檢驗
利用R v3.5.1[12]繪制Q-Q圖(Quantile-Quantile plot)進行群體分層檢驗。以此來判斷樣本群體是否存在群體分層及分析性狀顯著相關的SNPs。
1.6 候選基因分析策略
基于芯片對應的Bos_taurus genome UMD 3.1[4]在線數據庫(http://genome-asia.-ucsc. edu/),在顯著SNPs的上、下游各50 kb區域內進行候選基因的篩查,并對基因功能進行注釋確定影響奶牛肢蹄結構和乳房形態性狀的候選基因。
2 結 果
2.1 全基因組關聯分析結果
通過對芯片數據進行嚴格的質量控制,共得到274頭母牛40 501個SNPs位點的基因型結構。對中國荷斯坦牛肢蹄結構、乳房形態這兩個性狀進行全基因組關聯分析。利用R(3.5.1)繪制全基因組范圍SNPs的-log10(P-value)分布曼哈頓圖,如圖1所示。通過Bonferroni校正,對于肢蹄結構共檢測到8個染色體水平顯著(P<1/40 501)SNPs位點,對于乳房形態共檢測到16個染色體水平顯著SNPs位點和1個全基因組水平顯著(P<0.05/40 501)位點。
2.2 群體分層檢驗結果
主成分分析結果如圖2,從圖中可以看出,樣本群體可分為3個組,在全基因組關聯分析中,把群體親緣關系作為效應考慮進模型中,能夠避免因群體分層導致的假陽性結果。從Q-Q 圖結果(圖3)中顯示通過SNPs關聯分析計算的χ2統計量分布與假設檢驗沒有過早偏離,說明采用的分析模型是合理的。而在圖形的右上角則是顯著較高的位點,是潛在與性狀相關的候選位點。
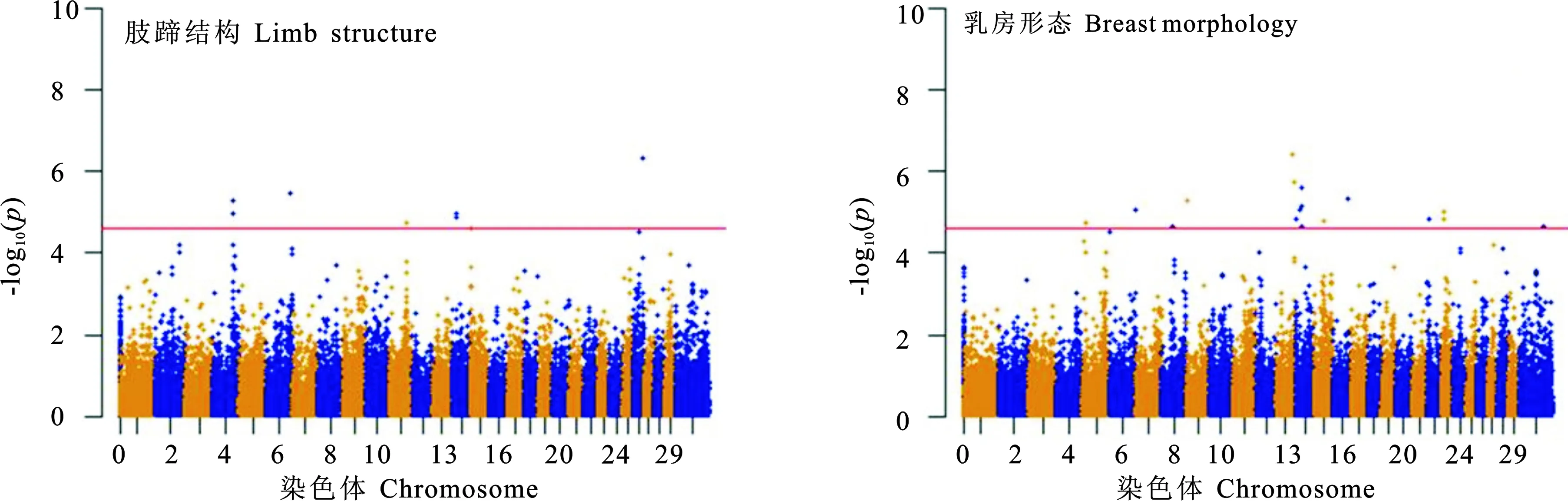
圖1 全基因組SNPs分布散點圖
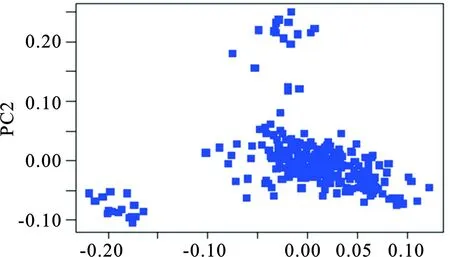
圖2 主成分分析圖
2.3 顯著SNPs及相關基因
通過Bonferroni校正,共檢測出25個顯著SNPs,其中8個與肢蹄結構相關,顯著SNP分別位于4(2個)、6(1個)、11(1個)、14(2個)、15(1個)、26(1個)等6條染色體上;17個與乳房形態相關,顯著SNP分別位于5(1個)、6(1個)、8(1個)、9(1個)、13(2個)、14(5個)、15(1個)、16(1個)、22(1個)、23(3個)等10條染色體上,其中2個位于基因內部。對25個顯著SNPs位點進行分析注釋,得到22個與其距離最近的候選基因。對候選基因進行注釋,其中有3個編碼microRNA,19個蛋白質編碼類基因(見表2)。
3 討 論
3.1 預期傳遞力
在GWAS中,表型校正的方式有所不同,有些研究采用EBV[12-14]; 然而美國研究機構的大部分學者采用PTA[15-17]育種值作為表型進行關聯分析。本研究采用PTA作為表型,可以有效排除其它環境效應的干擾[18],通過計算PTA估計基因組的遺傳效應。出生奶牛的初始PTA為父母的均值,隨著后期成長,綜合利用表型及多層面信息來校正PTA,PTA的參考群體每五年進行更新以保證數據的可靠性。
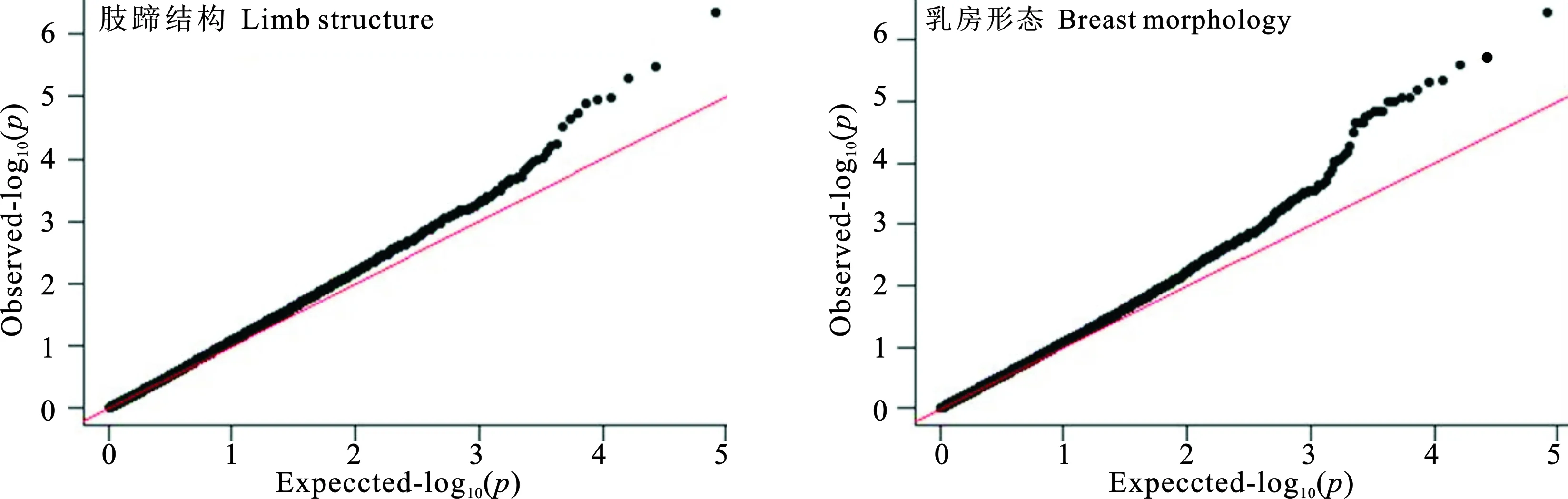
圖3 EMMAX方法結果的QQ圖
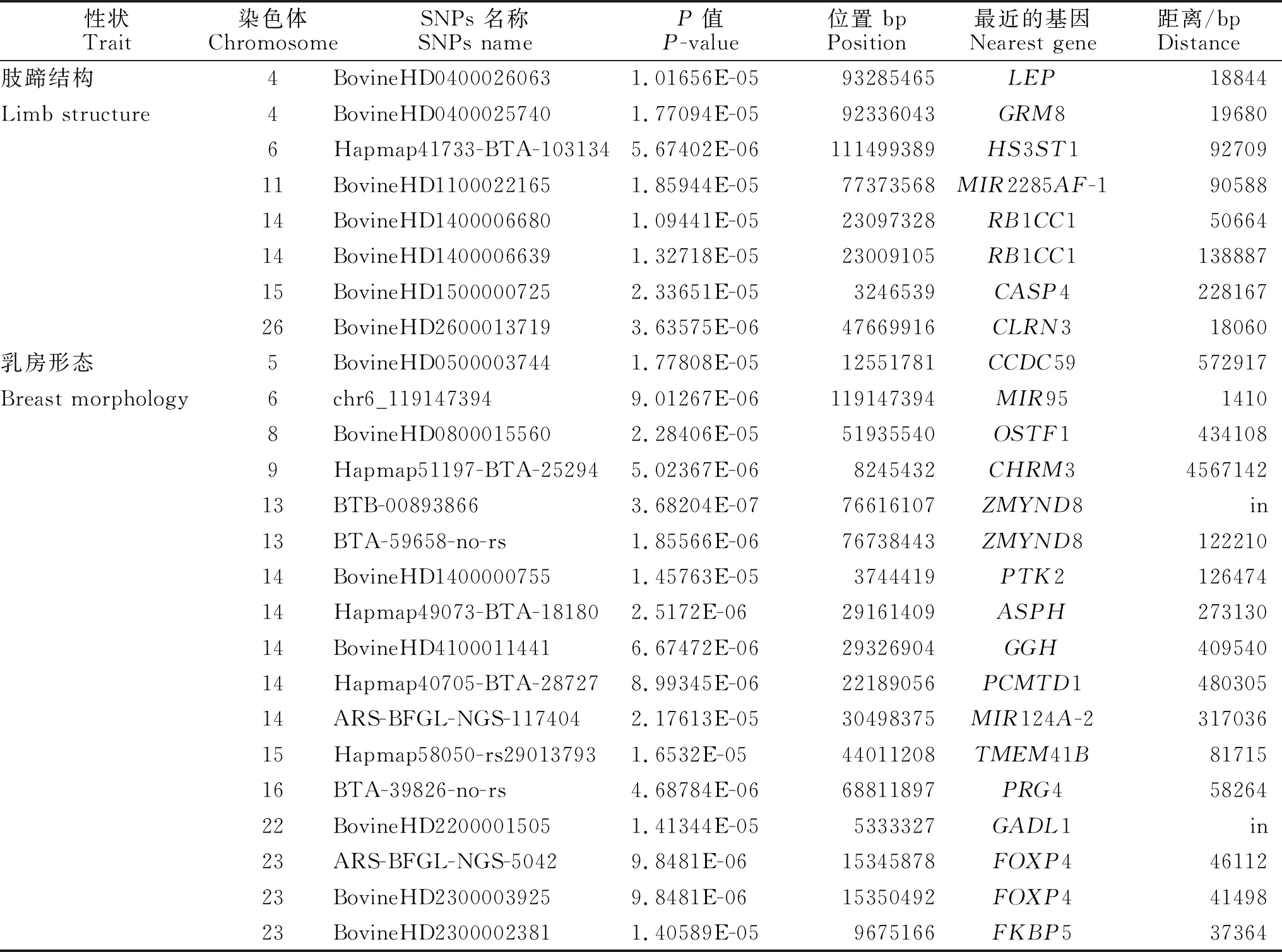
表2 中國荷斯坦牛肢蹄結構和乳房形態全基因組關聯分析顯著SNPs和相關基因
3.2 全基因組關聯分析
本研究采用GeneSeek Genomic Profiler Bovine 50 K SNP芯片對中國荷斯坦牛的肢蹄結構和乳房形態兩個性狀進行了全基因組關聯分析。利用PLINK軟件進行主成分分析,前兩個主成分累計大于93%,基于前兩個組分進行群體分層分析,研究發現樣本個體大致可分為3組。因此,為了提高檢測多效應遺傳變異的效力及避免關聯分析的假陽性[19],在GWAS分析中,本研究將群體分層和SNP效應作為固定效應,親緣關系矩陣作為隨機效應[20-21]有效地提高了關聯分析的檢測效力。
3.3 候選基因功能注釋
通過數據庫對候選基因進行功能注釋,發現多個涉及代謝通路調節的候選基因,例如:LEP、HS3ST1、RB1CC1、ASPH與生長發育相關,OSTF1、FKBP5參與免疫調節,ZMYND8、PTK2與產奶性狀相關。
LEP(瘦素)是一種主要由脂肪細胞合成和分泌的肽激素,位于4號染色體上,在胎盤、胃和骨骼肌等組織中表達,參與多種信號通路,如脂質代謝、葡萄糖轉運、胰島素分泌[22]等,在調節能量穩態中起著至關重要的作用。例如:LEP參與調節5'-AMP活化的蛋白激酶(AMPK)的信號傳導。AMPK作為能量傳感器,響應AMP與ATP比率的增加而激活。活化的AMPK通過調節脂肪酸生物合成-乙酰輔酶A羧化酶(ACC)的活性來調節脂肪酸生物合成。LEP表達量高的動物,其日增重可能比其他飼喂量和營養狀況相似的動物要低,產犢期也可能更長[23],在牛皮膚組織中,LEP通過旁分泌參與控制表皮生長和毛囊循環[24]。
OSTF1在細胞溶菌產物中表達,具有特殊的吸收功能,參與某些局部炎癥病灶的吸收。在荷斯坦奶牛中,OSTF1有5種基因型(AA、AB、BB、AC、CC),其中BB型的牛奶中具有較低的體細胞,表明OSTF1的BB基因型可以作為乳房炎抗性遺傳標記[25]。
ZMYND8是一個蛋白質編碼基因,轉錄調控網絡的關鍵組成部分。在同源小鼠中,注射ZMYND8過表達的浸潤性乳腺癌細胞,腫瘤體積和重量減小,與正常組織相比,乳腺癌組織中ZMYND8顯著下調[26]。Venturini等[27]通過GWAS發現有一個SNP位于ZMYND8基因內,并與產奶量、乳脂量、乳蛋白量顯著相關。
PTK2作為整合素信號傳導的主要介質,已被發現對乳腺上皮細胞的存活、增殖和分化具有重要作用[28],可能參與幾種信號轉導途徑,如細胞運動[29]、微管穩定[30]、細胞與細胞連接的調節[31]。此外,在維持乳腺發育和體內功能方面發揮著重要作用[32]。Wang等[33]在GWAS分析中也鑒定出了PTK2與產奶性狀相關,并通過后續的試驗驗證了不同的突變體對PTK2的表達量有顯著相關,在所有檢測到的組織中均有表達,在乳腺,子宮和腎臟中表達水平較高,TT基因型的乳腺中表達量最高。
此外,Kim等[34]鑒定出HS3ST1與牛的胴體重量相關,Magalhaes等[35]鑒定出RB1CC1與生長和肌肉發育相關。ASPH在牛中發現與肌肉肥大有關[36],并且是胴體性狀的候選基因[37]。FKBP參與TNFα/NF-kB信號通路,是宿主對疾病和其它有害應激物免疫反應的主要途徑[14]。
4 結 論
本研究通過全基因組關聯分析,檢測了影響中國荷斯坦牛肢蹄結構和乳房形態的候選基因,包括LEP、HS3ST1、RB1CC1、ASPH、OSTF1、FKBP5、ZMYND8、PTK2等,為揭示奶牛肢蹄結構和乳房形態的分子遺傳基礎提供了重要參考。

