茶樹Gro/Tup1基因家族鑒定及外源激素和非生物脅迫下表達分析
詹冬梅 朱晨,2 周承哲,2 黃雪婷 賴鐘雄,2 郭玉瓊,2
(1. 福建農林大學園藝學院/茶學福建省高校重點實驗室/茶產業研究院,福州 350002;2. 福建農林大學園藝植物生物工程研究所,福州 350002)
植物中存在大量輔轉錄調控因子,其包含輔抑制因子和輔激活因子,能夠輔助控制植物基因表達、調控植物生長發育以及抵御不利環境影響[1-3]。Groucho/Thymidine uptake 1(Gro/Tup1)家族是其中一類重要的輔轉錄調控因子,Gro/Tup1蛋白缺乏獨立與DNA結合能力,只能通過被轉錄因子招募來輔助調節靶基因的表達[4]。Gro/Tup1包含兩個亞家族,分 別 為 TPL/TPR(TOPLESS/TOPLESS-related) 和LUG/LUH(LEUNIG/LEUNIG HOMOLOG)。兩個亞家族成員都具有N端富含谷氨酰胺的lissencephaly homology(LisH) 結 構 域(PF08513) 和 C端 的WD40結構域(PF00400),區別在于兩個家族成員的蛋白質互作區域結構不同,TPL/TPR具有CTLH結構域(PF10607),而LUG/LUH結構中央具有更多的谷氨酰胺(Q)富集區域(Q-rich)[5-7]。
目前,在擬南芥[8]、番茄[9]、玉米[10]等植物中已對Gro/Tup1家族基因進行鑒定分析,結果表明,在外源MeJA噴施處理下,玉米嫩枝ZmTPL3表達水平上調,ZmLUG2,ZmLUG5,ZmLUG6表達水平下調。在蛋白水平上同樣驗證了Gro/Tup1家族參與多種激素信號轉導途徑,如TPL蛋白亞型可與大多數Aux/IAA蛋白相互作用,在缺乏生長素的情況下,AUX/IAA蛋白通過招募TPL來抑制生長素應答基因表達[11]。TPL通過與AFP蛋白相互作用參與ABA信號通路,而ABA與JA信號通路在干旱時協同抑制植物生長和產量[12]。TPL蛋白還可以與JAZ以及NINJA蛋白形成JAZ-NINJA-TPL蛋白復合物抑制JA信號轉導通路中的MYC2轉錄因子的表達[13],而LUH正向調控依賴于MYC2的JA應答基因的轉錄[14]。大量研究表明Gro/Tup1參與植物應激反應,如擬南芥TPL相互作用組的研究表明,TPR3和TIR-NB-LRR SNC1蛋白之間相互作用,且TPL/TPR家族參與非生物脅迫響應[15]。擬南芥lug突變體的全基因組轉錄組比較研究發現,一些非生物脅迫基因的表達水平發生顯著變化[16]。HOS15、LUH和TPR1的相關研究也證明植物Gro/Tup1參與應答鹽脅迫、滲透調節及凍害防御過程[17-19]。
由此可見,在基因水平和蛋白水平上都證明Gro/Tup1家族成員在植物激素轉導及抵御外界不良環境影響過程中都具有重要作用。茶樹(Camellia sinensis)是我國重要的經濟作物,對我國的農業和國民經濟發展具有重要作用[20],隨著茶樹栽培面積的擴大,由干旱、寒冷等環境脅迫引起的茶芽萌發遲、長勢差,營養積累困難等問題增加,導致茶葉產量減少和品質下降[21-22]。目前在茶樹中尚未見Gro/Tup1家族相關報道,而茶樹基因組測序數據的發布有利于從全基因組水平對茶樹Gro/Tup1家族成員進行鑒定分析[23-27],基于茶樹基因組所進行的基因家族鑒定的研究已有許多[28-29]。本研究利用茶樹基因組數據[30],鑒定茶樹中所有的Gro/Tup1基因,明確在茶樹中的結構特點與進化特征,通過實時熒光定量(RT-qPCR)分析其在多種外源植物激素(MeJA,GA3和ABA)和非生物脅迫(干旱和低溫)處理下的表達模式,以期探究該基因家族成員在茶樹中的作用機制,同時為進一步研究Gro/Tup1家族基因在茶樹激素信號傳導途徑中的作用奠定基礎。
1 材料與方法
1.1 材料
2019年11月,以8年生盆栽鐵觀音茶樹為供試材料,置于溫度(25±2)℃、相對濕度70%、光暗周期為14 h/10 h的人工氣候箱中培養。隨后參考Zhou等[31]的方法,對茶樹進行外源激素、低溫和干旱處理。在對照未處理、外源激素處理和低溫處理后的0、3、6、12、24 h分別收集茶樹第二葉位為試驗材料。上述取樣進行3次重復,所有處理樣品采集后迅速用液氮固樣,然后轉移至-80℃冰箱保存待用。具體處理如下:
(1)激素處理:將若干盆茶樹分為4組,其中3組分別對茶樹葉片噴灑1 mmol/L MeJA、1 mmol/L GA3和100 μmol/L ABA新鮮制備的工作溶液,剩余一組不做處理,對照組。
(2)低溫處理:其他條件保持不變,將盆栽茶樹移入保持在4℃的人工氣候室中。
(3)干旱處理:若干盆栽茶樹分成4組,分別在正常供水(土壤水分含量為19.50%;CK)、輕度干旱脅迫(土壤水分=15.20%;T1)、中度干旱脅迫(土壤水分= 10.17%;T2)和重度干旱脅迫(土壤水分= 5.54%;T3)下培養10 d后,收集葉片。
1.2 方法
1.2.1 茶樹Gro/Tup1基因家族成員鑒定 在擬南芥基因組數據庫 TAIR(https://www.arabidopsis.org/)下載擬南芥Gro/Tup1氨基酸序列,從TPIA(http://tpia.teaplant.org/)數據庫下載茶樹基因組數據。用已鑒定出的擬南芥Gro/Tup1氨基酸序列為種子序列,利用本地BLASTP對茶樹基因組進行對比搜索(E-value≤ 10-5),得到候選茶樹Gro/Tup1成員氨基酸序列。利用HMMER 3.0軟件對Gro/Tup1在Pfam數據庫中的功能域(PF08513)隱馬可夫模型進行鑒定。最后通 過 CDD(https://www.ncbi.nlm.nih.gov/St-ructure/cdd/wrpsb.cgi?tdsourcetag=s_pctim_aiomsg)和SMART(http://smart.embl-heidelberg.de/) 驗 證 候 選 蛋 白結構域,以確定其含有LisH(PF08513)和WD40(PF00400)保守結構域。利用Proparam(https://www.expasy.org/)工具對茶樹Gro/Tup1蛋白進行理化性質分析;通過WoLF PSORT Web網站(https://wolfpsort.hgc.jp/)對其編碼蛋白的亞細胞定位進行預測。
1.2.2 茶樹Gro/Tup1基因家族進化分析 從phytozome數據庫下載擬南芥(Arabidopsis thalian)、番 茄(Solanum lycopersicum)、 水 稻(Oryza sativa)的Gro/Tup1氨基酸序列,從茶樹基因組數據庫(TPIA)下載茶樹Gro/Tup1氨基酸序列。利用ClustalW對上述物種和茶樹Gro/Tup1蛋白序列進行多序列比對,通過MEGA X軟件構建上述物種系統進化樹,進化樹構建方法為鄰近法(neighbor-joining,NJ),校驗參數為Bootstrap=1 000,并在iTOL網站(https://itol.embl.de/)進行可視化處理。
1.2.3 茶樹Gro/Tup1基因家族結構分析 利用MEGA X軟件構建茶樹和擬南芥Gro/Tup1系統進化樹,參數同1.2.2;利用MEME網站(http://meme-suite.org/tools/meme)分析茶樹Gro/Tup1家族成員的保守基序,基序數量設置為10;從TPIA數據庫中下載茶樹基因結構注釋文件(GFF3);最后通過TBtools軟件對Gro/Tup1系統進化樹、基序分布以及內含子-外顯子分布進行可視化處理。
1.2.4 茶樹Gro/Tup1基因家族順式作用元件分析 通過Tbtools軟件提取茶樹Gro/Tup1家族成員起始密碼子上游2 000 bp的基因序列,利用啟動子在線預測軟件PlantCARE(http://bioinformatics.psb.ugent.be/webtoo-ls/plantcare/html/)對其順式元件進行預測分析。
1.2.5 茶樹Gro/Tup1基因家族表達分析 使用RNAprep Pure Plant Kit(天根生化科技(北京)有限公司)提取樣品總RNA;使用EasyScript One-Step gDNA Removal and cDNA Synthesis SuperMix Kit(上海翊圣生物科技有限公司)試劑盒逆轉錄合成cDNA;使用Primer Premier v6.0軟件設計Gro/Tup1的特異性引物,并使用DNAMAN 9軟件進行驗證,引物序列見表1。以CsSAND作為干旱處理組樣品的內參基因,Csβ-actin作為低溫和激素處理組的內參基因。利用LightCycler 480(Roche應用科學公司)儀器進行RT-qPCR反應,反應體系使用Hieff qPCR SYBR Green Master Mix(上海翊圣生物科技有限公司)試劑盒的方法,設置3次生物學重復。用2-△△Ct算法計算Gro/Tup1基因相對表達量。用EXCEL 2013、GraphPad Prism v6.01和SPSS v22.0軟件進行統計分析。

表1 RT-qPCR所用引物序列Table 1 Primer sequences used in RT-qPCR
2 結果
2.1 茶樹Gro/Tup1基因家族鑒定
根據擬南芥Gro/Tup1蛋白的序列信息,在茶樹基因組中檢索并分析,最終獲得13個茶樹Gro/Tup1基因。通過CCD和Tbtools軟件對所獲得的蛋白序列進行分析(圖1),所有的候選蛋白的N端都含有LisH結構域,C末端含有WD40或WD40 superfamily結構域,而CsTPL1-9在LisH結構域后還含有CTLH。根據蛋白結構的分布以及前人研究,將茶樹Gro/Tup1分為TPL/TPR和LUG/LUH兩個亞家族。
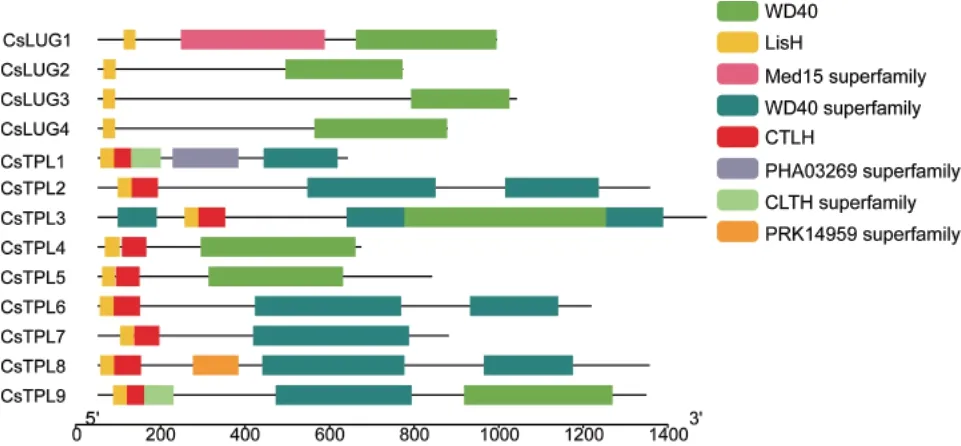
圖1 茶樹Gro/Tup1蛋白保守結構域分布Fig. 1 Conserved domains of CsGro/Tup1 proteins in C. sinensis
根據scaffold 編號順序和結構域組成,將其以CsLUG1-4、CsTPL1-9的方式進行命名(表2)。分析表明,CsGro/Tup1氨基酸長度在540-1 319aa,分子量在60.182-145.850 kD,等電點在6.08-8.17。其中CsTPL1、3、4、6和9共5種蛋白質不穩定指數小于40,屬于穩定蛋白質,其余8種蛋白質指數均大于40,屬于不穩定蛋白質。CsGro/Tup1家族蛋白均為親水性蛋白。亞細胞定位預測結果顯示,CsLUG1位于質體,CsTPL6位于內質網,CsTPL4和CsTPL8位于葉綠體中,其余的9種蛋白質均位于細胞核,推測Gro/Tup1家族蛋白主要分布于茶樹細胞核中。
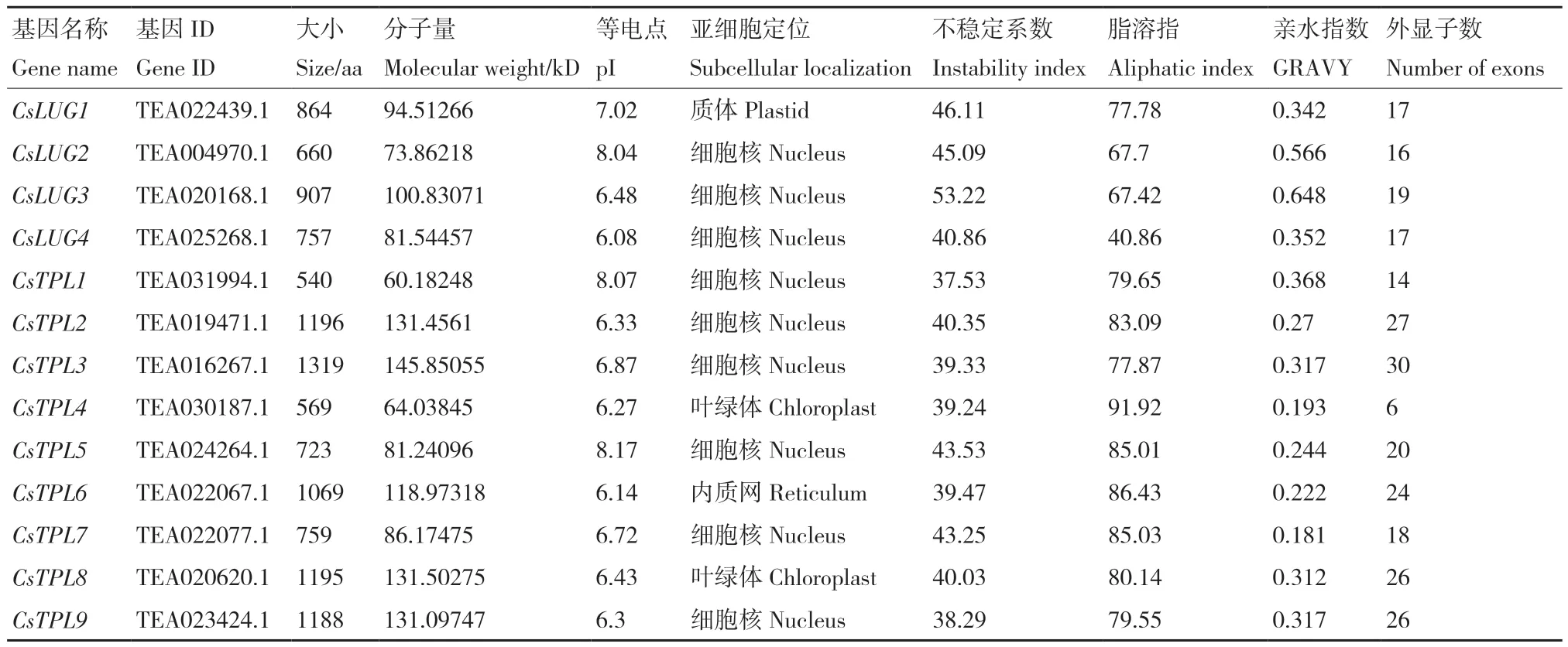
表2 茶樹Gro/Tup1蛋白質序列特征Table 2 Feature of CsGro/Tup1 protein in C. sinensis
2.2 茶樹Gro/Tup1家族進化及結構分析
為了解Gro/Tup1蛋白在不同物種間的進化關系,采用MEGA X軟件對茶樹(13)、擬南芥(12)、番茄(17)以及水稻(11)中共53個Gro/Tup1氨基酸序列進行系統進化分析(圖2)。結果表明53個Gro/Tup1成員被分成兩組:TPL/TPR和LUG/LUH兩個亞家族。雙子葉植物茶樹、擬南芥及番茄的大部分Gro/Tup1成員聚在同一分支上,單子葉植物水稻的Gro/Tup1大部分成員單獨聚為一個小分支。其中單子葉植物中未鑒定到TPL/TPR成員,僅有11個LUG/LUH成員;雙子葉植物中TPL/TPR成員數量多于LUG/LUH,如擬南芥具有9個TPL/TPR,3個 LUG/LUH, 番 茄 具 有 10個 TPL/TPR,7個LUG/LUH,茶樹具有9個TPL/TPR,3個LUG/LUH。與玉米Gro/Tup1基因家族研究一致,雙子葉植物的TPL/TPR成員數量多于LUG/LUH,單子葉植物反之[10]。
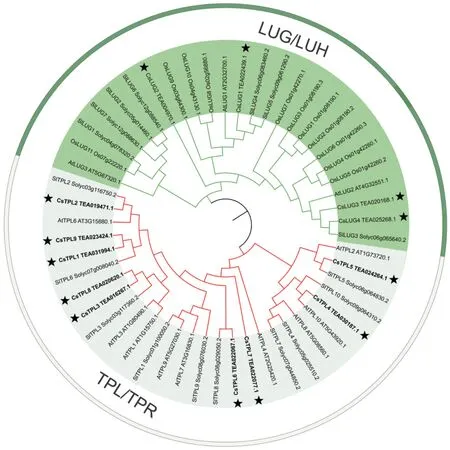
圖2 茶樹、擬南芥、番茄和水稻Gro/Tup1蛋白的進化關系與分類Fig. 2 Phylogenetic relationships and classification of Gro/Tup1 proteins from C. sinensis,A. thaliana,S. lycopersicum,and O. sativa
為進一步分析Gro/Tup1家族成員進化關系,利用MEGA X軟件構建13條茶樹Gro/Tup1蛋白和12條擬南芥Gro/Tup1蛋白的進化樹(圖3-A),結果顯示,茶樹與擬南芥的Gro/Tup1蛋白都存在3個分支,茶樹中CsLUG1-4為一支,CsTPL4和5為一支,剩余CsTPL成員為一支。通過在線軟件MEME分析茶樹Gro/Tup1蛋白序列的10個基序(圖3-B)發現,motif 8和10是其中較為典型的保守基序,存在于大部分Gro/Tup1家族成員和全部LUG/LUH亞家族中。在TPL/TPR亞家族中,motif 1-10均較為保守,但CsTPL4和CsTPL5的保守基序與其他CsTPL成員差異大。CsTPL4和CsTPL5的氨基酸長度更短,CsTPL4只具有motif 1、5、8和10,CsTPL5只具有motif 8。綜上,推測LUG/LUH和TPL/TPR兩個亞家族的作用功能可能不同,CsTPL4和CsTPL5蛋白可能功能不同于其它CsTPL/TPR蛋白。
基因結構與植物的進化關系密切,為進一步了解Gro/Tup1基因特征,對其外顯子及內含子長度和數量進行分析。外顯子-內含子結構(圖3-C)結果表明,擬南芥Gro/Tup1基因長度在5 000 bp左右,與擬南芥相比,茶樹Gro/Tup1長度更長,其基因長度最短的為CsTPL2,除比AtTPL5略短些,都長于剩余擬南芥Gro/Tup1。茶樹Gro/Tup1家族成員的外顯子和內含子數量差異很大,外顯子數量從6個到30個不等。與茶樹TPL/TPR亞家族相比,LUG/LUH亞家族成員間外顯子數量差異較小,在16-19個之間,說明LUG/LUH亞家族在基因結構上比TPL/TPR亞家族更保守。
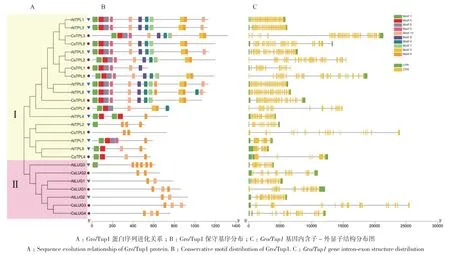
圖3 茶樹和擬南芥的Gro/Tup1Fig. 3 Gro/Tup1 in C. sinensis and A. thaliana
利用在線軟件STRING v10.5將所有茶樹Gro/Tup1蛋白與擬南芥的同源序列進行比對,建立Gro/Tup1蛋白間的相互作用網絡(圖4)。結果表明,擬南芥中CsLUG1、2與LUH同源;CsLUG3、4與LUG同源;CsTPL1、2、9與WSIP2同源;CsTPL3與TPL同源;CsTPL6、7、8與T21F11.18同源。其中,WSIP2與LUG、LUH,TPL與LUH、LUG之間存在較強的互作,它們可能共同表達或形成蛋白復合體以輔助抑制或激活靶基因的表達。
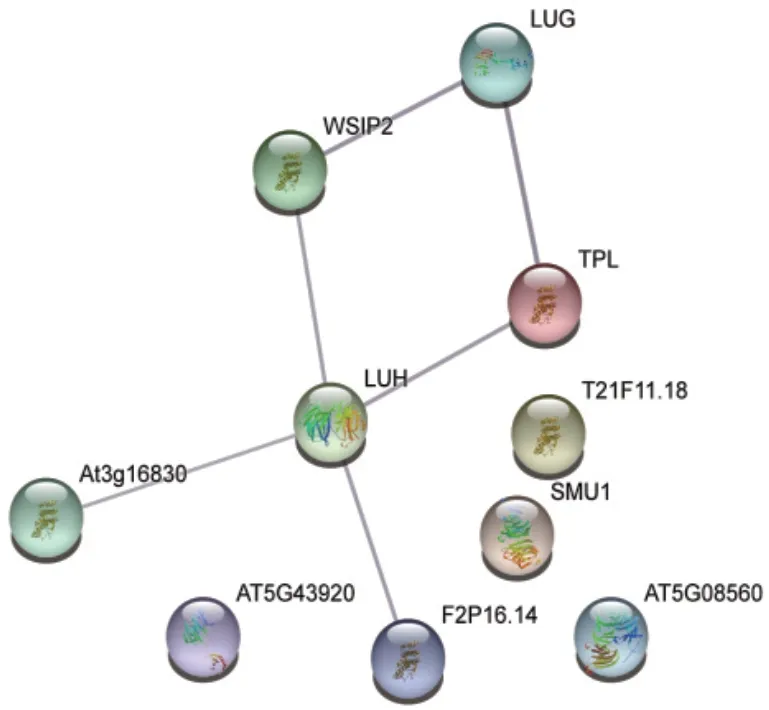
圖4 Gro/Tup1蛋白間的相互作用網絡Fig. 4 Interaction network of Gro/Tup1 proteins
2.3 茶樹Gro/Tup1啟動子順式作用元件分析
為進一步分析茶樹Gro/Tup1家族成員潛在的應答機制,使用在線軟件 PlantCARE預測茶樹Gro/Tup1基因起始密碼子上游2 000 bp啟動子區域的順式作用元件,結果表明,其啟動子區域富集大量與植物激素響應、植物生長發育、次生代謝、脅迫響應相關的順式作用元件(圖5)。茶樹Gro/Tup1基因中具有10種植物激素響應元件(CGTCA-motif、ABRE、TGACG-motif、P-box、TATC-box、TCA-element、GARE-motif、TGA-element、TGA-box和AuxRR-core),其數量和種類最多;5種脅迫響應元件(LTR、ARE、GC-motif、MBS 和 WUN-motif);3種植物生長發育相關元件(Circadian、MSA-like和CAT-box);2種次生代謝相關元件(MBSI和CCAAT-box)。其中 CGTCA-motif 和 TGACG-motif元件與JA響應有關,P-box、TATC-box和GARE-motif元件與GA響應有關,ABRE元件與ABA響應有關,MBS元件與干旱脅迫響應有關,LTR元件與低溫響應相關。所有Gro/Tup1基因都響應激素或脅迫中的一個或多個,推測可能在激素響應和脅迫應對方面的作用更為突出。
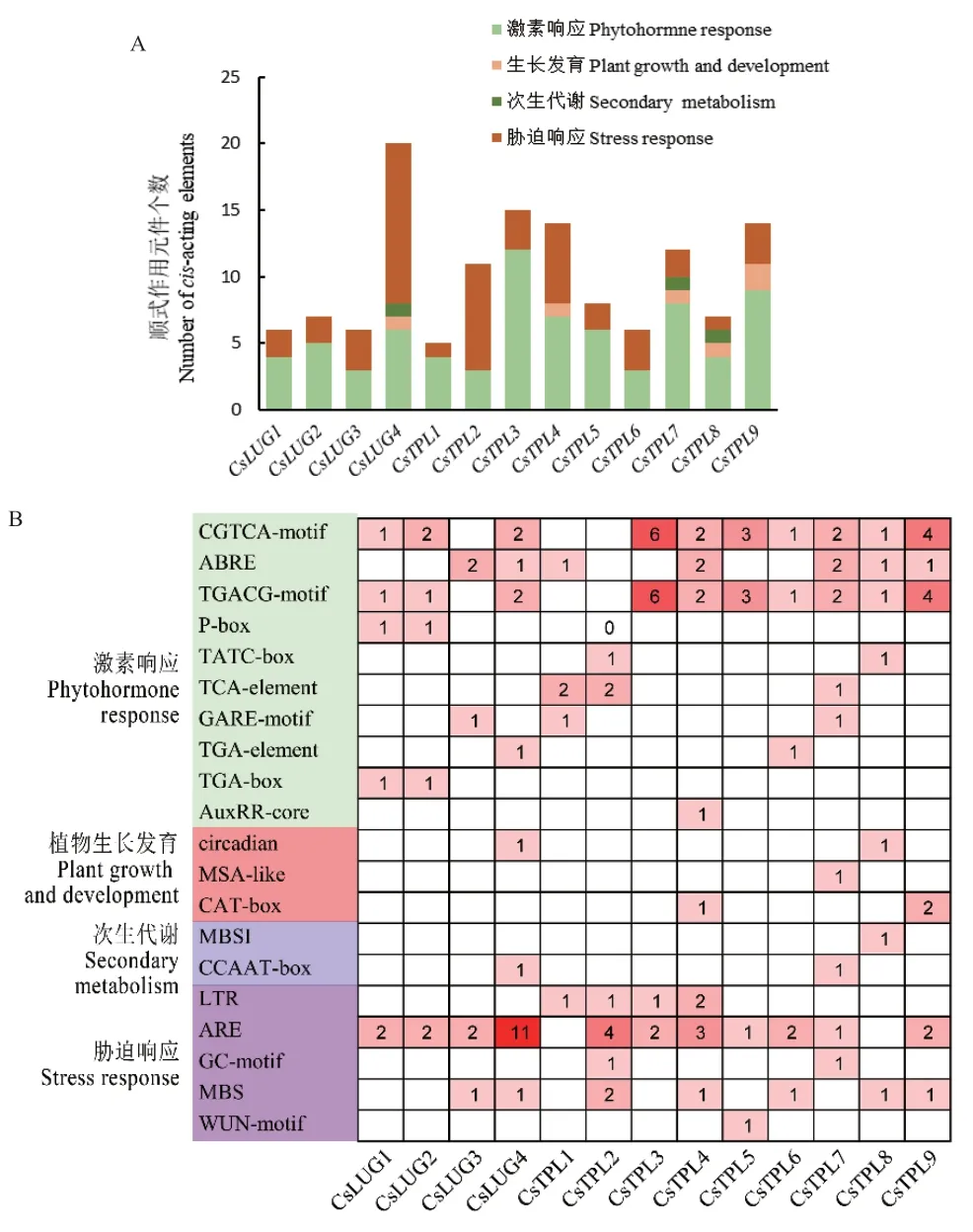
圖5 茶樹Gro/Tup1啟動子順式作用元件預測Fig. 5 Prediction of the cis-acting regulatory elements in the promoters of CsGro/Tup1
2.4 茶樹Gro/Tup1在不同組織中表達模式
為進一步了解茶樹Gro/Tup1家族成員在茶樹不同組織的表達模式,對‘鐵觀音’不同組織部位(根、莖、花、果、頂芽、幼葉、成熟葉和老葉)的茶樹Gro/Tup1成員表達模式進行分析。結果(圖6)表明,大部分Gro/Tup1家族成員在葉組織上表達量較高,不同成熟度葉組織成員表達水平有所差異;在根組織上表達水平較低。具體如下:除CsLUG1、CsTPL6和CsTPL9外,其余基因均在成熟葉與老葉高表達;CsLUG4、CsTPL2、CsTPL3、CsTPL4、CsTPL5 和CsTPL7在幼葉表達量較高;CsLUG2和CsLUG3在頂芽較高表達;CsLUG2、CsTPL2、CsTPL6和CsTPL9在 果 上 較 高 表 達 ;CsLUG1、CsLUG2、CsLUG3、CsTPL3、CsTPL4和CsTPL8在花上表達水平較高;CsLUG1、CsTPL1和CsTPL6在莖部位上較高表達。
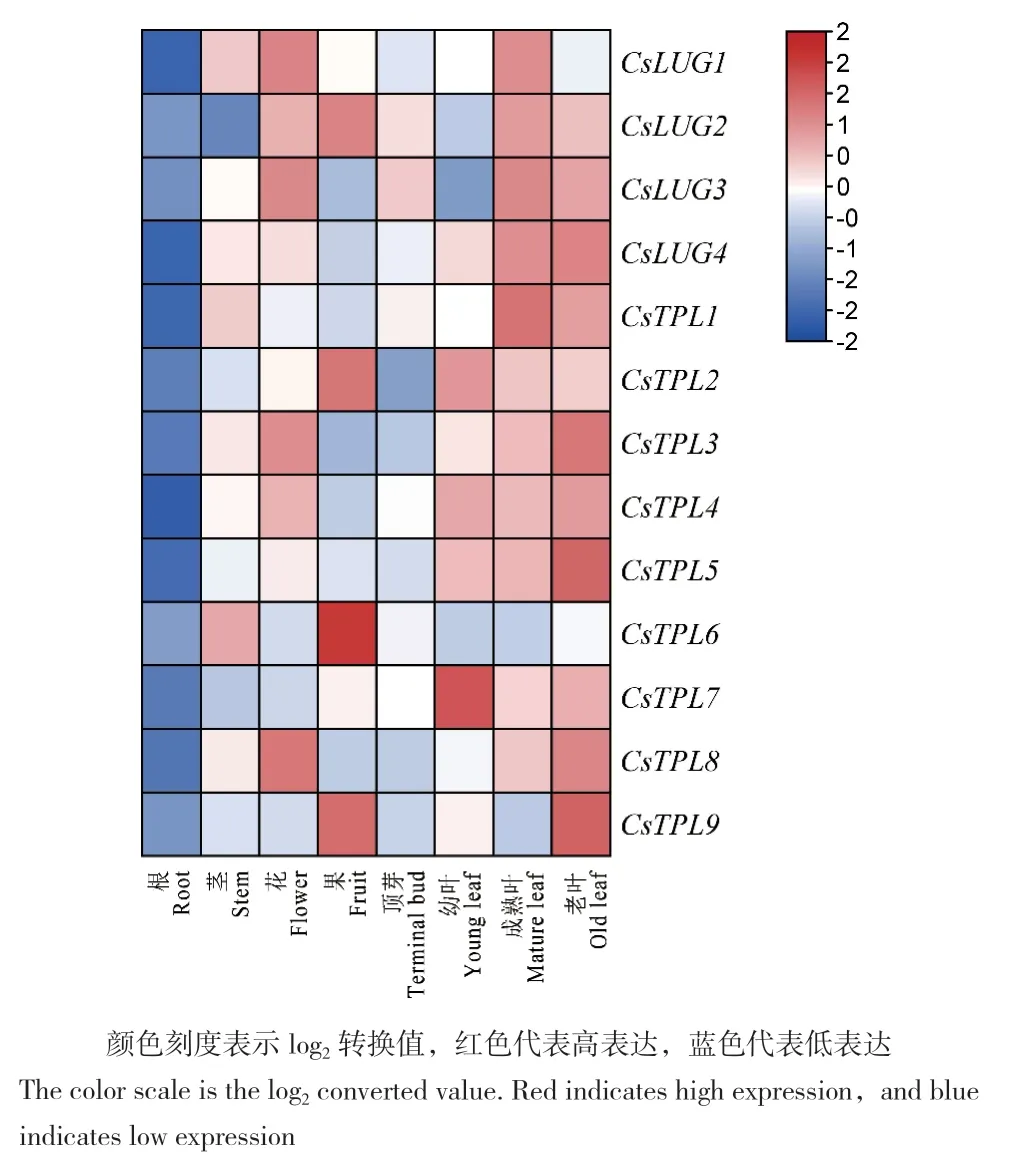
圖6 Gro/Tup1家族成員在‘鐵觀音’組織部位的表達譜Fig.6 Expression profiles of CsGro/Tup1 family genes in the tissues of ‘Tieguanyin’ cultivar
2.5 茶樹Gro/Tup1在外源激素處理和非生物脅迫下表達模式
基于順式作用元件預測分析,通過實時熒光定量分析茶樹Gro/Tup1在干旱、低溫、GA3、ABA和MeJA處理下相對表達量(圖7),結果表明茶樹Gro/Tup1在不同處理下呈多種表達模式。
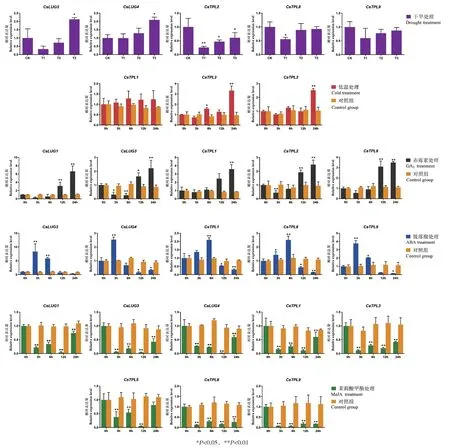
圖7 干旱、低溫、GA3、ABA和MeJA處理下茶樹Gro/Tup1相對表達量Fig. 7 Expression pattens of CsGro/Tup1 in C. sinensis under drought,cold,GA3,ABA,MeJA treatment
干旱處理下,CsLUG3和CsLUG4表達水平在重度干旱時(T3)上調,而CsTPL2表達水平在干旱時受到抑制,CsTPL8和CsTPL9相對表達量穩定,不受干旱調控。在低溫(4℃)處理下,CsTPL1的表達水平穩定,不受低溫調控,CsTPL8和CsTPL9基因表達量呈上調趨勢,并在24 h達到峰值。
外源GA3處理下,CsLUG1基因表達量最高,與CsLUG3、CsTPL1、CsTPL2、CsTPL8基因都呈上調趨勢,并在24 h達到峰值。外源ABA處理下,CsLUG1基因表達水平最高,與CsTPL1、CsTPL8、CsTPL9均呈現先上調后下調趨勢。外源MeJA處理下,CsLUG1、CsLUG3、CsLUG4、CsTPL1、CsTPL3、CsTPL5、CsTPL8和CsTPL9基因表達均受到極顯著抑制。
3 討論
3.1 茶樹Gro/Tup1家族的進化特征
Gro/Tup1是茉莉酸信號轉導途徑中一類重要的輔轉錄調控因子,調控植物生長發育的許多重要環節,從而響應逆境脅迫。本研究在茶樹基因組中鑒定出13個Gro/Tup1基因家族成員,在系統進化分類中,茶樹Gro/Tup1家族成員被劃分為兩個亞家族,與擬南芥[8]和番茄[9]的研究結果一致,與玉米研究結果有所不同,它將LUG/LUH亞家族細分為LUG/LUH和LUG/LUH-like,并認為LUG/LUH-like可能具有與LUG/LUH蛋白行使不同功能[10]。值得注意的是,茶樹的CsTPL4和CsTPL5在系統進化樹上與TPL/TPR成員分離,聚在另一個小分支,其保守基序和基因結構也與其余Gro/Tup1成員存在顯著差異。CsTPL4缺少motif 3和motif 6,外顯子數目最少且布局分散,CsTPL5缺少motif 1、motif 3、motif 5和 motif 6,外顯子布局分散。蛋白互作預測結果表明,茶樹中CsTPL4和CsTPL5分別與AT5G43920和SMU1同源,這兩個蛋白與其他蛋白不存在互作關系,推測CsTPL4和CsTPL5蛋白質在茶樹中的作用可能與其余Gro/Tup1成員作用不同。前人研究表明基因功能發生分化和產生新功能基因的主要原因是基因復制,包括全基因復制、片段復制、串聯復制以及轉座復制[32],CsTPL4和CsTPL5是否具有不同功能以及功能分化的原因是否與其基因復制相關還有待進一步研究。CsTPL8和CsTPL9的外顯子數量都為26個,CsTPL8和CsTPL9的氨基酸長度相近,基序構成及分布都極其相似,氨基酸序列相似度為72.98%,其啟動子區域順式元件預測結果表明兩者響應元件類型與數量相近,最后發現CsTPL8和CsTPL9在干旱處理、脫落酸和茉莉酸甲酯分別處理下的表達模式類似,據此推測兩者可能存在功能冗余。
結構域是蛋白質結構單位,也是功能單位,即功能區域。在本研究中,所有的Gro/Tup1都與擬南芥中的一致,具有保守的N端LisH結構域,高度保守的C端WD40重復結構域以及相對保守性差的中間結構域。每個WD40域是由40個氨基酸殘基組成,序列一定間隔會特征性出現一個色氨酸(W)和天冬氨酸(D)殘基,該重復序列能夠介導Gro/Tup1與轉錄因子的相互作用[33]。LisH主要是促進蛋白質之間相互作用,能夠介導蛋白質二聚化[34]。LUG是植物中第一個被發現的Gro/Tup1成員,除上述特點外,它有一個LUFS結構域,其內含LisH基序。LUFS結構域是LUG/LUH蛋白與接頭蛋白SEUSS相互作用被招募到轉錄因子的必需結構域,此外它還能夠與轉錄因子YABBY直接作用[35]。TPL/TPR是Gro/Tup1的另一個亞家族,具有TPD結構域,其包含了LisH和CTLH位點,該結構域是其與轉錄因子WUS相互作用位點,是通過組蛋白脫乙酰化實現的[7]。此外,TPL的TPD結構域能夠與接頭蛋白NINJA的EAR基序相互作用,與JAZ轉錄因子共同作用抑制JA靶標基因表達[13,36-37]。
通常,真核蛋白編碼基因序列都具有內含子-外顯子結構,其中內含子是基因內部不編碼區域,不出現在成熟mRNA中[38]。過去內含子常常被認為是 “垃圾序列”,近年來的研究發現內含子是分子進化過程中不可或缺的推動者,是基因表達中的調控者[39]。研究表明在整個基因組水平上,內含子數量和內含子長度均與基因表達水平呈負相關,在內含子數目極少(不超過5個)和內含子長度較短的情況下,二者與基因表達水平呈正相關,內含子過多、過長會造成轉錄能量浪費,只有在內含子數量和長度適中時才最有利于基因表達[40]。在進化過程中,外顯子是較為穩定,而內含子的序列和長度是迅速隨機突變的,對于種群量相當大的物種來說,其內含子數量是在進化過程中經過優化,其基因內含子數目變化大。Gro/Tup1家族廣泛存在于真菌、動物和植物中,是一個古老的基因家族,其基因的結構差異大。外顯子-內含子結構預測結果表明,茶樹Gro/Tup1的結構呈現多樣性,主要表現在內含子數量差異很大,從5個到29個不等。LUG/LUH亞家族成員平均內含子數目為16.25,數量最少的為15(CsLUG3), 最 多 的 為 18(CsLUG3);TPL/TPR亞家族成員平均內含子數目為20.34,數量最少為5(CsTPL4),數量最多為29(CsTPL3)。表明茶樹Gro/Tup1家族成員基因結構多樣化,能夠使茶樹在遭受不利環境影響時做出高效有利的反應,使其存活下來。
3.2 茶樹Gro/Tup1家族成員響應外源激素及非生物脅迫
基因轉錄往往受到精細的調控,轉錄因子和順式作用元件上的結合位點作用而調控基因轉錄的精確起始和轉錄效率。啟動子區域順式作用元件預測結果表明,茶樹Gro/Tup1家族成員的表達受多種激素及逆境脅迫調控,其在MeJA、ABA、GA3響應及干旱和低溫下的表達模式,證實茶樹中Gro/Tup1參與激素響應,與茶樹的逆境響應密切相關。在MeJA處理下茶樹Gro/Tup1基因表達水平均顯著被抑制,與玉米研究中嫩梢的ZmLUG2、ZmLUG5和ZmLUG6表達在JA處理后下調[10]結果類似。前人研究表明,Gro/Tup1蛋白的兩個亞家族分別在茉莉酸信號轉導通路中的不同階段起到不同的作用。在靜息階段,JAZ蛋白招募輔抑制因子TPL,繼而和NINJA共同形成三元復合物抑制JA信號轉導通路中轉錄因子myelocytomatosis2(MYC2)靶基因的表達;在激活階段,LUH通過Q-rich域與MED25蛋白相互作用,從而將LUH和HAC1招募到MYC2靶基因啟動子上,該過程中LUH能夠促進MED25和HAC1相互作用以及MED25和MYC2相互作用,最后激活MYC2靶基因表達,即 JA 應答基因表達[13-14,41]。GA3處理的Gro/Tup1基因表達水平均在12 h和24 h顯著上調,ABA處理的Gro/Tup1基因表達水平均在3 h和6 h顯著上調,MeJA處理的Gro/Tup1基因表達水平均顯著被抑制,表明Gro/Tup1基因能夠應答GA3、ABA和MeJA,對ABA和JA應答更快。
非生物脅迫是植物生長發育和作物產量穩定的主要威脅,許多植物研究表明Gro/Tup1家族成員參與了許多非生物脅迫應激反應,例如擬南芥的LUH和HOS15已被證明分別參與鹽、凍害防御[17-18],玉米中TPL和LUG都對干旱和低溫進行應答[10]。在本研究中,茶樹Gro/Tup1家族成員在干旱和低溫處理下的表達量均表現出動態變化。CsLUG3和CsLUG4基因表達量均在重度干旱(T3)時顯著上調,TPL2表達水平則顯著被抑制,表明LUG和TPL基因對干旱刺激的應答模式不同。在低溫處理下,CsTPL2和CsTPL3基因表達量顯著上調,推測TPL基因是應答低溫刺激的主要基因,且與應答干旱時的表達模式相反。這意味著這些基因的蛋白在茶樹抗旱及抵御低溫方面發揮重要作用。
4 結論
Gro/Tup1基因家族在進化上是保守的,其中個別基因如CsTPL4和CsTPL5可能在進化過程中發展出不同的功能,CsTPL8和CsTPL9的功能冗余,其具體功能有待進一步研究。同其他植物一樣,茶樹Gro/Tup1家族成員能夠響應非生物脅迫及外源激素,其基因應答呈現不同表達譜,表明不同脅迫下Gro/Tup1基因應答方式不同,其具體生物學功能有待一進步研究。