橡膠樹產膠生物學研究進展
唐朝榮


摘? 要:天然橡膠(順式-1,4-聚異戊二烯)是一種重要的工業原料,主要來自橡膠樹。以天然橡膠的生物合成與產量形成為主要內容的產膠生物學研究為橡膠樹高產遺傳改良提供理論指導,近10年取得了重要進展。本文從橡膠樹基因組測序、橡膠轉移酶、轉基因、以及轉錄組和蛋白質組等4個方面介紹產膠生物學研究主要進展,并討論了相關領域研究存在的問題,對未來5~10年需重點關注的研究內容提出了建議。在介紹橡膠轉移酶時,同時概述其他幾種產膠植物的相關研究進展。
關鍵詞:橡膠樹;產膠生物學;基因組測序;橡膠轉移酶
中圖分類號:S794.1? ? ? 文獻標識碼:A
Abstract: Natural rubber (cis-1, 4-polyisoprene) is an important industrial raw material, commercially harvested mainly from rubber tree (Hevea brasiliensis). Important progresses have been made in the past decade on Hevea biology of rubber production, focusing on rubber biosynthesis and latex production. The present review summarized the major progress in four research areas with relation to rubber production, i.e. Hevea genome sequencing, rubber transferase, transgenic research, and transcriptomics & proteomics. Moreover, the problems were discussed and a few of research emphasis in the next 5 to 10 years were proposed. When describing the studies of rubber transferase, the recent progress in several other rubber-producing plants were included.
Keywords: Hevea brasiliensis (para rubber tree); biology of rubber production; genome sequencing; rubber transferase
DOI: 10.3969/j.issn.1000-2561.2020.10.003
天然橡膠是一種重要的工業原料,在交通運輸、醫療衛生、國防軍工等領域應用廣泛,其優異的綜合性能迄今仍無法被人工合成橡膠完全替代[1]。我國是世界最大的天然橡膠消費國,年消費量(約550萬t)約占世界天然橡膠總產量(約1400萬t)的40%,但自給率僅約15%[2]。目前,天然橡膠的商業來源幾乎全部來自巴西橡膠樹這一個熱帶樹種,無論是理論預測[3]或是生產上超高產橡膠樹單株的發現[4]都顯示橡膠樹的產膠潛力巨大,可在現有最高產橡膠樹品種單產水平(年產約2500 kg/hm2)的基礎上提升3~5倍。產膠生物學基礎研究,將為橡膠樹高產分子育種提供理論依據與靶標基因。
近10年,隨著高通量、低成本的二代和三代測序技術以及蛋白質組學技術的飛速發展,橡膠樹產膠生物學研究取得了顯著進展。本文從橡膠樹基因組、橡膠轉移酶、轉基因以及轉錄組和蛋白質組學等4個方向介紹產膠生物研究領域的主要進展,并對該研究領域存在的問題和發展方向提出了思考,以期增進研究者對本領域發展現狀和未來工作重點的了解。
1? 橡膠樹基因組測序研究
2013年,馬來西亞研究者公布了第一個橡膠樹基因組[5],此后包含筆者團隊在內的不同研究者又陸續報道了4個版本[6-9](表1)。從拼接質量上看,筆者團隊在2016年發表的版本[6]僅次于最近以三代測序為主完成的版本[9],但明顯優于其他3個同樣以二代測序技術為主的版本(表1)[10]。從對產膠生物學問題的闡述上看,筆者團隊發現了橡膠樹物種高產橡膠的遺傳線索、提出乙烯刺激產膠新機制等重要產膠生物學問題[6],是迄今最受關注和認可的橡膠樹基因組版本。
1.1? 橡膠樹基因組的大小
最早基于孚爾根染色微密度測定法(Feulgen microdensitometry)測定橡膠樹基因組的大小為2.15 Gb[11],而筆者團隊用二代測序k-mer分析法估算橡膠樹‘熱研7-33-97等6個品種的基因組大小在1.41~1.55 Gb之間[6],這與53份橡膠樹栽培種質基因組大小的流式細胞分析結果(1.46~1.60 Gb, 平均1.53 Gb)[12]一致。結合幾個較高質量橡膠樹基因組的實際拼接大小[6-9](表1),我們認為橡膠樹這個物種基因組的大小應為1.5 Gb左右。
1.2? 橡膠生物合成單體IPP的來源
在橡膠樹的產膠細胞乳管中存在2種異戊烯基焦磷酸(IPP)的合成途徑,即細胞質胞漿的甲羥戊酸途徑(MVA)和質體的2-C-甲基-D-赤蘚糖醇-4-磷酸途徑(MEP)。多數研究認為MVA途徑可能是橡膠生物合成單體IPP的主要來源[13],但MEP途徑是否參與橡膠生物合成長期存在爭議[14-15]。
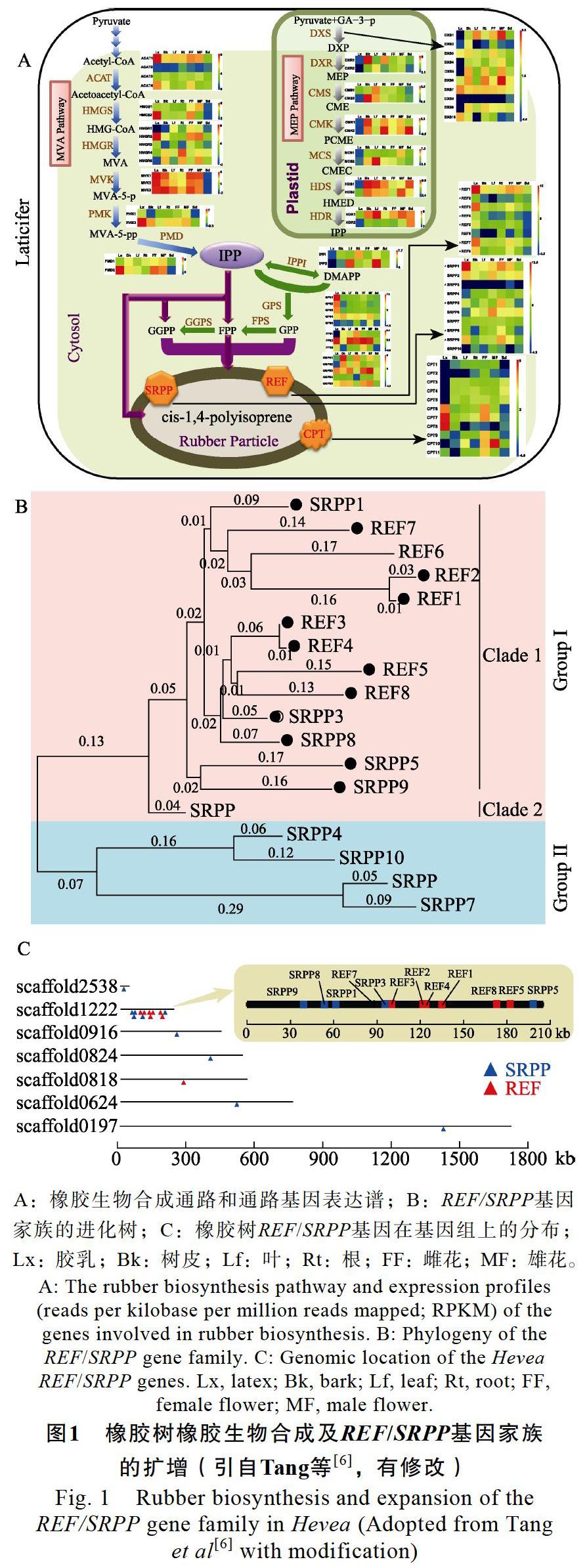
筆者團隊在橡膠樹基因組中鑒定到18個MVA和22個MEP途徑家族基因(圖1A)。組織表達分析發現,MVA途徑的6個催化酶中都至少有1個在膠乳(乳管細胞的細胞質)中特異高表達的基因,而MEP途徑中僅DXS有2個編碼基因在膠乳中的表達量相對較高,其他6個酶的編碼基因在膠乳中表達量很低或明顯低于其他組織。這些結果表明,MVA途徑是橡膠樹中橡膠生物合成IPP的主要來源,MEP途徑可能對橡膠生物合成的貢獻很小。
1.3? 橡膠樹高產橡膠的遺傳線索
在已知的2500余種產膠植物中,為何橡膠樹具有優異的產膠能力,筆者團隊從基因組研究上獲得了科學線索。
通過與其他17種植物基因組比較,我們發現橡膠樹中橡膠延伸因子/小橡膠粒子蛋白(REF/ SRPP)基因家族發生了顯著擴增,共有18個基因,數目遠高于其他植物[6],類似發現在另外2個關于橡膠樹基因組的研究[7, 9]中也有報道。在橡膠樹基因組上,有12個REF/SRPP基因形成一個跨度約160 kb的基因簇,并且在膠乳中特異高表達的4個基因(REF1、SRPP1、REF3和REF7)都在這個基因簇上;從進化關系上看,膠乳中表達量最高的REF1基因可能是由表達量較低的REF/SRPP基因逐步進化而來(圖1)。從蛋白序
列比對結果來看,REF1類似于一個C-端被截斷的SRPP1(膠乳表達量僅次于REF1基因)。有趣的是,僅在橡膠樹中存在與REF1編碼蛋白(138 aa)大小相當的REF/SRPP基因,而其他植物中只有編碼大小與SRPP1(204 aa)相當或更大的REF/SRPP蛋白。
按粒徑尺寸將橡膠粒子分為大(>400 nm)、小(<400 nm)兩類,雖然大橡膠粒子的數目只占膠乳中橡膠粒子總數的6%,卻貢獻了93%的橡膠產量[16]。SRPP和REF在橡膠粒子上的分布模式存在明顯差異,SRPP主要位于小橡膠粒子膜的表面,而REF主要鑲嵌在大橡膠粒子的膜中[17]。綜合上述結果,我們推測REF/SRPP基因家族的顯著擴增和乳管特異性功能分化,特別是乳管中超高表達特異REF1的出現可能是橡膠樹進化出優異高產橡膠性狀的根本原因,這種性狀有利于橡膠樹在熱帶叢林中抵御蛀食性害蟲的侵害[18]。
1.4? 乙烯刺激橡膠樹產膠的新機制
在橡膠樹的樹干上涂抹乙烯利(2-氯乙基磷酸,乙烯釋放劑)或直接施用氣體乙烯可刺激橡膠樹排膠、顯著提高膠乳產量,是橡膠樹生產上的一項重要增產措施[18]。在過去40多年中,國內外發表了大量關于乙烯刺激橡膠樹膠乳產生機制的論文,發現乙烯處理增強了乳管細胞中蔗糖的吸收和降解[19-22]、促進水分吸收[23]和能量代謝[24]、導致胞漿pH堿性化[24]、促進氮同化[25]和引起防御應答[26]等,這些過程與膠乳再生或膠乳排出直接或間接相關,但都未能從根本上回答乙烯處理能顯著刺激橡膠樹乳管產膠的原因。
利用RNAseq深度測序,筆者團隊共鑒定出500多個受乙烯調控的膠乳差異表達基因。進一步分析發現,乙烯合成關鍵酶ACO家族基因在膠乳中不表達或表達量極低[6],結合膠乳中氧含量低[19]和ACO催化ACC氧化產生乙烯時需要氧氣的事實,推測橡膠樹乳管細胞中的乙烯合成能力很弱;同時,膠乳中乙烯信號傳遞和應答通路中的多個關鍵基因,包括4個ETR、2個EIN2和1個EIL1基因受乙烯刺激后顯著上調表達[6],表明乳管細胞中存在活躍的乙烯信號傳遞與應答通路。這一發現,從源頭上回答了在橡膠樹上使用外源乙烯刺激可顯著刺激膠乳增產的根本原因。
2? 橡膠轉移酶研究
1969年Archer等[27]將催化橡膠生物合成的酶稱為橡膠轉移酶(rubber transferase),其催化橡膠生物合成單體IPP逐個摻入到不斷延伸的橡膠烴(順式-1,4-聚異戊二烯)上,分布在膠乳的水相和橡膠粒子上。此后至今的50余年,人們對橡膠轉移酶的生化、分子特性和調控機制的認識不斷深入,特別是在2015—2016年出現了突破性進展。研究者分別以萵苣[28]、短角蒲公英[29]和橡膠樹[30]這3種產膠植物為研究對象,發現橡膠轉移酶不是一種單一蛋白,而是以順式異戊烯基轉移酶(cis-prenyl-transferase, CPT)為核心,并包含CPT結合蛋白(一種人NogoB受體類似蛋白,也稱CPT類似蛋白、橡膠轉移酶活化劑)、橡膠延伸因子(rubber elongation factor,REF)和小橡膠粒子蛋白(small rubber particle protein,SRPP)等多種蛋白組分,定位在橡膠粒子上催化橡膠烴大分子合成的一種復雜的蛋白復合體[31]。
2.1? 順式異戊烯基轉移酶
1989年,Light等[32]報道從橡膠樹膠乳中純化了橡膠轉移酶,在橡膠粒子存在時可催化橡膠烴的合成,在無橡膠粒子但有二甲基烯丙基焦磷酸(DMAPP)存在時可催化合成牻牛兒基焦磷酸(GPP)和法尼基焦磷酸(FPP)。但Cornish[33]隨后的研究發現Light等純化的不是橡膠轉移酶,而是在膠乳中催化橡膠合成起始分子FPP的可溶性反式異戊烯基轉移酶,真正的橡膠轉移酶是與橡膠粒子密切結合的順式異戊烯基轉移酶(CPT)。Asawatreratanakul等[34]首先克隆了2個在橡膠樹膠乳中特異表達的CPT基因(HRT1和HRT2),其中原核表達的HRT2重組蛋白在膠乳離心洗滌后的底層顆粒存在時可催化合成橡膠大分子。Takahashi等[35]在酵母和擬南芥細胞中表達HRT1和HRT2基因,但發現HRT重組蛋白并不能催化生成橡膠大分子,推測橡膠轉移酶活性需要一些特定的膠乳成分。Post等[36]證明短角蒲公英中乳管特異表達的CPT是橡膠生物合成所必需的,相關基因經RNAi沉默后,膠乳中的橡膠含量減低90%以上。利用蛋白質組學分析,Dai等[37]從橡膠樹膠乳的橡膠粒子上鑒定到6個CPT蛋白,證實了早期關于CPT與橡膠粒子緊密結合的報道[33]。Tang等[6]發現橡膠樹基因組上共有11個CPT基因,其中3個在膠乳中高豐度表達,這與Uthup等[38]認為橡膠樹中僅有3個與橡膠生物合成相關的CPT基因(RubCPT1、RubCPT2和RubCPT3)的結論一致。Uthup等[38]同時發現RubCPT1在不同橡膠品系中的單倍體類型與其基因表達水平和產膠性狀密切相關。最近,Ding等[39]利用轉錄組學和基因組學整合分析研究手段發現1個CPT基因(CPT2)是所鑒定的3個橡膠生物合成樞紐基因之一。
2.2? 橡膠延伸因子和小橡膠粒子蛋白
橡膠延伸因子(REF)和小橡膠粒子蛋白(SRPP)是橡膠樹膠乳中豐度最高的蛋白,同屬REF/SRPP蛋白家族。橡膠樹基因組中有18個REF/SRPP基因,是已報道植物基因組中家族基因數量最多的物種[6],其中一些膠乳特異高表達的家族基因在多個蛋白質組研究中被鑒定到[37, 40-42]。關于橡膠樹REF/SRPP蛋白參與橡膠生物合成的研究主要集中在2個膠乳特異高表達家族成員上,即REF1(138 aa, 14.6 kDa)和SRPP1(204 aa, 24 kDa)[6],它們具有較高的氨基酸序列同源性,都主要分布在膠乳中的橡膠粒子上,但與橡膠粒子的結合方式和生化特性存在明顯差別[43-44]:SRPP1松散地結合在小橡膠粒子膜的表面,而REF1同時存在于大、小橡膠粒子上且與膜結合緊密;在溶液中,REF1聚合成富含β-折疊的淀粉樣蛋白,并很快形成微米級別的大聚合體,SRPP1則形成穩定的納米級別的近球形多聚體;SRPP1與REF1可發生相互作用,SRPP1會抑制REF1的聚集。
Dennis等[13]發現橡膠粒子上的REF蛋白與橡膠烴分子的比例約1∶1,膠乳中REF蛋白的含量與橡膠含量正相關,同時在體外橡膠生物合成體系中加入REF抗體或去除橡膠粒子上的REF蛋白均會顯著抑制橡膠合成。Priya等[45]發現REF基因在橡膠樹高產品系膠乳中的表達水平高于低產品系,割膠和乙烯利刺激均能誘導REF基因的表達,表明REF基因表達與膠乳產量正相關。Oh等[46]發現在體外橡膠生物合成體系中添加SRPP重組蛋白可顯著促進橡膠合成,推測SRPP和REF可能都是橡膠生物合成系統的重要組成部分。
在2種產膠蒲公英——俄羅斯蒲公英和短角蒲公英中,SRPP/REF被RNA干擾下調表達后,根中的橡膠含量顯著降低,幅度可高達50%以上[47-49]。不同的是,在俄羅斯蒲公英干擾植株中,橡膠粒子的穩定性和橡膠分子量都顯著下降[47];而在短角蒲公英干擾植株中,橡膠粒子的穩定性、橡膠分子量和分子分散度則均不受影響[48-49]。Hillebrand等[48]推測,在短角蒲公英中SRPP是維持橡膠粒子穩定性的重要蛋白,RNA干擾植株中橡膠含量下降的主要原因是橡膠粒子的穩定性受到影響。奇怪的是,在另一種產膠植物萵苣中,RNA干擾膠乳中2個主要SRPP基因并不影響橡膠生物合成,干擾植株膠乳中的橡膠含量、橡膠分子量和分子分散度都不受影響[50]。
需要指出的是,系統進化分析結果顯示橡膠樹膠乳中特異高表達的REF/SRPP基因單獨聚為一簇,其他產膠植物中一些REF/SRPP基因聚為另一簇,而橡膠樹和其他產膠植物其余的REF/SRPP基因則分散開來與非產膠植物聚為不同的簇[6],這表明其他產膠植物膠乳中關于特異高表達REF/ SRPP基因的研究結果可能難以真實反映橡膠樹中相應基因的功能。最近,Ding等[39]研究鑒定了3個參與橡膠生物合成的樞紐基因,其中包括膠乳中表達豐度最高的2個REF/SRPP基因(REF1和SRPP1)。
2.3? 橡膠轉移酶復合體
Qu等[28]從萵苣中鑒定到一個在膠乳中高表達的CPT-like 2(CPTL2)蛋白,該蛋白與人NogoB受體和典型的CPT都有一定的同源性,但缺乏CPT的保守motif。萵苣中CPTL2基因經RNA干擾后,膠乳中的橡膠含量降至野生植株的5%;CPT3和CPTL2可直接互作,在煙草和酵母細胞中共表達CPT3和CPTL2基因時,原本在細胞溶質中表達的CPT3定位到內質網上;在酵母微粒體中添加CPT3/CPTL2重組蛋白后低聚合度順式-聚異戊二烯的合成能力增強,卻不能合成橡膠大分子。根據以上結果,作者推測CPTL2可能作為一種腳手架蛋白將CPT3拉到內質網上,進而形成橡膠生
物合成的細胞器——橡膠粒子。在短角蒲公英中,Epping等[29]從膠乳蛋白質組上鑒定了一個與萵苣CPTL2功能近似的NogoB受體蛋白,該蛋白在橡膠粒子膜上與CPT互作,是橡膠轉移酶復合物的必需組分;在該蛋白基因的RNA干擾的植株中,橡膠生物合成能力幾乎完全喪失,膠乳中檢測不到CPT蛋白,但多萜醇(dolichol)的含量和蛋白質的糖基化不受影響,因此將該蛋白命名為橡膠轉移酶活化劑(rubber transferase activator, RTA)。
Yamashita等[30]在橡膠轉移酶復合體研究中更進了一步,發現REF1也是橡膠轉移酶復合體的一個關鍵組分。利用去污劑(8 mmol/L CHAPS)處理,獲得了去除大部分膜蛋白的橡膠樹橡膠粒子(WRP),結合無細胞蛋白翻譯系統建立了橡膠體外合成反應的研究體系。在WRP懸浮液中同時表達橡膠樹CPT(HRT1)、REF和HRT1-REF橋梁蛋白(HRBP,系萵苣CPTL2和短角蒲公英RTA的同源蛋白)這3種蛋白,發現橡膠轉移酶的活性遠高于單獨表達HRT1或同時表達HRT1和HRBP的活性。橡膠粒子蛋白質組學和互作網絡的研究表明,HRT1、REF和HRBP三者可能在橡膠粒子上形成復合體,其中HRBP同時與HRT1和REF互作,在復合體形成中發揮橋梁作用。基于相關結果,提出了在橡膠樹膠乳中的橡膠粒子上進行橡膠合成以及橡膠粒子發生機制的模型圖(圖2)[30],指出在橡膠粒子上形成正確的HRT1蛋白復合體是進行橡膠大分子生物合成的關鍵。
3? 橡膠樹轉基因研究
橡膠樹遺傳轉化的效率低、周期長,轉基因研究進展緩慢。1994年Arokiaraj等[51]將GUS報告基因導入橡膠樹的基因組,但其后十幾年僅有幾篇進行橡膠樹轉基因體系優化研究的報道[52-56]。2012年發表了第一篇對轉基因橡膠樹植株表型和所轉基因的功能進行系統研究的文章[57],研究者在橡膠樹中過表達了其自身的細胞質胞漿CuZnSOD酶,轉基因植株在干旱脅迫處理時氣孔導度下降、脯氨酸含量增加,干旱耐受能力明顯增強,并發現這種增強與轉基因植株中活性氧清除能力的提高直接相關[57]。近幾年,橡膠樹轉基因研究取得可喜進展,有望通過轉基因手段明顯改進橡膠樹的抗逆和產量性狀[58-60]。
Lestari等[59]在橡膠樹中過表達1個擬南芥ERF1(ethylene response factor 1)的橡膠樹同源基因HbERF-IXc5,發現轉基因植株的根系發達,莖圍和株高明顯增加,植株干重顯著增大,這與以往報道的ERF1過表達植株矮化嚴重的現象明顯不同,表明HbERF-IXc5的功能可能有別于經典的植物ERF1;HbERF-IXc5轉基因植株對干旱、冷、鹽等非生物脅迫的耐受力有所增強,與相關植株生態生理指標的測定結果一致;轉基因植株主葉脈和綠色嫩莖中的初生乳管數量明顯增加,其中一個轉基因株系老化莖中的次生乳管數量也明顯增多,推測HbERF-IXc5基因可能通過參與乙烯和茉莉酸信號傳導間的“竄擾”(crosstalk),從而控制一些直接參與乳管分化的基因。Jayashree等[60]在橡膠樹中過表達IPP甲羥戊酸合成途徑的關鍵酶HMGR基因(hmgr1),結果令人振奮,所有轉基因植株的莖圍和膠乳產量均高于對照植株,膠乳產量最高可達對照的5倍,顯示通過轉基因手段培養高產甚至超高產橡膠樹的前景誘人。
最近,中國熱帶農業科學研究院橡膠研究所的科研人員[61]將體外組合的Cas9/sgRNA核蛋白導入橡膠樹原生質體中,實現了對橡膠樹靶標基因FT和TFL1的有效編輯,結合該團隊前期建立的橡膠樹原生質體植株再生體系[62],有望將無外源DNA導入的基因編輯技術應用到橡膠樹遺傳改良中。
4? 橡膠樹轉錄組與蛋白質組研究
橡膠樹基因組測序推動了與產膠直接或間接相關的轉錄組和蛋白質組研究。研究內容涉及以下方面:膠乳蛋白質組[63]與轉錄組的研究方法[10, 64-65];不同橡膠樹組織的轉錄組[66-69];膠乳[41, 70]及其不同亞細胞組分(橡膠粒子、c-乳清和黃色體)的蛋白組[37, 42, 63, 71-73];乙烯和茉莉酸刺激[68, 74],以及與死皮相關[75]的膠乳或樹皮轉錄組;乙烯刺激的膠乳蛋白質組[41, 70];不同產膠水平單株或品系[4, 76]以及排膠相關的膠乳轉錄組[77];基于轉錄組的產膠相關基因家族發掘[78-79]、分子標記開發與遺傳連鎖圖譜構建[61, 80];橡膠樹基因組與轉錄組的整合數據庫[81]等。這些研究產生了海量的以產膠組織膠乳為主的轉錄組和蛋白質組數據,加深了人們對橡膠樹的橡膠生物合成、激素應答和割膠脅迫等生物學問題的認識。但從整體上看,多數研究只是對相關組學數據進行初步分析,未能對所發現的產膠相關候選基因或蛋白進行深入探究。
最近幾年,在橡膠樹組學研究上有了一些新變化,取得一些新進展,這里簡要介紹幾項代表性的研究。Wang等[70]鑒定到143個表達豐度顯著受乙烯刺激調控的膠乳蛋白,進一步利用磷酸化蛋白質組學技術鑒定到59個應答乙烯處理的磷酸化蛋白,其中包括一些REF和SRPP的同源異構體(isoform),推測蛋白翻譯后修飾和isoform特異性磷酸化修飾可能在乙烯刺激產膠中發揮重要作用。Makita等[65]建立了橡膠樹不同組織的全長cDNA文庫,利用Sanger和Illumina兩種測序手段結合更新了5500個基因結構,新注釋了9500個轉錄起始位點,結合橡膠樹不同品種和組織的RNAseq數據,對橡膠生物合成、膠乳產量和抗病有了新認識。Chow等[10]利用PacBio三代測序技術對橡膠樹全長cDNA文庫進行轉錄組測序,共得到3.7萬余個平均長度約2 kb的全長轉錄本,這些轉錄本對應約1.5萬個基因座,與‘熱研7-33-97基因組比對后發現,超過一半的轉錄本可能是新的基因isoform。Ding等[82]利用公共數據庫中的129個RNAseq數據包進行基因共表達分析,共鑒定到25個基因共表達模塊,其中1個模塊基因注釋富集為類異戊二烯代謝,在膠乳中高豐度表達并顯著應答乙烯、茉莉酸處理和死皮發生,分析認為模塊中的SRPP1、CPT2和REF1是橡膠生物合成的中心(hub)基因;基因組進化分析發現REF/SRPP基因家族在橡膠樹物種中進化產生2個分別包含SRPP1和REF1基因的特異基因簇,推測與橡膠生物合成密切相關,這些結果與基因組研究[6]發現“REF1基因可能是橡膠樹物種進化出高產橡膠性狀的關鍵事件”一致。
5? 問題與展望
過去10年,橡膠樹產膠生物學研究取得了顯著進展,加深了人們對橡膠樹的產膠機制以及與產膠相關的抗逆與激素應答等重要生物學問題的認識,也為橡膠樹高產分子改良提供了思路和技術儲備。但與水稻等重要糧食作物相比,橡膠樹橡膠產量性狀形成的分子機制研究尚處于比較初級的水平,相關研究成果還無法對橡膠樹高產遺傳改良提供切實支持。根據橡膠樹高產分子育種研究與實踐發展的需要,筆者認為在未來5~10年,應對以下幾個方面予以重點關注:
(1)構建橡膠樹主要栽培品種和核心種質的泛基因組及膠乳等主要組織的泛轉錄組,揭示橡膠樹基因組物種水平的基因結構、序列變異和可變剪輯模式;
(2)利用各組學相結合的整合生物學研究手段以及豐富的橡膠樹栽培與種質材料,揭示橡膠產量形成的關鍵代謝途徑與節點基因;
(3)利用橡膠樹膠乳體外橡膠合成體系,以及酵母或植物懸浮細胞等表達體系,揭示橡膠轉移酶復合體的精準結構與作用機制;
(4)利用橡膠樹懸浮培養細胞以及產膠模式植物——橡膠草的轉基因與基因編輯研究,揭示產膠細胞器(橡膠粒子)的發生與發育機制;
(5)探索橡膠樹體胚發生與植株再生的分子調控機制,建立橡膠樹高效遺傳轉化和基因編輯技術平臺。
參考文獻
Van Beilen J B, Poirier Y. Establishment of new crops for the production of natural rubber[J]. Trends in Biotechnology, 2007, 25(11): 522-529.
莫業勇. 天然橡膠供需形勢和風險分析[J]. 中國熱帶農業, 2019(2): 4-6, 10.
Paardekooper E. Exploitation of the rubber tree[M]//Webster C, Baulkwill W. Rubber. New York: Longman Scientific and Technical, 1989.
Tang C, Xiao X, Li H, et al. Comparative analysis of latex transcriptome reveals putative molecular mechanisms underlying super productivity of Hevea brasiliensis[J]. PLoS One, 2013, 8(9): e75307.
Rahman A Y A, Usharraj A O, Misra B B, et al. Draft genome sequence of the rubber tree Hevea brasiliensis[J]. BMC Genomics, 2013, 14: 75.
Tang C, Yang M, Fang Y, et al. The rubber tree genome reveals new insights into rubber production and species adaptation[J]. Nature Plants, 2016, 2(6): 16073.
Lau N S, Makita Y, Kawashima M, et al. The rubber tree genome shows expansion of gene family associated with rubber biosynthesis[J]. Scientific Reports, 2016, 6(1): 28594.
Pootakham W, Sonthirod C, Naktang C, et al. De novo hybrid assembly of the rubber tree genome reveals evidence of paleotetraploidy in Hevea species[J]. Scientific Reports, 2017, 7: 41457.
Liu J, Shi C, Shi C C, et al. The Chromosome-based rubber tree genome provides new insights into spurge genome evolution and rubber biosynthesis[J]. Molecular Plant, 2020, 13(2): 336-350.
Chow K S, Khoo J S, Mohd-Zainuddin Z, et al. Utility of PacBio Iso-Seq for transcript and gene discovery in Hevea latex[J]. Journal of Rubber Research, 2019, 22(4): 169-186.
Bennett M D, Leitch I J. Nuclear DNA amounts in angiosperms-583 new estimates[J]. Annals of Botany, 1997, 80(2): 169-196.
柳? 覲, 牛迎鳳, 吳? 裕, 等. 巴西橡膠樹栽培種質基因組C值測定和變異分析[J]. 熱帶亞熱帶植物學報, 2018, 26 (5): 523-528.
Dennis M S, Light D R. Rubber elongation factor from Hevea brasiliensis. Identification, characterization, and role in rubber biosynthesis[J]. The Journal of Biological Chemistry, 1989, 264(31): 18608-18617.
Sando T, Takeno S, Watanabe N, et al. Cloning and characterization of the 2-C-methyl-D-erythritol 4-phosphate (MEP) pathway genes of a natural-rubber producing plant, Hevea brasiliensis[J]. Bioscience, Biotechnology and Biochemistry, 2008, 72(11): 2903-2917.
Chow K S, Matisa M N, Bahari A, et al. Metabolic routes affecting rubber biosynthesis in Hevea brasiliensis latex[J]. Journal of Experimental Botany, 2012, 63(5): 1863-1871.
Yeang H Y, Yip E, Hamzah S. Characterisation of Zone 1 and Zone 2 rubber particles in Hevea brasiliensis latex[J]. Journal of Natural Rubber Ressearch, 1995, 10: 108-123.
Berthelot K, Lecomte S, Estevez Y, et al. Rubber particle proteins, HbREF and HbSRPP, show different interactions with model membranes[J]. Biochimica et Biophysica Acta (BBA)-Biomembranes, 2014, 1838(1): 287-299.
dAuzac J, Jacob J L, Prév?t J C, et al. The regulation of cis-polyisoprene production (natural rubber) from Hevea brasiliensis[M]//Pandalai S G. Recent research developments in plant physiology. Trivandrum: Research Singpost, 1997.
Tupy J. Sucrose supply and utilization for latex production[M]//DAuzac J, Jacob J-L, Chrestin H. Physiology of rubber tree latex. Boca Raton: CRC Press, 1989.
Tang C, Huang D, Yang J, et al. The sucrose transporter HbSUT3 plays an active role in sucrose loading to laticifer and rubber productivity in exploited trees of Hevea brasiliensis (para rubber tree)[J]. Plant, Cell and Environment, 2010, 33(10): 1708-1720.
Dusotoit-Coucaud A, Kongsawadworakul P, Maurousset L, et al. Ethylene stimulation of latex yield depends on the expression of a sucrose transporter (HbSUT1B) in rubber tree (Hevea brasiliensis)[J]. Tree Physiology, 2010, 30(12): 1586-1598.
Liu S, Lan J, Zhou B, et al. HbNIN2, a cytosolic alkaline/neutral-invertase, is responsible for sucrose catabolism in rubber-producing laticifers of Hevea brasiliensis (para rubber tree)[J]. New Phytologist, 2015, 206(2): 709-725.
Tungngoen K, Kongsawadworakul P, Viboonjun U, et al. Involvement of HbPIP2; 1 and HbTIP1;1 aquaporins in ethylene stimulation of latex yield through regulation of water exchanges between inner liber and latex cells in Hevea brasiliensis[J]. Plant Physiology, 2009, 151: 843-856.
Amalou Z, Bangratz J, Chrestin H. Ethrel (ethylene releaser)-induced increases in the adenylate pool and transtonoplast delta pH within Hevea latex cells[J]. Plant Physiology, 1992, 98(4): 1270-1276.
Pujade-Renaud V, Clement A, Perrotrechenmann C, et al. Ethylene-Induced increase in glutamine synthetase activity and mRNA levels in Hevea brasiliensis latex cells[J]. Plant Physiology, 1994, 105(1): 127-132.
Putranto R A, Duan C, Kuswanhadi, et al. Ethylene response factors are controlled by multiple harvesting stresses in Hevea brasiliensis[J]. PLoS One, 2015, 10(4): e0123618.
Archer B L, Cockbain E G. Rubber transferase from Hevea brasiliensis latex[J]. Methods in Enzymology, 1969, 15: 476-480.
Qu Y, Chakrabarty R, Tran H T, et al. A lettuce (Lactuca sativa) homolog of human Nogo-B receptor interacts with cis-prenyltransferase and is necessary for natural rubber biosynthesis[J]. Journal of Biological Chemistry, 2015, 290(4): 1898-1914.
Epping J, Van Deenen N, Niephaus E, et al. A rubber transferase activator is necessary for natural rubber biosynthesis in dandelion[J]. Nature Plants, 2015, 1(5): 15048.
Yamashita S, Yamaguchi H, Waki T, et al. Identification and reconstitution of the rubber biosynthetic machinery on rubber particles from Hevea brasiliensis[J]. eLife, 2016, 5: e19022.
Cherian S, Ryu S B, Cornish K. Natural rubber biosynthesis in plants, the rubber transferase complex, and metabolic engineering progress and prospects[J]. Plant Biotechnology Journal, 2019, 17(11): 2041-2061.
Light D R, Dennis M S. Purification of a prenyltransferase that elongates cis-isoprene rubber from latex of Hevea brasiliensis[J]. Journal of Biological Chemistry, 1989, 264(31): 18589-18597.
Cornish K. The separate roles of plant cis and trans prenyl transferases in cis-1,4-polyisoprene biosynthesis[J]. European Journal of Biochemistry, 1993, 218(1): 267-271.
Asawatreratanakul K, Zhang Y W, Wititsuwannakul D, et al. Molecular cloning, expression and characterization of cDNA encoding cis-prenyltransferases from Hevea brasiliensis: a key factor participating in natural rubber biosynthesis[J]. European Journal of Biochemistry, 2003, 270(23): 4671- 4680.
Takahashi S, Lee H J, Yamashita S, et al. Characterization of cis-prenyltransferases from the rubber producing plant Hevea brasiliensis heterologously expressed in yeast and plant cells[J]. Plant Biotechnology, 2012, 29(4): 411-417.
Post J, van Deenen N, Fricke J, et al. Laticifer-specific cis-prenyltransferase silencing affects the rubber, triterpene, and inulin content of Taraxacum brevicorniculatum[J]. Plant Physiology, 2012, 158(3): 1406-1417.
Dai L, Kang G, Li Y, et al. In-depth proteome analysis of the rubber particle of Hevea brasiliensis (para rubber tree)[J]. Plant Molecular Biology, 2013, 82(1-2): 155-168.
Uthup T K, Rajamani A, Ravindran M, et al. Distinguishing CPT gene family members and vetting the sequence structure of a putative rubber synthesizing variant in Hevea brasiliensis[J]. Gene, 2019, 689: 183-193.
Ding Z, Fu L, Tan D, et al. An integrative transcriptomic and genomic analysis reveals novel insights into the hub genes and regulatory networks associated with rubber synthesis in H. brasiliensis[J]. Industrial Crops and Products, 2020, 153: 112562 .
Dai L, Nie Z, Kang G, et al. Identification and subcellular localization analysis of two rubber elongation factor isoforms on Hevea brasiliensis rubber particles[J]. Plant Physiology and Biochemistry, 2017, 111: 97-106.
Tong Z, Wang D, Sun Y, et al. Comparative proteomics of rubber latex revealed multiple protein species of REF/SRPP family respond diversely to ethylene stimulation among different rubber tree clones[J]. International Journal of Molecular Sciences, 2017, 18(5): 958.
Wang D, Sun Y, Chang L L, et al. Subcellular proteome profiles of different latex fractions revealed washed solutions from rubber particles contain crucial enzymes for natural rubber biosynthesis[J]. Journal of Proteomics, 2018, 182: 53-64.
Berthelot K, Lecomte S, Estevez Y, et al. Homologous Hevea brasiliensis REF (Hevb1) and SRPP (Hevb3) present different auto-assembling[J]. Biochimica et Biophysica Acta (BBA) - Proteins and Proteomics, 2014, 1844(2): 473-485.
Berthelot K, Lecomte S, Estevez Y, et al. Hevea brasiliensis REF (Hev b 1) and SRPP (Hev b 3): An overview on rubber particle proteins[J]. Biochimie, 2014, 106: 1-9.
Priya P, Venkatachalam P, Thulaseedharan A, et al. Differential expression pattern of rubber elongation factor (REF) mRNA transcripts from high and low yielding clones of rubber tree (Hevea brasiliensis Muell. Arg.) [J]. Plant Cell Reports, 2007, 26(10): 1833-1838.
Oh S K, Kang H, Shin D H, et al. Isolation, characterization and functional analysis of a novel cDNA clone encoding a small rubber particle protein from Hevea brasiliensis[J]. Journal of Biological Chemistry, 1999, 274(24): 17132- 17138.
Collinssilva J, Nural A T, Skaggs A, et al. Altered levels of the Taraxacum kok-saghyz (Russian dandelion) small rubber particle protein, TkSRPP3, result in qualitative and quantitative changes in rubber metabolism[J]. Phytochemistry, 2012, 79: 46-56.
Hillebrand A, Post J, Wurbs D, et al. Down-regulation of small rubber particle protein expression affects integrity of rubber particles and rubber content in Taraxacum brevicorniculatum[J]. PLoS One, 2012, 7(7): e41874.
Laibach N, Hillebrand A, Twyman R M, et al. Identification of a Taraxacum brevicorniculatum rubber elongation factor protein that is localized on rubber particles and promotes rubber biosynthesis[J]. The Plant Journal, 2015, 82(4): 609- 620.
Chakrabarty R, Qu Y, Ro D K. Silencing the lettuce homologs of small rubber particle protein does not influence natural rubber biosynthesis in lettuce (Lactuca sativa)[J]. Phytochemistry, 2015, 113: 121-129.
Arokiaraj P, Jones H, Cheong K F, et al. Gene insertion into Hevea brasiliensis[J]. Plant Cell Reports, 1994, 13(8): 425-431.
Arokiaraj P, Yeang H Y, Cheong K F, et al. CaMV 35S promoter directs β-glucuronidas expression in the laticiferous system of transgenic Hevea brasiliensis (rubber tree)[J]. Plant Cell Reports, 1998, 17: 621-625.
Montoro P, Rattana W, Pugade-Renaud V, et al. Production of Hevea brasiliensis transgenic embryogenic callus lines by Agrobacterium tumefaciens: roles of calcium[J]. Plant Cell Reports, 2003, 21: 1095-1102.
Blanc G, Baptiste C, Oliver G, et al. Efficient Agrobacterium tumefaciens-mediated transformation of embryogenic calli and regeneration of Hevea brasiliensis Mull Arg. plants[J]. Plant Cell Reports, 2006, 24: 724-733.
Montoro P, Lagier S, Baptiste C, et al. Expression of the HEV2.1 gene promoter in transgenic Hevea brasiliensis[J]. Plant Cell Tissue and Organ Culture, 2008, 94(1): 55-63.
Leclercq J, Lardet L, Martin F, et al. The green fluorescent protein as an efficient selection marker for Agrobacterium tumefaciens-mediated transformation in Hevea brasiliensis (Mull. Arg)[J]. Plant Cell Reports, 2010, 29: 513-522.
Leclercq J, Martin F, Sanier C, et al. Over-expression of a cytosolic isoform of the HbCuZnSOD gene in Hevea brasiliensis changes its response to a water deficit[J]. Plant Molecular Biology, 2012, 80: 255-272.
Rekha K, Nazeem P A, Venkatachalam P, et al. Development of osmotin transgenics in Hevea brasiliensis Muell. Arg. using explants of zygotic origin[J]. Journal of Tropical Agriculture, 2014, 52(1): 7-20.
Lestari R, Rio M, Martin F, et al. Overexpression of Hevea brasiliensis ethylene response factor HbERF‐IXc5 enhances growth and tolerance to abiotic stress and affects laticifer differentiation[J]. Plant Biotechnology Journal, 2018, 16(1): 322-336.
Jayashree R, Nazeem P A, Rekha K, et al. Over-expression of 3-hydroxy-3- methylglutaryl-coenzyme A reductase 1 (hmgr1) gene under super-promoter for enhanced latex biosynthesis in rubber tree (Hevea brasiliensis Muell. Arg.)[J]. Plant Physiology and Biochemistry, 2018, 127: 414-424.
Fan Y, Xin S, Dai X, et al. Efficient genome editing of rubber tree (Hevea brasiliensis) protoplasts using CRISPR/ Cas9 ribonucleoproteins[J]. Industrial Crops and Products, 2020, 146: 112146 .
戴雪梅, 黃天帶, 李? 季, 等. 不同外植體對橡膠樹原生質體分離和再生的影響[J]. 分子植物育種, 2014, 12(6): 1259-1264.
Wang X, Shi M, Lu X, et al. A method for protein extraction from different subcellular fractions of laticifer latex in Hevea brasiliensis compatible with 2-DE and MS[J]. Proteome Science, 2010, 8: 35.
Chow K, Ghazali A, Hoh C, et al. RNA sequencing read depth requirement for optimal transcriptome coverage in Hevea brasiliensis[J]. BMC Research Notes, 2014, 7(1): 69.
Makita Y, Ng K K, Singham G V, et al. Large-scale collection of full-length cDNA and transcriptome analysis in Hevea brasiliensis[J]. DNA Research, 2017, 24(2): 159-167.
Xia Z, Xu H, Zhai J, et al. RNA-Seq analysis and de novo transcriptome assembly of Hevea brasiliensis[J]. Plant Molecular Biology, 2011, 77(3): 299.
Li D, Deng Z, Qin B, et al. De novo assembly and characterization of bark transcriptome using Illumina sequencing and development of EST-SSR markers in rubber tree (Hevea brasiliensis Muell. Arg.)[J]. BMC Genomics, 2012, 13(1): 192.
Pirrello J, Leclercq J, Dessailly F, et al. Transcriptional and post-transcriptional regulation of the jasmonate signalling pathway in response to abiotic and harvesting stress in Hevea brasiliensis[J]. BMC Plant Biology, 2014, 14(1): 341.
Fang Y, Mei H, Zhou B, et al. De novo transcriptome analysis reveals distinct defense mechanisms by young and mature leaves of Hevea brasiliensis (para rubber tree)[J]. Scientific Reports, 2016, 6: 33151.
Wang X, Wang D, Sun Y, et al. Comprehensive proteomics analysis of laticifer latex reveals new insights into ethylene stimulation of natural rubber production[J]. Scientific Reports, 2015, 5: 13778.
Xiang Q, Xia K, Dai L, et al. Proteome analysis of the large and the small rubber particles of Hevea brasiliensis using 2D-DIGE[J]. Plant Physiology and Biochemistry, 2012, 60: 207-213.
Wang X, Shi M, Wang D, et al. Comparative proteomics of primary and secondary lutoids reveals that chitinase and glucanase play a crucial combined role in rubber particle aggregation in Hevea brasiliensis[J]. Journal of Proteome Research, 2013, 12(11): 5146-5159.
Habib M A H, Gan C Y, Othman F, et al. Proteomics analysis of latex from Hevea brasiliensis (clone RRIM 600)[J]. Biochemistry and Cell Biology, 2017, 95(2): 232-242.
Liu J, Zhuang Y, Guo X, et al. Molecular mechanism of ethylene stimulation of latex yield in rubber tree (Hevea brasiliensis) revealed by de novo sequencing and transcriptome analysis[J]. BMC Genomics, 2016, 17(1): 257.
Li D, Wang X, Deng Z, et al. Transcriptome analyses reveal molecular mechanism underlying tapping panel dryness of rubber tree ( Hevea brasiliensis)[J]. Scientific Reports, 2016, 6: 23540.
Chao J, Chen Y, Wu S, et al. Comparative transcriptome analysis of latex from rubber tree clone CATAS8-79 and PR107 reveals new cues for the regulation of latex regeneration and duration of latex flow[J]. BMC Plant Biology, 2015, 15: 104.
Wei F, Luo S, Zheng Q, et al. Transcriptome sequencing and comparative analysis reveal long-term flowing mechanisms in Hevea brasiliensis latex[J]. Gene, 2015, 556(2): 153-162.
Piyatrakul P, Yang M, Putranto R A, et al. Sequence and expression analyses of ethylene response factors highly expressed in latex cells from Hevea brasiliensis[J]. PLoS One, 2014, 9(6): e99367
Nie Z, Kang G, Li Y, et al. Whole-transcriptome survey of the putative ATP-binding cassette (ABC) transporter family genes in the latex-producing laticifers of Hevea brasiliensis[J]. PLoS One, 2015, 10(1): e0116857.
Shearman J R, Sangsrakru D, Jomchai N, et al. SNP identification from RNA sequencing and linkage map construction of rubber tree for anchoring the draft genome[J]. PLoS One, 2015, 10(4): e0121961.
Makita Y, Kawashima M, Lau N S, et al. Construction of Pará rubber tree genome and multi-transcriptome database accelerates rubber researches[J]. BMC Genomics, 2018, 19(Suppl 1): 922.
Ding Z, Fu L, Tan D, et al. An integrative transcriptomic and genomic analysis reveals novel insights into the hub genes and regulatory networks associated with rubber synthesis in H. brasiliensis[J]. Industrial Crops and Products, 2020, 153: 112562.

