水稻Ossp1基因的亞細胞定位及其干旱條件下的表達
騰海艷
摘要:sp1基因是葉綠體蛋白質輸入調控的關鍵基因,本研究通過生物信息學分析、Real time PCR分析、瞬時表達分析等方法,對sp1在水稻中的同源基因Ossp1進行了結構和功能預測、組織表達和干旱響應性分析以及亞細胞定位分析。生物信息學分析結果顯示,水稻Ossp1基因位于7號染色體,基因登錄號為Os07g0647800,序列全長1 032 bp,SP1蛋白由343個氨基酸殘基組成,信號肽序列位于氮端,蛋白質分子中有2個跨膜區域。Real time PCR分析結果顯示,Ossp1在水稻葉片中表達量最高,其次為葉鞘,根中最低,此外,Ossp1在葉片中的表達具有明顯的干旱響應性,受到干旱脅迫誘導后,表達量顯著提高。采用瞬時表達進行亞細胞定位結果顯示,SP1蛋白定位于水稻葉綠體。上述結果顯示了Ossp1基因與水稻葉綠體及干旱脅迫響應的關系,為Ossp1基因功能的深入研究提供了基礎。
關鍵詞:水稻;Ossp1基因;亞細胞定位;干旱脅迫;基因表達
中圖分類號:Q786文獻標識碼:A文章編號:1000-4440(2020)03-0529-06
Subcellular localization and expression under drought conditions of rice Ossp1 gene
TENG Hai-yan
(School of Chemistry and Bioengineering, Yichun University, Yichun 336000, China)
Abstract:The? sp1 is a key gene for the regulation of chloroplast protein import, and Ossp1 is the homologous gene of sp1 in rice. In this study, the structure and function prediction, tissue expression, drought-responsiveness analysis and subcellular localization analysis of Ossp1 were carried out by bioinformatics analysis, realtime PCR analysis and transient expression analysis. The results of bioinformatics analysis showed that Ossp1 was located on chromosome 7(gene accession number: Os07g0647800), and the sequence was 1 032 bp in length. The SP1 protein was composed of 343 amino acid residues, the signal peptide was located at the N-terminus, and there were two transmembrane regions. The results of real time PCR analysis showed that the expression level of Ossp1 was highest in rice leaves, followed by leaf sheaths, and lowest in roots. In addition, the expression of Ossp1 could be significantly induced by drought stress, showed obvious drought responsiveness. Subcellular localization analysis of SP1 protein was performed by transient expression assay, and the results indicated that the SP1 protein was localized in rice chloroplasts. The above results show the relationship between gene Ossp 1 and rice chloroplast and drought responsiveness, and provide the basis for further research on the function of gene Ossp 1.
Key words:rice;Ossp1;subcellular localization; drought stress;gene expression
植物葉綠體含有大約3 000種蛋白質,其中絕大部分由核基因編碼,少數由葉綠體自身基因組編碼。核基因編碼的葉綠體蛋白質在細胞質合成后,經葉綠體外膜和內膜上的轉運蛋白TOC(Translocon of the outer membrane of chloroplasts)和TIC(Translocon of the inner membrane of chloroplasts)轉運到葉綠體內發揮功能[1-2]。研究結果[3-4]表明,擬南芥是通過控制外膜TOC蛋白的降解進行葉綠體蛋白輸入調控的,降解由SP1(Suppressor of ppi1 locus1)、SP2(Suppressor of ppi1 locus2)及CDC48(Cell division cycle 48)蛋白協作完成,其中,SP1蛋白負責催化TOC發生泛肽化,再由SP2和CDC48介導泛肽化的蛋白質由葉綠體外膜回到細胞質,被細胞質中的泛肽-蛋白酶體系統(Ubiquitin-proteasome system,UPS)降解,這個對TOC蛋白進行識別、泛肽化和降解的機制稱為葉綠體相關蛋白降解(Chloroplast-associated protein degradation,CHLORAD)[4]。CHLORAD機制不僅參與正常生長條件下的葉綠體蛋白輸入調控,還與植物的脅迫響應有關。在氧化脅迫條件下,擬南芥通過提高sp1基因表達促進TOC蛋白降解,從而降低葉綠體蛋白輸入量,減慢葉綠體內光反應速率和氧氣生成,避免加劇氧化脅迫[5-6]。在擬南芥中過表達sp1后,植物的抗逆能力相比野生型明顯增強,而相同條件下的sp1突變體則表現出發育滯后,抗逆性下降等表型,TOC等蛋白質的含量變化與SP1含量也表現出直接相關性,表明SP1是蛋白質由細胞質向葉綠體輸入的重要調節因子,與植物的生長發育和逆境響應機制密切相關[4,7]。
sp1在水稻中的同源基因目前尚未見報道。水稻是中國最重要的糧食作物之一,也是單子葉植物研究的模式植物,本研究選擇sp1在水稻中的同源基因(命名為Ossp1)作為研究對象,通過生物信息學分析、組織表達、干旱響應性分析和亞細胞定位,確定Ossp1基因在水稻基因組的序列信息、組織表達和干旱響應特性及SP1蛋白的亞細胞定位情況,以期為后續深入研究該基因的功能和闡明水稻葉綠體蛋白的輸入調節機制奠定基礎。
1材料與方法
1.1試驗材料
本研究所用水稻(Oryza sativa L.)品種為粳稻品種中花11,試驗材料種植于宜春學院苗圃溫室,常規管理。
1.2水稻sp1基因(Ossp1)及其編碼蛋白質的生物信息學分析
參照擬南芥(Arabidopsis thaliana)的sp1基因(AT1G63900)編碼序列和蛋白質氨基酸序列,在NCBI(https://www.ncbi.nlm.nih.gov/)網站通過BLAST功能查找其在水稻基因組中的同源序列。通過DNAMAN軟件進行水稻和擬南芥SP1蛋白氨基酸序列同源性分析。利用SignalP軟件進行水稻SP1蛋白信號肽位置分析,TMHMM軟件進行跨膜區位置預測,SOPMA軟件進行二級結構預測。
1.3RNA提取、反轉錄和Ossp1克隆
正常生長21 d的土培水稻苗,取倒數第二片葉,平行取樣3份,混合后快速放入液氮中,然后置于-80 ℃冰箱保存,經Magen試劑盒提取總RNA,Vazyme反轉錄試劑盒反轉錄后,cDNA產物作為模板,根據Ossp1的上下游序列設計引物(Ossp1-F:5′-ATGTTGATCCCATGGGGCGG-3′,Ossp1-R:5′-TCAATGGCGGAAAGTTCTC-3′)進行PCR擴增,擴增產物經瓊脂糖凝膠電泳檢測后,切膠回收目的條帶,并將回收產物連接至pMD-18T載體,轉化大腸桿菌TOP10感受態細胞,菌液PCR鑒定后,送生物公司測序。
1.4水稻植株的栽培和干旱處理
用于組織表達分析的中花11野生型水稻幼苗,木村營養液培養,每3 d更換1次營養液,培養至三葉一心,沖洗掉根部營養液成分,吸水紙吸干多余水分,每株分別剪下倒數第二片葉片、葉鞘、根,用于RNA提取和組織表達分析。
用于干旱處理的中花11野生型水稻幼苗,木村營養液培養至至一葉一心,栽種于砂土基質(砂與水稻種植用土各一半,施加適量尿素)中繼續生長至三葉一心(從發芽起共約21 d),停止供水,開始進行干旱處理,至所有葉片完全卷起和葉片全卷后16 h各取樣1次,每次3株,每株剪下倒數第二片葉后快速放入液氮中,然后置于-80 ℃冰箱保存,用于RNA提取和干旱響應性分析。
1.5Real time PCR分析
葉片、葉鞘和根樣品按方法1.3的方法進行RNA提取和反轉錄,cDNA產物用ddH2O稀釋10倍,作為模板,以OsActin1(基因登錄號:Os03g07181000)作為內參基因,以未經干旱處理的樣品作為對照,進行目的基因的Real-time PCR擴增,數據處理采用2-△△Ct法[8],Excel軟件和Sigma Plot軟件進行分析和作圖。Real time PCR反應體系(10 μl):2×SYBR mix(TaKaRa) 50 μl,cDNA 10 μl,F、R引物各05 μl,ddH2O 30 μl。Real time PCR反應條件:95 ℃ 3 min;95 ℃ 15 s,60 ℃ 30 s,40個循環。Real time PCR擴增使用引物:Ossp1-1210-F:5′-ACTTTCCGCCATTGACA-3′;Ossp1-1379-R:5′-TGCTTCGGCGCAATAC-3′;OsActin1-F:5′-CTTCATAGGAATGGAAGCTGCGGGTA-3′;OsActin1-R:5′-CGACCACCTTGATCTTCATGCTGCTA-3′。
1.6Ossp1-gfp瞬時表達載體的構建
以帶有Ossp1編碼序列的pMD-18T載體為模板,擴增不含終止密碼子TGA的Ossp1編碼序列(擴增引物及酶切位點:Ossp1-Spe I-F:5′-TGACACTAGTATGTTGATCCCATGGGGC-3′;Ossp1-Hind-R:5′-TCAGAAGCTTTAGGCGGAAAGTTCTCAC-3′),擴增產物經瓊脂糖凝膠電泳檢測后,用回收試劑盒回收DNA片段,限制性酶切后,將Ossp1序列連入到pOX載體的35S啟動子和gfp序列之間,構建p35S-sp1-gfp-nos表達框。連接產物轉化大腸桿菌TOP10菌株,經菌液PCR和核酸序列測定獲得陽性克隆。
1.7原生質體的分離和瞬時表達分析
水稻葉鞘細胞原生質體的分離:取生長7 d狀態良好的水稻苗,按Zhang等[9]的方法制備原生質體。帶有p35S-sp1-gfp-nos表達框的質粒20 μl(總質量10 μg以上),采用PEG介導法[9]轉入新鮮制備的水稻原生質體,同時轉化帶有p35S-gfp表達框的質粒作為對照,28 ℃避光孵育18 h,250 g離心5 min,保留沉淀和少許上清,輕輕搖勻,激光共聚焦顯微鏡(Carl Zeiss, LSM7 DUO)觀察結果并拍照。
2結果與分析
2.1Ossp1基因堿基序列和SP1蛋白氨基酸序列特征
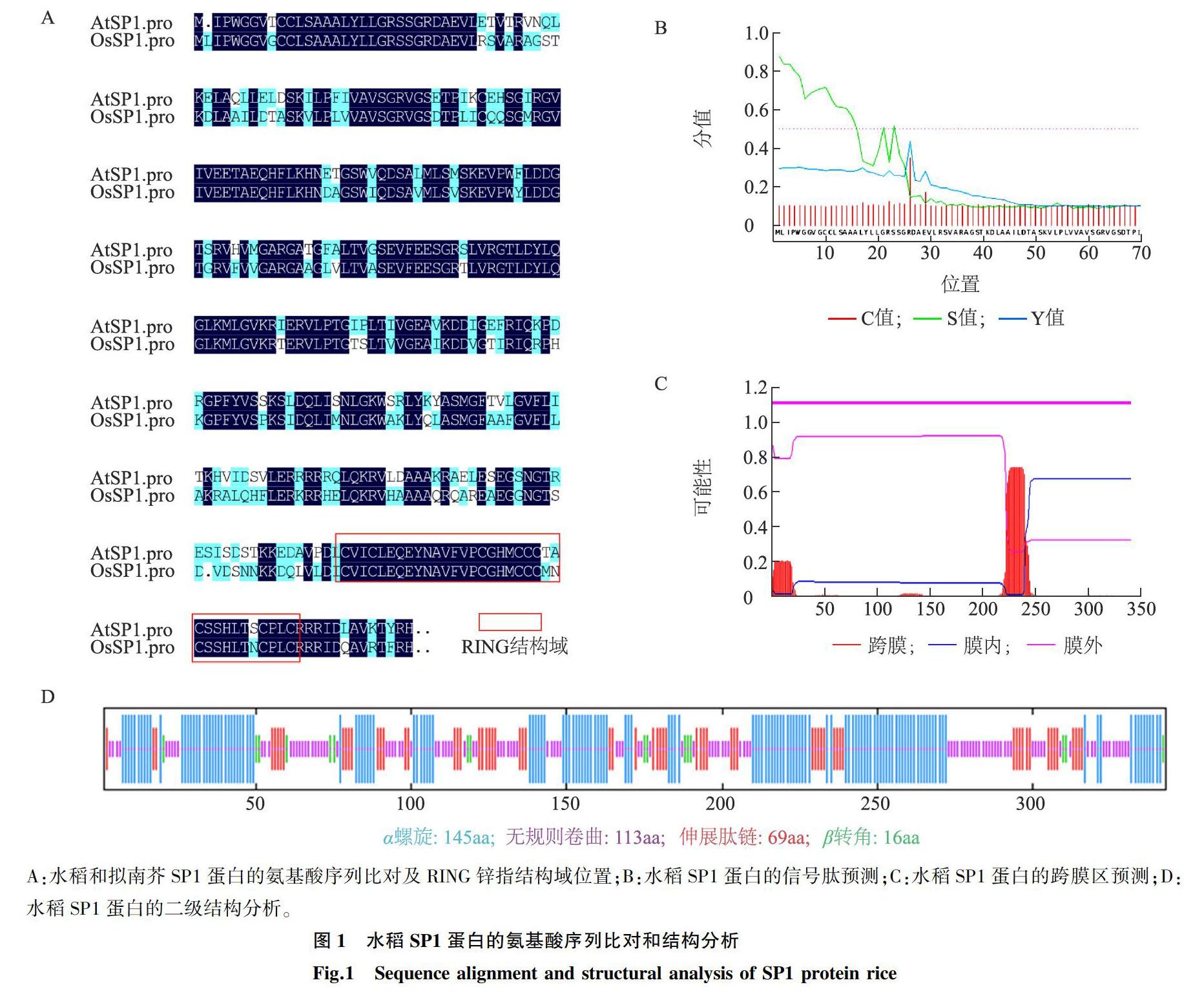
以擬南芥sp1基因的堿基序列和蛋白質氨基酸序列作為參照,經BLAST分析,確定水稻中的同源基因位于7號染色體,基因登錄號為Os07g0647800,基因堿基序列全長1 032 bp。水稻SP1蛋白質氨基酸序列由343個氨基酸殘基組成,與擬南芥SP1氨基酸殘基數相同。DMAMAN等軟件分析結果顯示,水稻和擬南芥SP1蛋白的氨基酸序列相似度為7118%,其中靠近羧基端的35個氨基酸殘基構成RING(Really interesting new gene)型鋅指結構域(圖1A),水稻SP1蛋白共有2個跨膜區,一個位于氨基端,含25個氨基酸殘基(圖1B,圖1C),為SP1蛋白的信號肽序列,該信號肽與擬南芥SP1信號肽序列基本相同,只有2個氨基酸殘基差異,推測SP1蛋白也定位于葉綠體外膜,另一個跨膜區域位于第220~250氨基酸殘基處,2個跨膜區之間的肽段位于膜外,第二個跨膜區后的肽段(羧基端)位于膜內側(圖1C)。二級結構預測顯示SP1蛋白中含有多段α螺旋結構,占4227%,其余為無規卷曲、伸展性結構以及少量β轉角(圖1D),分別占3294%、2012%和466%。
2.2Ossp1基因克隆
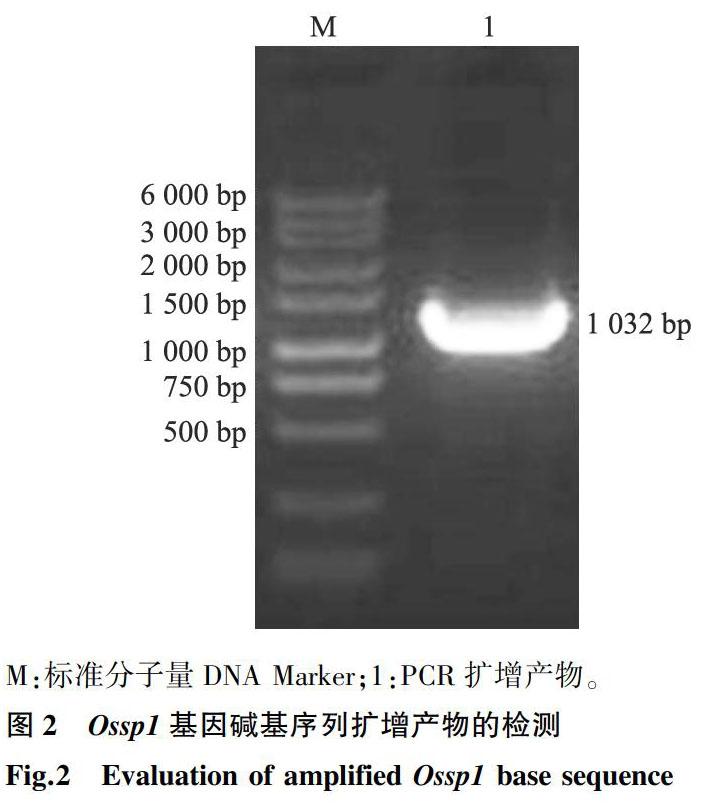
根據生物信息學分析結果,在開放讀碼框兩側設計特異性引物進行PCR擴增,以反轉錄獲得的cDNA為模板,PCR擴增獲得Ossp1的全長堿基序列,產物經回收和測序驗證,確定該序列與BLAST分析獲得的mRNA序列一致,片段長度為1 032 bp,電泳檢測結果見圖2。
2.3基因Ossp1的組織表達和干旱響應性分析
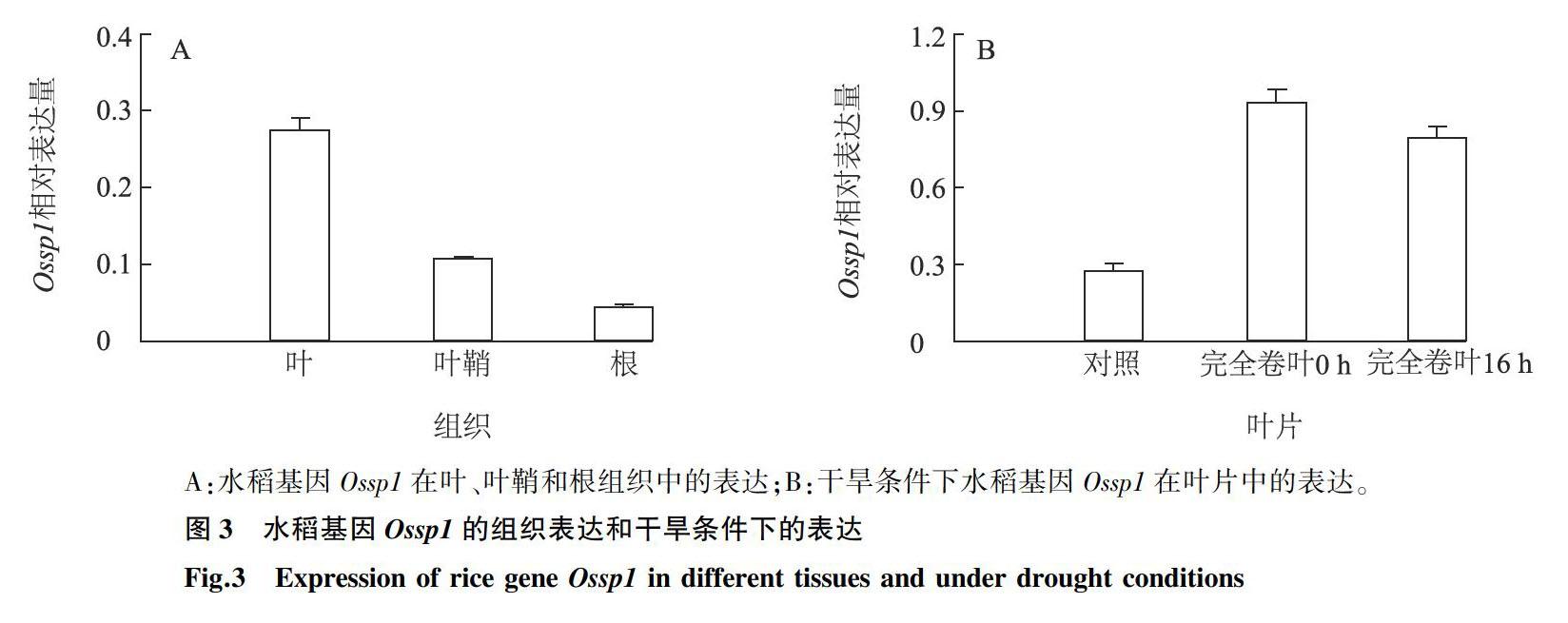
野生型水稻幼苗在自然生長條件下經營養液培養至三葉一心后,取根、葉鞘和倒數第二片葉片分析基因Ossp1的組織表達情況。Real time PCR結果顯示,目的基因在3種組織中的表達存在差異,在葉片中的表達水平最高,而在根中表達水平最低。Ossp1在正常生長條件下的表達水平均低于內參基因OsActin1,葉中表達水平為OsActin1的0.27倍,葉鞘和根中的表達水平分別為OsActin1的011倍和004倍(圖3A)。
在干旱條件下,基因Ossp1的表達水平顯著提高。干旱處理至所有葉片完全卷葉時,基因Ossp1的表達水平接近內參基因OsActin1的表達水平,持續干旱至卷葉時間達到16 h時,基因Ossp1表達水平較葉片剛完全卷葉時有所下降,但相比對照仍然較高(圖3B)。
2.4Ossp1-gfp載體的構建
為確定SP1蛋白在細胞內的定位情況,我們以綠色熒光蛋白基因gfp作為報告基因,構建了Ossp1與gfp融合的瞬時表達載體。根據SP1蛋白的信號肽分析結果,該蛋白的信號肽位于氨基端,因此我們構建了gfp堿基序列位于Ossp1堿基序列3′端的融合表達載體(羧基端融合),以防止GFP遮蔽信號肽而影響定位。經序列擴增、酶切、連接、轉化大腸桿菌并測序后,獲得Ossp1堿基序列(不含終止密碼子TAG)插入到gfp上游的融合表達載體陽性克隆,載體結構見圖4。
2.5水稻SP1蛋白的亞細胞定位
為了分析水稻SP1蛋白在細胞內的定位情況,用構建好的帶有p35S-sp1-gfp-nos表達框的質粒轉化水稻葉鞘原生質體,同時用p35S-gfp-nos質粒轉化水稻葉鞘原生質體作為對照。激光共聚焦顯微鏡檢測結果顯示,作為對照的p35S-gfp-nos質粒轉化后,GFP蛋白全部定位于細胞質,而p35S-sp1-gfp-nos轉化后,SP1-GFP融合蛋白出現的區域與葉綠素自發熒光區域完全重合,表明融合蛋白已經定位到葉綠體(圖5),可以確定SP1蛋白定位于葉綠體。
3討論
葉綠體是植物最重要的細胞器之一,是植物進行光合作用的場所,此外還涉及植物的氨基酸代謝、脂肪酸代謝和脅迫信號ROS的產生[10]。葉綠體是半自主性細胞器,其自身基因組負責約100種蛋白質的合成,其余絕大多數蛋白質通過核基因編碼,在細胞質合成后轉運到葉綠體內[11]。
在過去的幾十年里,核基因編碼的蛋白質向葉綠體的轉運過程已經大致研究清楚。葉綠體的外膜和內膜上共結合有十幾種轉運蛋白,外膜上的轉運蛋白統稱為TOC,內膜上的轉運蛋白統稱為TIC,兩類蛋白均有多個成員,這些成員在分子量、分子結構和轉運功能方面均有差別[12]。TOC和TIC形成的轉運復合體可以識別葉綠體蛋白質的N端信號肽,引導蛋白質進入葉綠體,并進一步定位到葉綠體基質、類囊體膜、類囊體基質等部位[13]。
在植物生長發育過程中,以及對各種脅迫作出響應的過程中,葉綠體都需要對其蛋白質組及時進行動態調整,以適應體內體外生理生化條件的變化[14]。一直以來,盡管人們對葉綠體蛋白質的轉入機制已經有深入研究,但是,對葉綠體蛋白質輸入調控的過程了解較少。最近幾年,Ling等[6-7]陸續報道了擬南芥葉綠體蛋白質輸入調控基因sp1的功能,并于2019年報道了完整的擬南芥葉綠體蛋白質輸入調控機制,確定植物對葉綠體蛋白質輸入的調控是通過控制葉綠體外膜上TOC蛋白的降解實現的[4]。SP1蛋白對TOC蛋白的降解具有關鍵作用,sp1基因表達量的提高能夠促進外膜上TOC數量的下降,并減少葉綠體蛋白質的輸入量[4]。水稻Ossp1基因堿基序列長度、水稻SP1蛋白多肽鏈的長度均與擬南芥sp1基因和SP1蛋白相同,2種蛋白質的一級結構也具有高度相似性,BLAST分析結果顯示,Ossp1是水稻基因組中唯一與sp1具有高度相似性的基因,因此,可以確定Ossp1為sp1在水稻中的同源基因。
擬南芥SP1蛋白是泛肽E3連接酶家族成員[15],并且屬于RING(Really interesting new gene)型E3連接酶,酶分子中均含有RING結構域,這是一種鋅指結構域,與酶分子和其他蛋白質的互作功能及泛肽化功能有關,多數RING型E3連接酶都參與植物的脅迫響應[16-17]。擬南芥sp1基因的表達可在脅迫誘導下提高,sp1過表達植株對鹽脅迫耐受性高于野生型植株,而sp1突變植株則出現耐受性下降表型,表明該基因不僅參與正常生長條件下的葉綠體蛋白質輸入調控,還參與植物的逆境脅迫響應。本研究通過序列對比,確定水稻SP1具有與擬南芥SP1相同的RING結構域,因此可以確定水稻SP1也屬于RING型E3連接酶。轉錄分析結果表明,Ossp1的表達具有明顯的干旱響應性特征,在干旱條件下,Ossp1的表達量可以提高到正常條件下的4倍左右,表明該基因參與了水稻的干旱響應過程。此外,根據亞細胞定位結果,結合信號肽預測結果,可以確定水稻SP1定位在葉綠體,并且信號肽位于肽鏈N端,與擬南芥SP1的信號肽位置和定位相同。
水稻是中國最重要的糧食作物之一,干旱、高鹽等各類逆境條件對水稻的生長和產量影響嚴重[18-19]。本研究結果顯示了基因Ossp1與水稻葉綠體及水稻干旱脅迫響應之間的關系,為進一步的基因功能研究和揭示水稻葉綠體蛋白質輸入調控機制奠定了基礎。目前,我們已經通過轉基因技術開展了Ossp1基因過量表達植株和轉基因植株的培育,以期通過轉基因植株對Ossp1基因的功能進行更為深入的研究。
參考文獻:
[1]JARVIS P, KESSLER F. Mechanisms of chloroplast protein import in plants[M].New York: Springer, 2014: 241-259.
[2]WATSON S J, SOWDEN R G, JARVIS P. Abiotic stress-induced chloroplast proteome remodelling: a mechanistic overview[J]. Journal of Experimental Botany, 2018, 69(11): 2773-2781.
[3]LEE S, LEE D W, LEE Y, et al. Heat shock protein cognate 70-4 and an E3 ubiquitin ligase, CHIP, mediate plastid-destined precursor degradation through the ubiquitin-26S proteasome system in Arabidopsis[J]. Plant Cell, 2009, 21(12):3984-4001.
[4]LING Q, BROAD W, TRSCH R, et al. Ubiquitin-dependent chloroplast-associated protein degradation in plants[J]. Science, 2019, 363(6429):4467.
[5]KESSLER F. Chloroplast delivery by UPS[J]. Science, 2012, 338(6107):622-623.
[6]LING Q, WEIHUA H, AMY B, et al. Chloroplast biogenesis is regulated by direct action of the ubiquitin-proteasome system[J]. Communicative & Integrative Biology, 2013, 338(2):655-659.
[7]LING Q, JARVIS P. Regulation of chloroplast protein import by the ubiquitin E3 ligase SP1 is important for stress tolerance in plants[J]. Current Biology, 2015, 25(19):2527-2534.
[8]SCHMITTGEN T D, LIVAK K J. Analyzing real-time PCR data by the comparative CT method[J]. Nat Protoc, 2008, 3(6):1101-1108.
[9]ZHANG Y, SU J, DUAN S, et al. A highly efficient rice green tissue protoplast system for transient gene expression and studying light/chloroplast-related processes[J]. Plant Methods, 2011, 7(1):30.
[10]BOSE J, MUNNS R, SHABALA S, et al. Chloroplast function and ion regulation in plants growing on saline soils: Lessons from halophytes[J]. Journal of Experimental Botany, 2017, 68(12): 3129-3143.
[11]LEE D W, HWANG I. Evolution and design principles of the diverse chloroplast transit peptides[J]. Molecules & Cells, 2018, 41(3):161-167.
[12]胥華偉,侯典云. 植物細胞中蛋白質向葉綠體轉運的研究進展[J]. 植物學報, 2018, 53(2): 264-275.
[13]CHOTEWUTMONTRI P, REDDICK L E, MCWILLIAMS D R, et al. Differential transit peptide recognition during preprotein binding and translocation into flowering plant plastids[J]. Plant Cell, 2012, 24(7):3040-3059.
[14]JARVIS P, LPEZ-JUEZ E. Biogenesis and homeostasis of chloroplasts and other plastids[J]. Nat Rev Mol Cell Biol, 2013, 14(12): 787-802.
[15]PAN R, HU J. Sequence and biochemical analysis of Arabidopsis SP1 protein, a regulator of organelle biogenesis[J]. Communicative & Integrative Biology, 2017, 10(4): e1338991.
[16]LIM S D, YIM W C, MOON J C, et al. A gene family encoding RING finger proteins in rice: their expansion, expression diversity, and co-expressed genes[J]. Plant Mol Biol,2010, 72(4/5):369-380.
[17]BORJA B, JOSE J, ALBERTO C, et al. ABA inhibits myristoylation and induces shuttling of the RGLG1 E3 ligase to promote nuclear degradation of PP2CA[J]. The Plant Journal, 2019, 98(5): 813-825.
[18]劉艷,孫文濤,雋英華. 控水對水稻生長發育及產量的影響[J].江蘇農業科學,2018,46(4):53-55.
[19]李婷,朱長波,李俊偉,等. 海水脅迫對海稻86種子萌發和幼苗生長的影響[J].南方農業學報,2018,49(7):1297-1303.
(責任編輯:陳海霞)

