新型冠狀病毒序列變異時空分析系統的設計與實現
李建勛,李 鑫,謝 康,徐德晨,李 杰
(哈爾濱工業大學 計算機科學與技術學院 生物信息學研究中心,哈爾濱 150001)
2019年12月出現以干咳、發熱、渾身乏力等為典型癥狀的新型肺炎疫情,中國國家衛生健康委員會暫時將其命名為新型冠狀病毒肺炎。2020年1月31日,世界衛生組織(WHO)宣布將新型冠狀病毒肺炎列入“國際經濟公共衛生事件”,并于2月11日將之命名為COVID-19[1]。同日,國際病毒分類委員會聲明,將引發疾病的新型冠狀病毒命名為SARS-CoV-2[2]。
1月份,新型冠狀病毒肺炎擴散到國內多個省份和地區,后續世界各國也爆發了疫情[1]。截止到4月16日,中國累計確診COVID-19共83 799例,但疫情已得到基本的控制,現存確診1 891例,其中境外輸入1 534例,占絕大部分[注]數據來源于國家及各省市地區衛健委,丁香醫生整理(https://ncov.dxy.cn/ncovh5/view/pneumonia?)。然而,與此同時,新型冠狀病毒已在世界多國相繼爆發,全球累計確診達2 047 104例,現存確診140 675例,世界整體形勢不容樂觀。另外,國內外有多位相關領域的醫學專家表示,新型冠狀病毒短期內不會消失,可能與人類長期共存。
冠狀病毒是一種包膜的單鏈RNA病毒,它的受體結合域能快速突變和重組,以適應并感染大量物種[3]。自病毒疫情爆發以來,學術界對于新型冠狀病毒的基本傳染數R0(Basic reproduction number)有許多研究和討論,在多篇論文中給出的取值呈越來越大的趨勢。4月7日,美國疾病管制局期刊《新興傳染病》發表了關于新型冠狀病毒的最新研究結論[4],研究人員通過兩種建模方法(到達模型和病例計數模型),最終計算得出新型冠狀病毒肺炎R0中位數為5.7(95%CI 3.8-8.9),也就是說,一名新型冠狀病毒肺炎患者可以傳染5.7人,是之前認為的2~3倍。
在這種背景下,對新型冠狀病毒序列的變異情況進行監控、分析及可視化,就顯得尤為重要[5-6]。國內外已有一些相關的軟件系統向用戶開放,但是這些已有系統沒有從時空角度進行分析的功能或相關功能仍有進一步完善的空間,例如北京志諾維思公司的“戰新冠”系統(https://fight-sars2.genowis.com/),提供了對病毒序列的變異分析及可視化,但沒有從時空角度進行分析的功能;國家生物信息中心開發的2019新型冠狀病毒信息庫線上系統(https://bigd.big.ac.cn/ncov/),提供了包含時空角度的病毒序列變異分析功能,但是其時空變異分析功能是使用熱力圖來實現,完全是靜態圖的形式,也不支持用戶對基準序列、時間區間和地域范圍進行選擇,功能上仍然需要擴展和進一步完善。本文所設計和實現的新型冠狀病毒變異時空分析系統用于解決這一問題,能夠動態顯示不同時間和地域病毒序列的變異境況(系統現已上線,網址http://bionet.org.cn/NCP-web/)。
1 系統的總體功能
新型冠狀病毒變異時空分析系統的主要功能有三個:病毒序列來源統計、病毒序列變異統計及病毒序列比對。
1.1 系統首頁
系統首頁主要包括系統導航欄、系統信息簡介、系統三個主要功能模塊的簡介。系統首頁的部分截圖如圖1所示。
圖1 系統首頁截圖展示Fig.1 Screenshot of the homepage
1.2 病毒序列來源統計
自新型冠狀病毒爆發以來,各國研究人員在SARS-CoV-2測序方面做了大量的工作[7-12]。全球共享流感數據倡議組織(GISAID)將各國研究人員提交的病毒序列數據進行了整理和匯總,并在其官方網站(https://www.gisaid.org/)進行公開。本文使用的源數據來自GISAID官網[13]。
新型冠狀病毒序列來源統計功能旨在對來自不同國家和地區的新型冠狀病毒序列的數量進行統計、以地圖和餅狀圖的形式進行可視化。
對新型冠狀病毒序列來源的統計和可視化有兩種形式——地圖和餅狀圖(見圖2)。
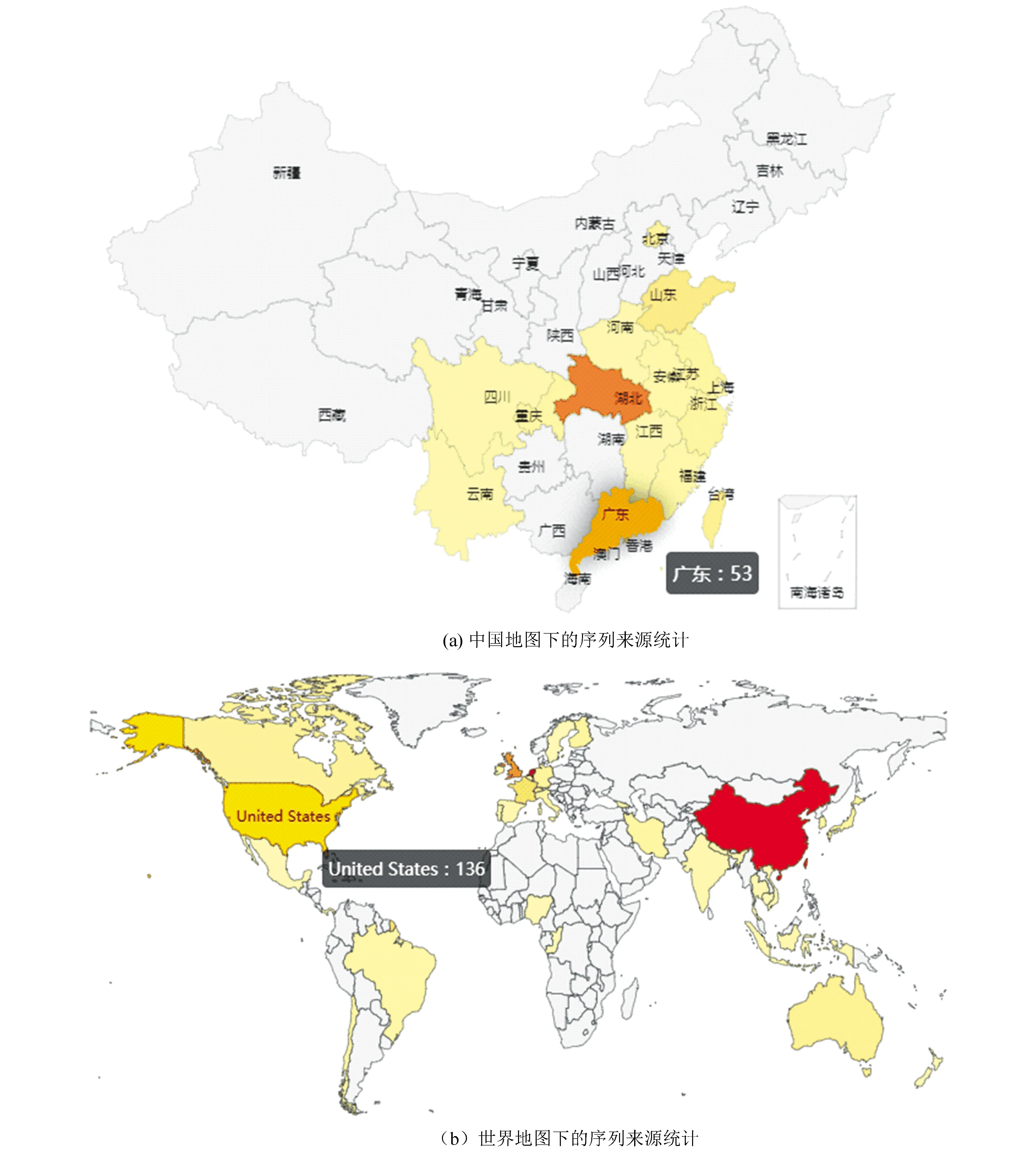
圖2 地圖形式的序列來源可視化Fig.2 Visualization of sequence sources in map form
圖2展示了中國地圖和世界地圖上的新型冠狀病毒序列來源統計,當用戶將鼠標懸浮在目標國家或者地區上時,會以彈框的形式展示該國家或地區已提交的序列總數。
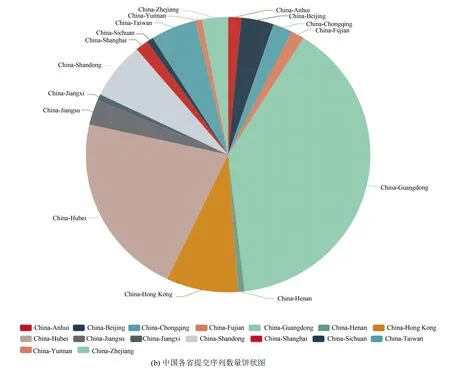
圖3展示了餅狀圖形式下世界各國提交序列數量統計和中國各省提交序列數量統計。此外,系統還提供將以上二者結合的餅狀圖可視化。同時,當用戶將鼠標懸浮在圖中的扇形上時,系統會以彈框的形式展示該國家或省份提交的序列數占總數的百分比。
根據系統給出的結果(截止到3月19日的數據),可以發現世界范圍內共提交SARS-CoV-2序列934條,中國提交的病毒序列最多,共267條,占28.59%;國內廣東省提交的序列最多,共90條,占33.71%,湖北省次之,共42條,占15.73%。希望世界各國及時共享更多的SARS-CoV-2序列,促進COVID-19的研究。
1.3 病毒序列變異統計
系統將來自GISAID的序列數據進行篩選和預處理之后,將所有序列轉換成等長的形式存入數據庫(具體的篩選與預處理過程及方法見3.1節病毒序列數據的處理)。在此基礎上,系統在第一次啟動時,會比對數據庫中所有序列,將每一個位點上頻次最高的核苷酸作為該位點的標準核苷酸,從而得到一條統計意義上的標準序列(Standard sequence),后續可以此標準序列為基準進行變異分析(也支持由用戶指定某一序列作為標準序列的變異分析)。
該模塊下又分為序列變異統計分析和序列變異時空分析兩個子功能模塊。
1.3.1 序列變異統計分析
圖4展示了系統為用戶提供的選擇入口,用戶可在該入口下指定標準序列、選擇待分析的目標序列集合、指定要分析的起始位點和終止位點。通過圖4的設置面板完成分析基本參數選定并點擊分析按鈕,系統會給出病毒變異統計分析的兩個結果,如圖5和圖6所示。

圖4 選擇基準序列與待分析序列Fig.4 Choice for base sequence and sequences to be analyzed
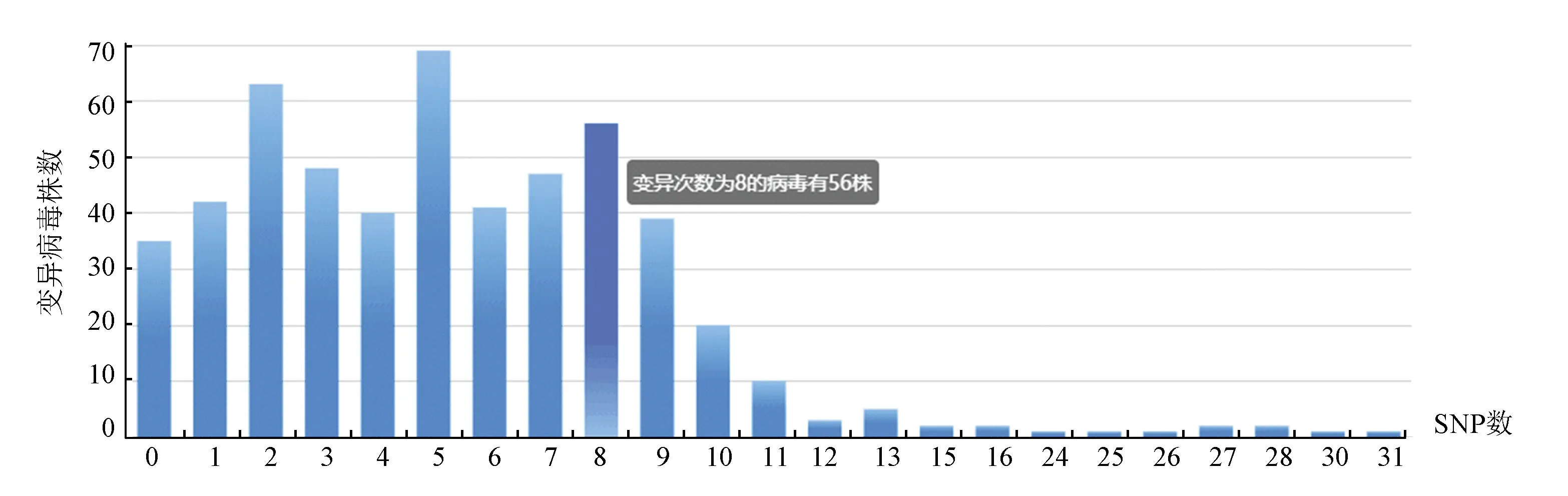
圖5 在選擇位點之間的變異病毒株數分布Fig.5 Distribution of variant virus strains between selected sites
圖5展示了用戶選定的目標序列集合與基準序列相比,具有不同變異位點數的病毒株數分布。除具有不同變異頻數的病毒株數分布統計(見圖5)之外,系統還會對病毒序列在不同時間和地區的平均變異率做統計(見圖6)。該功能模塊會將所有序列按照國家進行分組,每一組用不同的顏色加以區分,組內序列按照采樣時間進行排序,給出所在地區在每一個采樣日的平均變異率(每一個采樣日內,對所在地區當天采樣的所有病毒序列的變異率求取平均值)。該柱狀圖可以直觀地展示某一地區內病毒序列的變異率隨著時間遞增的變化情況。
根據系統給出的分析結果(截止到3月13日的病毒序列數據),由圖5可以發現,大部分序列的變異位點數少于10個,單條序列的最多變異數為31,有1條;由圖6可以發現,中國在2020年1月29日采樣的病毒序列的平均變異率最高,為0.09%,其余大部分序列變異率波動不大,絕大部分序列的變異率在0.04%以下。
1.3.2 序列變異時空分析
隨著疫情的擴散,病毒在不同地區的變異率差異、在不同時間點的變異率變化這兩個信息尤為重要,可以幫助科研人員對病毒進行更加精準的分析和研究,從而為疾病的研究和防治提供指導。該功能模塊可以實現這一目標。
與病毒序列變異統計模塊的參數面板相比,圖7所示的參數面板具有更豐富的選項,包括標準序列、要分析的時間區間(以采樣時間為準)、要分析的地域(以采樣地域為準,可選擇全世界的不同國家,也可以選擇全國的不同省份)。在提交參數選項之后,系統會給出兩個分析結果圖,如圖8和圖9所示。

圖7 變異時空分析的基本參數選項Fig.7 Basic parameter options for spatiotemporal analysis of variation
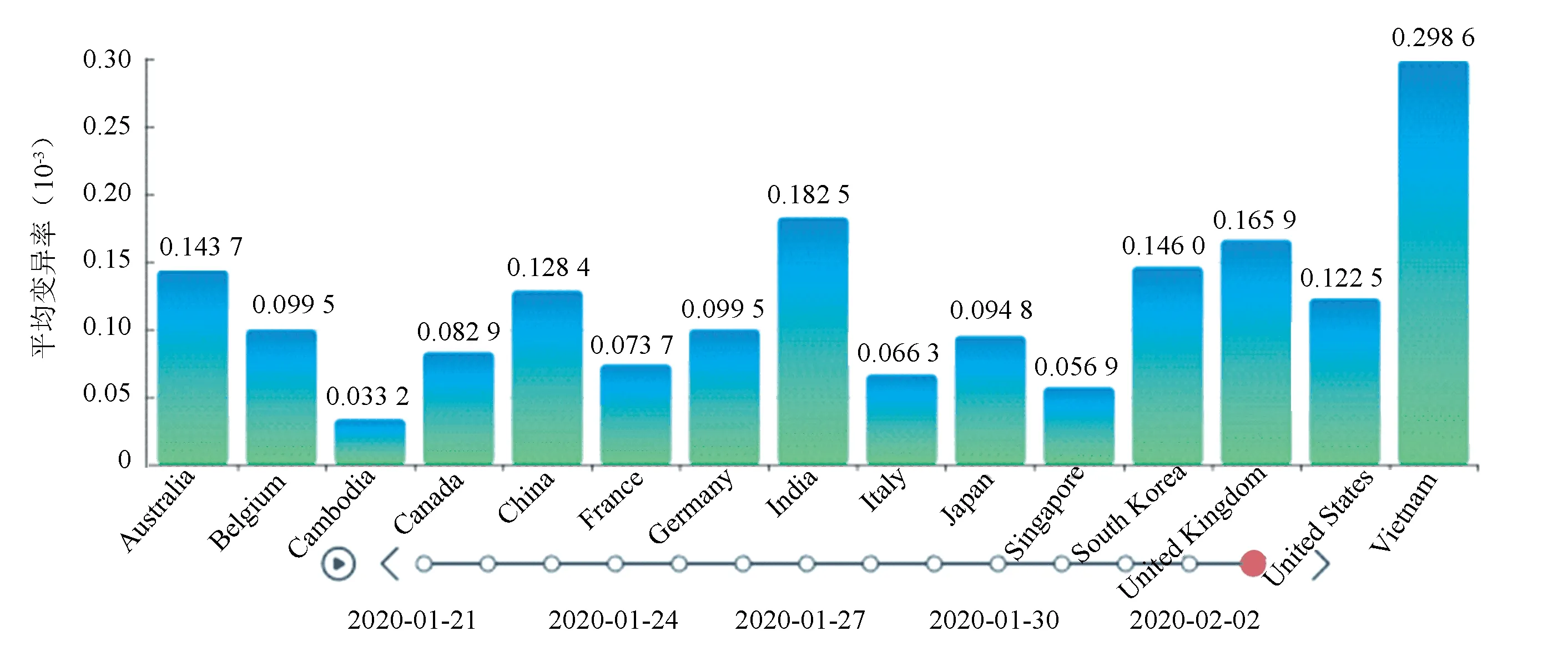
圖8 不同時間下各地區的變異率Fig.8 Variation rates in different regions at different times
圖8展示了在2020年1月21日到2020年2月3日之間十五個不同國家的病毒序列平均變異率(對所在國家或地區的截止到觀測日期所采樣的所有病毒序列的變異率求取平均值)的變化情況。在系統中實際操作時,該條形圖是時間為變量進行動態播放的,在播放到最后一個時間節點時就會停止輪播。根據圖8我們可以發現,在1月21日到2月3日之間,中國的平均變異率為0.013%,大致在平均水平左右;平均變異率最高的國家是越南,為0.030%;平均變異率最低的國家是柬埔寨,為0.003%。
圖9展示的是序列變異時空分析模塊給出的第二個分析結果。與圖8相同,該折線圖在實際操作時也是隨時間變化動態播放。該圖展示的是1月21日到2月3日之間,十五個不同國家整體的病毒序列變異情況。其中黃色折線為最大變異率、綠色折線為最小變異率、藍色折線為平均變異率(對全球所有國家或者國內所有省份的截止到觀測日期所采樣的所有病毒序列的變異率求取平均值),鼠標在圖上懸浮時會給出詳細信息。根據圖9可以發現,1月21日到2月3日之間,病毒序列的最大變異率為0.09%,結合1.3.1節的圖6,可知該序列在1月29日采樣于中國廣東;最小變異率為0%,該序列在1月23日被采樣;平均變異率在0.01%左右波動。
1.4 病毒序列比對
除病毒序列的來源統計和病毒序列變異統計兩個模塊外,系統還設有病毒序列比對模塊。該模塊下又細分為病毒序列基本信息展示和病毒序列差異比對兩個子模塊。
1.4.1 病毒序列基本信息展示
GISAID官網除了提供病毒序列之外,還給出了一些病毒序列相關的信息,其中包括病毒序列號、病毒名稱、采樣地點、采樣實驗室、提交實驗室、作者信息、采樣時間,系統以表格的形式對這些信息做了展示(見圖10)。表格的每一行對應一個病毒序列,所有病毒序列按照國家分組并按照采樣時間升序排序。同時,在實際操作系統時,表中每一行前面都有一個單選框,用戶在感興趣的單選框內打鉤,即可選中該行所對應的序列,進行病毒序列差異比對。
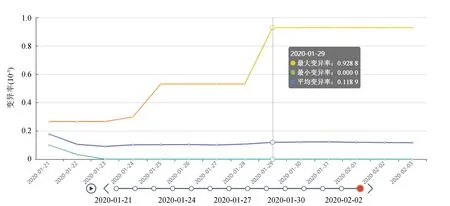
圖9 不同時間下所有地區的累計變異率Fig.9 Cumulative variation of all regions at different times

圖10 序列的基本信息表Fig.10 Sequence basic information table
1.4.2 病毒序列差異比對
圖11展示的是系統對EPI_ISL_403932、EPI_ISL_408484、EPI_ISL_410984、 EPI_ISL_415462 和EPI_ISL_415652共五條序列在1到30145這一位點區間上進行差異比對的結果,最終以表格的形式將所有存在差異的位點一一列出。根據表格可以發現,上述五條序列在298、1 247、3 094、8 839、9 495、11 143、13 111、14 468、15 384、19 251、23 467、24 098、24 926、26 793、28 141、28 208、29 159這些位點上存在差異,即這些位點都是可能發生了變異的位點。

圖11 病毒序列差異比對Fig.11 Virus sequence difference alignment
2 系統的設計與實現
2.1 數據庫
系統使用了關系型數據庫MySQL,以每一條序列作為一個實體,將所有數據都整合到一張表中。即表中綜合了每個序列的基本信息(序列名、病毒名、采樣地點、采樣時間和提交時間等)和核苷酸排布信息(以字符串形式存儲,在數據庫中字段的類型為不限長度的text類型)。表的主要字段如圖12所示。

圖12 表格主要字段Fig.12 Main fields of the data table
2.2 服務端
系統服務器端使用Spring、SpringMVC配合MyBatis框架作為主要的技術進行實現[14],并將項目部署在阿里云ECS服務器上提供服務。
2.3 客戶端
系統的客戶端主要采用jquery、bootstrap、echarts框架[15]進行開發。jquery框架作為一個快速簡潔的輕量型框架,提供了完備的功能接口,支持豐富的插件便于擴展,同時能夠較好的支持服務端與客戶端得數據交互。bootstrap框架具有響應式布局設計,能讓網站兼容不同分辨率設備,給用戶較好的體驗。而echarts框架能夠將我們的數據以圖表可視化的方式進行呈現,有利于用戶對數據進行閱讀和分析。
3 數據處理與分析
3.1 病毒序列數據的處理
為了尋找發生變異的位點,我們首先將新型冠狀病毒序列進行了多重序列比對。本研究中使用的多序列對比工具是MUSCLE[16],軟件版本為3.8.31。比起其它常用的序列對比工具,如ClustalW[17]、T-coffee[18],MUSCLE在保證了較高的準確度的同時,具有更快的多序列比對速度。在參數選擇上,我們設置了參數-diags來適用于高度同源的新型冠狀病毒序列,同時最大迭代次數(-maxiters)選擇16來保證多序列對比的準確性,而其它參數選擇了默認設置。
多重序列比對之后,多個新型冠狀病毒序列間同源序列的相同核苷酸將處在相同的位點上,而如果原始序列在該位點中不存在核苷酸,將用字符“-”來表示。經過多重序列比對的序列都將擁有相同的序列長度。
對于對比后的序列,根據編碼區異常字符(N)的數量以及異常缺失位點數量,我們設置30個為病毒序列的篩選條件,異常位點大于篩選條件值的序列將被遺棄。
將篩選后的比對結果轉化為核苷酸矩陣,每條序列為行,每個位點為列,尋找每一列是否有超過一種核苷酸,如果結果為是,則該位點為有變異。
經如上方法對來自GISAID的數據進行預處理之后,即可將所有病毒序列轉換為等長的形式,在此基礎上即可按照2.1節所述內容進行數據的整合和數據庫表的設計與填充。
3.2 病毒變異的時空分析
隨著時間線的延長,各國提交的序列也逐漸增多,相應的各地區病毒序列的平均變異率也在逐漸增加,如圖9所示。這也符合包膜的陽性單鏈RNA病毒在傳播過程中容易變異的特點。我們也基于圖8所對應的功能模塊對截止到3月13日的不同國家或地區的平均變異率做了比較分析,找出了平均變異率較高和較低的部分國家以及國內部分省份,分別如表1和表2所示。可以看出,到目前為止,病毒序列的平均變異率處于一個較小的波動范圍內,病毒仍然處于較為穩定的狀態。

表1 病毒變異率較高和較低的部分國家Table 1 Countries with higher and lower virus variation rates

表2 病毒變異率較高和較低的中國部分省份Table 2 Provinces in China with higher and lower virus variation rates
結合在選擇位點之間的變異病毒株數分析結果(圖5所對應的功能模塊),大部分有變異發生的病毒序列的變異位點數量小于12個,這也印證了目前病毒變異率較小這一結果。
進一步比較不同地區不同時間的病毒變異率,發現隨著地區與時間的不同,病毒平均變異率也有差異,但總體仍處于在較小范圍內波動的狀態。
對于病毒序列變異分析,用戶可以選擇感興趣的起始位點和終止位點對序列進行分析,比如針對病毒的S蛋白編碼區,在本次多重序列對比中對應起始與終止位置分別為21 627和25 448,在系統中選擇后,可以看到變異數量的信息,其中變異位點數量(SNP)為0和1的病毒數量最多,對應病毒株數分別為302和194,其余SNP數分別為2、3和7,對應病毒株數分別為28、6和1,大部分病毒序列均未發生明顯改變。
4 總結
本系統對來源于全球共享流感數據倡議組織(GISAID)的新型冠狀病毒數據進行整合與分析,主要實現了以下功能:
1)對數據的來源進行可視化分析。
2)對序列的基本信息進行展示,并支持選定序列之間核苷酸比較。
3)以動態圖的形式呈現不同時間下各地區的病毒變異率,以及所有地區的累計變異率,使用戶能夠直觀感受病毒的時空變異情況。
4)對選擇位點之間的變異病毒株數進行統計,并以柱狀圖的形式對病毒的變異率進行時間空間上的展示,方便用戶對病毒的變異情況進行分析。
該系統以一種更為直觀的方式呈現病毒的具體信息,同時以圖表的方式,對病毒的變異情況進行了展示,方便用戶對新型冠狀病毒的序列進行分析,對新型冠狀病毒防治的研究具有重要的參考意義。