Hsa-miR-210-5p靶基因預測及其相關信號通路的生物信息學分析
蔡丹平,龍鼎新
(南華大學 船山學院, 湖南 衡陽 421001)
microRNAs(MiRNAs)是一類長約18-25個核苷酸的非編碼RNA,與mRNA轉錄本的3’非翻譯區(3’-UTR)結合,可以對靶基因的表達進行調控[1]。它們對各種重要的生物學過程具有調控作用,如細胞發育、細胞增殖、細胞分化及細胞凋亡等有關[2],因此準確預測miRNA的靶基因并對其靶基因進行系統的生物學分析是研究其作用機制的重要環節。本研究前期為了探討miRNAs在三鄰甲苯基磷酸酯(Tri-o-cresyl phosphate,TOCP)誘發神經細胞自噬中的作用機制,以人神經母細胞瘤細胞SK-N-SH細胞為體外細胞自噬模型,應用miRNA基因芯片、RT-PCR等實驗技術檢測了自噬相關miRNA表達情況。綜合芯片和熒光實時定量PCR的結果,篩選出了miR-210-5p顯著性差異表達的自噬基因。
Hsa-miR-210-5p屬于miR-210基因家族,定位于人11號染色體568 150-568 171。研究表明低氧誘導的miR-210參與細胞循環、細胞分化、DNA修復、細胞凋亡、膜轉運、氧化應激/糖酵解等調控信號通路,可作為非小細胞癌、乳腺癌、胰腺癌、肺細胞癌、食管鱗狀細胞癌等癌癥的診斷參考指標和預后指標[3],其過表達與晝夜節律過程、神經元發育、GTP酶信號轉導和光感受相關的途徑有關[4],但目前國內外對miR-210-5p的報道較少。本研究運用生物信息學分析,預測hsa-miR-210-5p的靶基因,繪制韋恩圖得靶基因集合,并對其靶基因集合進行蛋白質互作網絡分析、GO(Gene Ontology)分析和KEGG Pathway(Kyoto encyclopedia of genes and genomes)分析預測結果中的靶基因集合作用機制,并注釋其靶基因的生物學功能,為展開miR-210-5p的靶基因鑒定及生物學功能研究提供理論基礎。
1 材料與方法
1.1 miR-210-5p序列的保守性分析
使用miRbase[5](http://www.mirbase.org/)、RNAcentral(https://rnacentral.org/)數據庫在線查找各物種已知成熟miR-210-5p堿基序列,對比分析miR-210-5p序列在各物種間的保守性。
1.2 miR-210-5p靶基因的預測
利用miRDB[5](www.mirdb.org/),TargetSc-an[5](http://www.targetscan.org/)和DIANA TO-OLS[5](http://diana.imis.athena-innovation.gr/) 3個在線數據庫預測miR-210-5p的靶基因,用在線軟件Veney 2.1.0 (http//bioinfogp.cnb.csic.es/tools/Venny/index.html)畫韋恩圖,得到3個數據庫預測結果的交集,該交集為靶基因集合,以降低假陽性率。
1.3 預測靶基因集合編碼蛋白質之間的相互作用
將該靶基因集合使用String 11.0(Search Tool for the retrieval of interacting genes)在線數據庫(https://string-db.org/)分析,繪制互作網絡鄰接編碼蛋白數目柱狀圖,再利用Cytoscape_v3.6.1繪制靶基因集合編碼蛋白質之間相互作用(protein-protein interaction, PPI)網絡圖。
1.4 miR-210-5p靶基因的GO分析
使用DAVID(https: //david. ncifcrf. gov/)數據庫對預測到的miR-210-5p靶基因集合進行GO功能注釋,以人的所有基因為背景基因,顯著性閾值取P< 0.05,獲得相對hsa-miR-210-5p具有統計學意義的GO分析。GO分析由細胞組分(Cellular component)、分子功能(Molecular function)、生物調節(Biological process)3個部分組成。用R軟件繪制miR-210-5p靶基因交集的GO功能注釋圖。
1.5 miR-210-5p靶基因的KEGG Pathway分析
使用DAVID(https://david.ncifcrf.gov/)數據庫對預測到的miR-210-5p靶基因集合進行KEGG Pathway富集分析,以人的所有基因為背景基因,顯著性閾值取P< 0.05,獲得相對于hsa-miR-210-5p具有統計學意義的基因集合信號轉導通路。用R軟件繪制miR-210-5p靶基因交集的KEGG Pathway富集通路圖。
2 結果分析
2.1 miR-210-5p參與的疾病
miR-210-5p在骨質疏松[6]、骨關節炎[7]、自閉癥障礙[8],心血管疾病[9]中起上調作用(見表1)。

表1 miR-210-5p調控靶基因參與人類的部分疾病Table 1 miR-210-5p target genes involved in parts of human diseases
*注:該靶基因是小鼠模型試驗獲得,尚無人類試驗驗證。
Note:The target genes was were obtained by mouse model test and have not been verified by human test.
2.2 各物種成熟miR-210-5p序列的保守性分析
使用RNAcentral和miRBase數據庫進行對比分析,可知其基因序列號為MIMAT002475,定位于11號染色體568 150-568 171。對獼猴(mml)、小鼠(mmu)、褐家鼠(rno)等8個物種對比分析,可知hsa-miR-210-5p的成熟堿基序列“agccccugcccaccgcacacug”在各物種間高度保守(見表2)。

表2 各物種miR-210-5p的成熟堿基序列Table 2 Mature base sequences of miR-210-5p of various species
2.3 預測靶基因集合編碼蛋白質之間的相互作用使用數據庫miRDB,TargetScan和DIANA TOOLS預測miR-210-5p的靶基因,預測的個數分別為401、4 048和87,然后用在線軟件Veney 2.1.0可得三個數據庫預測靶基因的交集23個,占各預測軟件預測靶基因總和的0.6%(見圖1)。
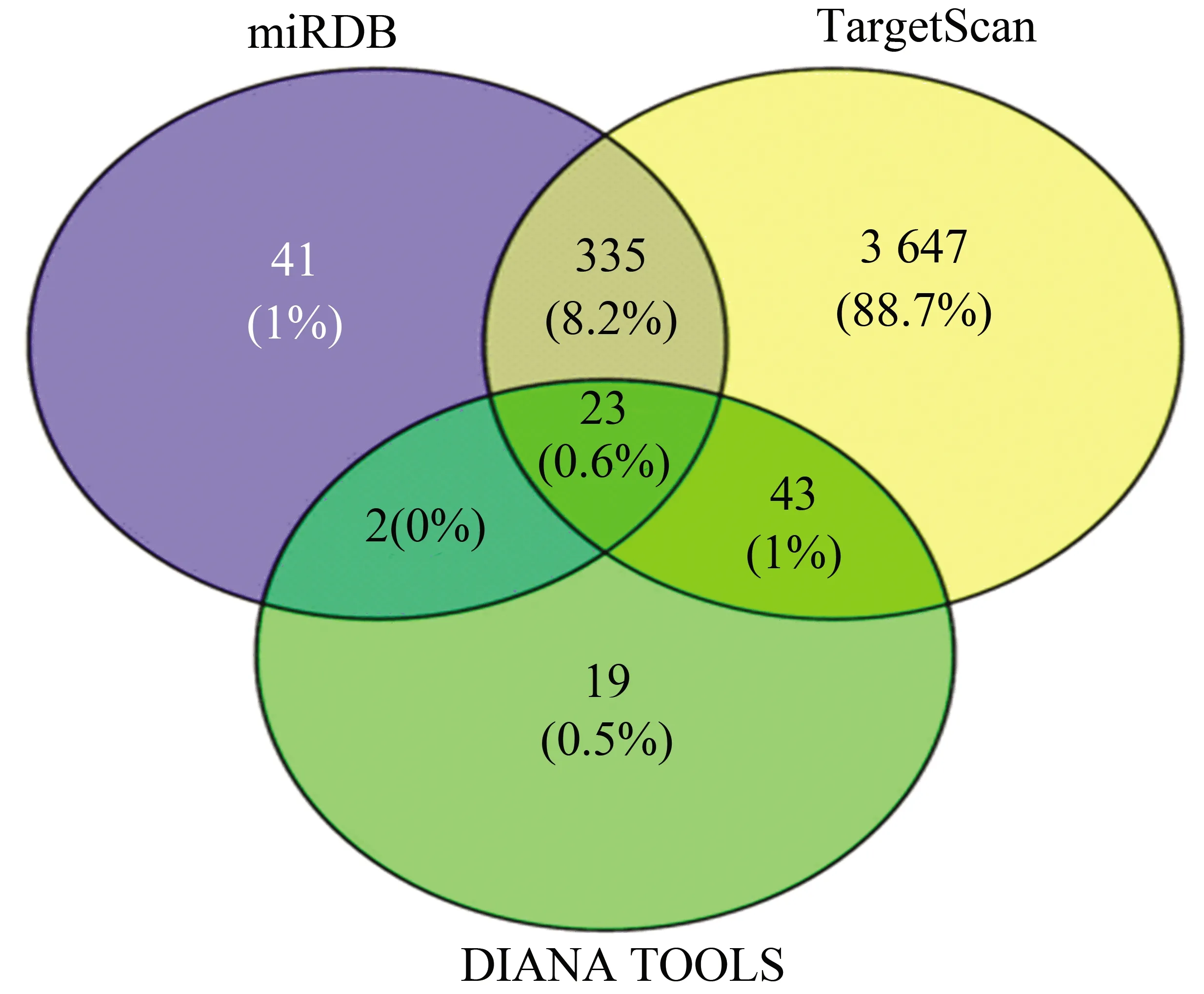
圖1 miR-210-5p的預測靶基因個數Fig.1 Prediction of the number of target genes for miR-210-5p
2.4 預測靶基因集合編碼蛋白質之間的相互作用
將三個數據庫得到的靶基因集合使用String 11.0數據庫分析,繪制出互作網絡鄰接編碼蛋白數目柱狀圖(見圖2)。結果顯示,靶基因CDK8、MED18、MED13、MED12、CCNC、MED14、MED23、CDK19、MED1、MED15和MED13L的編碼蛋白質所占比重較大。運用Cytoscape_ v 3.6.1軟件,繪制出PPI網絡圖(見圖3)。結果表明,miR-210-5p的靶基因集合編碼蛋白質之間的相互作用關系較復雜,有10個編碼蛋白質之間的互作關系較穩定。
2.5 miR-210-5p靶基因的GO功能注釋
使用DAVID數據庫對預測到的miR-210-5p靶基因集合進行GO功能注釋,以人的所有基因為背景基因(P< 0.05),可得其主要富集的細胞組分是細胞質、細胞漿和突觸前活動區;分子功能是參與鳥苷酸合成;參與突觸小泡胞吐的調控、鳥苷酸蛋白介導的信號轉導、鈣離子調節的神經遞質胞吐和膜電位調節等生物學過程(見表3),用R軟件繪制miR-210-5p靶基因交集的GO功能注釋圖(見圖4)。
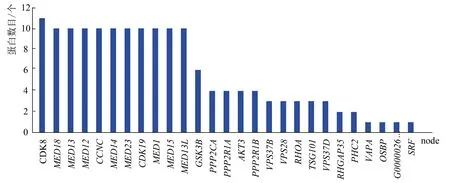
圖2 互作網絡鄰接編碼蛋白數目柱狀圖Fig.2 Adjacency coding protein number histogram of interaction networks
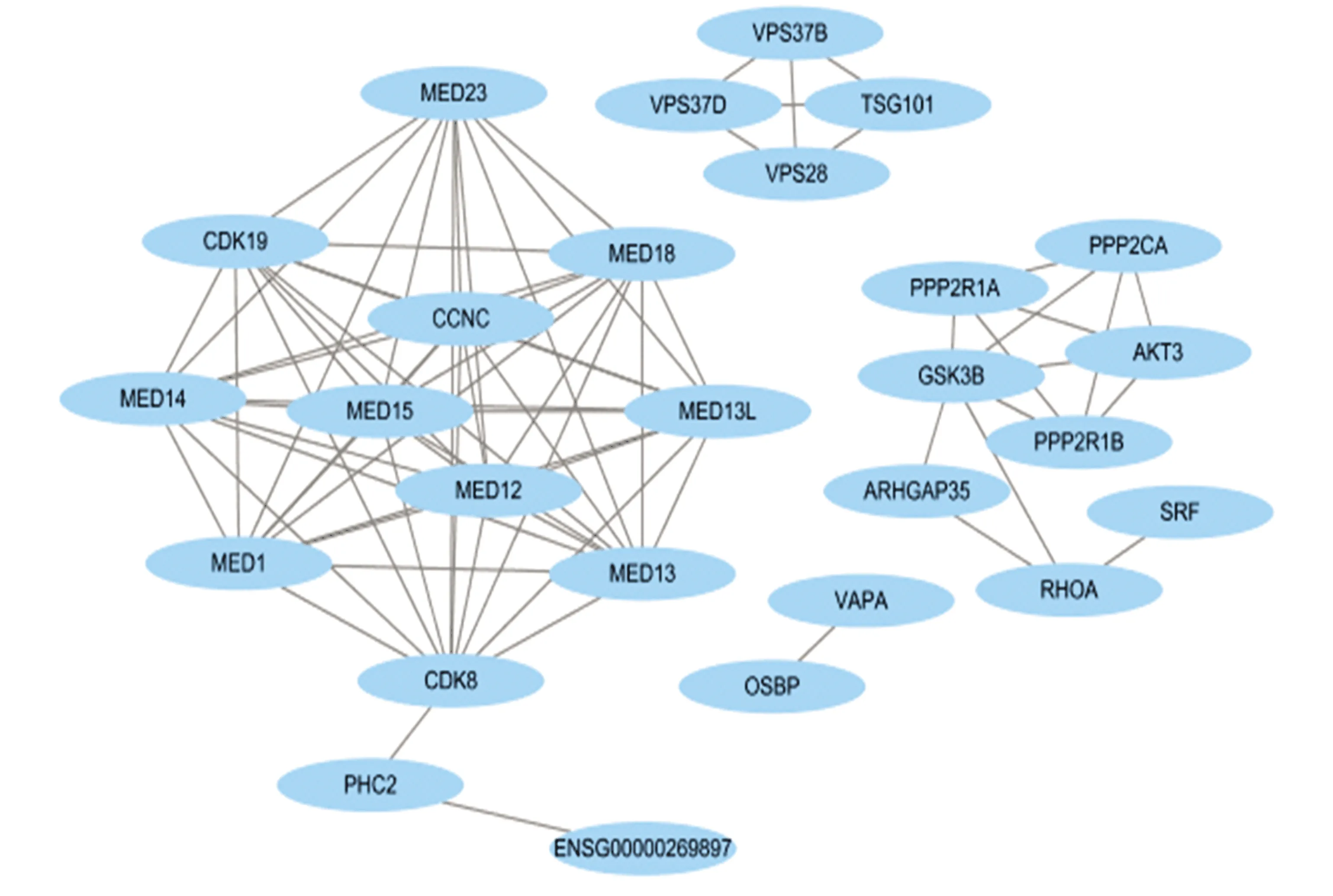
圖3 miR-210-5p預測靶基因集合所編碼蛋白質之間的相互作用Fig.3 Protein-protein interaction network of the target genes of miR-210-5p
表3 miR-210-5p靶基因交集的GO功能注釋
Table 3 GO functional annotation for intersection of miR-210-5p target genes
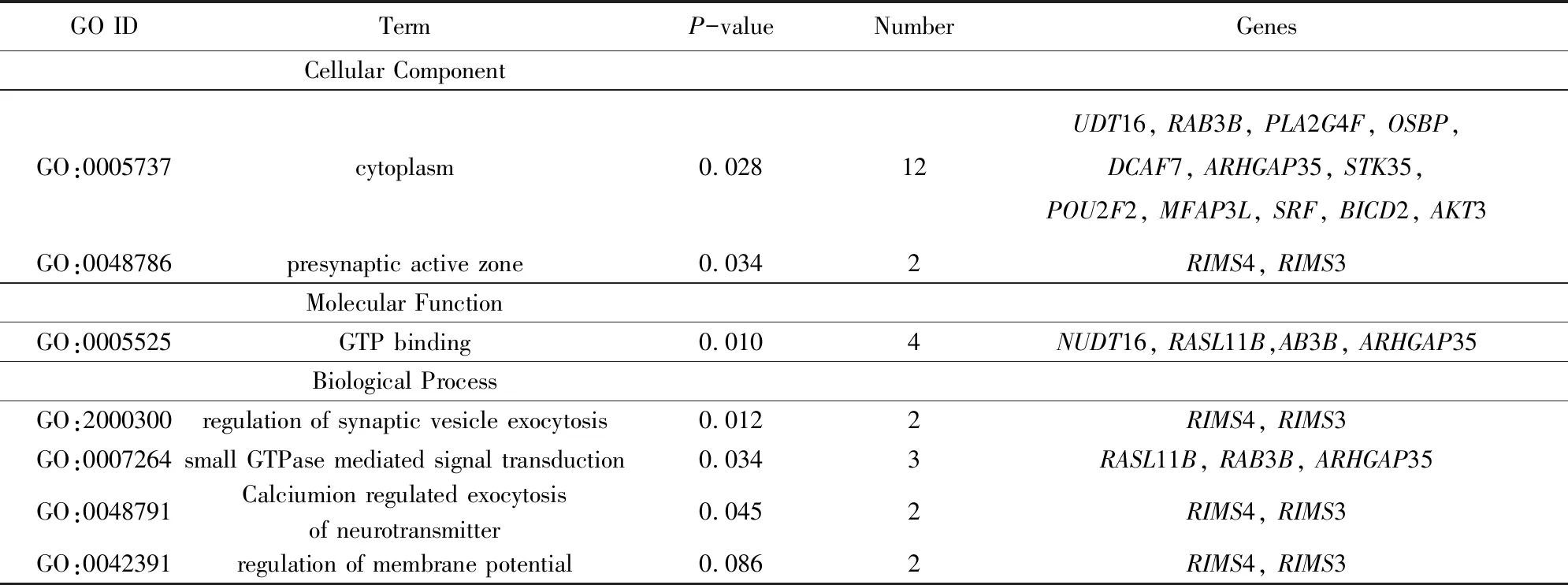
GO IDTermP-valueNumberGenesCellular ComponentGO:0005737cytoplasm0.02812UDT16, RAB3B, PLA2G4F, OSBP, DCAF7, ARHGAP35, STK35, POU2F2, MFAP3L, SRF, BICD2, AKT3GO:0048786presynaptic active zone0.0342RIMS4, RIMS3Molecular FunctionGO:0005525GTP binding0.0104NUDT16, RASL11B,AB3B, ARHGAP35Biological ProcessGO:2000300regulation of synaptic vesicle exocytosis0.0122RIMS4, RIMS3GO:0007264small GTPase mediated signal transduction0.0343RASL11B, RAB3B, ARHGAP35GO:0048791Calciumion regulated exocytosisof neurotransmitter0.0452RIMS4, RIMS3GO:0042391regulation of membrane potential0.0862RIMS4, RIMS3
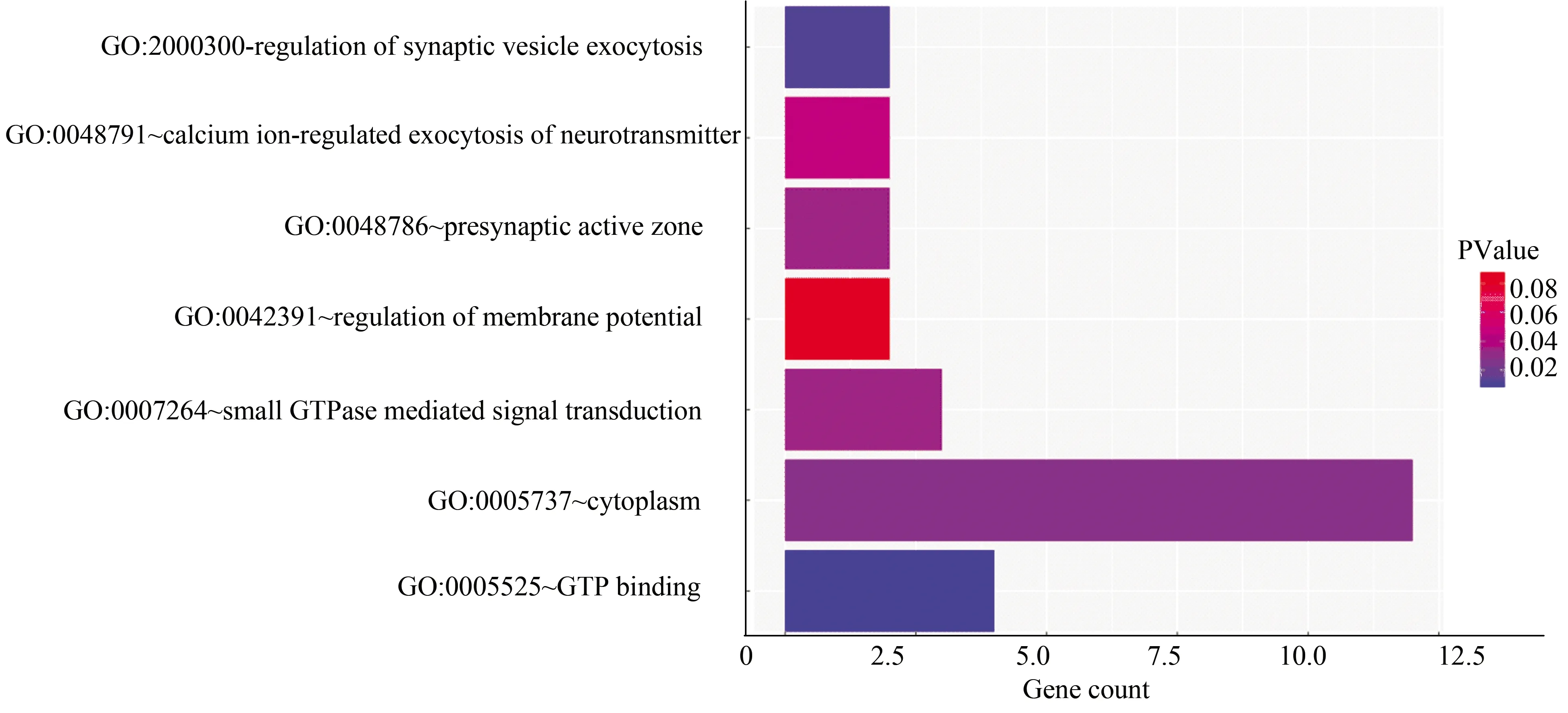
圖4 miR-210-5p靶基因交集GO功能注釋Fig.4 GO functional annotation for intersection of miR-210-5p target gene
2.6 miR-210-5p靶基因的KEGG Pathway分析
使用DAVID數據庫對預測到的miR-210-5p靶基因集合進行KEGG Pathway分析,以人的所有基因為背景基因(P< 0.05),可得miR-210-5p的靶基因集合參與血小板活化、MAPK信號通路、VEGF信號通路、癌癥通路、甲狀腺激素信號通路等(見表4),用R軟件繪制miR-210-5p靶基因交集的KEGG pathway通路圖(見圖5)。
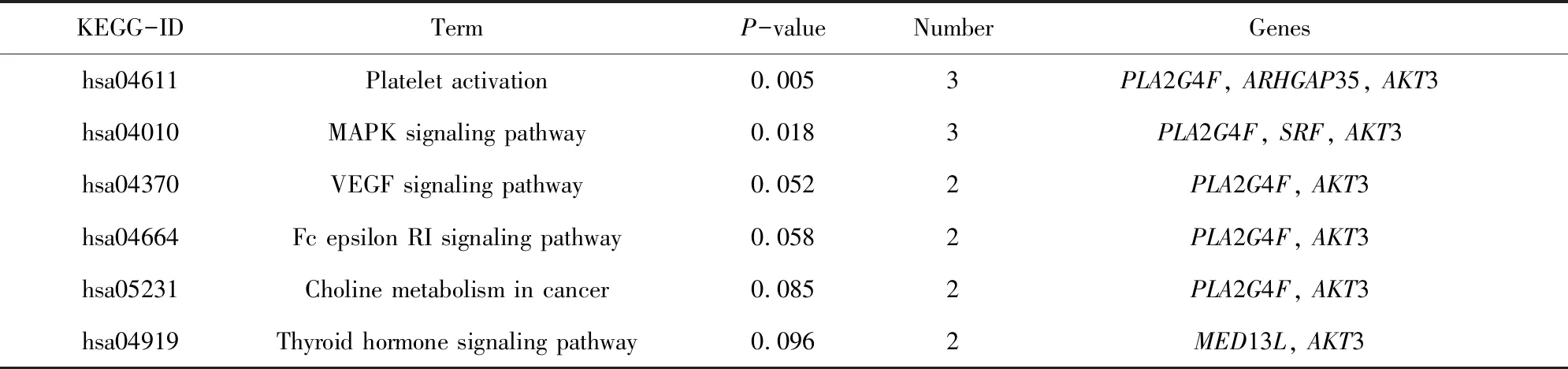
表4 miR-210-5p靶基因交集的KEGG Pathway分析Table 4 KEGG pathway analysis for intersection of miR-210-5p target genes
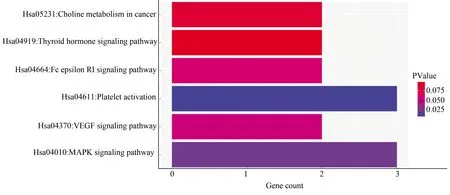
圖5 miR-210-5p靶基因交集的KEGG Pathway分析Fig.5 KEGG pathway analysis for intersection of miR-210-5p target genes
3 討 論
miR-210在腫瘤、心血管系統,神經系統疾病中起發揮重要作用。miR-210能上調Kaposi肉瘤相關的皰疹病毒和感染[11]、慢性鼻-鼻竇炎鼻息肉患者黏膜黏蛋白0型聚糖生物合成途徑[12]、人骨肉瘤[13],小鼠感覺軸突再生[14]和脊髓再生[15],可通過在動脈粥樣硬化的情況下直接靶向PDK1誘導內皮細胞凋亡有下調作用[16],能抑制滋養層細胞侵襲,是先兆子癇的血清生物標志物[17]。hsa-miR-210-5p屬于miR-210基因家族,miRNA-210-5p在神經功能調節中報道較少。
考慮到靶基因預測過程中miRNA與靶基因結合位點的序列匹配、mRNA與miRNA雙鏈特異結合的熱穩定性及序列的保守性等因素,采用miRDB、TargetScan,DIANA TOOLS三個數據庫對miR-210-5p靶基因進行預測,得到了可信度較高的靶基因集合,進行編碼蛋白質互作分析,GO分析和KEGG pathway分析。研究發現miR-210-5p能促進成骨細胞的分化[6]、成骨細胞胞外體的表達[7]、調節鈣通道[8]、促進細胞缺氧凋亡[9]、能結合位點突變,降低熒光酶活性[10]、參與細胞的免疫反應[18]、阻斷胚狀體細胞擴增[19]、對孕婦發生先兆子癇[20]、腎細胞癌[21],在副溶血性孤菌感染的文昌魚的免疫反應中起著重要調節作用[22]。提示miR-210-5p可能調控癌癥的發生與轉歸、鈣離子信號通路、血小板活化,免疫系統和神經系統等生物學過程。miR-210-5p靶向TNS1和STC1調節缺氧時心肌細胞發生的變化,進而調控心血管疾病[9]。在TargetScan Human 7.2可查到基因TNS1與miR-210-5p種子區域的配對類型為7mer-m8、7mer-A1和8mer,基因STC1與miR-210-5p種子區域的配對類型為7mer-m8,并且它們與miR-210-5p非種子區域3'端存在不同長度的互補位型(見表5)。miR-210-5p種子區域與TNS1 和STC1基因的3’UTR完全匹配可以增強miR-210-5p對TNS1和STC1基因的沉默效果,故可用miR-210-5p與TNS1和STC1基因的高特異性來研究miR-210-5p對TNS1和STC1基因富集的疾病通路如心血管疾病的治療是具有發展意義的。
表5TNS1 和STC1基因3'UTR結合miR-210-5p的位點
Table 5 3’UTR sequences ofTNS1 andSTC1 targeted by miR-210-5p

4 結 論
選用三種數據庫預測miR-210-5p靶基因,取其交集分析,不能避免預測結果的假陽性,需通過實驗驗證該靶基因集合里23個靶基因為miR-210-5p的靶基因,進一步探討目標靶基因3’UTR結合miR-210-5p的位點及其具體分子機制來降低假陽性率。運用生物信息學分析方法,較全面的分析了hsa-miR-210-5p所參與的生物學過程,為后續研究提供了方向。