棉花CRVW的克隆與抗黃萎病功能分析
王秋瑩,王偉巧,張艷,王國寧,吳立強,張桂寅,馬峙英,楊君,王省芬
?
棉花的克隆與抗黃萎病功能分析
王秋瑩,王偉巧,張艷,王國寧,吳立強,張桂寅,馬峙英,楊君,王省芬
(河北農業大學農學院/教育部華北作物種質資源研究與利用重點實驗室,河北保定 071001)
【】黃萎病(Verticillium wilt)是棉花生產上的重要病害,嚴重影響棉花的產量和品質。棉花基因組測序工作的完成為抗病基因挖掘提供了重要的信息資源。通過對一個尚未有功能注釋的陸地棉基因(cotton resistance to Verticillium wilt)進行克隆與抗病功能驗證,為棉花基因組信息完善、抗病機制解析和分子育種等方面奠定基礎。根據參考基因組序列設計引物,同源克隆陸地棉()農大601(ND601)中的開放讀碼框(open reading frame,ORF)。利用在線工具ProtParam預測蛋白氨基酸組成、分子量、理論等電點、不穩定指數和總平均親水性等性質;應用PSIPRED v3.3預測蛋白二級結構;在線工具ProtComp v. 9.0進行亞細胞定位預測;PlantCARE在線軟件分析順式作用元件。構建與綠色熒光蛋白基因融合表達載體,通過基因槍介導法轉化洋蔥表皮細胞,觀察的表達位置。利用qRT-PCR檢測在棉花不同組織、黃萎病菌脅迫條件下不同抗、感品種間,以及水楊酸(salicylic acid,SA)誘導處理條件下的表達模式。構建沉默載體,應用病毒誘導的基因沉默(virus-induced gene silencing,VIGS)技術進一步驗證該基因在棉花中的抗病功能。檢測沉默后一些與植物抗病調控相關標志基因的表達變化,分析其介導的抗病通路。從陸地棉品種ND601中克隆到的ORF,其全長780 bp,編碼259個氨基酸殘基,分子量約為30.2 kD,理論等電點9.59;蛋白二級結構含69.50%不規則卷曲、17.76% α-螺旋、11.20%延伸鏈和1.54% β-卷曲。綜合生物信息學預測和熒光觀察結果,顯示CRVW主要存在于植物細胞膜和細胞質。在棉花根、莖和葉中都有表達,但在根中的表達量最高。的ORF上游序列()中包括響應乙烯(ethylene)、SA、生長素(auxin)和脫落酸(abscisic acid)等4種激素信號的順式作用元件。另外,還包括一些與傷害、防御、脅迫、病菌、干旱和低溫等相關的順式作用元件。SA噴灑處理后,顯著上調表達。黃萎病菌脅迫后,在抗病品種ND601和感病品種中棉所8號(CCRI8)中均顯著上調表達,但在感病品種中上調表達的發生時間明顯滯后。黃萎病菌處理20 d后,沉默組棉苗表現出比對照(CK)組更明顯的黃化、萎蔫和落葉等黃萎病病癥。進一步統計分析顯示,沉默組病指顯著高于CK組,表明沉默顯著降低了棉苗對黃萎病菌的抗性。沉默后,棉苗中SA含量顯著降低;(isochorismate synthase 1)、(enhanced disease susceptibility 1)、(phytoalexin deficient 4)、(nonexpresser of PR gene 1)和(pathogenesis-related protein 1)等與SA積累和信號調控相關的標志基因均發生顯著下調表達。CRVW定位于細胞質和細胞膜,主要在棉花根部表達,可能通過SA信號通道參與棉花抗黃萎病反應過程。
棉花;黃萎病;CRVW;克隆;病毒誘導的基因沉默;抗性
0 引言
【研究意義】棉花是重要的纖維作物和油料作物,也是關系國計民生的重要戰略物資。在生產上,黃萎病(Verticillium wilt)對棉花產量和品質危害嚴重,藥劑難于防治。實踐表明,培育和種植抗病品種是防治該病的有效途徑[1]。因此,篩選和鑒定抗病基因,不僅有利于棉花抗黃萎病機制的解析,還能為棉花分子育種提供候選基因。【前人研究進展】黃萎病是由大麗輪枝菌()引起的土傳真菌維管束病害[2]。組織結構抗性和生理生化抗性被認為在棉花抗黃萎病過程中發揮重要作用[3-5]。隨著分子生物學的發展,越來越多的抗性基因獲得鑒定,使得在分子水平解析棉花黃萎病抗性機制有了長足的發展。在組織結構抗性方面,棉花主要通過提高木質素積累和改變細胞壁組成增強對黃萎病菌的抗性,其中一些重要參與基因包括:與果膠代謝相關的(pectin methylesterase inhibitor)[6]、與木質素合成相關的(laccase)[7]和[8]、與細胞壁組成相關的(hybrid proline-rich protein)[9]等。在生理生化抗性方面,棉花能夠通過(polyamine oxidase)調控對黃萎病菌具有抑制活性的精胺(spermine)和植保素(camalexin)的積累[10],依賴(enhanced disease susceptibility 1)調控水楊酸(salicylic acid,SA)信號通路、引發活性氧爆發(reactive oxygen species,ROS)和酚類物質積累等[11]。由南京農業大學等單位完成的陸地棉()TM-1基因組scaffold總長度為2.4 Gb,包含70 478個編碼蛋白基因,通過與其他植物基因組進行比對,其中66 434個基因獲得了注釋[12]。相對于其他已測序植物,棉花基因組較大,基因多倍化明顯,并且生長周期長,以至于通過棉花遺傳轉化大規模分析基因功能明顯滯后于擬南芥、煙草、水稻等植物[12]。病毒誘導的基因沉默(virus-induced gene silencing,VIGS)技術避免植物轉化,能夠在侵染植物當代對目標基因進行功能分析[13]。因此,VIGS技術一經建立,即成為植物基因功能分析的強有力工具,得到了廣泛的應用[14]。目前,該技術已經成功應用于棉花生長發育、抗病反應、代謝調控等功能基因研究[15-17]。棉花基因組研究已進入功能基因組時代,快速鑒定目標基因功能已成為未來重要的研究內容之一。【本研究切入點】河北農業大學棉花品種創新與產業化團隊前期利用RNA-Seq技術構建了黃萎病菌侵染處理的海島棉()和陸地棉表達譜[11, 18],發現其中一個無功能注釋的基因表達受黃萎病菌顯著誘導,可能參與棉花抗黃萎病菌反應過程,故將其命名為(cotton resistance to Verticillium Wilt)。【擬解決的關鍵問題】本研究通過對進行克隆、生物信息學分析、亞細胞定位、組織表達特異性分析、SA誘導和黃萎病菌脅迫處理后的表達模式分析、以及抗病功能鑒定等方面的研究,為進一步揭示其抗病機制奠定基礎。
1 材料與方法
試驗于2017—2019年在河北農業大學教育部華北作物種質資源研究與利用重點實驗室進行。
1.1 試驗材料
陸地棉抗病品種農大601(ND601)和感病品種中棉所8號(CCRI8)由河北農業大學棉花品種創新與產業化團隊提供,種植于光照周期16 h(光照)/8 h(黑暗),溫度25℃,相對濕度60%—70%的植物生長室中。
試驗所用黃萎病菌為臨西2-1,由河北農業大學棉花品種創新與產業化團隊分離鑒定并繼代保存于PDA(potato dextrose agar)培養基[19]。
植物亞細胞定位載體pCamE由河北農業大學棉花品種創新與產業化團隊構建并保存[20]。棉花VIGS載體pTRV1和pTRV2由清華大學劉玉樂教授饋贈[13]。沉默對照載體pTRV2-CLA1(cloroplastos alterados 1 gene)和農桿菌GV3101由河北農業大學棉花品種創新與產業化團隊保存。
EASYspin Plus植物RNA提取試劑盒購自北京艾德萊生物科技有限公司;EasyScript?First-Strand cDNA Synthesis SuperMix購自北京北京全式金生物技術有限公司;2×PhantaTMMaster Mix購自南京諾唯贊生物科技有限公司;pGM-T載體、T4 DNA連接酶、2×Taq PCR Master Mix、大腸桿菌DH5α感受態細胞、瓊脂糖凝膠DNA回收試劑盒、質粒小提試劑盒購自天根生化科技有限公司;DNA Marker、PrimeScriptTMRT reagent Kit with gDNA Eraser和EraserSYBR?TM試劑盒購自寶生物工程有限公司。
引物合成和測序工作由生工生物工程股份有限公司完成。
1.2 基因克隆
按照ZHANG等[18]的方法將ND601培養于MS培養基(murashige and skoog medium)上。取約14日齡的棉苗,按照EASYspin Plus植物RNA提取試劑盒說明書進行總RNA提取。依據EasyScript?First- Strand cDNA Synthesis SuperMix試劑盒說明書合成cDNA。根據已測序陸地棉TM-1編號為Gh_A06G1566的基因上下游序列設計擴增引物CRVW-F(5- GATTCTTTGATTTTCAAGAGGTG-3)和CRVW-R(5-CGATTAAAAGTAACAAATTATCGT-3)。目的基因PCR擴增體系(20 μL)為1 μL cDNA、1 μL CRVW-F、1 μLCRVW-R、10 μL 2×PhantaTMMaster Mix和7 μL去離子水。擴增程序為94℃ 5 min;94℃ 30 s,56℃ 30 s,72℃ 1 min,35個循環;72℃ 10 min。1%瓊脂糖凝膠電泳檢測后,按照瓊脂糖凝膠DNA回收試劑盒操作說明對目的片段進行回收,與pGM-T載體過夜連接(16℃)后,通過熱激法轉化大腸桿菌DH5α感受態細胞,菌落PCR篩選陽性克隆并送測序。
1.3 生物信息學分析
基因序列處理軟件為BioEdit 7.0.4(http://www. mbio.ncsu.edu/bioedit/bioedit.html);陸地棉TM-1基因組序列信息和RNA-seq數據來源于CottonFGD網站(https://cottonfgd.org/);使用在線工具ProtParam(http://web.expasy.org/protparam)預測蛋白氨基酸組成、分子量、理論等電點、不穩定指數和總平均親水性等性質;應用PSIPRED v3.3(www.psipred.net)預測蛋白二級結構;應用ProtComp v. 9.0(http://www. softberry.com/berry.phtml?topic=protcomppl&group=programs&subgroup=proloc)預測亞細胞定位;應用PlantCARE在線軟件(http://bioinformatics.psb.ugent. be/webtools/plantcare/html/)預測分析順式作用元件。
1.4 亞細胞定位載體構建與熒光觀察
利用引物CRVW-SF(5-ATGCAACAA ATCAAAGATTCATGG-3,下劃線處為Ⅰ酶切位點)和CRVW-SR(5-TTTCCACCCTAAA ACCTTAAGC-3,下劃線處為Ⅰ酶切位點)擴增的ORF(去除終止密碼子),并插入植物表達載體pCamE多克隆位點,由CaMV 35S啟動子驅動,與編碼綠色熒光蛋白(green fluorescent protein,gfp)基因融合表達。洋蔥上皮細胞的制備、基因槍轉化和熒光觀察等操作參照YANG等[21]方法。
1.5 熒光定量PCR分析
按照EASYspin Plus植物RNA提取試劑盒說明書對棉苗組織進行總RNA提取。根據PrimeScript? RT reagent Kit with gDNA Eraser試劑盒所提供方法合成cDNA。以棉花(catalytic subunit of protein phosphatase 2A)作為內參(PP2A1-F:5-GATCCTTG TGGAGGAGTGGA-3;PP2A1-R:5-GCGAAACAGTT CGACGAGAT-3)[22]。表達檢測引物設計為qCRVW-F(5-TCCCGCTCCTCCTTCGATTT-3)和qCRVW-R(5-TGCATTTCAGCCTCTGGGATT-3)。棉花SA合成和信號通路標志基因檢測引物按照SUN等[23]研究報道進行合成。熒光定量PCR(real-time quantitative polymerase chain reaction,qRT-PCR)反應體系為cDNA模版1.0mL、正反向引物各0.8mL(10 μmol·L-1)、2×SYBR mix 10 μL和7.4 μL滅菌雙蒸水。使用Bio-Rad CFX96 Real-Time PCR Detection System進行熒光定量檢測,具體程序為95℃ 15 s;95℃ 10 s,58℃ 10 s,72℃ 15 s,40個循環。采用2-ΔCt法進行相對定量分析。3次生物學重復檢測。
1.6 SA誘導處理
參照YANG等[9]的方法,配制100 μmol·L-1的SA溶液,用噴霧器均勻噴灑于7日齡棉苗子葉,并以透明罩覆蓋保濕。分別于處理后6、12、24和36 h后取子葉,-80℃保存備用。以噴灑蒸餾水組作為對照。每個時間點取3株棉苗混樣,共重復3次。
1.7 黃萎病菌脅迫處理
按照YANG等[9]的方法制備濃度為1×107cfu/mL的黃萎病菌孢子懸浮液。采用“六棱塑料缽定量 接種法”對棉苗進行接菌處理[24],接種菌液體積為缽體的1/10。按5級標準統計病情,并計算病情指數[9]。
1.8 棉花VIGS
設計引物CRVW-VF(5-TTCCCGCTCC TCCTTCGATT-3,下劃線處為RⅠ酶切位點)和CRVW-VR(5-AACCTCCACAAACCCTTG CC-3,下劃線處為Ⅰ酶切位點)。通過PCR擴增400 bp的片段并插入pTRV2多克隆位點RⅠ和Ⅰ間。利用凍融法將構建好的載體轉入農桿菌GV3101[25]。農桿菌介導的棉花VIGS操作按照GAO等[26]方法進行。
1.9 SA含量測定
通過對約15日齡的棉苗進行接菌處理,在0、12和24 hpi(hours post inoculation)時,收集3棵長勢一致的棉苗第一片和第二片真葉進行混樣,作為一個生物學重復,每個處理共采集3個生物學重復。采集的組織于液氮中研磨,按照Verberne等[27]方法提取SA,應用Agilent HPLC 1200 Series檢測SA(流動相為甲醇﹕水﹕乙酸=80﹕19﹕1,流速為1 mL·min-1,上樣量為20 μL,檢測波長302 nm)。根據標準曲線計算SA含量。
1.10 數據分析
應用Graphpad Prism?6.02軟件進行統計分析并作圖。基因在不同時間和不同樣本內的相對表達量進行兩因素方差分析(Two-way ANOVA)和Sidak-test(<0.05)檢測。
2 結果
2.1 CRVW克隆與生物信息學分析
提取陸地棉ND601總RNA(圖1),反轉錄后的cDNA作為模板,利用引物CRVW-F和CRVW-R進行PCR擴增,產物經電泳檢測顯示約為800 bp(圖1)。PCR產物經膠回收后與T載體連接,轉化大腸桿菌并提取質粒。測序后,目的基因ORF全長780 bp,與參考基因組TM-1編號為Gh_A06G1566的序列相似性100%,編碼蛋白含259個氨基酸殘基,尚無法預測明顯的功能域,為putative uncharacterizedprotein。后續研究表明該基因/蛋白與棉花黃萎病抗性相關,故命名為CRVW(cotton resistance to Verticillium wilt)。

圖1 棉苗RNA提取與CRVW的PCR擴增
經預測,CRVW分子量約為30.2 kD,理論等電點9.59,分子式C1332H2061N383O395S14;在組成蛋白的20種氨基酸中,絲氨酸(Ser)所占比例最高(12.0%),而組氨酸(His)所占的比例最低(0.8%);含32個負電荷氨基酸殘基,42個正電荷氨基酸殘基;不穩定指數為61.41,屬于不穩定蛋白;總平均親水性-0.975,屬于疏水蛋白。二級結構預測顯示CRVW含有69.50%不規則卷曲、17.76% α螺旋、11.20%延伸鏈和1.54% β-折疊。
2.2 CRVW亞細胞定位
ProtComp v. 9.0在線工具預測顯示,CRVW最有可能位于細胞膜(基于Neural Nets算法)和細胞質(基于Pentamers算法)。為進一步確定CRVW在細胞中的位置,將其與GFP蛋白進行融合表達。熒光顯微觀察顯示,單獨表達GFP時,熒光信號存在于在細胞核、細胞質和細胞膜中;而對于CRVW與GFP融合表達的細胞,熒光主要出現在細胞膜和細胞質(圖2)。綜合生物信息學預測和熒光顯微觀察結果,表明CRVW主要存在于植物細胞膜和細胞質。
2.3 CRVW的組織表達特異性
已有RNA-seq數據顯示,在棉花根、莖和葉中均有表達,且葉中最高。為進一步明確的組織表達情況,利用qPCR技術對該基因在陸地棉ND601根、莖和葉中的表達進行了檢測。結果顯示,在以上3種組織中均有表達,但在根中的表達量最高,大約是莖中的10倍和葉中的4倍(圖3)。RNA-seq和qPCR檢測結果均確認在棉花根、莖和葉中都有表達,但存在組織表達差異。
2.4 CRVW受SA誘導表達
從已測序棉花TM-1基因組調取ORF上游2 000 bp序列(染色體A06:100117602—100119601)作為潛在的啟動子,命名為。預測分析顯示,含有響應4種激素信號的順式作用元件,包括乙烯(ethylene,ET)、SA、生長素(auxin)和脫落酸(abscisic acid,ABA)。另外,還包括一些與傷害、防御、脅迫、病菌、干旱和低溫等相關的作用元件(表1)。根據TM-1上游序列設計引物,對抗病品種ND601和感病品種CCRI8中上游序列進行同源克隆,并未發現抗感品種在所含順式作用元件的位置和數量上存在差異(結果未顯示)。

圖2 CRVW在洋蔥表皮細胞中的亞細胞定位
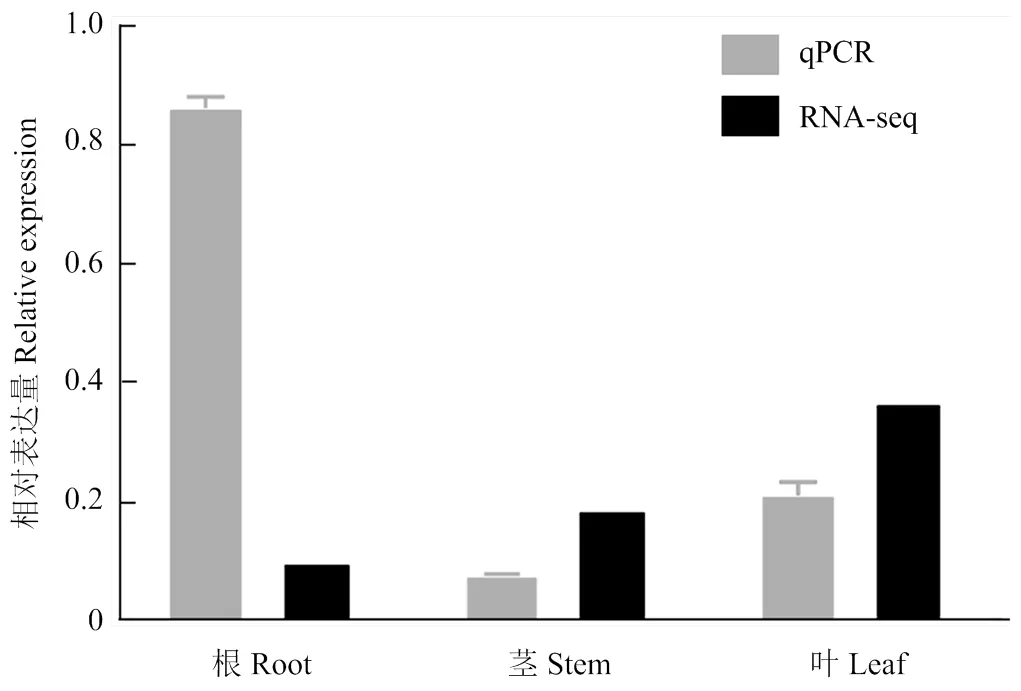
圖3 棉花不同組織中CRVW的表達分析
進一步對棉苗進行SA誘導處理,結果顯示,與對照相比,在所有4個時間點的表達量均發生顯著上調(圖4),表明受SA信號通路調控。

CK:對照組;SA:水楊酸處理組;***表示差異在P<0.001水平上具有顯著性。下同

表1 CRVW-P中的順式作用元件預測
2.5 CRVW受黃萎病菌誘導顯著上調表達
以抗病品種ND601和感病品種CCRI8根組織為分析對象,利用qPCR檢測在黃萎病菌脅迫下的表達模式。黃萎病菌脅迫后,在抗病品種中顯著上調表達(與同時間水處理對照相比)(圖5)。而對于感病品種CCRI8,在黃萎病菌脅迫后的6和12 hpi,的表達水平未發生顯著變化,直到24 hpi其表達才發生顯著上調(圖6)。由此可見,黃萎病菌脅迫處理后,不管是抗病品種還是感病品種,均發生顯著上調表達,但其在抗病品種中發生響應的時間早于感病品種。

VD:大麗輪枝菌處理組;CK:對照組;*表示差異在P<0.05水平上具有顯著性;**表示差異在P<0.01水平上具有顯著性。下同

VD:大麗輪枝菌處理組;CK:對照組
2.6 沉默CRVW顯著降低棉花黃萎病抗性
對尚未長出真葉的棉苗進行VIGS處理,以沉默(cloroplastos alterados 1)作為技術對照。在VIGS操作7 d后,發生沉默并表現為新生真葉白化(圖7-A),表明技術體系成功建立。同時對沉默情況通過qRT-PCR確認,并進行黃萎病菌脅迫處理。在接菌處理20 d后,野生(wild type,WT)組(既無VIGS處理也未接菌)生長正常;CK組(VIGS處理中注射pTRV2空載體,同時接菌處理)表現為葉片黃化和植株萎蔫;沉默組(注射攜帶序列片段的pTRV2載體,同時接菌處理)表現出比CK組更明顯的黃化、萎蔫和落葉等黃萎病病癥(圖7-B)。病指統計分析顯示,沉默組病指顯著高于CK組(圖7-C),進一步表明沉默顯著降低了棉苗對黃萎病菌的抗性。
2.7 沉默CRVW導致SA合成和信號通路相關基因表達顯著降低
在黃萎病菌侵染后的12和24 hpi,對照棉苗中SA含量顯著增加(與0 hpi對照相比)。而對于被沉默的棉苗,其體內SA含量僅在12 hpi顯著增加(與0 hpi沉默棉苗相比)。與對照相比,在0、12和24 hpi 3個時間點,沉默棉苗中SA含量均出現顯著降低(圖8),約為對照的1/2。為進一步確認是否參與SA信號通路,對與SA合成和信號通路相關的5個標志基因進行表達檢測。結果顯示,(isochorismate synthase 1)、(enhanced disease susceptibility 1)(phytoalexin deficient 4)(nonexpresser of PR gene 1)和(pathogenesis-related protein 1)在沉默組中的表達均顯著降低,特別是在12 hpi,這些基因在沉默組中的表達比對照降低得最為明顯(圖9)。以上結果表明的沉默能夠顯著影響SA的積累,降低SA信號通路相關基因的表達。
3 討論
3.1 CRVW是潛在重要的棉花抗黃萎病基因
高通量轉錄組測序(RNA-seq)技術為棉花抗病基因挖掘提供了新的平臺。應用該技術,大量潛在的與黃萎病菌脅迫相關的棉花基因被發現[11,18,28]。棉花基因組測序的完成更是為克隆這些基因提供了極大地便利[12],使得鑒定這些基因功能成為科研工作者重要的研究內容。最初,河北農業大學棉花品種創新與產業化團隊從棉花受黃萎病菌脅迫處理后的RNA-aeq數據中發現,為一個差異表達基因,但無功能注釋[11,18]。本研究通過qRT-PCR對該基因的表達進行了分析,確認受黃萎病菌誘導后顯著上調表達,且其在抗病品種中表達變化早于感病品種(圖5和圖6),表明參與棉花和黃萎病菌間的互作。通過VIGS技術對進行沉默,發現棉花對黃萎病的抗性顯著降低(圖7),進一步證明參與棉花對黃萎病菌的抗病過程。本研究雖然成功克隆了(圖1),但其編碼蛋白無明顯的功能域。因此,目前還無法通過生物信息學對其進行注釋。亞細胞定位和組織表達特異性分析明確了CRVW在棉花組織中的分布(圖2和圖3),為進一步注釋其生理生化功能提供了依據。

圖8 黃萎病菌侵染后棉苗體內水楊酸的積累

圖9 黃萎病菌脅迫后SA合成和信號通路相關基因在對照和CRVW沉默棉苗中的表達分析
3.2 CRVW通過SA信號通路參與棉花對黃萎病菌的抗性
植物抵御病原菌侵染是一個復雜的免疫過程,激素是調控該過程的重要信號分子[29]。在已知的植物激素中,SA、茉莉酸(jasmonic acid,JA)和ET是參與免疫反應最為核心的成員[29]。在中存在與ET、SA、auxin和ABA等激素信號響應相關的的順式作用元件(表1),表明可能通過以上4種激素信號通路參與棉花的生長發育和抗逆反應。棉花被黃萎病菌侵染后,體內SA會發生顯著積累[11]。SA信號通路上的標志性基因發生沉默后,棉花對黃萎病的抗性也會顯著降低[11],表明SA在棉花抗黃萎病過程中發揮重要的調控功能。病原菌誘導的SA合成主要經異分支酸合酶(isochorismate synthase,ICS)途徑,發生在葉綠體中[30]。另外,與SA積累相關的重要標志基因有和[31]。是SA信號通路的重要調節基因。NPR1接收到SA信號后會由多聚體(oligomer)變為單體(monomer),從胞質進入細胞核,與TGA(TGACG motif-binding factor)互作,進而調節相關抗病基因的表達,特別是病程相關蛋白PR1的積累[32]。本研究檢測了SA誘導后的表達情況,發現SA能夠誘導顯著上調表達(圖4)。沉默后,棉苗體內SA含量顯著降低(圖8),、、、和等5個與SA積累和信號調控相關的標志基因均發生了顯著下調表達(圖9)。據此,推測可能通過SA信號通路參與棉花對黃萎病菌的抗性。
3.3 植物未知功能基因研究展望
目前,在蛋白家族數據庫(Pfam)中未知功能結構域蛋白家族約占26.5%[33],意味著在植物界存在數量巨大的未知功能基因。大量組學數據分析都表明,這些未知功能基因在植物生長發育和逆境脅迫過程中發揮重要作用。因此,對這些基因功能的研究將有助于在全新層面上透徹地了解生物體復雜的生命活動機制[34]。本文對進行了表達模式分析,并證明其參與了棉花對黃萎病脅迫的響應,但其具體的調控機制還需深入研究。為進一步完善的功能注釋,解析CRVW蛋白三維構象、尋找其互作蛋白、明確其參與的信號通路等都將是未來重要的研究內容。
4 結論
棉花CRVW無明顯結構域,且無生理生化功能注釋。CRVW定位于細胞質和細胞膜,主要在棉花根部表達;可能通過SA信號通路參與棉花抗黃萎病,是一個潛在重要的抗病基因。
[1] 馬存, 簡桂良, 鄭傳臨. 中國棉花抗枯、黃萎病育種50年. 中國農業科學, 2002, 35(5): 508-513.
MA C, JIAN G L, ZHENG C L. The advances in cotton breeding resistance to fusarium and Verticillium wilts in China during past fifty years., 2002, 35(5): 508-513. (in Chinese)
[2] KLOSTERMAN S J, ATALLAH Z K, VALLAD G E, SUBBARAO K V. Diversity, pathogenicity, and management of Verticillium species., 2009, 47: 39-62.
[3] XU L, ZHU L F, TU L L, LIU L L, YUAN D J, JIN L, LONG L, ZHANG X L. Lignin metabolism has a central role in the resistance of cotton to the wilt fungusas revealed by RNA-Seq-dependent transcriptional analysis and histochemistry., 2011, 62(15): 5607-5621.
[4] FRADIN E F, THOMMA B P. Physiology and molecular aspects of Verticillium wilt diseases caused byand., 2006, 7(2): 71-86.
[5] 潘家駒, 張天真, 蒯本科, 郭小平, 王謚. 棉花黃萎病抗性遺傳研究. 南京農業大學學報, 1994, 17(3): 8-18.
PAN J J, ZHANG T Z, JING B K, GUO X P, WANG S. Studies on the inheritance of resistance toin cotton., 1994, 17(3): 8-18. (in Chinese)
[6] LIU N N, SUN Y, PEI Y K, ZHANG X Y, WANG P, LI X C, LI F G, HOU Y X. A pectin methylesterase inhibitor enhances resistance to Verticillium wilt., 2018, 176(3): 2202-2220.
[7] HU Q, MIN L, YANG X Y, JIN S X, ZHANG L, LI Y Y, MA Y Z, QI X W, LI D Q, LIU H B, LINDSEY K, ZHU L F, ZHANG X L. Laccase GhLac1 modulates broad-spectrum biotic stress tolerance via manipulating phenylpropanoid pathway and jasmonic acid synthesis., 2018, 176(2): 1808-1823.
[8] ZHANG Y, WU L Z, WANG X F, CHEN B, ZHAO J, CHEN J, LI Z K, YANG J, WU L Q, WU J H, ZHANG G Y, MA Z Y. The cotton laccase gene GhLAC15 enhances Verticillium wilt resistance via an increase in defence-induced lignification and lignin components in the cell walls of plants., 2019, 20(3), 309-322.
[9] YANG J, ZHANG Y, WANG X F, WANG W Q, LI Z K, WU J H, WANG G N, WU L Q, ZHANG G Y, MA Z Y. HyPRP1 performs a role in negatively regulating cotton resistance tovia the thickening of cell walls and ROS accumulation., 2018, 18(1): 339.
[10] MO H J, WANG X F, ZHANG Y, ZHANG G Y, ZHANG J F, MA Z Y. Cotton polyamine oxidase is required for spermine and camalexin signalling in the defence response to., 2015, 83(6): 962-975.
[11] ZHANG Y, WANG X F, RONG W, YANG J, LI Z K, WU L Q, ZHANG G Y, MA Z Y. Histochemical analyses reveal that stronger intrinsic defenses inthan inare associated with resistance to., 2017, 30(12): 984-996.
[12] ZHANG T Z, HU Y, JIANG W K, FANG L, GUAN X Y, CHEN J D, ZHANG J B, SASKI C A, SCHEFFLER B E, STELLY D M, HULSE-KEMP A M, WAN Q, LIU B L, LIU C X, WANG S, PAN M Q, WANG Y K, WANG D W, YE W X, CHANG L J, ZHANG W P, SONG Q X, KIRKBRIDE R C, CHEN X Y, DENNIS E, LLEWELLYN D J, PETERSON D G, THAXTON P, JONES D C, WANG Q, XU X Y, ZHANG H, WU H T, ZHOU L, MEI G F, CHEN S Q, TIAN Y, XIANG D, LI X H, DING J, ZUO Q Y, TAO L N, LIU Y C, LI J, LIN Y, HUI Y Y, CAO Z S, CAI C P, ZHU X F, JIANG Z, ZHOU B L, GUO W Z, LI R Q, CHEN Z J. Sequencing of allotetraploid cotton (L. acc. TM-1) provides a resource for fiber improvement., 2015, 33(5): 531-537.
[13] LIU Y, SCHIFF M, MARATHE R, DINESH-KUMAR S P. Tobacco Rar1, EDS1 and NPR1/NIM1 like genes are required for N-mediated resistance to tobacco mosaic virus., 2002, 30(4): 415-429.
[14] 姚丹青, 張微微, 原麗華, 潘俊松, 何歡樂, 蔡潤. VIGS: 植物功能基因組學研究的革命. 分子植物育種, 2009, 7(1): 155-161.
YAO D Q, ZHANG W W, YUAN L H, PAN J S, HE H L, CAI R. VIGS: the revolution of plant function genomics research., 2009, 7(1): 155-161. (in Chinese)
[15] GAO X Q, WHEELER T, LI Z, KENERLEY C M, HE P, SHAN L B. Silencingandcompromises cotton resistance to Verticillium wilt., 2011, 66(2): 293-305.
[16] GAO W, LONG L, ZHU L F, XU L, GAO W H, SUN L Q, LIU L L, ZHANG X L. Proteomic and virus-induced gene silencing (VIGS) analyses reveal that gossypol, brassinosteroids, and jasmonic acid contribute to the resistance of cotton to., 2013, 12(12): 3690-3703.
[17] MA Z Y, HE S P, WANG X F, SUN J, ZHANG Y, ZHANG G, WU L, LI Z, LIU Z, SUN G, YAN Y, JIA Y, YANG J, PAN Z, GU Q, LI X, SUN Z, DAI P, LIU Z, GONG W, WU J, WANG M, LIU H, FENG K, KE H, WANG J, LAN H, WANG G, PENG J, WANG N, WANG L, PANG B, PENG Z, LI R, TIAN S, DU X. Resequencing a core collection of upland cotton identifies genomic variation and loci influencing fiber quality and yield., 2018, 50(6): 803-813.
[18] ZHANG Y, WANG X F, DING Z G, MA Q, ZHANG G R, ZHANG S L, LI Z K, WU L Q, ZHANG G Y, MA Z Y. Transcriptome profiling ofinoculated withprovides a resource for cotton improvement., 2013, 14: 637.
[19] 王國寧, 趙貴元, 岳曉偉, 李志坤, 張艷, 張桂寅, 吳立強, 王省芬, 馬峙英. 河北省棉花黃萎病菌致病性與ISSR遺傳分化. 棉花學報, 2012, 24(4): 348-357.
WANG G N, ZHAO G Y, YUE X W, LI Z K, ZHANG Y, ZHANG G Y, WU L Q, WANG X F, MA Z Y. Pathogenicity and ISSR genetic differentiation ofisolates from cotton growing areas of Hebei province. Cotton Science, 2012, 24(4): 348-357. (in Chinese)
[20] 吳立柱, 王省芬, 李喜煥, 馬峙英. 通用型植物表達載體pCamE的構建及功能驗證. 農業生物技術學報, 2014, 22(6): 661-671.
WU L Z, WANG X F, LI X H, MA Z Y. Construction and function identification of universal plant expression vector pCamE., 2014, 22(6): 661-671. (in Chinese)
[21] YANG J, JI L L, WANG X F, ZHANG Y, WU L Q, YANG Y, MA Z Y. Overexpression of 3-deoxy-7-phosphoheptulonate synthase gene fromenhancesresistance to Verticillium wilt., 2015, 34(8): 1429-1441.
[22] ARTICO S, NARDELI S M, BRILHANTE O, GROSSI-DE-SA M F, ALVES-FERREIRA M. Identification and evaluation of new reference genes infor accurate normalization of real-time quantitative RT-PCR data., 2010, 10: 49.
[23] SUN L Q, ZHU L F, XU L, YUAN D J, MIN L, ZHANG X L. Cotton cytochrome P450 CYP82D regulates systemic cell death by modulating the octadecanoid pathway., 2014, 5: 5372.
[24] 馬峙英, 王省芬, 張桂寅, 李興紅, 劉叔倩, 吳立強, 劉占國, 孫濟中, 劉金蘭. 河北省棉花黃萎病菌致病性的研究. 棉花學報, 1997, 9(1): 15-20.
MA Z y, Wang X f, Zhang G y, LI X H, LIU S J, WU L Q, LIU Z G, SUN J Z, LIU J L. Study on pathogenicity ofin Hebei Province., 1997, 9(1): 15-20. (in Chinese)
[25] CHEN H, NELSON R S, SHERWOOD J L. Enhanced recovery of transformants ofafter freeze-thaw transformation and drug selection., 1994, 16(4): 664-668, 670.
[26] GAO X Q, BRITT R J, SHAN L B, HE P.-mediated virus-induced gene silencing assay in cotton., 2011(54): e2938.
[27] VERBERNE M C, BROUWER N, DELBIANCO F, LINTHORST H J M, BOL J F, VERPOORTE R. Method for the extraction of the volatile compound salicylic acid from tobacco leaf material., 2002, 13: 45-50.
[28] SUN Q, JIANG H Z, ZHU X Y, WANG W N, HE X H, SHI Y Z, YUAN Y L, DU X M, CAI Y F. Analysis of sea-island cotton and upland cotton in response toinfection by RNA sequencing., 2013, 14: 852.
[29] PIETERSE C M J, LEON-REYES A, VAN DER ENT S, VAN WEES S C M. Networking by small-molecule hormones in plant immunity., 2009, 5(5): 308-316.
[30] WILDERMUTH M C, DEWDNEY J, WU G, AUSUBEL F M. Isochorismate synthase is required to synthesize salicylic acid for plant defence., 2001, 414(6863): 562-565.
[31] ZHENG X Y, ZHOU M, YOO H, PRUNEDAPAZ J L, SPIVEY N W, KAY S A, DONG X. Spatial and temporal regulation of biosynthesis of the plant immune signal salicylic acid., 2015, 112(30): 9166-9173.
[32] YASUOMI T D, STEVEN H S, KAROLINA P M, ZHONG L M, SONG J Q, WANG C, ZUO J R, DONG X N. Plant immunity requires conformational charges of NPR1 via S-nitrosylation and thioredoxins. 2008, 321(5891): 952-956.
[33] PUNTA M, COGGILL P C, EBERHARDT R Y, MISTRY J, TATE J, BOURSNELL C, PANG N, FORSLUND K, CERIC G, CLEMENTS J, HEGER A, HOLM L, SONNHAMMER E L, EDDY S R, BATEMAN A, FINN R D. The Pfam protein families database., 2012, 40(Database issue): D290- D301.
[34] 羅成科, 肖國舉, 李明. 不同未知功能結構域蛋白家族(DUFs)基因在植物中的生物學功能. 植物生理學報, 2015, 51(2): 153-158.
LUO C K, XIAO G J, LI M. Biological functions of different domains of unknown function protein families (DUFs) genes in plants., 2015, 51(2): 153-158. (in Chinese)
Cloning and Functional Characterization of GeneInvolved in Cotton Resistance to Verticillium Wilt
WANG QiuYing, WANG WeiQiao, ZHANG Yan, WANG GuoNing, WU LiQiang, ZHANG GuiYin, MA ZhiYing, YANG Jun, WANG XingFen
(College of Agronomy, Hebei Agricultural University/North China Key Laboratory for Crop Germplasm Resources, Ministry of Education, Baoding 071001, Hebei)
【】Verticillium wilt is an important disease in cotton production, and it seriously affects the yield and quality of cotton. Genome sequences ofprovide valuable information resources for searching for resistance genes. In this study, an uncharacterized gene, designed as(cotton resistance to Verticillium wilt), was cloned and identified for disease resistance. The results will lay a foundation for upgrading cotton genomic information, further studying the resistance mechanism and molecular breeding. 【】 The open reading frame (ORF) ofwas cloned from upland cotton cultivar ND601 using the primers, which were designed according to the reference genome sequence. The online software ProtParam was used to predict protein properties, including amino acid composition, molecular weight, the theoretic isoelectric point, instability index and grand average of hydropathicity. PSIPRED v3.3 was used to predict the protein secondary structure. The prediction of protein subcellular localization and-acting elements in the promoter was performed using ProtComp v. 9.0 and PlantCARE, respectively. To elucidate the subcellular localization of the CRVW protein, the CRVW-GFP fusion construct was transformed into onion epidermal cells by particle bombardment. qRT-PCR was performed using normal cotton tissues and tissues that were treated with exogenous application of salicylic acid (SA) andstress. The function ofinvolving in cotton resistance towas further verified by the technology of virus-induced gene silencing (VIGS). To preliminarily analyze the disease resistance pathway mediated by, the expression of some marker genes related to plant disease resistance was assayed in-silenced plants.【】A 780 bp ORF ofwas successfully cloned fromND601.encodes a putative protein of 259 amino acids with a molecular mass of 30.2 kD and an isoelectric point of 9.59. The protein secondary structure of CRVW contains 69.50% random coil, 17.76% α-helical, 11.20% extension and 1.54% β-sheet. By bioinformatics prediction and fluorescence observation, we found that CRVW was mainly located in the cell membrane and cytoplasm.was expressed in the roots, stems and leaves of cotton, but the highest expression occurred in the roots. The upstream sequence ofORF () contains-acting elements in response to four kinds of hormones, including ethylene, SA, auxin and abscisic acid. Additionally,includes a few other elements relating to injury, defense, stress, disease, drought and low temperature. The expression ofwas significantly upregulated in the leaves sprayed with SA. After inoculated with,was dramatically upregulated both in resistant cultivar ND601 and susceptible cultivar CCRI8, but the upregulated expression in susceptible cultivar lagged behind in the resistant cultivar. After 20 days inoculated with,silenced cotton seedlings showed more clearly chlorosis, wilting and defoliating comparing to CK. Further statistical analysis showed thatsilenced cotton seedlings had higher disease index than the CK, suggesting that the silence ofsignificantly reduced the resistance of cotton seedling to. Endogenous SA content insilenced cotton seedlings was significantly lower than in CK. The expression of marker genes related to SA accumulation and signal regulation, including(isochorismate synthase 1),(enhanced disease susceptibility 1),(phytoalexin deficient 4)(nonexpresser of PR gene 1) and(pathogenesis- related protein 1), were significantly down-regulated after silencing.【】CRVW is located in the cytoplasm and the cell membrane, mainly expressed in cotton roots, and involved in the process of cotton resistance to Verticillium wilt, perhaps through SA-mediated defense pathway.
cotton; Verticillium wilt; CRVW; clone; virus-induced gene silencing; resistance
10.3864/j.issn.0578-1752.2019.11.002
2019-01-08;
2019-03-22
河北省自然科學基金(C2016204098)、河北省科技支撐計劃(16226307D)
王秋瑩,Tel:0312-7528415;E-mail:wangqiuying9308@163.com。通信作者楊君,Tel:0312-7528401;E-mail:yang22181@163.com。通信作者王省芬,Tel:0312-7528401;E-mail:cotton@mail.hebau.edu.cn
(責任編輯 李莉)

