基于RAPD的白色金針菇菌株遺傳多樣性分析
楊和川,譚一羅,蘇文英,秦裕營,馬 騰,浦漢春,周振玲
(連云港市農業科學院,江蘇 連云港 222000)
金針菇(Flammulinavelutipes)又名毛柄金錢菌、構菌、樸菇、冬菇等,在自然界中分布廣泛,是我國最早開始人工栽培的食用菌之一,具有廣闊的市場前景[1]。金針菇鮮菇的顏色是其銷售的重要分級指標之一,早期生產企業多采用黃色金針菇菌種,但由于黃色金針菇菌株在栽培中易發生褐變等問題,嚴重影響了金針菇的品質、銷售及加工。因此,近年來金針菇工廠化生產企業均以白色金針菇的生產為主[2-3]。由于金針菇可以通過無性繁殖進行菌種生產,且菌種分離難度低,因此市場上金針菇菌種自行編號或命名現象普遍,菌種生產、銷售混亂,極易出現同種異名、異種同名的情況,既給金針菇生產中菌種管理帶來了困難,又影響了育種者的權益,同時,劣質菌種流入生產還會造成巨大的經濟損失。因此,研究目前市場上白色金針菇種質資源的遺傳多樣性,對金針菇的種性鑒定、遺傳育種等基礎科研和產業體系建立等具有重要意義。
傳統的菌株鑒別方法,如子實體形態特征和拮抗試驗等易受外界環境的影響,同一菌株在不同栽培條件下表現出較大的差異,不能鑒別出親緣關系較近的菌株[4-6]。近年來,DNA指紋技術被廣泛應用于真菌的系統學研究及其分類鑒定,隨機擴增多態性DNA(randomly amplified polymorphic DNA,RAPD)、擴增片段長度多態性(Amplified Fragment Length Polymorphism,AFLP)、簡單重復序列(Simple Sequence Repeats,SSR)、簡單重復序列區間(Inter-Simple Sequence Repeats,ISSR)、特征性片段擴增區域(Sequence Characterized Amplified Region,SCAR)等技術[7-11]在食用菌種質資源的評價、菌株鑒別、遺傳多樣性的研究中取得了顯著成效。其中,RAPD分子標記技術具有操作簡便、對基因組DNA濃度和試劑要求低、成本低廉的優點。為了避免在實際生產中品種混亂,降低栽培風險,本研究對15種白色金針菇菌株進行了RAPD分子標記分析,以期為金針菇的種性研究、良種選育及種質資源管理提供參考和依據。
1 材料與方法
1.1 供試菌株
15株白色金針菇菌株的編號和來源如表1所示。
表1 供試菌株編號及來源
1.2 試劑和儀器
DreamTaq PCR Master Mix(2×)購自賽默飛世爾科技公司,DNA提取試劑盒購自康為世紀生物科技有限公司,DNA marker、引物序列購自北京鼎國昌盛生物技術有限責任公司,其余試劑購自北京奧博星生物技術有限責任公司。PCR儀為德國艾本德股份公司產品,電泳儀、電泳槽為伯樂生命醫學產品(上海)有限公司產品。
1.3 培養基
MYG固體培養基:葡萄糖5 g、酵母浸粉5 g、麥芽糖10 g、瓊脂糖15 g、水1000 mL、自然pH值。該培養基用于金針菇菌絲培養。
1.4 試驗方法
1.4.1 總DNA的提取 將供試菌株分別接種于MYG固體培養基平皿,25 ℃恒溫培養10 d,刮取平皿上菌株氣生菌絲體于1.5 mL離心管中,放入液氮研磨成粉末,采用DNA提取試劑盒提取金針菇的基因組DNA,提取的DNA于-20 ℃下保存備用。
1.4.2 RAPD-PCR的擴增 采用的20條RAPD引物序列見表2。PCR反應體系20 μL,包含模板DNA 1 μL、引物1μL、DreamTaq PCR Master Mix(2×)10 μL、ddH2O 8 μL。PCR反應程序:92 ℃預變性5 min;92 ℃變性1 min;35 ℃退火1 min;72 ℃延伸2 min,循環40次,72 ℃延伸10 min,擴增產物于-20 ℃下保存。從20條RAPD引物中篩選譜帶清晰、多態性豐富的引物,采用1%瓊脂糖凝膠電泳,拍照記錄電泳結果。
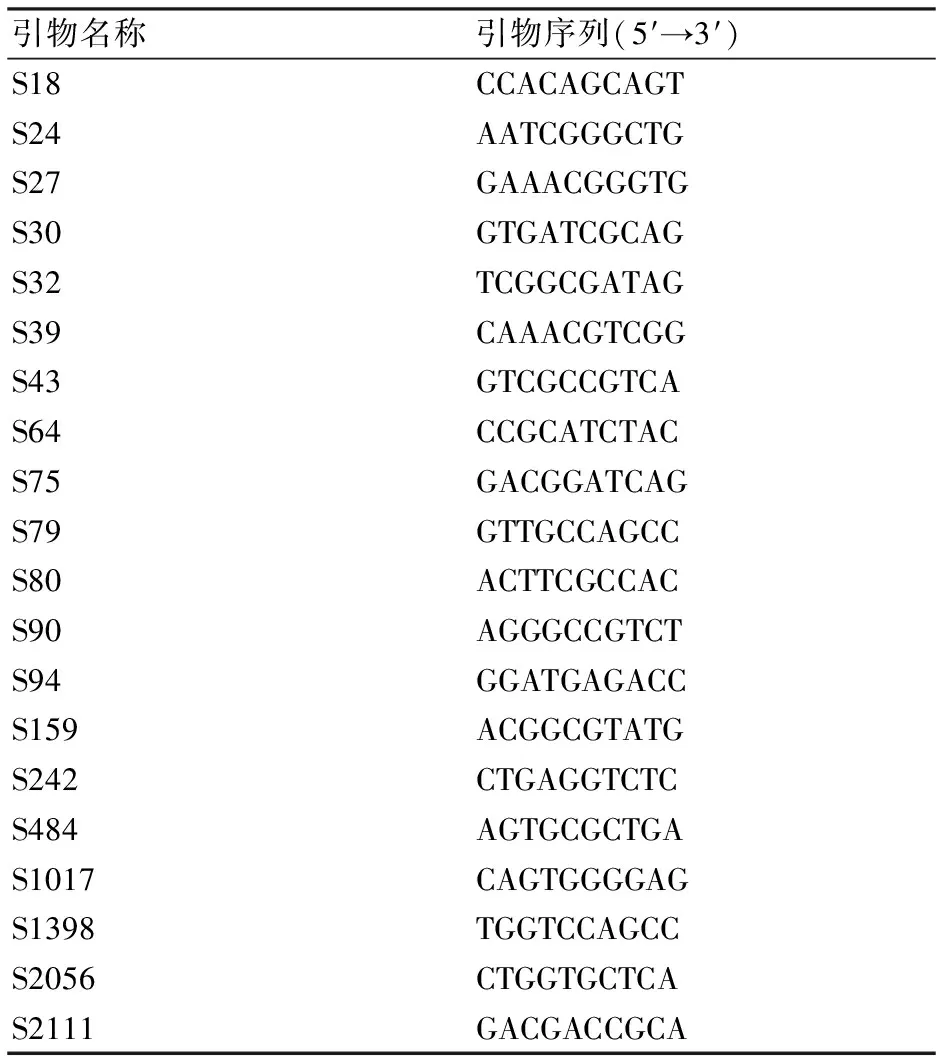
表2 RAPD擴增使用的引物
1.4.3 數據處理 對電泳圖譜進行條帶統計,相同位移上有條帶記為“1”,無條帶記為“0”,轉換為0/1矩陣。利用NTSYSpc 2.11軟件對0/1矩陣數據進行分析,通過非加權配對算數平均法(Unweighted Pair-group Method with Arithmetic means,UPGMA)進行聚類分析,并基于原始0/1矩陣進行主成分分析(Principal Component Analysis,PCA)。
2 結果與分析
2.1 RAPD-PCR擴增
從20條RAPD引物中篩選出8條譜帶清晰、多態性豐富的引物,分別為引物S43、S75、S79、S80、S90、S484、S1398、S2111。使用這8條引物對15株白色金針菇菌株進行多態性分析(圖1),共計擴增出45條片段,其中多態性片段33條,多態性比率為73.3%,說明RAPD分子標記有較好的多態性。
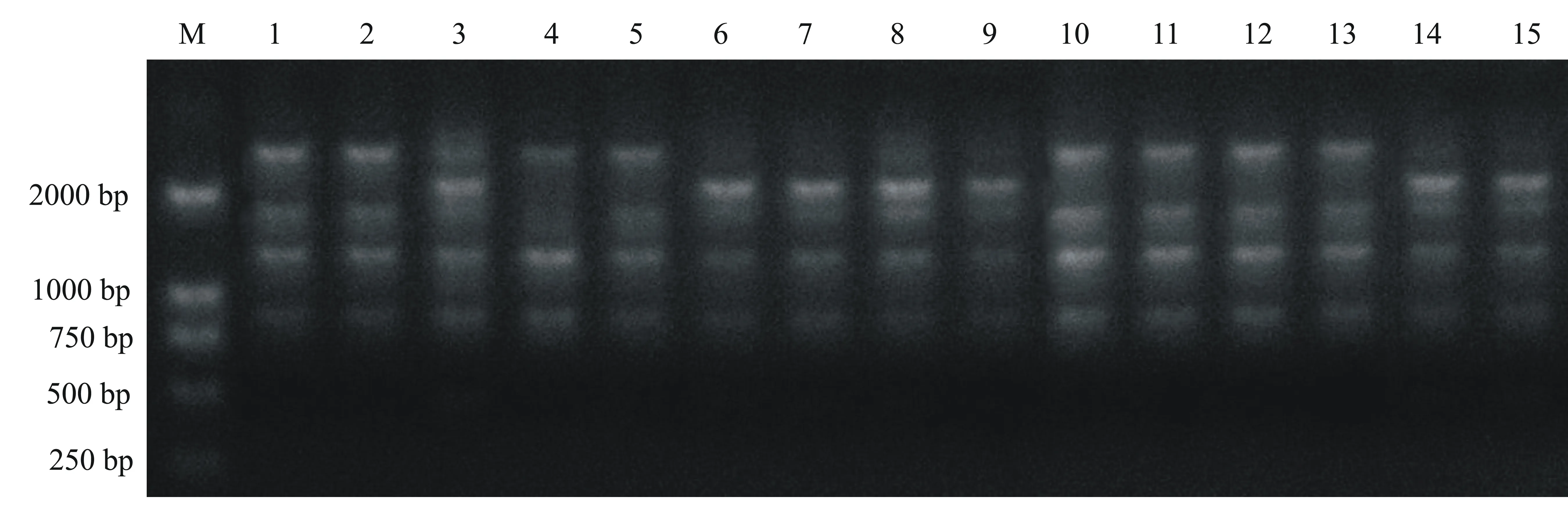
1~15為供試菌株編號,與表1中品種編號一致;M為DL2000 Marker。下同。
2.2 供試菌株遺傳相似性分析
通過NTSYSpc 2.11軟件對15株白色金針菇菌株間遺傳相似系數進行了計算,結果表明:供試菌株的遺傳相似系數在0.560~0.984之間,2號菌株與15號菌株的遺傳相似系數最小,12號菌株與13號菌株的遺傳相似系數最大,說明2號菌株與15號菌株的親緣關系較遠,12號菌株與13號菌株的親緣關系較近(表3)。
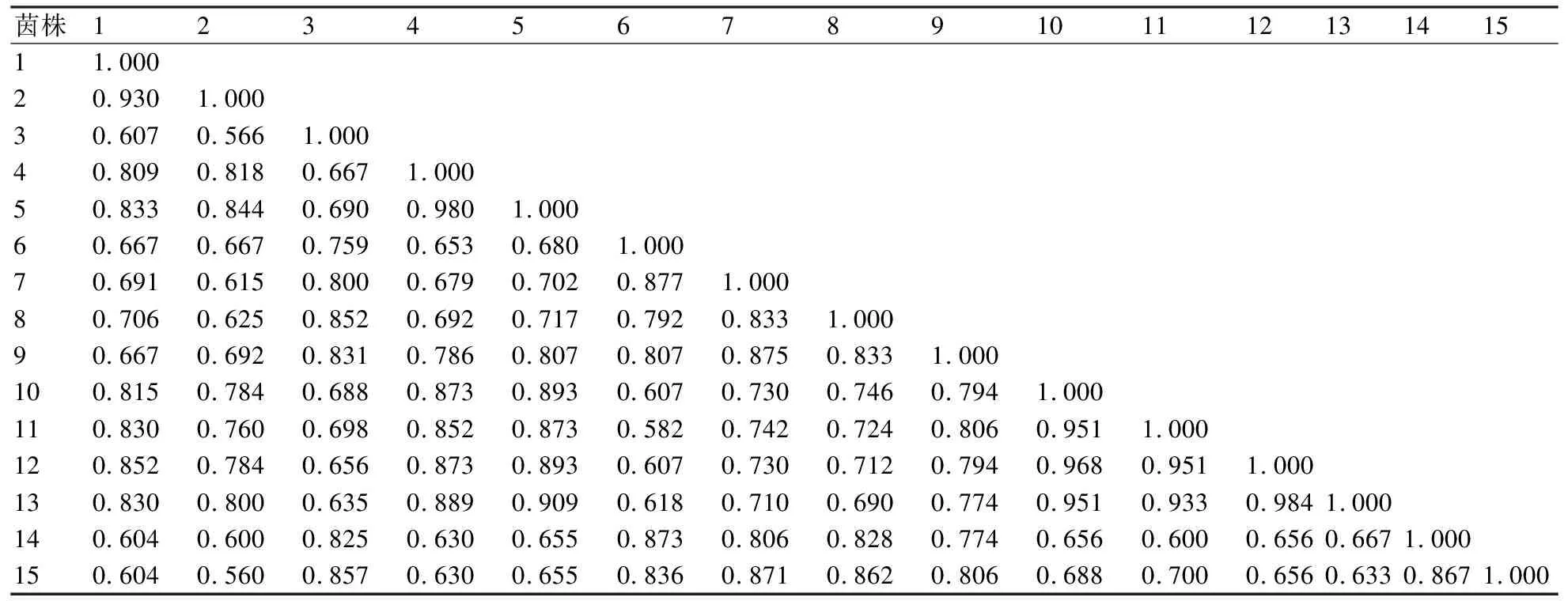
表3 基于RAPD的供試菌株的遺傳相似系數矩陣
注:1~15為供試菌株編號,與表1中品種編號一致。下同。
2.3 供試菌株UPGMA聚類分析
采用UPGMA法進一步聚類進行分析,構建遺傳親緣系數樹狀圖(圖2)。當遺傳相似系數在0.68時,15株供試菌株被聚為1類;在0.830水平上被聚為4類,其中,類群Ⅰ包含1號和2號菌株;類群Ⅱ包含4、5、10、12、13、11號菌株;類群Ⅲ包含3、8、9號菌株;類群Ⅳ包含6、7、14、15號菌株。
2.4 供試菌株的主成分分析
對15株供試金針菇種質的原始矩陣進行主成分分析,第1主坐標和第2主坐標的方差貢獻率分別為45.31%和15.40%,以第1、第2主坐標的二維圖排序繪制主成分分析二維聚類圖(圖3)。可以看出,二維聚類圖將15個白色金針菇菌株分為4類,比較圖2和圖3,主成分分析結果與遺傳相似系數0.830水平上聚類分析結果相似,聚類圖中部分菌株間的遺傳多樣性較高,因此對應在主成分分析圖中的分布相對分散。
3 討論
利用菌株的拮抗反應和農藝性狀比較是鑒別食用菌菌株的常見辦法。但是,一方面,遺傳背景相似或親緣關系很近的菌株則難以區分,且拮抗試驗不具有傳遞性;另一方面,金針菇子實體農藝性狀受環境因素的影響大,栽培流程復雜、耗時耗工,因此采用分子生物學技術研究金針菇菌株間的親緣關系成為一種重要方法。其中,RAPD技術不需要供試菌株的基因組序列信息,而是采用隨機引物,因此,該方法具有流程簡單、成本低廉的優勢。
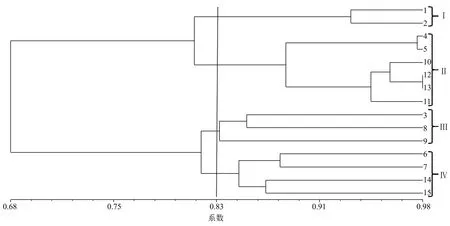
圖2 15株供試菌株的RAPD分子標記聚類圖
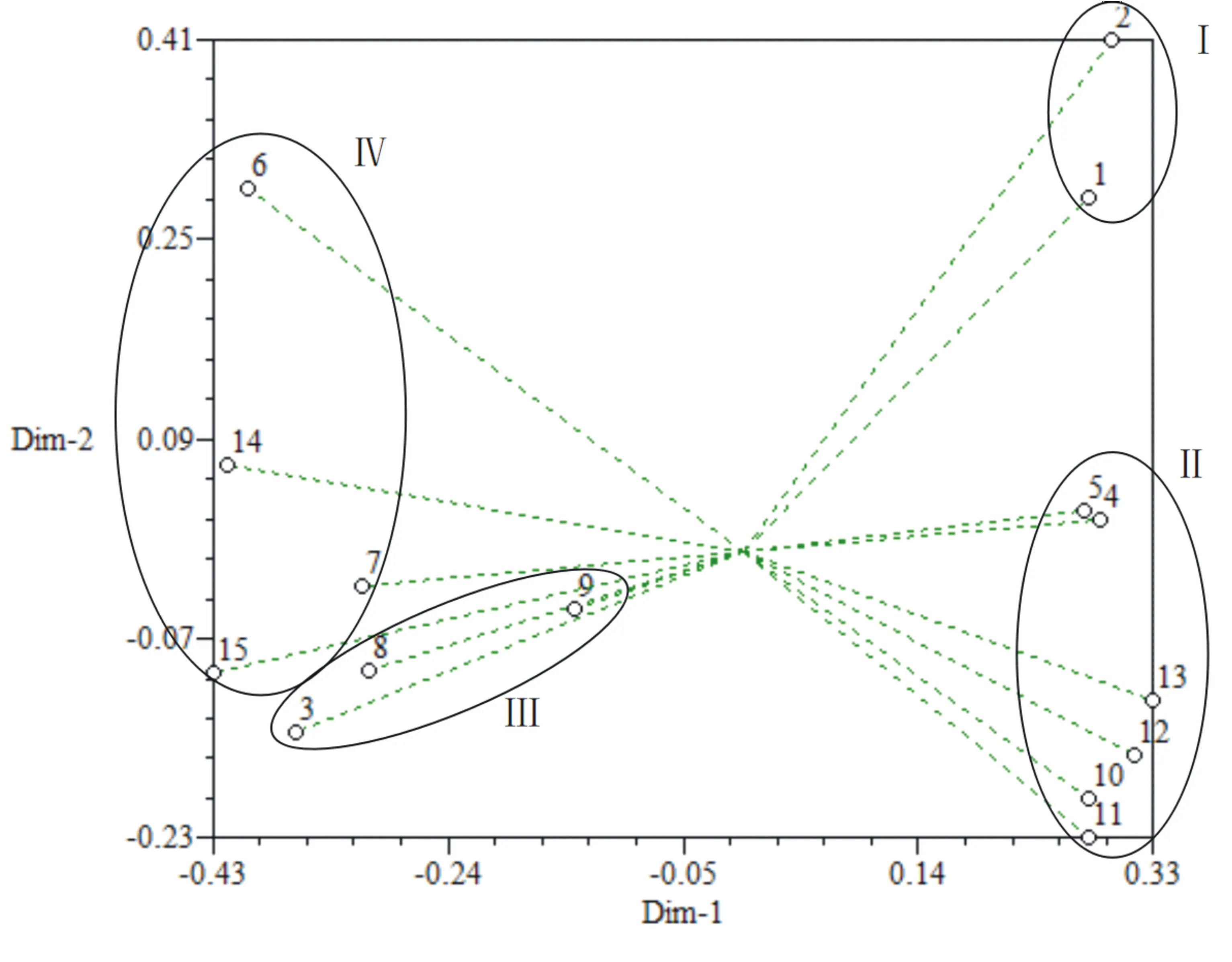
圖3 供試菌株二維主成分分析圖
基于RAPD技術對供試菌株進行遺傳多樣性的分析,試驗結果能明顯反映供試菌株間的遺傳差異和親緣關系。15株白色金針菇菌株根據子實體顏色又可以分為乳白色和白色2類,聚類分析結果雖然可將供試菌株分為4類,但是無法將白色和乳白色菌株子實體按顏色區分,乳白色金針菇菌株可能來源于不同顏色菌株的雜交。對照UPGMA聚類圖和主成分分析圖,在主成分分析圖中類群Ⅰ和類群Ⅱ能明顯劃分,且類群內的菌株分布較近,說明類群內菌株的親緣關系較近,2個類群間的親緣關系較遠;而類群Ⅲ、類群Ⅳ內菌株間分布較分散,說明類群內菌株彼此親緣關系相對較遠,與UPGMA聚類結果一致。本研究結果表明:RAPD分子標記是研究金針菇菌株遺傳多樣性的一個有效方法,菌株DNA指紋圖譜的建立對知識產權保護具有重要意義。但是,單一分子標記技術在計算遺傳相似系數時存在偏差,因此同時結合其他分子標記技術綜合分析金針菇菌株的遺傳多樣性,開發SCAR和SSR等能特異鑒別金針菇菌株的分子標記,對金針菇菌株的準確鑒定、保護知識產權、育種親本選擇、提高育種效率均具有重要意義。

