基于靶向二代測序技術檢測肝細胞癌線粒體DNA突變
劉洋 郭姍姍劉曼玲 李藝杰 賈永峰 郭旭
線粒體是幾乎存在于所有真核細胞中的重要細胞器,每個細胞都包含多個拷貝的線粒體基因組。有研究報道線粒體DNA(mitochondrial DNA,mtDNA)突變引起的線粒體功能障礙與腫瘤發展和轉移關系密切[1-2]。對肝癌患者全外顯子測序(whole exosome sequencing,WES)數據中的mtDNA測序數據進行分析,發現了92個體細胞突變[3]。因線粒體占據了肝細胞胞質體積的20%[4],且在肝代謝中起重要作用,因此推測肝細胞癌(hepatocellular carcinoma,HCC)的發生可能與mtDNA突變密切相關。目前傳統測序技術已被廣泛用于檢測癌癥的體細胞mtDNA突變[5-10],但操作復雜,檢測靈敏度低,臨床應用受限。本研究利用全基因組測序(whole genome sequencing,WGS)技術檢測mtDNA拷貝數,并基于靶向二代測序(next generation sequencing,NGS)技術,在匹配的 HCC 癌組織、癌旁組織、外周血細胞與血漿樣本中進行了深覆蓋度的mtDNA測序并分析mtDNA突變的一致性與差異性,以進一步闡明血漿中mtDNA的突變情況,以及可能在HCC早期診斷、預后評估中的作用。
1 材料與方法
1.1 標本來源
收集2017年9月23日至2017年12月18日在空軍軍醫大學第一附屬醫院經病理確診的HCC患者癌組織及癌旁組織(距癌組織≤3 cm)、外周血細胞及血漿樣本,其中癌組織及癌旁組織為多點取材。樣本篩選標準:癌組織中腫瘤細胞比例>90%且沒有壞死,癌旁組織中腫瘤細胞比例<5%;所有樣本提取的DNA均于-20℃下保存。本研究最終共納入5例HCC患者,所有病例術前未經放療、化療。5例HCC患者的臨床特征見表1。
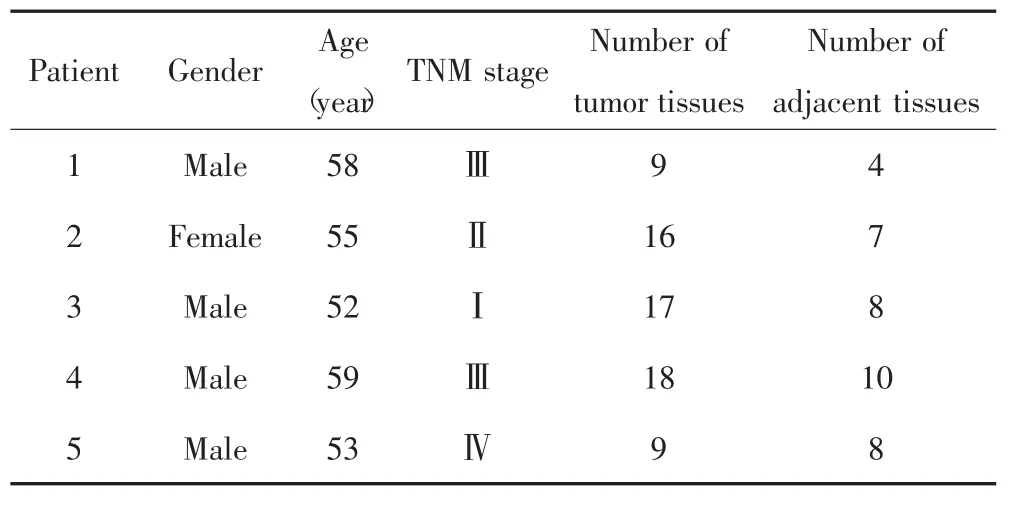
表1 5例肝細胞癌患者的臨床病理特征Tab.1 Clinicopathological characteristics of five HCC patients
1.2 主要試劑與儀器
E.N.Z.A組織DNA提取試劑盒和E.N.Z.A血液DNA 提取試劑盒購自 Omega(New York,USA),QIAamp Circulating Nucleic Acid Kit購自 Qiagen(Dusseldorf,Germany),NEB Ultra v2 Kit購自 New England Biolabs(Ipswich,US),Dynabeads 購 自 Life(Massachusetts,USA)。Illumina HiSeq×Ten,Qubit 3.0 熒光定量儀和Nonodrop分光光度計購自Thermo Fisher Scientific(Massachusetts,USA),Agilent 2100 bioanalyzer system購自Agilent(California,USA)。
1.3 DNA的提取和定量
按照E.N.Z.A組織和血液DNA提取試劑盒說明書分別提取HCC患者癌組織、癌旁組織和相應外周血細胞中的DNA,并用Nonodrop分光光度計檢測DNA的濃度和質量。采用QIAamp Circulating Nucleic Acid Kit提取HCC患者血漿中的游離DNA(cell free,cfDNA),并用Qubit 3.0熒光定量儀檢測cfDNA的濃度和質量。
1.4 DNA測序文庫構建
從HCC癌組織、癌旁組織及外周血細胞樣本中分別取5 μg DNA,按照超聲打斷、末端修復、加A尾、接頭連接、擴增順序進行文庫構建;同時取1 μg血漿cfDNA,根據NEB Ultra v2 Kit試劑盒說明書進行文庫構建。將構建的全基因組文庫置于Illumina HiSeq×Ten平臺進行雙端150 bp測序。
1.5 靶向捕獲測序
將自制生物素標記的mtDNA探針[11]與DNA文庫進行雜交。根據鏈霉親和素磁珠與生物素特異性結合的原理,用mtDNA探針結合目標片段,經磁珠純化后獲得特異性產物。將捕獲的特異性產物置于Illumina HiSeq×Ten平臺進行雙端150 bp測序。
1.6 測序數據分析
對測序數據進行質量控制,條件包括:⑴去除接頭污染的Reads;⑵去除低質量的Reads;⑶去除含N堿基比例>5%的Reads。將質控后的數據通過BWA-mem軟件比對至人類參考基因組hg19,提取比對至線粒體參考基因組(the revised cambridge reference sequence,rCRS)的數據,計算mtDNA拷貝數和片段分布并進行突變位點分析。mtDNA拷貝數=2×mtDNA depth/nuclear depth。mtDNA異質性突變頻率=含突變位點的Reads/總 Reads。異質性突變分析的過濾條件包括:⑴兩條鏈上的次等位基因頻率(minor allele frequency,MAF)≥2%;⑵每條鏈上攜帶突變的Reads≥3;⑶總測序深度≥100×。將MAF≥98%的突變定義為同質性突變。本研究將癌組織和癌旁組織的多點取材數據分別合并進行突變分析。
1.7 統計學分析
使用Graphpad prism 5.0對數據進行統計分析。采用配對的Wilcoxon秩和檢驗分析癌組織和血漿樣本中拷貝數和突變頻率的差異。以P<0.05為差異有統計學意義。
2 結果
2.1 HCC患者癌組織與血漿樣本mtDNA拷貝數的比較
全基因組測序結果顯示,血漿樣本中mtDNA拷貝數平均值為 4.30,中位值為 2.87(1.92~7.84);癌組織中mtDNA拷貝數平均值為868.60,中位值為887.56(517.71~1 086.00)。HCC患者血漿中的mtDNA拷貝數較癌組織明顯降低(Z=-2.023,P=0.008),見圖 1。
2.2 HCC患者癌組織、癌旁組織、外周血細胞與血漿樣本中的突變位點。
對5例HCC患者癌組織、癌旁組織、外周血細胞、血漿樣本進行靶向捕獲測序,平均測序深度分別為 4 683×、5 447×、4 223×、3 335×,見表 2。靶向捕獲測序結果顯示,癌組織、癌旁組織、外周血細胞和血漿樣本可分別鑒定出181、180、181、179個同質性突變和 33、42、3、78 個異質性突變,見表 3。其中,4 種類型樣本可檢測到相同的同質性突變位點;癌組織與血漿樣本中可檢測到相同的異質性突變位點,平均突變個數為4.2;但癌旁組織與血漿樣本中檢測不到相同的異質性突變位點,見圖2。癌組織、癌旁組織、血漿中可分別檢測到10、39、56個特異性突變位點。
2.3 HCC患者癌組織與血漿樣本中突變頻率的比較
根據上述5例HCC患者癌組織及血漿樣本的突變結果分析其異質性突變頻率,結果顯示,癌組織平均異質性突變頻率為0.0888,中位值為0.0837(0.0420~0.1563);血漿樣本平均異質性突變頻率為0.0679,中位值為 0.0596(0.0351~0.1055),兩者比較差異無統計學意義(Z=-1.367,P=0.219),見圖 3。
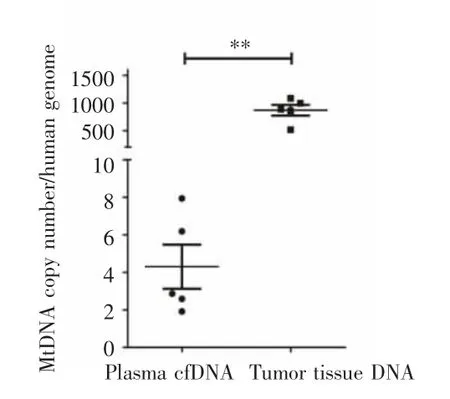
圖1 血漿及腫瘤組織中mtDNA拷貝數的比較Fig.1 Comparison of mtDNA copy numbers in plasma and tumor tissues
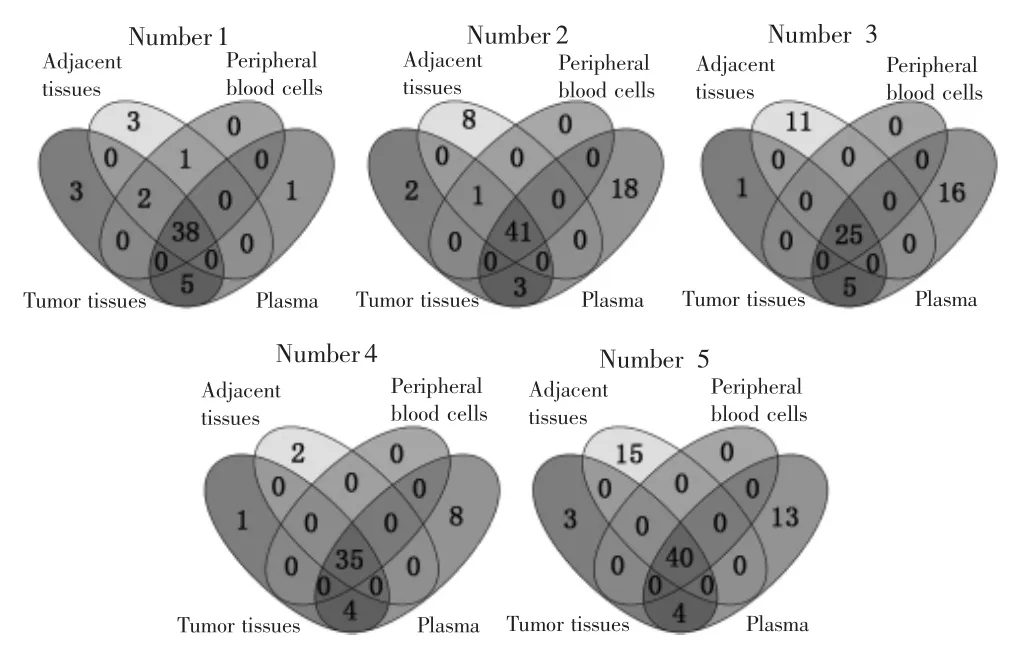
圖2 癌組織、癌旁組織、外周血細胞和血漿樣本中的突變位點數量Fig.2 The number of mutation sites in tumor tissues,adjacent tissues,peripheral blood cells and plasma
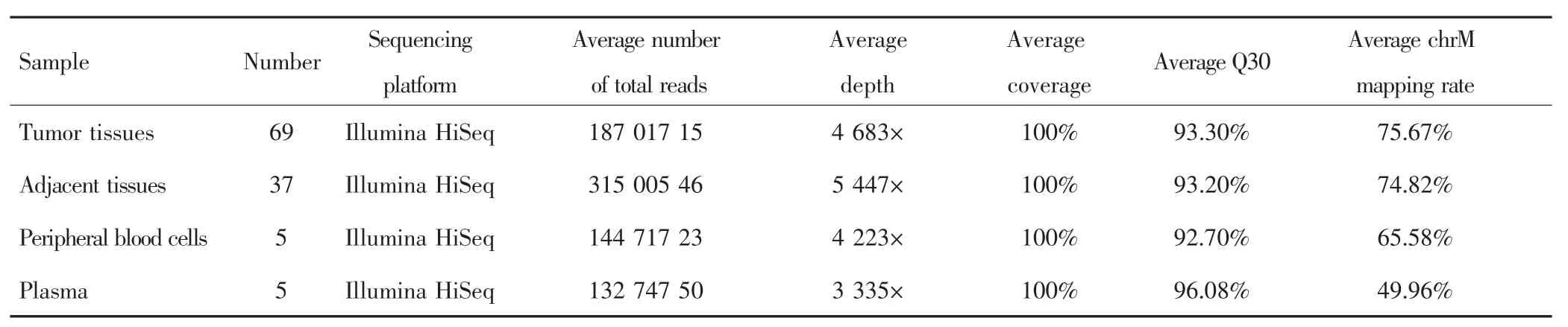
表2 mtDNA測序數據概況Tab.2 Summary of mtDNA sequencing data
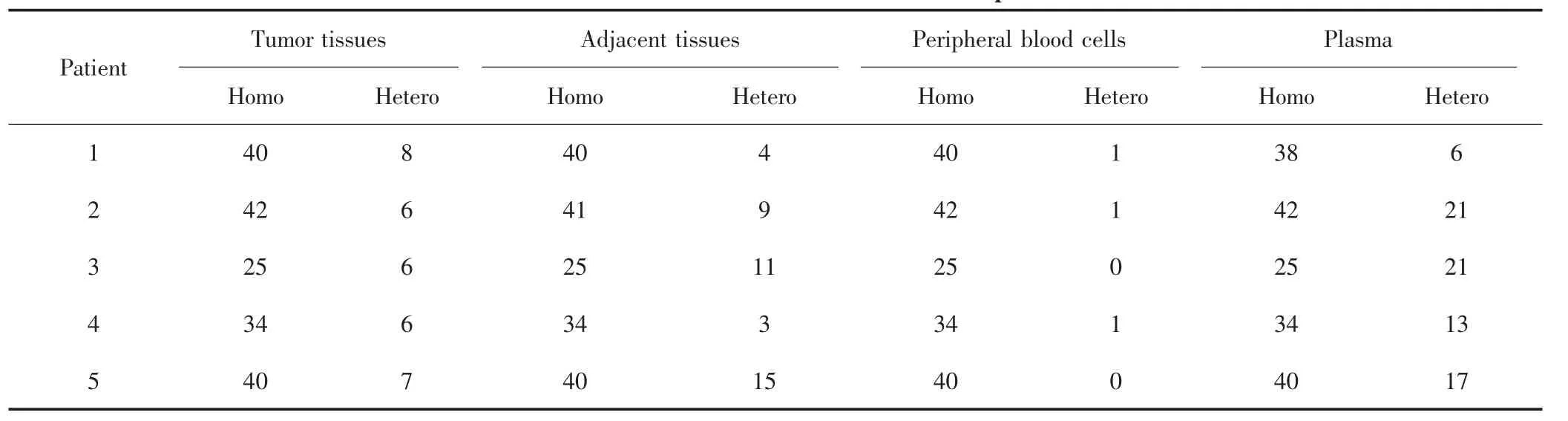
表3 5例肝細胞癌患者的突變位點數量Tab.3 The number of variants for five HCC patients
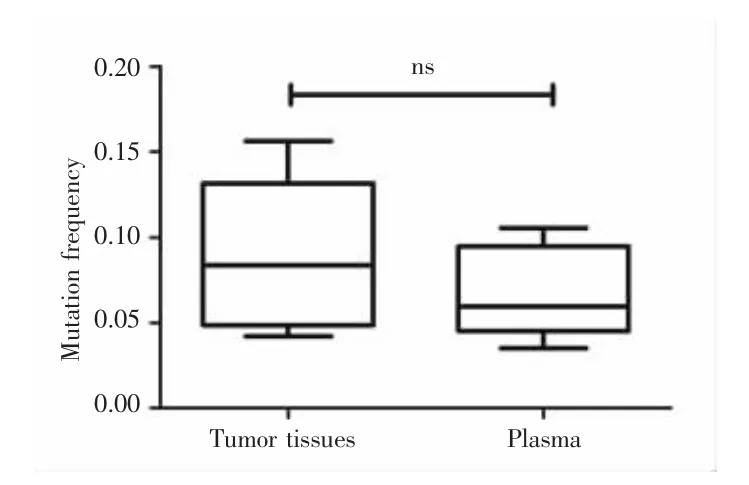
圖3 癌組織與血漿樣本中的突變頻率Fig.3 The mutation frequency between tumor tissues and plasma
3 討論
mtDNA突變在腫瘤發生發展過程中起重要作用[12]。mtDNA是一段雙鏈閉合環狀結構,包括編碼區和非編碼區。與核DNA相比,檢測mtDNA突變及拷貝數變異有以下獨特優勢[13-14]:⑴線粒體DNA全長僅有16 569 bp,測序產生的數據量小,檢測快速、成本低;⑵mtDNA拷貝數高,一個細胞中可含幾百至幾千個mtDNA的拷貝;⑶腫瘤細胞中mtDNA常存在于高水平活性氧環境下,且缺少組蛋白保護和有效的DNA修復系統,突變頻率較核基因組高10倍左右。目前傳統測序技術如Sanger測序已被廣泛應用于檢測癌癥的體細胞mtDNA突變[8-10],但操作復雜,檢測靈敏度低,臨床應用受限。NGS技術為系統檢測多種類型癌癥中的mtDNA異質性突變提供了便利,已越來越多地應用于mtDNA突變檢測中。由于線粒體基因組小,在測序之前,應從包括基因組DNA和mtDNA在內的總細胞DNA中分離mtDNA,以降低測序成本并簡化數據分析流程。富集mtDNA包括兩種主要策略:一種是首先通過密度梯度離心從細胞中分離線粒體,然后通過DNA提取試劑盒從分離的線粒體中提取DNA,但該方法不適用于血漿樣本;另一種是從細胞中提取總DNA,然后通過基于靶向擴增或靶向捕獲的方法獲得mtDNA。本研究基于靶向NGS技術系統全面分析了HCC患者癌組織、癌旁組織、外周血細胞及血漿中的mtDNA突變。測序結果發現,該方法不僅實現了100%的覆蓋度,測序質量Q30高于90%,且mtDNA的富集效率即mtDNA比對率>50%,同時可以準確地檢測突變頻率低至2%的mtDNA突變。此外,還通過4種配對樣本的數據準確識別了與HCC相關的mtDNA突變,說明該方法有效、可行,且能夠準確地檢測血漿中的低頻突變。
目前大多研究集中于檢測HCC患者組織樣本或外周血細胞中的mtDNA突變[14],鮮見檢測cf-mtDNA突變的研究。有研究使用單分子實時(single-molecule real-time,SMRT)測序技術對來自8例癌癥患者的19例組織樣本和9例血漿樣本的mtDNA進行了突變分析,結果在血漿中幾乎檢測不到腫瘤特異性mtDNA突變[15]。本研究在血漿中檢測到了較癌組織中明顯增多的mtDNA突變,說明血漿樣本能夠克服腫瘤組織異質性。此外,在血漿中觀察到的mtDNA突變頻率與癌組織中的mtDNA突變頻率無明顯差異,提示血漿樣本很大程度上可反映腫瘤細胞中的mtDNA突變特征,可代替癌組織樣本進行mtDNA突變分析。在HCC患者癌組織與血漿樣本中還觀察到mtDNA拷貝數存在顯著性差異,為后續的實驗及數據分析提供了重要的理論依據。值得注意的是,在實驗和數據分析過程中,要根據不同的樣本類型設置不同參數,例如探針的使用量、探針的長度、擴增次數,數據質控條件等。
綜上所述,本研究使用基于靶向NGS技術在HCC患者血漿中檢測到腫瘤來源的mtDNA突變,同時還準確地檢測出血漿樣本中的低頻突變;血漿中的mtDNA有望成為HCC的潛在診斷或預后評估生物標志物,可作為組織活檢的有力補充。但本研究樣本量較小且未分析血漿中mtDNA突變與HCC生存的關系。因此,該法的臨床實用性有待在大型HCC患者隊列中進一步評估。

