基于植物轉座子的分子標記研究進展
何虎翼, 樊吳靜, 唐洲萍, 楊 鑫, 李麗淑, 譚冠寧, 何新民
(廣西農業科學院經濟作物研究所,廣西南寧 530007)
1 植物轉座子
植物轉座子是廣泛分布于植物基因組中的一類能從染色體的一個位置跳躍到另一個位置或者另一條染色體的DNA序列。根據轉座機制的不同可分為DNA轉座子和反轉錄轉座子。DNA轉座子是一類由DNA介導,通過“剪切-復制”進行轉座的可移動元件,包括微型反向重復轉座元件(miniature inverted repeat transposable elements,簡稱MITEs)和Helitron,其插入位點多態性豐富、拷貝數多。MITEs兩端由末端反向重復序列(terminal inverted repeat,簡稱TIR)和靶位點重復序列(target site duplication,簡稱TSD)組成,富含 T/A,其轉座主要依靠自主型DNA轉座子的轉座酶。反轉錄轉座子是一類廣泛分布于植物基因組的由RNA介導,通過“復制-粘貼”進行轉座的可移動遺傳因子。根據是否具有長末端重復元件(long terminal repeat,簡稱LTR)又可分為LTR反轉錄轉座子和非LTR反轉錄轉座子,其中LTR反轉錄轉座子包括2種類型——Ty1-copia和Ty3-gypsy,非LTR反轉錄轉座子包括長散在核重復序列(long interspersed nuclear element,簡稱LINE)和短散在核重復序列(short interspersed nuclear element,簡稱SINE)。植物轉座子具有拷貝數豐富、插入位點多態性、高度異質性等特點,非常適合用來開發分子標記。
2 基于植物轉座子的分子標記類型
目前基于反轉錄轉座子的分子標記技術主要包括序列特異擴增多態性(sequence-specific amplification polymorphisms,簡稱S-SAP)、反轉錄轉座子序列間擴增多態性(inter-retrotransposon amplified polymorphism,簡稱IRAP)、反轉錄轉座子-微衛星擴增多態性(retrotransposon-microsatellite amplified polymorphism,簡稱REMAP)、基于反轉錄轉座子的插入多態性(retrotransposn-based insertion polymorphisms,簡稱RBIP)。S-SAP標記技術來源于擴增片段長度多態性(amplified fragment length polymorphism,簡稱AFLP),由限制性內切酶Pst1和Mse1消化基因組DNA,通過連接接頭進行預擴增和選擇性擴增,在同一位置片段的有無有判斷其多態性。不僅可以檢測反轉錄轉座子內部變異,還可以用來檢測插入位點的多態性[1]。IRAP標記技術是利用基于反轉錄轉座子LTR區域設計的引物,通過PCR擴增出相鄰同一家族的反轉錄轉座子成員間的片段,從而檢測反轉錄轉座子插入位點間的多態性[2]。REMAP標記技術是利用基于反轉錄轉座子LTR區域和微衛星序列設計的引物,通過PCR擴增出反轉錄轉座子與相鄰微衛星間的片段,從而檢測反轉錄轉座子與簡單重復序列間的多態性[2]。RBIP標記技術是以PDR1反轉錄轉座子設計引物,篩選基因組文庫,通過測序獲得該反轉錄轉座子及兩側翼的堿基序列,再通過設計特異引物組合A/E或C/E產生PCR擴增產物[3]。這些基于轉座子的分子標記技術普遍具有共顯性、高多態性和高重復性等特征,已廣泛應用于遺傳多樣性分析、變異鑒定和遺傳連鎖圖譜構建等方面。
3 基于植物轉座子的分子標記應用
3.1 遺傳多樣性分析
基于PDR1的S-SAP標記技術顯示豌豆屬可分為Pisumfulvum、Pisumabyssinicum、Pisumspp.等3組[4]。基于Tps12、Tps19和PDR1的S-SAP標記揭示了55份不同豌豆種間和種內的關系[5]。基于Tms1元件的S-SAP標記技術非常適合用來研究紫花苜蓿的遺傳多樣性[6]。基于BARE-1、BAGY-1、BAGY-2、Sabrina、Nikita、Sukkula等6個反轉錄轉座子家族的S-SAP分子標記可以用來比較大麥的遺傳多樣性[7]。基于Vine-1的S-SAP擴增能有效分析葡萄的遺傳多樣性[8]。基于Ty1-copia 類反轉錄轉座子的S-SAP標記技術可用于檢測不同生態型黃瓜材料間的多態性[9]。利用不同酶切組合S-SAP引物建立了適用于糯玉米的DNA圖譜[10]。利用10個S-SAP引物組對28份柿屬基因型種質進行遺傳多樣性分析,發現MseI與反轉錄轉座子引物組擴增性能最優[11]。基于反轉錄轉座子的25對S-SAP引物將28份柿屬植物聚為2組[12]。利用S-SAP標記技術檢測短芒大麥突變系種群的遺傳多樣性,發現種群遺傳變異主要存在于種群內[13]。利用基于楊梅Ty1-copia類反轉錄轉座子LTR序列設計的18個S-SAP引物將48份楊梅試材分為3個組,分組結果與地域來源基本一致[14]。
基于2個不同類copia反轉錄轉座子的IRAP標記技術將克萊門柚分成北非和西班牙2組,分組結果與地理來源基本一致[15]。利用基于反轉錄轉座子IRAP標記技術可以將59份烤煙品種分為14個類群[16]。為分析茄子遺傳多樣性,根據馬鈴薯和煙草反轉錄轉座子LTR區域設計引物并建立了茄子IRAP分子標記體系[17]。用基于馬鈴薯和煙草反轉錄轉座子LTR區域設計的2組引物將34份茄子材料分為3大類群[18]。通過優化各影響因素,建立了穩定的香菇IRAP分子標記技術體系[19]。與ISSR方法相比,用IRAP標記分析煙草種質資源遺傳多樣性的品種間遺傳相異系數和多態性比率更高[20]。利用基于小麥反轉錄轉座子Wis2-1A的IRAP標記技術可將21個小麥品種聚成6大類群[21]。沈玉英等利用基于梅Ty1-copia反轉錄轉座子的IRAP標記技術體系研究了梅的遺傳多樣性和親緣關系[22]。杜曉云等利用IRAP標記技術揭示了部分柿屬種間倍性進化關系[23]。基于Ty1-copia類反轉錄轉座子的IRAP標記技術也適用于檢測火龍果IRAP多態性[24]。利用IRAP標記可以區分36份小麥材料的抗性強弱關系[25]。李慧等利用IRAP標記技術研究顯示,湖北省建始縣的“關口葡萄”與歐美雜交雜種親緣關系最近,其來源可能與“尼加拉”和“白香蕉”有關[26]。崔博文等利用29條IRAP引物將12份馬尾松種質分為3類,并構建了DNA指紋圖譜[27]。王燕等利用IRAP標記技術將48份柿屬植物區分為野生君遷子居群、柿品種和浙江柿種質3組[28]。賈春平等基于4個稻屬反轉錄轉座子的LTR區域設計引物,用IRAP標記方法分析新疆粳稻種質資源的遺傳多樣性,發現多態性比率為93.4%的Houba/Tos5/Osr13引物最適合于DNA指紋圖譜數據庫構建,以0.55為閥值將37份新疆粳稻品種(系)分為6大類群[29]。10個IRAP引物可將49份江西柿屬種質資源分為柿、油柿、野柿3種[30]。
沈玉英等利用REMAP標記技術將24個梅品種聚為3組[31]。通過Ty1-copia類反轉錄轉座子LTR保守區域和ISSR設計引物,王東等建立了甜瓜屬REMAP分子標記技術體系[32]。為鑒定中國水仙種質資源遺傳多樣性,林曉紅等建立了優化的REMAP技術體系[33]。馬忠友等建立了一種基于MITEs的分子標記方法,可廣泛應用到水稻及其他植物上[34]。利用AhMITE轉座子標記技術可以鑒定花生栽培種及高世代材料的親緣關系[35]。基于植物轉座子的分子標記在遺傳多樣性中的應用詳見表1。
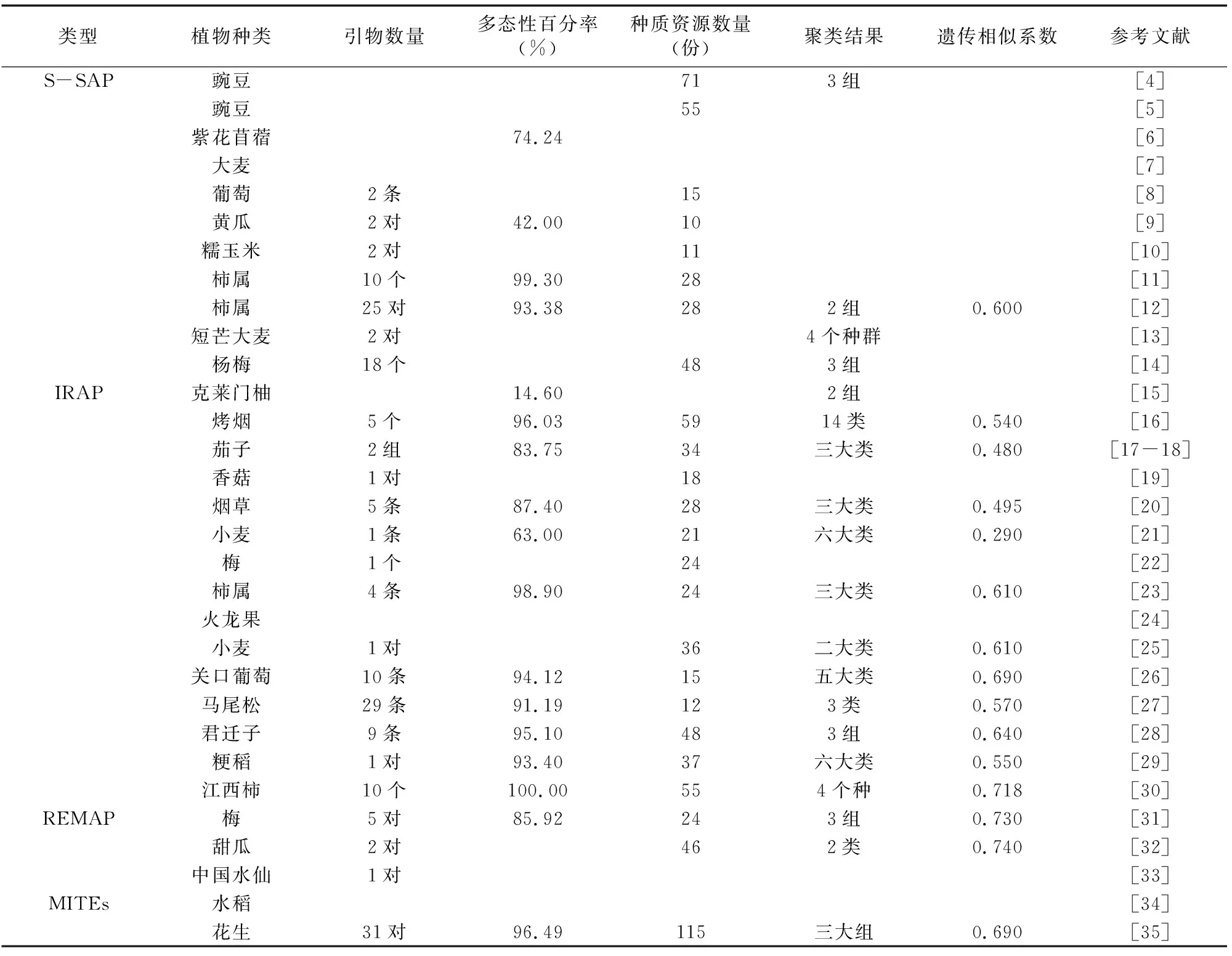
表1 基于植物轉座子的分子標記在遺傳多樣性中的應用
3.2 變異鑒定
利用S-SAP標記技術發現富士蘋果芽變品種產生可能與反轉錄轉座子的插入有關[36]。利用基于蘋果Ty1-copia類反轉錄轉座子建立S-SAP標記技術可以區分元帥蘋果芽變[37]。一個基于蘋果查爾酮合成酶基因啟動子的特異性片段可以用來鑒定芽變材料發生的性狀變異[38]。何平等用7對基于反轉錄轉座子的S-SAP標記引物將15個蘋果芽變品種區分為嘎拉系、元帥系和富士系[39]。基于普通歐柑特異片段的S-SAP標記技術可鑒別青甌柑品種[40]。
基于反轉錄轉座子的單引物IRAP-PCR可以有效建立33份柿屬植物基因型的DNA指紋圖譜,可以區分芽變品種[41]。基于反轉錄轉座子序列開發的分子標記可用來鑒定族毛麥染色質,提高小麥-族毛麥易位系的利用價值[42]。基于蘋果反轉錄轉座子LTR保守區域設計引物的IRAP標記技術可以鑒定部分芽變品種[43]。通過利用IRAP引物鑒定元帥蘋果和富士蘋果無性系芽變,揭示轉座插入和反轉錄轉座子間重組是蘋果無性系變異的重要機制[44]。利用IRAP標記技術構建的蘋果指紋圖譜,可以作為短枝和著色芽變鑒定的依據[45]。基于反轉錄轉座子引物IRAP標記可鑒定遺傳背景高度相似的磨盤柿芽變[46]。林曉紅等利用IRAP標記技術研究中國水仙自然變異體,發現轉座子插入和反轉錄轉座子間重組是中國水仙變異的重要機制[47]。IRAP引物標記可用于分析金棗柿實生后代的遺傳變異[48]。利用基于旱酥梨及其紅皮芽變Ty1-copia反轉錄轉座子的IRAP標記技術可以建立DNA指紋圖譜[49]。
成文革等用REMAP標記技術分析吉生羊草體細胞無性系變異,發現其遺傳相似性系數范圍比ISSR技術要大[50]。馮榮芳等用基于反轉錄轉座子的REMAP標記技術證實紅翎菜科海藻存在較高程度變異[51]。基于AhMITE1轉座子標記技術可以有效鑒定花生F1代雜種的真實性[52]。利用AhMITE1轉座子標記可對栽培種花生F1代雜交種子的真偽進行鑒定[53]。基于植物轉座子的分子標記在變異鑒定中的應用詳見表2。
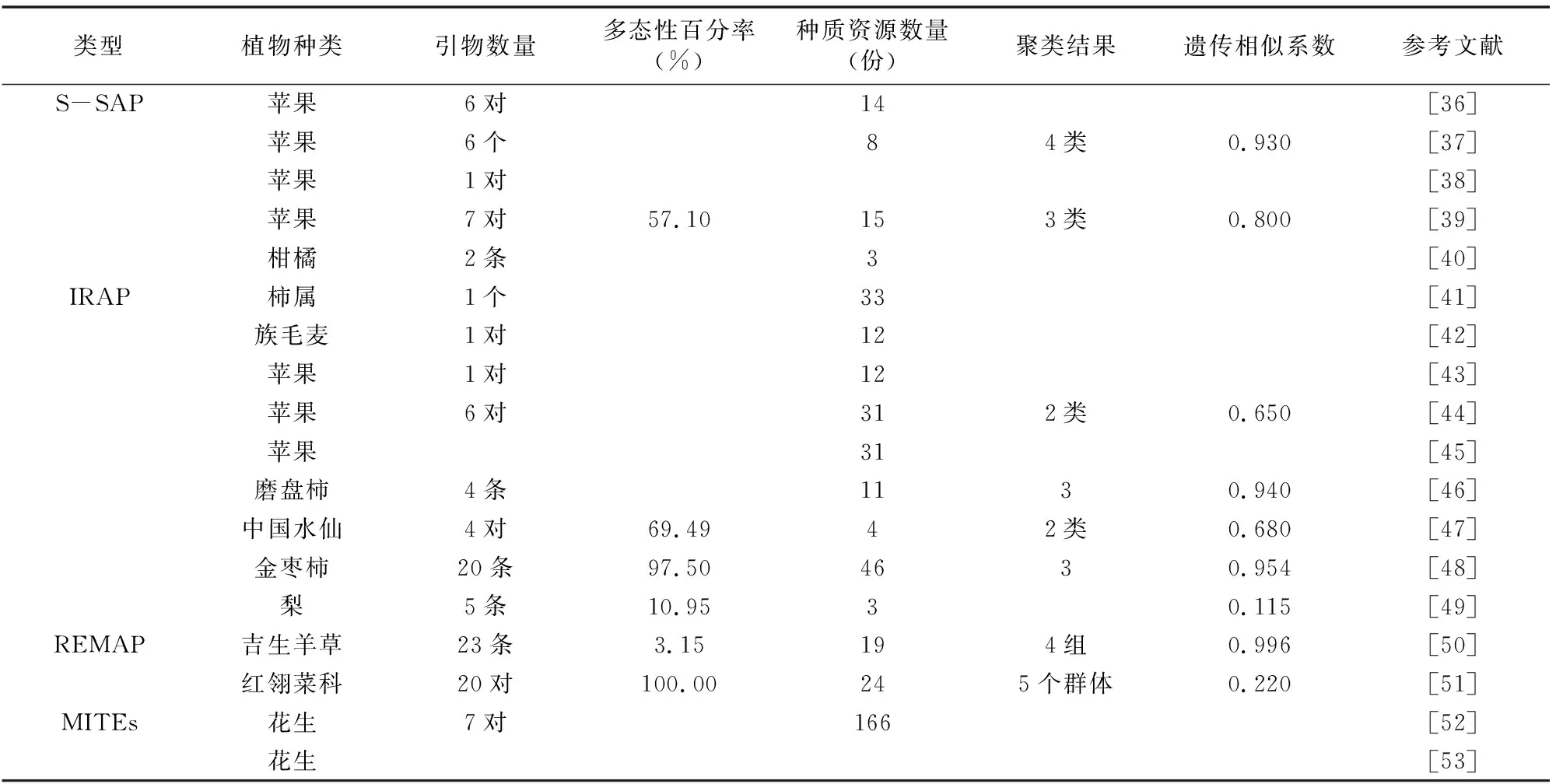
表2 基于植物轉座子的分子標記在變異鑒定中的應用
3.3 遺傳連鎖圖譜構建
基于Ty1-copia反轉錄轉座子LTR區域設計的引物可以分析棉屬不同種,構建棉花高密度遺傳圖譜[54]。王利英等根據大麥和煙草反轉錄轉座子Bare-1、Tto1的LTR區域設計引物,建立了茄子IRAP和REMAP分子標記技術體系[55]。用基于煙草反轉錄轉座子Tnt1的LTR區域設計的引物可將32個百合品種完全區分[56]。李芳等利用基于蘿卜Ty1-copia 反轉錄轉座子的IRAP標記技術構建了14個蘿卜品種指紋圖譜[57]。在基于MITEs結構特征開發的119條引物中,21個MITEs位點間多態性(inter-MITE polymorphism,簡稱IMP)標記可以整合定位到1個SSR標記的遺傳連鎖圖譜上[58]。基于植物轉座子的分子標記在遺傳連鎖圖譜構建中的應用詳見表3。
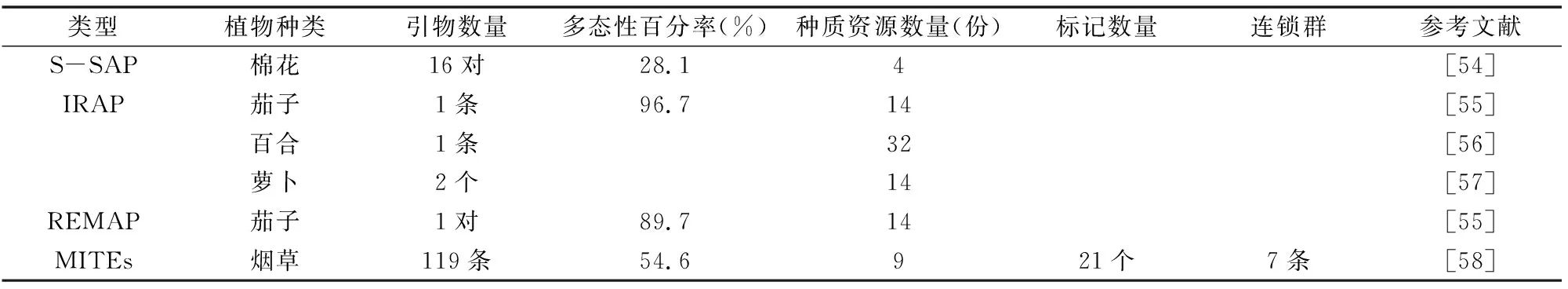
表3 基于植物轉座子的分子標記在遺傳連鎖圖譜構建中的應用
4 結論
植物轉座子是廣泛分布于植物基因組中的一類可移動元件,可分為DNA轉座子和反轉錄轉座子。植物轉座子拷貝數豐富、插入位點多態性、高度異質性等特點,非常適合用來開發分子標記。本研究在了解國內外研究現狀的基礎上,分析基于植物轉座子的S-SAP、IRAP、REMAP、RBIP等分子標記差異,歸納了這些標記在遺傳多樣性分析、變異鑒定以及遺傳連鎖圖譜構建等方面的應用。隨著越來越多植物全基因組序列的發布,在全基因組水平上鑒定植物轉座子、高通量開發基于植物轉座子的分子標記將成為可能。

