利用全局轉錄調控工程提高菌株耐受性研究進展
郭學武,張 玉,陳葉福,肖冬光
(工業發酵微生物教育部重點實驗室,天津市工業微生物重點實驗室,天津科技大學生物工程學院,天津 300457)
微生物在環境變化過程中的反應能力對于其在自然環境下的生存至關重要,因為其可能經常處于熱或冷、極端 pH、滲透壓改變、營養饑餓和有毒化合物的極端環境下.同樣,在工業生產中微生物也會面臨極端環境,例如,通過利用木質纖維素水解產物作為碳源生產生化物質、生物燃料以及生物材料,木質纖維素水解產物(呋喃衍生物、酚類化合物和有機酸)和抑制產物(乙醇、丁醇和異丁醇)中的有毒物質會顯著抑制微生物發酵,因此嚴重阻礙了工業規模的生物煉制[1-3].另外,當產物本身是疏水性并且有細胞毒性時,微生物細胞本身會受到產物的壓力,在這些壓力下,產物的生成途徑不能正常工作,導致生產效率降低.盡管維持細胞內環境的穩定可以確保細胞內適當的穩態,但是當細胞外環境改變破壞酶的最佳活性和代謝使細胞結構不穩定時,也可以造成細胞內環境產生波動,導致生產力和微生物活性降低.因此,在脅迫條件下,研究者們不斷尋找新的方法以提高微生物的耐受性以及保持代謝活性并促進其高效生產.
近年來,提高菌株的耐受性成為人們的研究熱點,廣泛用于提高耐受性的方法為過表達微生物耐性相關基因如溶劑外排泵和熱休克蛋白等相關內源或外源基因、長期適應性進化、人工誘變或基因組改組等[4-7]以提高微生物對藥物、滲透壓等各種壓力條件的耐受性.Patnaik等[8]通過全基因組重排的方法提高了乳酸菌(Lactobacillu)的耐酸性,使乳酸的產量提高了3倍;Fiocco等[9]通過過表達熱激蛋白(sHSP)改善了胚牙乳桿菌(Lactobacillus plantarum)的抗寒性,同時發現菌株對乙醇和丁醇的耐受性也得到提高;Borden等[10]通過在丙酮丁醇梭菌(Clostridium acetobutylicum)ATCC824中過表達轉錄調節因子CAC1869,發現菌株的丁醇耐受性提高了 81%;Tomas等[11]在硫解酶啟動子下過表達蛋白 groESL提高了丙酮丁醇梭菌(Clostridium acetobutylicum)ATCC824的丁醇耐受性;Park等[12]通過突變含鋅指基因的人工轉錄因子,篩選出了具有耐熱性的大腸桿菌(E.coli);Alper 等[13]通過σ因子的全局轉錄調控工程(σ70),使大腸桿菌對乙醇的耐受質量濃度高達60,g/L,并且能夠同時耐受十二烷基磺酸鈉(SDS);Gorsich 等[14]在ORFs缺失突變文庫中篩選出了具有糠醛耐受性的釀酒酵母.但在大多數情況下,因為抗脅迫性是一個復雜的多基因表型,通過簡單地過表達某些功能基因或途徑,雖然在一定程度上有效,但不一定能夠達到所需的耐受水平.盡管長期的適應性進化和人工誘變或基因組改組方法沒有這樣的限制,但是非常耗時且費力,并且所得到的表型難以轉化為新的菌株.而且,這些方法的固有缺點是獲得所需表型的總體具有隨機性,導致獲得所需耐受突變體的可能性降低.為打破以上局限,人們嘗試并建立了許多依賴特定的轉錄調控因子或 DNA結合序列改造微生物耐受性的方法,但由于仍局限在某一或特定基因簇中而達不到所期望的效果[15-17].
由于細胞能夠通過調整自身全局轉錄機制的某些組件,從而精細調節自身成百乃至上千個基因,基于該理論建立了全局轉錄調控工程(global transcription machinery engineering,gTME)的方法能突破以上限制.gTME是一種全新的能在整體水平上改變細胞基因組轉錄而獲得目的細胞表型的定向進化方法,能在全局水平上強化微生物細胞的目標性能,是在基因和細胞水平上改造微生物細胞的新途徑[18].幾種轉錄因子包括含鋅指基因的人工轉錄因子[19-21]、σ因子[13,22]、Spt15[18]、H-NS[23]、Hha[24]和 cAMP[25-26]都是通過全局轉錄工程進行修飾,以改善菌株耐受性及獲得其他優良表型.
gTME 中研究較多的是σ因子,σ因子是 DNA依賴型 RNA 聚合酶的活性中心,與α,2ββ′ω亞基單元(RNA 聚合酶核心酶)構成全酶,參與胞內所有RNA的合成,其功能如圖 1[27]所示.σ因子能夠特異性地識別基因啟動子的-35區和-10區,并激活轉錄過程的起始.σ因子全局轉錄調控工程的實施策略如圖2所示.隨著gTME方法的發展,目前已經成功建立了利用轉錄因子工程提高菌種耐受各種抑制劑的方法,在提高菌種耐受性方面取得了一定的進展.本文僅就全局轉錄調控工程提高菌株耐受性的研究包括乙醇耐受性、有機溶劑耐受性、耐酸性、氧化應激耐受性以及耐糖性進行綜述.
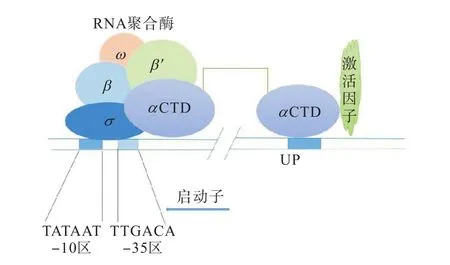
圖1 全局轉錄調控因子σ的功能示意圖Fig. 1 Function diagram of global transcriptional regulatory factor σ
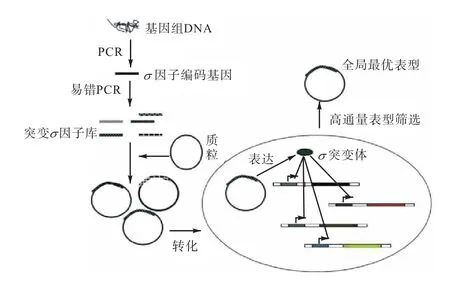
圖2 σ因子全局轉錄調控工程的實施策略Fig. 2 Implementation strategy of global transcription regulation project with factor σ
1 提高乙醇耐受性
生物的代謝途徑呈網絡體系,細胞中基因與表型之間并非線性對應關系,細胞的特定表型往往由多個基因控制,因而只有在基因組水平上同時對控制目的表型的多個基因進行修飾才有可能使得目的表型得到較高的表達,gTME 通過對調控基因轉錄的轉錄元件(σ因子和各種轉錄因子等)進行合理化改組修飾(如易錯 PCR、DNA shuffling 等),使得整個基因組的轉錄發生全局性改變,從而導致許多由多種基因控制的復雜的細胞表型得到改變,最終獲得需要的優良表型.因此,將 gTME應用于大腸桿菌(E.coli),通過對全局轉錄調控因子σ70(起始大部分基因在指數生長細胞中的轉錄)的誘變會改變啟動子的識別,進而從全局轉錄水平上調節轉錄組的轉錄特征[28-29].將具有低、中和高突變率的σ70因子的易錯PCR產物混合在一起,并通過連續傳代培養以提高乙醇的耐受性.獲得σ70的突變株可以耐受 70,g/L的乙醇溶液,而親本只能耐受40,g/L的乙醇溶液.在同一研究中,用不同的方法成功篩選出了耐乙醇和十二烷基硫酸鈉(SDS)的多重耐受性表型[30].Alper等[13]也證明了在大腸桿菌中σ70的全局轉錄調控工程可以提高菌株的乙醇耐受性,使乙醇的耐受性達到 60,g/L,同時菌株的 SDS耐受性也得到了提高,并可以大量生產番茄紅素.cAMP受體蛋白(CRP)在大腸桿菌中調節400多種基因,從而可以在轉錄水平上改善大腸桿菌的乙醇耐受性,Chong等[25]通過改造大腸桿菌的全局轉錄因子CRP以提高乙醇耐受性,在易錯PCR庫中篩選出了乙醇耐受性最好的 CRP突變體E2(M59T),在62,g/L乙醇中的生長速率為0.08,h-1,而對照菌株的僅為 0.06,h-1.隨后,將 M59T突變體整合到基因組中以產生突變體 iE2.當暴露于150,g/L乙醇中時,15,min后iE2的存活率約為12%,而對照菌株的存活率為 0.01%,.對 CRP調節的 444個基因進行的逆轉錄 PCR分析(RT-PCR)揭示了在不含乙醇的情況下iE2中差異表達的基因有203個,而 92個基因在乙醇脅迫時顯示差異表達.這些基因屬于中樞中間代謝(aceE、acnA、sdhD、sucA)、鐵離子運輸(entH、entD、fecA、fecB)和一般應激反應(osmY、rpoS)等多種功能群.全局轉錄調控工程不只應用到大腸桿菌中,在其他細菌中也同樣適用.Tan等[31]在運動發酵單胞菌中通過隨機誘變全局轉錄因子RpoD蛋白(σ70),從易錯 PCR庫中篩選出了耐乙醇的突變菌株,培養54,h后殘糖量為0.64%,,而對照菌株的殘糖量接近 5.43%,;在 9%,乙醇壓力下,突變菌株在30~54,h時乙醇產量為 13.0~14.1,g/L,對照菌株僅為 6.6~7.7,g/L.其研究結果表明,運動發酵單胞菌中RpoD在提高乙醇耐受性方面發揮了重要作用,通過 gTME操縱 RpoD可以為復雜表型菌株的改良提供借鑒.
對于較復雜的真核微生物釀酒酵母的轉錄機制而言,gTME用于靶向RNA聚合酶Ⅱ轉錄因子D的兩個組成部分,即 TATA結合蛋白復合體的成分SPT15和 TATA相關因子 TAF25.在此基礎上構建了兩個易錯 PCR基因文庫,并在葡萄糖和乙醇復合壓力下進行篩選.研究[18]發現:SPT15突變文庫中乙醇耐受性提高的突變體SPT15-300中有3個氨基酸發生突變,在 6%,乙醇溶液和 100,g/L葡萄糖溶液中生長 20,h后,生長速度與野生型菌株相比提高了 13倍;在 10%~20%,乙醇體積分數下,突變體的乙醇耐受性也有顯著提高.SPT15是酵母細胞存活必需的轉錄因子,通過結合 RNA聚合酶Ⅱ和十幾種基本的轉錄因子,可以轉錄基因組中大部分 mRNA,通過代謝工程方法也可以同時改變細胞中多個基因的轉錄水平.Yang等[32]應用類似的方法篩選出了兩株乙醇耐受性提高的釀酒酵母菌株,在生物反應器中用 20%,葡萄糖發酵 24,h 產生的乙醇比對照菌株提高 25%,,乙醇的產量和生產率也大大提高,分別由原來的0.31,g/g 和 2.6,g/(L·h)提高到 0.39,g/g 和 3.2,g/(L·h).由此研究表明,gTME篩選出的菌株不但增強了乙醇耐受性,還提高了乙醇產量.趙心清等[33]對釀酒酵母負責脅迫相關基因轉錄的 SAGA復合體成分 SPT3編碼基因進行易錯 PCR隨機突變,篩選得到了 1株在 10%,乙醇溶液中生長較好的突變株 M25.該突變株在 125,g/L葡萄糖中進行乙醇發酵時,最終乙醇的產量比對照菌株提高了11.7%,.
全局轉錄調控工程可通過改變全局轉錄調控因子對多基因控制的表型進行有效的系統工程改造,而乙醇耐受性正是受多基因調控的.因此,全局轉錄工程是獲得乙醇耐受性高產菌株十分有效的方法之一.
2 提高有機溶劑耐受性
在全細胞生物催化中,如果存在不溶或難溶于水的底物或產物,通常采用非水系統(如有機溶劑)來促進疏水底物和(或)產物的溶解[34],然而有機溶劑對于大多數微生物是有毒的.這些有機溶劑通過摻入細胞膜脂質與膜結合使蛋白質變性,破壞膜的基本功能[35],導致細胞死亡并降低全細胞生物催化效率.據報道[36]大腸桿菌幾乎不耐受log,P值低于3.4的有機溶劑,例如,即使體積分數低至 0.1%,,甲苯對大腸桿菌也有毒性[37].因此,在含水/有機溶劑兩相體系中開發工業應用的有機溶劑耐性(organic solvent tolerance,OST)微生物是至關重要的.
Matsui等[38]通過對有機溶劑耐受性的釀酒酵母進行分析,在轉運蛋白編碼基因的啟動子區中發現了轉錄調控因子 Pdrlp發生了突變;隨后通過對轉錄因子PDR1進行單點突變,成功篩選出了具有有機溶劑耐受性的菌株.該菌株能夠在YPD培養基/異辛烷兩相系統中將 3-氧代丁酸丁酯還原成 3-羥基丁酸丁酯.大腸桿菌中涉及 OST的幾個基因(例如glpC、purR和manXYZ)與葡萄糖代謝有關,因此有學者對OST所必需的碳水化合物代謝的調控因子進行了研究.在正己烷或正己烷與環己烷混合物的壓力下篩選大腸桿菌的 7個敲除突變體(acrA、acrB、cra、crp、cyaA、fnr和mlc),發現腺苷酸環化酶(CyaA)和全局調控因子cAMP受體蛋白(CRP)的敲除使得突變體增強了OST,這兩種耐受性突變體顯示出細胞內己烷濃度降低和對有機溶劑相的附著減少[39].Basak等[40]通過全局轉錄調控因子CRP以提高大腸桿菌的OST,選擇甲苯作為有機溶劑壓力,通過易錯PCR產生突變的crp基因,從隨機誘變文庫中富集篩選出了甲苯耐受突變體 M2,在 0.23%,甲苯中 M2的生長速率為 0.51,h-1,而野生型菌株的生長被完全抑制.與野生型相比,M2在其他有機溶劑如正己烷、環己烷和對二甲苯中也表現出優異的生長性能.Zhang等[41]在環己烷的壓力下,篩選 rpoD突變文庫,旨在篩選出全局轉錄調控因子σ70突變體有機溶劑耐受性提高的大腸桿菌,結果顯示含有σ70突變體 C9的大腸桿菌 JM109菌株具有耐受 69%,環己烷的能力,篩選出OST的大腸桿菌可以潛在地應用于非水相生物催化和生物燃料的生產.用 2D-PAGE檢測攜帶 C9大腸桿菌 JM109菌株產生的總蛋白質差異,并且在不同的溶劑壓力下,204個高豐度蛋白質顯示出超過兩倍的差異,其研究還表明了幾個基因(gapA、sdhB、pepB和dppA)在增強大腸桿菌 OST方面起著關鍵作用,主要涉及在溶劑脅迫下維持較高的細胞內能量水平.以上研究說明全局轉錄調控工程是用于增強OST表型工程菌株的有效方法之一.
3 提高耐酸性
某些生物催化過程在低 pH下,特別是在有機酸的生產過程中或利用木質纖維素酸水解產物生產生物燃料過程中,細胞的活力會逐漸下降.因此,篩選低 pH或耐酸突變體在工業生產中具有重要的意義[42-45].此前的研究[46-48]發現,大腸桿菌在酸壓力下應激反應的關鍵調控因子之一是受CRP調控的σ因子 RpoS.這些發現表明,對自身 CRP的修飾可能會改變大腸桿菌在低 pH下的耐受性.Basak等[49]試圖從轉錄水平改善菌株耐酸性,采用易錯 PCR方法突變大腸桿菌的全局轉錄調控因子 CRP,根據其生長性能,從隨機誘變文庫中篩選出耐酸突變體 AcM1.在pH 4.2時,AcM1的生長速率(0.113,h-1)與對照菌株(0.062,h-1)相比有大幅度增加,并且該菌株在48,℃時也具有較好的耐熱性.實時定量 PCR結果顯示,負責細胞耐酸性的關鍵酶——谷氨酸脫羧酶在AcM1突變體中的活性比親本菌株提高了 2倍,AcM1突變型CRP與Ⅰ型、Ⅱ型和Ⅲ型CRP依賴型啟動子的差異性結合表明,對自身 CRP的修飾可能導致全局轉錄水平的改變.
Klein-Marcuschamer等[22]應用 gTME技術對植物乳桿菌的σ因子進行改造,發現僅突變了σ因子中的1個氨基酸位點(Q345K)就明顯提高了植物乳桿菌對乳酸和高濃度鹽的耐性.張力[50]基于細胞全局轉錄水平,采用融合 PCR技術獲得 RNA聚合酶(RANP)的首要亞基σ因子的表達序列,并采用易錯PCR技術引入突變,獲得轉錄突變庫,篩選獲得乳酸耐受性提高的優勢菌株 LA5.由于氨基酸的替換及C末端27個氨基酸的缺失,導致了σ因子的空間結構發生了變化,產生了一定的調控效果,提高了其在高酸環境下的生長速度.
Gao等[51]采用隨機插入缺失鏈交換誘變(RAISE)的三步法突變大腸桿菌全局調節因子Sigma D(RpoD)以改善其耐酸性.從隨機誘變文庫中篩選出了耐酸突變體Ⅶ,其在pH低至3.17時的生長速率(0.22,h-1)比親本菌株(0.15,h-1)高得多.為了更好地理解RpoD對調節網絡的整體影響,對其進行的轉錄組學分析表明,除了耐酸性外,大腸桿菌中有 95個基因(2.1%,)被誘導,178個(4.0%,)基因被抑制,包括對海藻糖生物合成、核苷酸生物合成、碳代謝以及氨基酸利用;還包括主要調控因子(ArcA、EvgA、HNS和 RpoS)和基因/操縱子特異性轉錄因子(GadX、GadW、AppY、YdeO 和 KdgR).這些結果更好地說明了 RpoD在大腸桿菌的生長階段充當了全局轉錄調控因子.
Tanaka等[52]發現 Haa1(釀酒酵母中參與適應弱酸的轉錄激活因子)的過表達導致了乙酸耐受性的提高,Haa1的誘導削弱了參與乙酸進入細胞的基因表達或者增加了乙酸輸出細胞的基因表達.Masuda等[53]通過響應調節因子 EvgA的過表達可以在細胞生長指數階段賦予大腸桿菌耐酸性,在 LB培養基(pH 2.5)中處理 1,h后,EvgA高表達菌株的存活率比對照提高了 103~104倍,過表達 EvgA 和敲除EvgA大腸桿菌的微陣列分析顯示,EvgA直接或間接調節至少 37個基因,其中包含結構基因gadABC和hdeAB以及轉錄調節基因ydeO、yhiE和gadX.ydeO的過表達增強了菌株的耐酸性,而ydeO的缺失能部分降低菌株的耐酸性.
4 提高氧化應激耐受性
氧化應激是由活性氧(ROS)和非自由基產生和清除之間不平衡引起的,如羥基自由基(·OH)、超氧陰離子(O2??)、過氧化氫(H2O2)和單線態氧的氧化程度超過氧化物的清除[54].ROS會對細胞成分(DNA、脂質和蛋白質)以及細胞氧化還原平衡造成嚴重損害[55],因此篩選對 ROS具有較高耐受性表型的菌株對于工業規模的經濟生產是非常重要的.Zhao等[56]利用全局轉錄調控機制在兩個質粒突變文庫(spt15和taf25分別編碼TBP 和Taf(Ⅱ)25)中成功地篩選出了兩株由 H2O2引起的氧化應激耐性的酵母菌株.在溫和H2O2壓力下(≤3.5,mmol/L),氧化耐受菌株的細胞生長性能與 H2O2濃度呈正相關.發酵結果表明突變株 taf25-3與對照菌株相比延滯期較短,taf25-3對 H2O2誘導的氧化應激適應能力提高,發酵效率提高.這表明一般轉錄因子(spt15和taf25)中的幾個氨基酸被取代可以修飾細胞氧化防御系統并提高釀酒酵母的抗氧化能力.CodYst是調節鏈球菌代謝網絡的全球轉錄調節因子,Lu等[57]構建了codYst缺失的突變體,Wang等[58]比較了嗜熱鏈球菌ST2017及codYst缺陷型菌株中在不同H2O2應激下的細胞存活率.結果顯示:在 2,mmol/L H2O2的溫和氧化脅迫下,野生型菌株和codYst缺陷型突變體的細胞存活率相似,并且與未處理的菌株相比僅減少了1個數量級.當受到 10,mmol/L H2O2的強烈氧化應激時,codYst缺陷型突變體的細胞存活率急劇下降,比野生型菌株低大約3個數量級,說明全局轉錄因子codYst在維持嗜熱鏈球菌ST2017的氧化應激中具有重要的作用.Basak等[59]通過改造大腸桿菌中的全局調控因子CRP以改善大腸桿菌在氧化應激條件下的耐受性,這些蛋白可以直接或間接調節氧化還原感應調節因子 SoxR和 OxyR等,在 H2O2壓力下通過易錯PCR篩選出了具有氧化應激耐受性的最佳突變體OM3.該突變體菌株可在12,mmol/L H2O2中生長,生長速率為 0.6,h-1,而在此 H2O2濃度下野生型菌株的生長被完全抑制.OM3還具有異丙苯氫過氧化物耐受性和高溫耐受性.細胞內的ROS決定細胞的活性,對活性氧測定發現不管是否在 H2O2的壓力下,OM3菌株中活性氧的濃度都低于野生菌株.Pan等[60]在大腸桿菌中過表達全局轉錄調控因子 IrrE提高了對滲透壓、熱和氧化應激的耐受性.像IrrE這樣的全局轉錄調控因子經常激活多種途徑,其中一些途徑可能不是針對靶向應激耐受表型的特異性途徑.
5 提高耐糖性
肺炎克雷伯氏菌(K.pneumoniae)五碳糖利用性能優良,在木糖生物轉化方面極具開發潛力,但其木糖耐受力較低,阻礙了其工業化應用.本課題組[61]利用易錯 PCR的方法對產 2,3-丁二醇的肺炎克雷伯氏菌(KPG)全局轉錄調控因子σ的編碼基因rpoD進行突變,將突變產物連接 pDK7質粒后轉入親本菌株,構建突變菌株文庫,從突變文庫中篩選出了具有木糖耐受性并高產 2,3-丁二醇的肺炎克雷伯氏菌株kpC.與親本菌株相比,木糖的消耗速率提高了169%,2,3-丁二醇的產量提高了216%,,且親本菌株在48,h之后基本不耗糖,說明對全局轉錄調控因子σ的突變提高了肺炎克雷伯氏菌對木糖的耐受能力.
6 展 望
目前,gTME主要關注天然的σ70因子,只有鋅指蛋白已被用于設計人工調控因子,而大部分的全局轉錄調控因子尚未被開發研究.大腸桿菌本身的 7~14個全局轉錄調控因子中被大量研究的主要是σ70因子,到目前為止,只有 CRP能突變產生不同的表型. 生存在惡劣環境條件下,微生物的外源調節因子構成了可能賦予重組宿主抗性的轉錄調節蛋白庫,對于 IrrE這樣的外源全局調控因子來說,在不同的宿主或者相似的宿主中可能會成為新的全局轉錄調控因子的演化原型.特別是在轉錄組學、蛋白質組學和DNA與蛋白質相互作用(PDI)分析的幫助下,深入剖析由全局轉錄調控工程帶來的轉錄/翻譯譜的變化,會使人們認識到識別和靶向直接負責脅迫耐受表型具體的基因和途徑,進而應用到提高菌株耐性的研究.總之,全局轉錄調控工程方法應該擴展到更多天然的、外源的和人工調控的因子,并結合新的合成生物學的方法,使人們更深刻地了解如何人為調控工業微生物的基因組表達,在工業生產中使微生物在相關的壓力條件下具有更好的適應性.
[1] Attfield P V. Stress tolerance:The key to effective strains of industrial baker's yeast[J]. Nature Biotechnology,1997,15(13):1351-1357.
[2] Shima J,Ando A,Nakamura T. Environmental stress tolerance of yeast:Importance in industrial uses and molecular mechanisms[J]. Nippon Shokuhin Kogyo Gakkaishi,2010,57(6):225-231.
[3] Nicolaou S A,Gaida S M,Papoutsakis E T. A comparative view of metabolite and substrate stress and tolerance in microbial bioprocessing:From biofuels and chemicals,to biocatalysis and bioremediation[J]. Metabolic Engineering,2010,12(4):307-331.
[4] Bajwa P K,Shireen T,D'Aoust F,et al. Mutants of the pentose-fermenting yeastPichiastipitiswith improved tolerance to inhibitors in hardwood spent sulfite liquor[J]. Biotechnology & Bioengineering,2009,104(5):892-900.
[5] Geddes C C,Mullinnix M T,Nieves I U,et al. Simplified process for ethanol production from sugarcane bagasse using hydrolysate-resistantEscherichia colistrain MM160[J]. Bioresource Technology,2011,102(3):2702-2711.
[6] Shi D J,Wang C L,Wang K M. Genome shuffling to improve thermotolerance,ethanol tolerance and ethanol productivity ofSaccharomyces cerevisiae[J]. Journal of Industrial Microbiology & Biotechnology,2009,36(1):139-147.
[7] Tomas C A,Beamish J,Papoutsakis E T. Transcriptional analysis of butanol stress and tolerance inClostridium acetobutylicum[J]. Journal of Bacteriology,2004,186(7):2006-2018.
[8] Patnaik R,Louie S,Gavrilovic V,et al. Genome shuffling ofLactobacillusfor improved acid tolerance[J].Nature Biotechnology,2002,20(7):707-712.
[9] Fiocco D,Capozzi V,Goffin P,et al. Improved adaptation to heat,cold,and solvent tolerance inLactobacillus plantarum[J]. Applied Microbiology and Biotechnology,2007,77(4):909-915.
[10] Borden J R,Papoutsakis E T. Dynamics of genomiclibrary enrichment and identification of solvent tolerance genes forClostridium acetobutylicum[J]. Applied and Environmental Microbiology,2007,73(9):3061-3068.
[11] Tomas C A,Welker N E,Papoutsakis E T. Overexpression of groESL inClostridium acetobutylicumresults in increased solvent production and tolerance,prolonged metabolism,and changes in the cell’s transcriptional program[J]. Applied and Environmental Microbiology,2003,69(8):4951-4965.
[12] Park K S,Jang Y S,Lee H,et al. Phenotypic alteration and target gene identification using combinatorial libraries of zinc finger proteins in prokaryotic cells[J]. Journal of Bacteriology,2005,187(15):5496-5499.
[13] Alper H,Stephanopoulos G. Global transcription machinery engineering:A new approach for improving cellular phenotype[J]. Metabolic Engineering,2007,9(3):258-267.
[14] Gorsich S W,Dien B S,Nichols N N,et al. Tolerance to furfural-induced stress is associated with pentose phosphate pathway genes ZWF1,GND1,RPE1,and TKL1 inSaccharomyces cerevisiae[J]. Applied Microbiology and Biotechnology,2006,71(3):339-349.
[15] Gerber H P,Schaffner W. Transcriptional activation modulated by homopolymeric glutamine and proline stretches[J]. Science,1994,263(5148):808-811.
[16] Kim J S,Kim J,Cepek K L,et al. Design of TATA boxbinding protein/zinc finger fusions for targeted regulation of gene expression[J]. Proceeding of the National Academy of Sciences of the United States of America,1997,94(8):3616-3620.
[17] Park K S,Lee D K,Lee H,et al. Phenotypic alteration of eukaryotic cells using randomized libraries of artificial transcription factors[J]. Nature Biotechnology,2003,21(10):1208-1214.
[18] Alper H,Moxley J,Nevoigt E,et al. Engineering yeast transcription machinery for improved ethanol tolerance and production[J]. Science,2006,314(5805):1565-1568.
[19] Lee J Y,Sung B H,Yu B J,et al. Phenotypic engineering by reprogramming gene transcription using novel artifi-cial transcription factors inEscherichia coli[J]. Nucleic Acids Research,2008,36(16):e102.
[20] Park K S,Lee D K,Lee H,et al. Phenotypic alteration of eukaryotic cells using randomized libraries of artificial transcription factors[J]. Nature Biotechnology,2003,21(10):1208-1214.
[21] Tol N V,Zaal B J V D. Artificial transcription factormediated regulation of gene expression[J]. Plant Science,2014,225(8):58-67.
[22] Klein-Marcuschamer D,Stephanopoulos G. Assessing the potential of mutational strategies to elicit new phenotypes in industrial strains[J]. Proceedings of the National Academy of Sciences of the United States of America,2008,105(7):2319-2324.
[23] Hong S H,Wang X,Wood T K. Controlling biofilm formation,prophage excision and cell death by rewiring global regulator H-NS ofEscherichia coli[J]. Microbial Biotechnology,2010,3(3):344-356.
[24] Hong S H,Jintae L,Wood T K. Engineering global regulator Hha ofEscherichia colitocontrol biofilm dispersal[J]. Microbial Biotechnology,2010,3(6):717-728.
[25] Chong H,Huang L,Yeow J,et al. Improving ethanol tolerance ofEscherichia coliby rewiring its global regulator cAMP receptor protein(CRP)[J]. PLoS One,2013,8(2):e57628.
[26] Zhang H,Chong H,Chi B C,et al. Engineering global transcription factor cyclic AMP receptor protein ofEscherichia coli,for improved 1-butanol tolerance[J]. Applied Microbiology & Biotechnology,2012,94(4):1107-1117.
[27] 喬志新,于群. 全局轉錄調控及其在代謝工程中的應用[J]. 生物技術通訊,2009,20(5):689-691.
[28] Gao G,Tian B,Liu L,et al. Expression ofDeinococcus radiodurans,PprI enhances the radioresistance ofEscherichia coli[J]. DNA Repair,2003,2(12):1419-1427.
[29] Gardella T,Moyle H,Susskind M M. A mutantEscherichia colisigma 70 subunit of RNA polymerase with altered promoter specificity[J]. Journal of Molecular Biology,1989,206(4):579-590.
[30] Santos C N S,Stephanopoulos G. Combinatorial engineering of microbes for optimizing cellular phenotype[J]. Current Opinion in Chemical Biology,2008,12(2):168-176.
[31] Tan F,Wu B,Dai L,et al. Using global transcription machinery engineering(gTME)to improve ethanol tolerance ofZymomonas mobilis[J]. Microbial Cell Factories,2016,15(1):4.
[32] Yang J,Bae J Y,Lee Y M,et al. Construction ofSaccharomyces cerevisiaestrains with enhanced ethanol tolerance by mutagenesis of the TATA-binding protein gene and identification of novel genes associated with ethanol tolerance[J]. Biotechnology & Bioengineering,2011,108(8):1776-1787.
[33] 趙心清,姜如嬌,李寧,等. 利用 SPT3 的定向進化提高工業釀酒酵母乙醇耐受性[J]. 生物工程學報,2010,26(2):159-164.
[34] Heipieper H J,Neumann G,Cornelissen S,et al. Solventtolerant bacteria for biotransformations in two-phase fermentation systems[J]. Applied Microbiology & Biotechnology,2007,74(5):961-973.
[35] Sikkema J,de Bont J A,Poolman B. Mechanisms of membrane toxicity of hydrocarbons[J]. Microbiological Reviews,1995,59(2):201-222.
[36] Inoue A,Horikoshi K. Estimation of solvent-tolerance of bacteria by the solvent parameter logP[J]. Journal of Fermentation & Bioengineering,1991,71(3):194-196.
[37] Sardessai Y,Bhosle S. Tolerance of bacteria to organic solvents[J]. Research in Microbiology,2002,153(5):263-268.
[38] Matsui K,Teranishi S,Kamon S,et al. Discovery of a modified transcription factor endowing yeasts with organic-solvent tolerance and reconstruction of an organicsolvent-tolerantSaccharomyces cerevisiaestrain[J]. Applied & Environmental Microbiology,2008,74(13):4222-4225.
[39] Okochi M,Kurimoto M,Shimizu K,et al. Effect of global transcriptional regulators related to carbohydrate metabolism on organic solvent tolerance inEscherichia coli[J]. Journal of Bioscience & Bioengineering,2008,105(4):389-394.
[40] Basak S,Song H,Jiang R. Error-prone PCR of global transcription factor cyclic AMP receptor protein for enhanced organic solvent(toluene)tolerance[J]. Process Biochemistry,2012,47(12):2152-2158.
[41] Zhang F,Qian X,Si H,et al. Significantly improved solvent tolerance ofEscherichia coli,by global transcription machinery engineering[J]. Microbial Cell Factories,2015,14(1):175.
[42] Morel F,Delmas F,Jobin M P,et al. Improved acid tol-erance of a recombinant strain ofEscherichia coli,expressing genes from the acidophilic bacteriumOenococcus oeni[J]. Letters in Applied Microbiology,2001,33(2):126-130.
[43] Suthers P F,Cameron D C. Production of 3-hydroxypropionic acid in recombinant organisms:US6852517 B1[P]. 2005-02-08.
[44] Vemuri G N,Eiteman M A,Altman E. Effects of growth mode and pyruvate carboxylase on succinic acid production by metabolically engineered strains ofEscherichia coli[J]. Applied & Environmental Microbiology,2002,68(4):1715-1727.
[45] Taherdazeh M J,Karimi K. Acid-based hydrolysis processes for ethanol from lignocellulosic materials:A review[J]. Bioresources,2007,2(3):472-499.
[46] Small P,Blankenhorn D,Welty D,et al. Acid and base resistance inEscherichia coliandShigella flexneri:Role of rpoS and growth pH[J]. Journal of Bacteriology,1994,176(6):1729-1737.
[47] Cheville A M,Arnold K W,Buchrieser C,et al.rpoSregulation of acid,heat,and salt tolerance inEscherichia coliO157:H7[J]. Applied & Environmental Microbiology,1996,62(5):1822-1824.
[48] Henggearonis R. Signal transduction and regulatory mechanisms involved in control of the sigma(S)(RpoS)subunit of RNA polymerase[J]. Microbiology &Molecular Biology Reviews,2002,66(3):373-395.
[49] Basak S,Geng H,Jiang R. Rewiring global regulator cAMP receptor protein(CRP)to improveE. colitolerance towards low pH[J]. Journal of Biotechnology,2014,173(1):68-75.
[50] 張力. 全局轉錄工程選育耐受乳酸產物的基因工程菌研究[D]. 武漢:華中科技大學,2011.
[51] Gao X,Jiang L,Zhu L,et al. Tailoring of global transcription sigma D factor by random mutagenesis to improveEscherichia colitolerance towards low-pHs[J].Journal of Biotechnology,2016,224:55-63.
[52] Tanaka K,Ishii Y,Ogawa J,et al. Enhancement of acetic acid tolerance inSaccharomyces cerevisiaeby overexpression of the HAA1 gene,encoding a transcriptional activator[J]. Applied & Environmental Microbiology,2012,78(22):8161-8163.
[53] Masuda N,Church G M.Escherichia coligene expression responsive to levels of the response regulator EvgA[J]. Journal of Bacteriology,2002,184(22):6225-6234.
[54] Storz G,Christman M F,Sies H,et al. Spontaneous mutagenesis and oxidative damage to DNA inSalmonella typhimurium[J]. Proceedings of the National Academy of Sciences of the United States of America,1987,84(24):8917-8921.
[55] Spirin A S,Lim V I. Hyperoxia results in transient oxidative stress and an adaptive response by antioxidant enzymes in goldfish tissues[J]. International Journal of Biochemistry & Cell Biology,2005,37(8):1670-1680.
[56] Zhao H,Li J,Han B,et al. Improvement of oxidative stress tolerance inSaccharomyces cerevisiaethrough global transcription machinery engineering[J]. Journal of Industrial Microbiology & Biotechnology,2014,41(5):869-878.
[57] Lu W W,Wang Y,Wang T,et al. The global regulator CodY inStreptococcus thermophiluscontrols the metabolic network for escalating growth in the milk environment[J]. Applied & Environmental Microbiology,2015,81(7):2349-2358.
[58] Wang Y,He H Y,Li H H,et al. The global regulator CodY responds to oxidative stress by the regulation of glutathione biosynthesis inStreptococcus thermophilus[J]. Journal of Dairy Science,2017,100(11):8768-8775.
[59] Basak S,Jiang R. EnhancingE. colitolerance towards oxidative stress via engineering its global regulator cAMP receptor protein(CRP)[J]. PLoS One,2012,7(12):e51179.
[60] Pan J,Wang J,Zhou Z,et al. IrrE,a global regulator of extreme radiation resistance inDeinococcus radiodurans,enhances salt tolerance inEscherichia coliandBrassica napus[J]. PLoS One,2009,4(2):e4422.
[61] 郭學武,肖冬光,張玉,等. 一株高木糖耐性肺炎克雷伯氏菌株及其構建方法:201611156954.7[P]. 2017-07-25.

