基于交叉預測的蛋白質交互識別
閔慶凱,蔡松成
(南京航空航天大學 計算機科學與技術學院,江蘇 南京 211106)
1 概 述
蛋白質是生物細胞最重要的組成成分,通過交互作用執行著細胞內多數重要的分子過程。蛋白質交互作用(protein-protein interaction,PPI)的研究以及蛋白質交互網絡的建立是生物信息學研究的重要內容。目前,已有的交互關系數據庫,例如HPRD[1]、BIND[2]、DIP[3]、InAct[4]和MINT[5],均由生物醫學專家通過人工識別的方法從醫學文獻中搜集得到。然而,隨著生物醫學文獻的急劇增加,人工抽取的方法變得越來越不切實際,因此利用計算機信息抽取技術自動地從自然語言文本中抽取PPI成為一項重要的研究內容。
目前,用于PPI抽取的技術主要包括基于詞共現的方法[6]、基于規則的方法[7]和基于統計機器學習的方法[8-9]。基于詞共現的方法通過統計兩個蛋白質名稱在句子中共同出現的概率來判斷是否存在交互關系,這種方法召回率高但很難發現詞典外的PPI[10];基于規則的方法利用模式匹配的思想,可以取得較高的精確率,但泛化能力差,而且通過手動建立規則的方法需要大量的人力物力[11];基于統計機器學習的方法通過將關系抽取問題轉換為分類問題,同時結合自然語言處理方法,較好地解決了上述兩種方法存在的問題,目前廣泛用于PPI的抽取。這類方法又可分為基于特征的方法和基于核函數的方法。其中,基于特征的方法從句子中提取大量的語言學特征,包括詞法、語法和語義等特征來表示關系實例[12],能夠簡單有效地完成關系抽取任務;而基于核函數的方法通過設計核函數代替特征向量內積運算計算PPI間的相似度,具有良好的復合特性,在關系抽取領域也取得了不錯的效果[13]。
上述機器學習方法均基于有監督的思想,語料庫中的句子所包含的實體對及其關系都由人工標注完成,其性能非常依賴于訓練樣本的數量,當訓練語料不足時,關系抽取效果就會大打折扣。但人工標注大規模文本需要耗費大量的人力物力,因此出現了基于遠監督的方法:假設關系知識庫中的一對實體存在某種關系,那么包含這對實體的句子則表達了實體對的這種關系,通過將知識庫中的實體對與文本中的實體進行匹配,啟發式地產生大量的標記數據[14]。遠監督很好地解決了標注數據不足的問題,利用遠監督得到大規模標注文本結合基于特征的方法在PPI抽取上也取得了很好的效果[15-16]。然而,與有監督下人工精確標注的方法相比,遠監督采取的是相對粗糙的匹配方式,得到的標注數據并不總是正確的。如圖1所示,第一個句子確實表達了Michael Jackson和Gary之間的place_of_birth關系,而第二個句子并不能表達這種關系,這種實際上被錯誤標記的句子即被視為標注語料中的噪音,這種噪音會對最終的關系抽取效果造成很大的影響。針對訓練數據中存在的噪音,提出一種交叉預測的方法,并通過人工標注數據進行驗證。
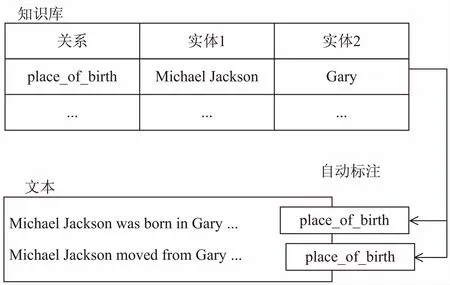
圖1 遠監督的自動標注
2 交叉預測去噪音
該方法以遠監督為基礎,首先搜索大規模醫學文獻獲取包含目標蛋白質對的句子作為原始訓練數據,從中提取特征,構建向量空間模型(vector space model,VSM),將每個句子映射為一個n維的特征向量;然后采用交叉預測識別出訓練數據中的噪音,消除噪音并重新形成訓練數據;最后利用訓練得到的分類器對另一有人工標注的數據進行預測。
2.1 構建特征向量
文中采用向量空間模型來表示文本,每一個句子表示向量空間中的一個向量,選取句子中重要的一元詞作為特征項,具體處理方法為:
(1)對句子進行分詞,去除無意義的標點符號以及停止詞;
(2)選取句子中兩個蛋白質之間的單詞作為特征項;
(3)選取第一個蛋白質左邊2個單詞和第二個蛋白質右邊2個單詞,作為特征項。
所得到每一個不同的特征項對應于向量空間中的一個維度,若句子中出現了該特征項,那么句子向量的對應維設為1,否則為0。
2.2 遠監督
遠監督:如果兩個實體之間存在某種關系,那么包含這兩個實體的句子就表達了這種關系。文中采用的知識庫分為兩部分:有交互關系的蛋白質對和無交互關系的蛋白質對,基于遠監督得到訓練數據的步驟如下:
(1)將知識庫中的蛋白質對與大規模醫學文本中的蛋白質進行匹配,篩選出所有包含知識庫中蛋白質對的句子;
(2)所有包含有交互關系的蛋白質對的句子標注為訓練數據中的正例即有交互關系;
(3)所有包含無交互關系的蛋白質對的句子標注為訓練數據中的負例即無交互關系。
將得到的訓練數據中的句子通過向量空間模型構建為特征向量,訓練分類器,然后對人工標注的測試集進行測試。
2.3 交叉預測去噪音
交叉預測的方法是在遠監督的基礎上,如圖2所示,將遠監督得到的訓練數據隨機分為k組,取1組數據作為預測集,其余k-1組數據作為訓練集進行訓練,依次輪換訓練集和預測集k次,對每組數據進行預測并去噪,具體步驟如下:
(1)隨機將遠監督得到的訓練數據S劃分為k個不相交的子集,假設S中句子個數為m,那么每個子集中有m/k個句子,相應的子集為{S1,S2,…,Sk};

圖2 交叉預測去噪音
將去噪后的訓練數據S'中的句子通過向量空間模型構建為特征向量,訓練分類器,然后對人工標注的測試集進行測試。
3 實驗及結果分析
3.1 實驗數據及設置
文中采用的知識庫中包含578對有交互關系的蛋白質對和576對無交互關系的蛋白質對。有交互關系的蛋白質對均直接來源于專業PPI數據庫HPRD,HPRD是現有國際上最大的人類PPI數據庫,數據可靠性高;而對于無交互關系的蛋白質對,采用生物醫學領域常用方法,將HPRD中的蛋白質進行隨機組合,去除其中已經包含在HPRD中的蛋白質對組合,剩余蛋白質對作為知識庫中的無交互關系的蛋白質對。
提取的大規模醫學文本來自PubMed數據庫,PubMed是生物醫學領域最具影響力的文獻檢索系統,內容豐富。通過將知識庫中的蛋白質對與PubMed數據庫中的文本進行匹配,可得到去噪前訓練數據共11 147個句子,其中有交互的句子5 477個,無交互的句子5 670個。
通過對實驗結果進行調整,采用五組交叉預測,即k=5,每組數據有2 229個句子,包括1 095個有交互的句子和1 134個無交互的句子。文中采用邏輯回歸分類器對每組訓練數據中的句子進行預測分類,并對人工標注的測試集進行測試,邏輯回歸模型簡單高效,易于實現,計算代價不高,在進行大規模線性分類時較為方便。
文中選取了AIMed語料中的1 000個標注作為測試數據。AIMed語料來自于PubMed摘要,是PPI實驗中最具代表性的專家標注語料。實驗采用的性能評價指標是當前PPI抽取系統主要使用的三個指標:精確度(Precision)、召回率(Recall)和F值。
Precision=TP/TP+FP
(1)
Recall=TP/(TP+FN)
(2)
F-Score=2×P×R/(P+R)
(3)
3.2 實驗結果及討論
遠監督與交叉預測去噪后得到的訓練數據如表1所示。
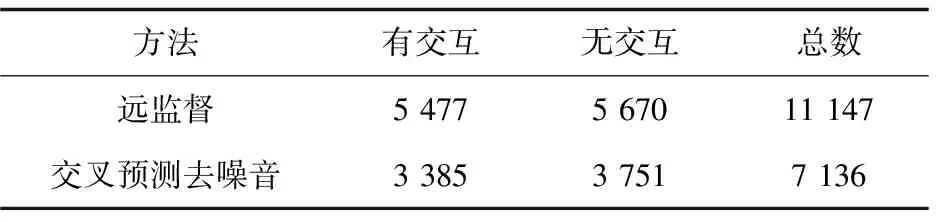
表1 訓練數據
從表1可以看出,相比于遠監督得到的訓練數據,經過五折交叉預測去噪后,訓練數據中有交互的句子數量減少了38%,無交互的句子數量減少了34%,句子總數減少了36%。由此可得,交叉預測較好地識別出了訓練數據中的噪音,且對于有交互和無交互的句子噪音數量識別相差不大,保證了訓練數據的平衡性。
分別使用遠監督和交叉預測去噪得到的訓練數據去訓練得到模型,然后對AIMed語料進行預測,結果如表2所示。

表2 測試結果對比
從表2可以看出,去噪后的模型在保持精確度的同時,召回率提高了8%,提升效果明顯,并且整體F-score也提高了2%。說明交叉預測的方法對訓練集中的噪音消除的效果較為明顯,有效提高了模型的性能。
4 結束語
詳細分析了基于遠監督產生大規模文本進行蛋白質交互關系抽取的方法,針對訓練數據存在噪音的問題,提出了一種交叉預測去噪的方法。通過對訓練數據進行分組預測來清除其中的噪音,并通過人工標注語料進行測試。實驗結果表明,同遠監督相比,交叉預測有效清除了訓練數據中的噪音,提高了模型的識別效果。
參考文獻:
[1] PRASAD T S K,GOEL R,KANDASAMY K,et al.Human protein reference database-2009 update[J].Nucleic Acids Research,2009,37:767-772.
[2] BADER G D,DONALDSON I,WOLTING C,et al.BIND:the biomolecular interaction network database[J].Nucleic Acids Research,2001,29(1):242-245.
[3] SALWINSKI L,MILLER C S,SMITH A J,et al.The database of interacting proteins:2004 update[J].Nucleic Acids Research,2004,32:449-451.
[4] KERRIEN S,ALAMFARUQUE Y,ARANDA B,et al.Int Act-open source resource for molecular interaction data[J].Nucleic Acids Research,2007,35:561-565.
[5] CEOL A,ARYAMONTRI A C,LICATA L,et al.MINT,the molecular interaction database:2009 update[J].Nucleic Acids Research,2010,38:532-539.
[6] BUNESCU R, MOONEY R, RAMANI A,et al.Integrating co-occurrence statistics with information extraction for robust retrieval of protein interactions from Medline[C]//Proceedings of the workshop on linking natural language processing and biology:towards deeper biological literature analysis.[s.l.]:Association for Computational Linguistics,2006:49-56.
[7] KOIKE A, KOBAYASHI Y, TAKAGI T. Kinase pathway database:an integrated protein-kinase and NLP-based protein-interaction resource[J].Genome Research,2003,13(6a):1231-1243.
[8] 楊志豪,洪 莉,林鴻飛,等.基于支持向量機的生物醫學文獻蛋白質關系抽取[J].智能系統學報,2008,3(4):361-369.
[9] 唐 楠,楊志豪,林鴻飛,等.基于多核學習的醫學文獻蛋白質關系抽取[J].計算機工程,2011,37(10):184-186.
[10] GRIMES G R,WEN T Q,MEWISSEN M,et al.PDQ Wizard:automated prioritization and characterization of gene and protein lists using biomedical literature[J].Bioinformatics,2006,22(16):2055-2057.
[11] ANANIADOU S,KELL D B,TSUJII J.Text mining and its potential applications in systems biology[J].Trends in Biotechnology,2006,24(12):571-579.
[12] NIU Y,OTASEK D,JURISICA I.Evaluation of linguistic features useful in extraction of interactions from PubMed;application to annotating known, high-throughput and predicted interactions in I2D[J].Bioinformatics,2010,26(1):111-119.
[13] HAUSSLER D.Convolution kernels on discrete structures[R].California:University of California at Santa Cruz,1999.
[14] MINTZ M,BILLS S,SNOW R,et al.Distant supervision for relation extraction without labeled data[C]//Proceedings of the joint conference of the 47th annual meeting of the ACL and the 4th international joint conference on natural language processing of the AFNLP.[s.l.]:Association for Computational Linguistics,2009:1003-1011.
[15] 王宇偉,牛 耘.基于關系相似性的蛋白質交互作用識別[J].計算機技術與發展,2015,25(2):42-46.
[16] 吳紅梅,牛 耘.基于特征加權的蛋白質交互識別[J].計算機技術與發展,2016,26(2):114-117.

