面向腫瘤精準醫學的綜合數據資源TCGA及其相關在線分析工具推薦
, ,睿,, ,,
癌癥是一種極為復雜的人類疾病,涉及基因組的多種動態變化[1]。每種類型的癌癥,發生的遺傳畸變都是獨特的,包括體細胞突變、拷貝數變異、基因表達譜差異和表觀遺傳改變。因此需要更好地理解腫瘤的各種遺傳變化,才能更好地對其進行診斷、治療和預防。全基因組測序和生物信息技術的發展為癌癥基因組研究提供了新的線索[2]。典型的綜合數據資源是癌癥基因組圖譜(The Cancer Genome Atlas,TCGA)項目,它收集整理了大量癌癥基因組數據,并利用新的基因組分析技術以加速對癌癥的全面了解。
TCGA數據庫的目標是完成一套完整的與所有癌癥基因組改變相關的“圖譜”,旨在獲得癌癥生物學的新見解,從而有助于癌癥的治療。該項目是2006年由美國國立衛生研究院牽頭的一項大型癌癥基因組計劃,自2008年開始有階段性成果發表[3],2009年繼續投資2.75億美元,增加了多種類型的癌癥數據,到2014年已收集了36類癌癥數據,包括臨床數據、DNA、RNA、蛋白質等多層次的數據。在數據生成方面,該項目取得了無可爭議的成功。隨著樣品采集、測序和分析技術的快速發展,TCGA收錄的腫瘤相關數據呈指數增長。目前,新成立的NCI Genomics Data Commons將TCGA的數據整合在該門戶網站中,并且為基因組數據用戶提供了交互式支持和更清晰友好的界面。
我們可以用前所未有的微觀視角來看待癌癥,但是還沒有達到能夠解釋這種疾病的全貌的程度,對其發病機制亦不完全清楚。而TCGA數據已被用于發現新的突變,確定內在的腫瘤類型,確定泛癌相似性和差異性,同時收集腫瘤演變的證據。目前已經開發了大量針對TCGA數據的生物信息學工具,反映出TCGA數據資源的重要性。
1 TCGA數據介紹
為了全面分析癌癥基因組圖譜,TCGA應用基于微陣列和下一代測序方法的高通量技術,產生了癌癥的多種數據類型信息。
TCGA中的癌癥數據通過各種標識符(ID)進行識別和編目(表1),每種癌癥類型都包括體細胞突變、拷貝數、基因表達、miRNA表達、DNA甲基化、逆轉蛋白相位陣列(RPPA)和臨床信息。除原始排序文件外(表2),每種數據類型都包括可供公開下載的原始數據和已處理的數據。

表1 TCGA數據庫中的ID號

表2 數據類型和可獲取水平
2 TCGA數據在線分析工具
目前TCGA數據分析很復雜,涉及多個步驟,為獲得有意義的生物學結果,需要仔細考慮分析每個步驟,并將特定工具應用于某些實驗模型。為現有數據開發相關的探索工具,需要實驗科學家和計算科學家之間的協調。然而,實驗科學家很難使用計算科學家開發的計算工具,因為這些計算工具需要數據準備以及安裝和使用打包軟件,而且某些軟件往往只適用于某些特定平臺或操作系統。一些更高級的計算工具往往難以理解或使用,從而限制了其應用。不過有基于網絡的工具可以提供方便的計算解決方案,幫助實驗科學家使用和分析復雜的癌癥基因組數據。這些工具幫助無生物信息學背景的生物學家和醫學家獲得更多的生物學和醫學見解,但是選擇適當的工具并不是一項簡單的任務,對于沒有經驗的用戶來說尤其如此。
本文整理了一個基于網絡的可用于分析TCGA數據的公開工具列表,并將這些工具進行分類以便更好地進行查詢和使用。
表3顯示了基于網絡工具的32個在線分析資源,它們代表了當前可用于分析TCGA數據的主要資源。為了進一步區分和指導這些工具的選擇,本文將所有資源工具分為全局分析工具、目標分析工具和輔助分析工具三大類。
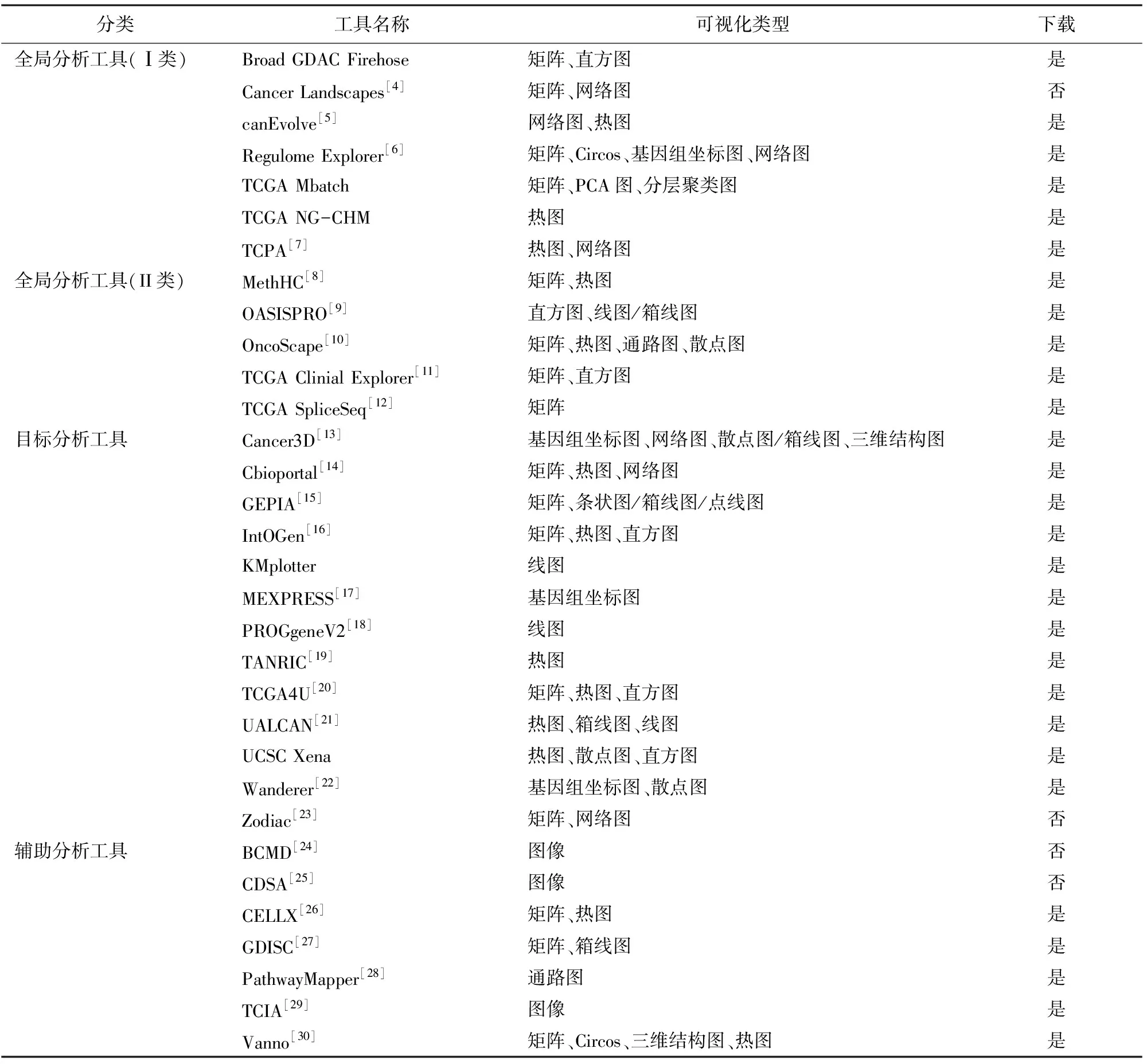
表3 針對TCGA數據的在線分析資源
全局分析工具能夠檢查癌癥基因組的整體特征,可以成為剛剛開始研究癌癥基因組數據研究人員的寶貴資源。全局分析工具有兩種類型即Ⅰ型和Ⅱ型,前者僅提供全局分析,后者則提供除全局分析之外的選定目標分析。
目標分析工具是研究人員最常使用的基于網絡的公共工具。這些工具可以令研究人員深入分析具體的基因或者基因集,甚至miRNA等研究對象,方便使用者調查癌癥數據中自己感興趣的目標。
基于公共網絡的輔助分析工具可以將TCGA數據轉換為易于訪問、瀏覽和下載的在線資源。這些數據可以幫助用戶補充實驗結果或者提供額外的證據和解釋,幫助研究人員更全面地分析自己的研究和促進生物學發現。
3 TCGA數據在線分析工具推薦
首先可以由本文的分類區分不同工具的使用類型,縮小選擇范圍;然后根據實際需要結合具體研究(如數據來源、數據類型、分析方法、研究目的),選擇具體的工具進行進一步的分析。以下是對TCGA數據進行不同分析時建議選擇的一些工具,但這些工具都不能完全取代先進的計算和統計方法,只是為研究人員提供一些使用幫助,擴展他們癌癥組學、癌癥復雜性和癌癥網絡等方面的相關知識。
3.1 突變分析
有10種在線工具(Broad GDAC Firehose,Cancer3D,cbioportal,CELLX,IntOGen,TANRIC,TCGA Clinical Explorer,TCGA4U,UCSC Xena和Vanno)可以進行突變分析。一般來說,推薦使用cbioportal,因其包含多種癌癥類型和多種可視化分析功能,功能強且易于使用。
3.2 相關性分析
有17種在線工具(Broad GDAC Firehose,Cancer Landscapes,canEvolve,cbioportal,CELLX,GDISC,GEPIA,MethHC,MEXPRESS,OASISPRO,Regulome Explorer,TANRIC,TCGA Clinical Explorer,TCGA NG-CHM,TCPA,Wanderer和Zodiac)可以進行相關性分析。總的來說,推薦使用麻省理工學院和哈佛大學Broad研究所研發的Broad GDAC Firehose,因其有多種分析算法供用戶使用,功能全面,且包含多種分析工具。
3.3 差異分析
有12種在線工具(Broad GDAC Firehose,canEvolve,cbioportal,CELLX,GEPIA,MEXPRESS,OncoScape,TANRIC,TCGA4U,TCPA,UALCAN和Wanderer)可以進行差異分析,一般推薦使用分析基因表達譜的工具GEPIA。差異分析是該工具的主要分析功能,其在線分析界面簡單易懂,非常易于理解和使用。
3.4 通路分析
有8種在線工具(Broad GDAC Firehose,Cancer Landscapes,canEvolve,MethHC,OncoScape,PathwayMapper,Regulome Explorer和TCGA NG-CHM)可以進行通路分析。推薦使用Broad GDAC Firehose和OncoScape,前者分析方法豐富,后者簡單直觀。
3.5 生存分析
有16種在線工具(Broad GDAC Firehose,Cancer Landscapes,canEvolve,cbioportal,CELLX,GDISC,GEPIA,KMplotter,OASISPRO,PROGgeneV2,TANRIC,TCGA Clinical Explorer,TCGA4U,TCPA,UALCAN和UCSC Xena)可以進行生存分析。如果僅想進行單一的生存分析,推薦使用PROGgeneV2,因其具有廣泛的數據來源和多種可選參數設置。
3.6 泛癌分析
有8種在線工具(Broad GDAC Firehose,CancerLandscapes,cbioportal,IntOGen,Regulome Explorer,TCGA NG-CHM,UCSC Xena和Zodiac)可以進行泛癌癥分析(pan-cancer analysis)。一般來說,推薦使用cbioportal和Cancer Landscapes,前者收集了來自泛癌研究的大量樣本且擁有強大的分析能力;后者的癌癥圖譜模型中包含了泛癌模型,可以直接用于分析。
4 總結
科學家們開發出多種生物信息學工具進行數據挖掘和分析,以便尋找新發現。不久的將來,新發現將有助于診斷、治療和預防癌癥。TCGA提供的癌癥基因組學數據可以系統地揭示癌癥分子生物學的新圖景。這些大量公開可用的數據,為世界各地的研究人員提供了癌癥遺傳學的知識來源,結合多種分析有助于開發個性化癌癥藥物。本文全面整理了基于網絡的公共可用的在線分析資源和工具,可以幫助研究人員方便地查找和使用合適的工具,增進他們對癌癥基因組學的理解。

