眼斑擬石首魚重復DNA序列的染色體定位
朱齊春 鄭 嬌 張 靜 劉賢德 王志勇 蔡明夷
(集美大學水產學院, 農業部東海海水健康養殖重點實驗室, 廈門 361021)
眼斑擬石首魚重復DNA序列的染色體定位
朱齊春 鄭 嬌 張 靜 劉賢德 王志勇 蔡明夷
(集美大學水產學院, 農業部東海海水健康養殖重點實驗室, 廈門 361021)
針對眼斑擬石首魚Sciaenops ocellatus染色體標記匱乏的問題, 利用熒光原位雜交(FISH)定位了眼斑擬石首魚的18S rDNA、5S rDNA和端粒序列。結果顯示, 眼斑擬石首魚的核型公式為2n=48t; 僅有1對18S rDNA位點, 位于第1對染色體的次縊痕部位; 有2對5S rDNA位點, FISH信號強度不等, 強信號位于第8對染色體的近著絲粒端, 弱信號位于第3對染色體的臂間。端粒FISH信號出現于所有染色體的兩端, 但表現出染色體兩端信號不平衡的特點, 著絲粒端FISH信號明顯強于遠端信號。這一特點為判定染色體的方向提供了便利。結合其他石首魚的核型數據可以推斷, 2n=48t的核型及單對近著絲粒分布的18S rDNA位點是石首魚的共同祖征; 在石首魚進化過程中, 曾發生活躍但不影響宏觀核型的小規模重排。研究結果豐富了眼斑擬石首魚染色體的辨識標記, 并為研究石首魚染色體進化提供了基礎數據。
眼斑擬石首魚; 核型; 染色體; 熒光原位雜交; 核糖體DNA; 端粒
染色體是遺傳物質的載體。染色體的形態和結構特征是研究真核生物的基因組結構、系統發生、進化、分類和育種的重要資料。1970年以來,魚類染色體核型數據不斷積累, 至今報道過核型的魚類大約有3425種或亞種, 約占現存魚類的12.2%[1,2]。近年來, 分子克隆、PCR和熒光原位雜交等新技術逐步應用于魚類染色體研究, 極大地拓展了魚類染色體研究內容和深度。
眼斑擬石首魚(Sciaenops ocellatus), 也稱紅姑魚或黑斑紅鱸, 屬鱸形目, 石首魚科, 擬石首魚屬,主要分布在從新澤西到佛羅里達的大西洋和墨西哥灣沿岸。1991年引入我國, 因其抗病力強、生長迅速、適溫和適鹽性廣、存活率高、耐低溫, 非常適合高密度養殖, 很快成為我國重要海水養殖魚類[3,4]。尤鋒等[3]和王曉艷等[5]分別分析了眼斑擬石首魚的核型(2n=48t, NF=48), 并將核仁組織區(NOR)定位于1號染色體的近著絲粒區, 但目前尚未見FISH定位相關報道。為了進一步揭示眼斑擬石首魚的核型特征, 本研究應用FISH技術在眼斑擬石首魚染色體上定位18S rDNA、5S rDNA和端粒序列3種重復序列, 以期為眼斑擬石首魚的染色體識別提供穩定有效的細胞遺傳學標記, 也為石首科魚染色體進化研究提供了基礎數據。
1 材料與方法
1.1 樣品和染色體制備
用于制備染色體的眼斑擬石首魚雌雄各5尾,購自福建省寧德市金鈴水產有限公司。魚體注射秋水仙堿和植物血凝素后, 取頭腎細胞制備染色體,具體的操作方法按陳紫瑩所描述的方法[6]。同時采集眼斑擬石首魚的鰭條固定于無水乙醇中備用。
1.2 探針制備
用DNA提取試劑盒(上海捷瑞生物工程有限公司)從無水乙醇固定的鰭條中提取全基因組DNA。18S rDNA部分編碼序列經PCR擴增獲得, 其上、下游引物序列分別為5′-CGCGCAAATTACCCAC TCCC-3′和5′-CTGAACGCCACTTGTCCCT-3′。5S rDNA的全部編碼序列和間隔序列同樣也用PCR擴增獲得, 所用上、下游引物序列為5′-GTCAGGC CTGGTTAGTACTTGGAT-3′和5′-GGGCGCATTCAGGGTGGTAT-3′。端粒重復序列(TTAGGG),通過引物(TTAGGG)5和(TAACCC)5無模板擴增獲得[7]。
18S rDNA、5S rDNA和端粒重復序列的PCR擴增均為20 μL反應體系[10×buffer (含Mg2+) 2 μL、2.5 mmol/L dNTPs 1.6 μL、10 mmol/L的上下游引物各1 μL、5 U/μL的EasyTaqDNA聚合酶 0.2 μL和50 ng基因組DNA 1 μL, 加ddH2O至20 μL]。不同目標產物對應PCR擴增條件略有不同。18S rDNA擴增條件如下: 94℃預變性4min; 94℃變性30S, 54℃退火1min, 72℃延伸1min, 循環30次; 72℃充分延伸5min。5S rDNA的擴增條件為: 94℃預變性2min, 54℃退火1min, 循環30次。端粒重復序列擴增條件為: 94℃預變性90S; 94℃變性45S, 52℃退火30S, 72℃延伸30S, 循環30次; 72℃充分延伸10min。
上述PCR擴增產物用缺口平移法制備生物素或地高辛標記探針, 操作方法參見試劑盒說明書(Roche)。
1.3 染色體載玻片制備和染色
采用蒸汽滴片法獲得染色體制片, 室溫風干后在60℃恒溫箱中老化3h后, 室溫存放備用, 具體操作同陳紫瑩等[8]的描述。部分制片用吉姆薩染液、4′,6-二脒基-2-苯基吲哚(DAPI)和碘化丙啶(PI)染色, 方法同鄭嬌等[9]的描述。
1.4 原位雜交、信號檢測及觀察
FISH操作參考前人描述[10,11], 包括制片和探針的變性、雜交過夜、嚴謹性洗滌、信號放大等步驟。其中, 生物素標記用親和素-Alexa fluor-488(Introgen)顯示綠色熒光信號, 地高辛標記用抗地高辛-諾丹明(Roche)顯示紅色熒光信號。最后, 滴加含PI或DAPI的抗褪色劑封片。
1.5 顯微觀察、測量與核型排列
在正置熒光顯微鏡(Olympus BX53)下觀察染色體, 用電荷藕合器件圖像傳感器(CCD, Olympus DP80)拍照。選取5個染色體完整、分散良好的中期相(PI染色)的數碼圖像, 應用Image-pro plus 6.0(IPP 6.0)軟件輔助排列并測量染色體的長度、寬度、面積和累積光密度(IOD), 方法參考前人的描述[6,9]。核型圖中的染色體按相對長度降序排列。
2 結果
2.1 核型特征
選取形態清晰、分散良好及數目完整的眼斑擬石首魚的中期分裂相顯微觀察統計, 結果顯示眼斑擬石首魚的細胞含有48條染色體, 核型公式為2n=48t (圖1), 且雄魚和雌魚的染色體數目沒有差異。根據染色體的形態特征和信號特征對其進行配對, 將配對的染色體按照相對長度降序排列并編號(圖1b)。取分散良好的5個中期相進行染色體配對, 并按降序排列成5幅核型圖, 利用Image-pro plus 6.0軟件對染色體的長度、面積、寬度和IOD進行測量, 通過Excel計算核型圖的同源染色體的相對長度、相對面積、相對寬度和相對IOD, 并統計5幅核型圖的平均值, 結果如表1所示。數據表明,眼斑擬石首魚染色體的相對長度在2.62%—5.45%、相對面積在2.20%—5.88%、相對寬度在3.63%—4.67%、相對IOD在2.05%—6.14%, 分布連續。值得注意的是, 吉姆薩染色在1號染色體近著絲粒區域有明顯的次縊痕, 而DAPI染色在相應位置出現陰性帶(圖2a、圖2b)。
2.2 重復序列的FISH定位
以生物素標記的端粒重復序列作探針做FISH,用PI對染色體進行染色, 染色體染為紅色, 端粒信號顯示綠色(圖1a)。綠色信號分布在染色體兩端。兩端端粒信號呈現強度不平衡的特點, 著絲粒端信號強, 遠端信號弱, 因而根據端粒信號強弱可以輕易的判斷染色體方向, 并直觀顯示眼斑擬石首魚含48條端部著絲粒染色體。18S和5S rDNA雙色FISH中, 18S rDNA用生物素標記, 5S rDNA用地高辛標記, 染色體用DAPI負染。結果顯示(圖2c), 染色體負染為藍色; 整個染色體組僅有一對18S rDNA信號, 呈綠色, 分布于1號染色體次縊痕部位; 2對5S rDNA信號, 均呈紅色, 但信號強度不同。強信號5S rDNA位點位于8號染色體近著絲粒區域, 弱信號5S rDNA位點位于3號染色體臂間。兩類rDNA位點不具同線性(圖2c、圖2d)。雌雄性個體間未見明顯的核型差異。
3 討論
3.1 宏觀核型的特征
目前認為, 2n=48t是硬骨魚核型的共同祖征[12]。在海水魚中, 大多數鱸形目魚類具有這一保守核型,石首魚是其中核型最為保守的一個科[1]。目前已知核型的石首魚約38種, 核型包含48條染色體有36種,核型公式為2n=48t約32種[2]。眼斑擬石首魚的核型公式為2n=48t, 與石首魚科的主流核型一致, 具硬骨魚原始核型特征
3.2 rDNA序列的定位

圖1 基于FISH的眼斑擬石首魚染色體核型圖Fig. 1 Karyotype of Sciaenops ocellatus based on FISH
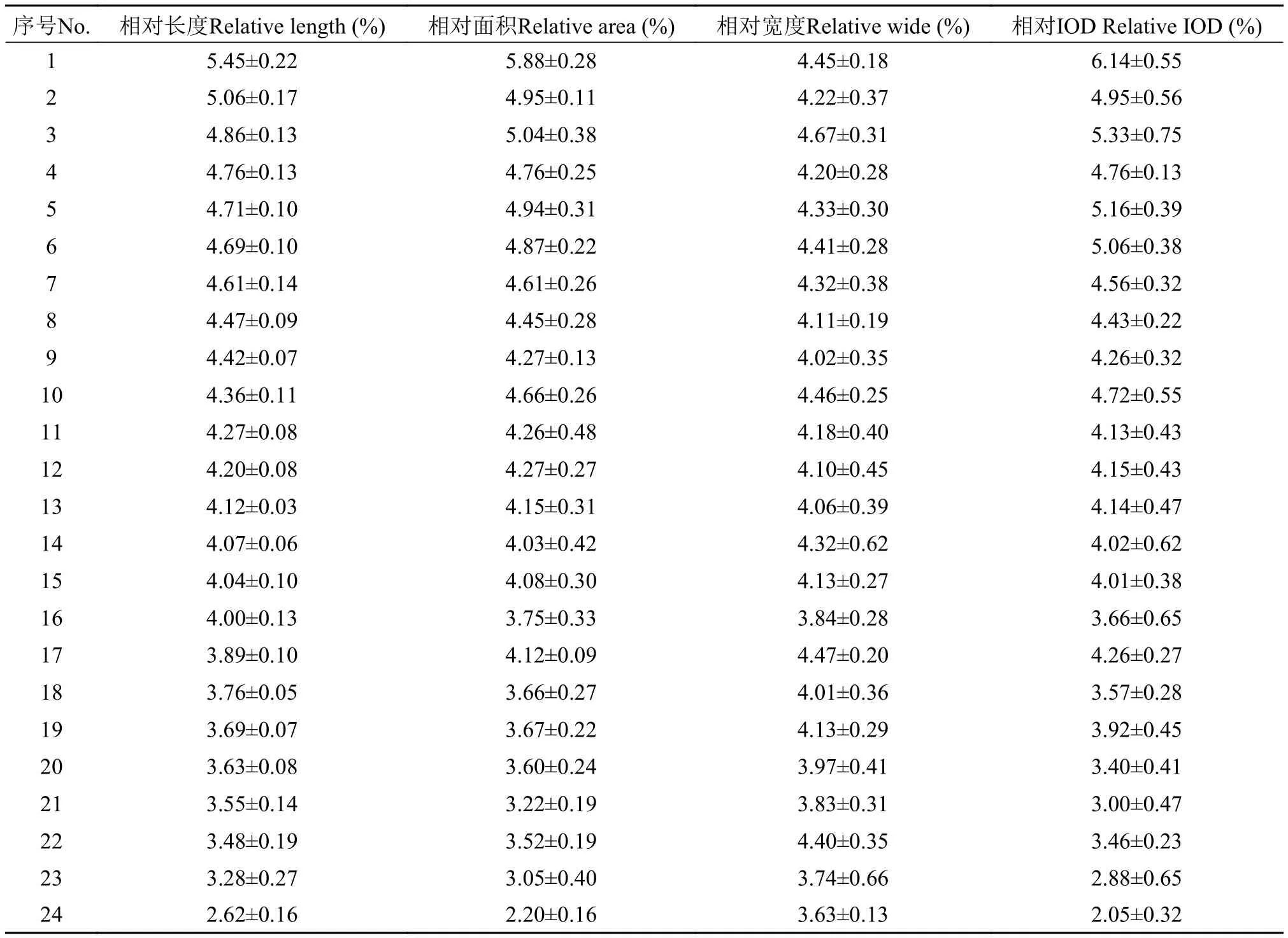
表1 眼斑擬石首魚染色體的相對長度、相對面積、相對寬度和相對IODTab. 1 The relative length, relative area, relative wide and relative IOD of the chromosomes of Sciaenops ocellatus

圖2 眼斑擬石首魚染色體中期相Fig. 2 Metaphase of Sciaenops ocellatus
宏觀核型穩定、保守是石首魚科的基本細胞遺傳學特征。然而, 在許多宏觀核型穩定的海水魚分類單元中常常觀察到微小的染色體重排, 特別是rDNA位點及其鄰近區域[13]。在真核生物細胞核中,rDNA組織為兩個獨立的基因家族, 串聯重復單元分別為45S rDNA和5S rDNA。其中, 45S rDNA編碼5.8S、18S和28S rRNA, 5S rDNA編碼5S rRNA[14]。45S rDNA在染色體上的分布可以通過銀染顯示核仁組織中心(NOR)、DAPI染色顯示陰性帶或CMA染色體顯示高亮帶等方法來顯示, 更直接的方法是以45S rDNA的部分序列(如18S rDNA)為探針作FISH來定位[15]。在石首魚中, 進行過45S rDNA (或NOR)定位研究共有12種(含本研究), 其中開展過FISH定位的有5種(表2)。12種石首魚均含有單對45S rDNA (或NOR)的位點, 但分布位置存在種間變異。其中, 廈門白姑魚(Argyrosomus amoyensis)、美洲無鰾石首魚(Menticirrhus americanus)、箕作黃姑魚(Micropogonias furnieri)、黃姑魚(N. albiflora)和眼斑擬石首魚等5種的分布模式一致, 均在1號染色體近著絲粒區域; 另外4種分布模式各不相同, 還有3種分布位置尚未明確。根據原始性狀的普遍性原則可以推斷, 單對45S rDNA單對分布于1號染色體近著絲粒位置是石首魚原始核型的特征。可見, 眼斑擬石首魚18S rDNA位點的染色體分布仍保留著石首魚共有祖先的特征。
5S rDNA位點的染色體分布模式只能通過FISH定位確定, 因此迄今開展過5S rDNA定位研究的石首魚僅有4種。然而, 現有數據已初步顯示, 石首魚5S rDNA位點在染色體上分布模式存在較高水平的種間變異: 廈門白姑魚具有1對信號; 大黃魚具有9—11對信號; 黃姑魚和眼斑擬石首魚均具有2對信號, 但分布位置不同(表2)。可見, 在石首魚進化進程中, 染色體曾發生過活躍的染色體重排, 但是這些重排沒有改變宏觀核型結構。這種宏觀核型穩定而rDNA位點分布模式高度變化的現象也見于其他硬骨魚類群。例如,科(Serranidae)中多數魚類核型包含48條單臂染色體, 但45S rDNA (或NOR)存在很大的種間變化; 銀鱸科尖口銀鱸屬(Eucinostomus)中也觀察到穩定的核型和兩種rDNA的位置變動[1,23]。導致rDNA位點位置差異的機制可能是轉座子的作用或同源染色不平衡交換等[24,25]。在石首魚中, 什么因素導致5S rDNA的種間變異尚需進一步研究。
雙色FISH直觀顯示, 眼斑擬石首魚的18S rDNA和5S rDNA位點不具有同線性。多數魚類的45S rDNA和5S rDNA位點分布在不同的染色體上,大多數的高等植物和動物也具有同樣的特征[14]。45S rDNA和5S rDNA位點分別位于不同染色體, 可以避免兩個基因簇之間的易位, 并使其可以獨立進化[26]。但是, 也有一些魚類的兩種rDNA位點位于同一條染色體上, 甚至在相鄰的位置上, 如黃姑魚(Nibea albiflora)有2對5S rDNA位點, FISH信號較強的一對與18S rDNA間隔分布在同一條染色體上[9];牙鲆(Paralichthys olivaceus)、大西洋鮭(Salmosalar)、雜斑盔魚魚(Coris julis)等魚類的兩類rDNA位點則串聯分布于同一染色體[27,28]。
3.3 端粒序列的定位
端粒是一種在動植物中廣泛分布的重復序列,是染色體的重要功能元件, 主要功能是維持染色體的完整性和獨立性[29]。目前在定位過端粒序列的80多種魚中, 存在端部以外端粒序列雜交信號的約42%[30]。眼斑擬石首魚的端粒序列雜交信號均出現在染色體的兩端, 未見臂間端粒信號, 說明眼斑擬石首魚在進化過程中沒有發生涉及端粒的染色體倒位或融合。值得注意的是, 眼斑擬石首魚的端粒信號呈現出一個有趣的現象, 各條染色體上著絲粒端的端粒信號較強, 而遠著絲粒端的端粒信號弱。這種染色體兩端端粒信號強度不一致的現象并未見于其他石首魚, 但偶見于其他脊椎動物和植物, 如人類和菠菜(Spinacia oleraceaL.)[29,31]。導致端粒信號強度不一致的可能原因有: (1)著絲粒端粒序列長度長于遠端著絲粒; (2)著絲粒DNA含有端粒序列, 兩種來源的端粒信號重疊。雖然, 導致眼斑擬石首魚兩端端粒信號不一致的原因仍然未明, 但這一現象可以直觀地呈現染色體的方向, 并證明眼斑擬石首魚的染色體全部為端部著絲粒染色體。

表2 石首魚科魚類的核型特點和rDNA位點定位研究概況Tab. 2 The karyotypes characteristics and summary of locations of rDNA on the chromosome of Sciaenidae
4 結論
本研究首次利用雙色FISH分析了眼斑擬石首魚的核型特征, 豐富了該物種的染色體辨識標記,并為研究石首魚染色體進化提供了基礎數據。結合其他石首魚核型數據, 可以推斷: 2n=48t的核型及單對近著絲粒分布的18S rDNA位點是石首魚的共同祖征。盡管開展過石首魚5S rDNA定位的石首魚僅有4種, 現有數據已初步揭示, 石首魚染色體進化過程中, 染色體上曾發生過活躍但不影響宏觀核型的小規模重排。FISH是分析核型特征和染色體進化的有力工具。以FISH為核心技術的分子細胞遺傳學是聯結信息學、分子生物學和細胞學的橋梁。在魚類基因組學和分子生物學研究飛速發展的今天, 魚類分子細胞遺傳學研究將迎來新的發展契機和更廣闊的應用前景。
[1]Galetti Jr P M, Aguilar C T, Molina W F. An Overview of Marine Fish Cytogenetics [M]. Marine Genetics. Springer Netherlands. 2000, 55—62
[2]Arai R. Fish Karyotypes: a Check List [M]. Springer Science amp; Business Media. 2011, 3—4
[3]You F, Liu J, Xu C. Study on the karyotype in the red drum,Sciaenops ocellatus[J].Marine Sciences, 1998,22(2): 51—53 [尤鋒, 劉靜, 徐成. 美國紅魚(Sciaenops ocellatus)的核型研究. 海洋科學, 1998, 22(2): 51—53]
[4]Meilahn C W, Davis D A, Arnold C R. Effects of commercial fish meal analogue and menhaden fish meal on growth of red drum fed isonitrogenous diets [J].The Progressive Fish-culturist, 1996, 58(2): 111—116
[5]Wang X Y, Wang S F, Zhang J S,et al. The Karyotype,Ag-NORs and C-banding ofSciaenops ocellata[J].Oceanologia et Limnologia Sinica, 2012, 43(1): 84—87[王曉艷, 王世鋒, 張建設, 等. 美國紅魚(Sciaenops ocellata)的核型、Ag-NORs和C-帶分析. 海洋與湖沼, 2012,43(1): 84—87]
[6]Chen Z Y. Cytogenetic Study onLarimichthys croceaandNibea albiflora[D]. Thesis for Master of Science. Jimei University, Fujian. 2013 [陳紫瑩, 大黃魚與黃姑魚細胞遺傳學初步研究. 碩士學位論文, 集美大學水產學院,福建. 2013]
[7]Ijdo J W, Wells R A, Baldini A,et al. Improved telomere detection using a telomere repeat probe (TTAGGG) n generated by PCR [J].Nucleic Acids Research, 1991,19(17): 4780
[8]Chen Z Y, Wang H H, Wang K W,et al. The procedure of DPI staining to display nucleolar organizer regions in Yellow Croaker (Nibea alibiflora) [J].Journal of Jimei University, 2014, 19(3): 161—167 [陳紫瑩, 王浩華, 王凱武, 等. DPI染色顯示黃姑魚NOR的方法研究. 集美大學學報: 自然科學版, 2014, 19(3): 161—167]
[9]Zheng J, Cao K, Yang A R,et al. Chromosome mapping using genomic DNA and repetitive DNA sequences as probes for somatic chromosome identification inNibea albiflora[J].Journal of Fisheries of China, 2016, 40(8):1156—1162 [鄭嬌, 曹款, 楊安冉, 等. 黃姑魚染色體識別與重復序列定位. 水產學報, 2016, 40(8): 1156—1162]
[10]Fujiwara A, Abe S, Yamaha E,et al. Uniparental chromosome elimination in the early embryogenesis of the inviable salmonid hybrids between masu salmon female and rainbow trout male [J].Chromosoma, 1997, 106(1):44—52
[11]Cai M Y, Liu X D, Chen Z Y,et al. Characterization of Pacific abalone (Haliotis discus hannai) karyotype by C-banding and fluorescencein situhybridization with rDNA[J].Journal of Fisheries of China, 2013, 37(7): 1002—1008 [蔡明夷, 劉賢德, 陳紫瑩, 等. 皺紋盤鮑染色體C帶和rDNA定位. 水產學報, 2013, 37(7): 1002—1008]
[12]Brum M J I, Galetti Jr P M. Teleostei ground plan karyotype [J].Journal of Computational Biology, 1997, 2(2):91—102
[13]de Mello Affonso P R A, Galetti Jr P M. Chromosomal diversification of reef fishes from genusCentropyge(Perciformes, Pomacanthidae) [J].Genetica, 2005, 123(3):227—233
[14]Martins C, Wasko A P. Organization and evolution of 5S ribosomal DNA in the fish genome [J].Focus on Genome Research, 2004: 335—363
[15]Gornung E. Twenty years of physical mapping of major ribosomal RNA genes across the teleosts: a review of research [J].Cytogenetic and Genome Research, 2013,141(2—3): 90—102
[16]Liao M X, Wang Z Y, Zhang J,et al. The comparison of chromosome mapping of rDNA inArgyrosomus amoyensisandLarimichthys crocea[J].Journal of Fisheries of China, 2017, 41(9): 1338—1344 [廖夢香, 王志勇, 張靜,等. 核糖體DNA在廈門白姑魚和大黃魚染色體上的比較定位. 水產學報, 2017, 41(9): 1338—1344]
[17]Gomes V, Vazzoler A E A D M, Phan V N. Estudos cariotípicos de peixes da familiaSciaenidae(Teleostei,Perciformes) da regi?o de Cananéia, SP, Brasil: 2. sobre o cariótipo deMenticirrhus americanus(Linnaeus, 1758)[J].Brazilian Journal of Oceanography, 1983, 32(2):187—191
[18]Reggi R, Périco E, Suninsky M,et al. Estudos citogenéticos em papa-terra,Menticirrhus litoralis(Perciformes, Serranidae) [J].Simpósio de Citogenética Evolutiva e Aplicada de Peixes Neotropicais,Botucatu,SP. Unesp,Botucatu, 1986, 57
[19]Gomes V, Vazzoler AEM, Phan VN. Estudos cariotípicos de peixes da famíliaSciaenidae(Teleostei, Perciformes)da regi?o de cananéia, sp, brasil, 1. sobre ocariótipo deMicropogonias Furnieri(Desmarest, 1823) [J].Brazilian Journal of Oceanography, 1823a, 32(2): 137—142
[20]Ojima Y, Kikuno T. Karyotypes of a gobiesociform and two perciform fishes (Teleostei) [J].Proceedings of the Japan Academy Ser B Physical amp; Biological Sciences,1987, 63(6): 201—204
[21]Accioly I V, Molina W F. Cytogenetic studies in Brazilian marine Sciaenidae and Sparidae fishes (Perciformes)[J].Genetics and Molecular Research, 2008, 7(2): 358—370
[22]Feldberg E, Porto J I R, Santos E B P,et al. Cytogenetic studies of two freshwater Sciaenids of the genusPlagioscion(Perciformes, Sciaenidae) from the Central Amazon[J].Genetics and Molecular Biology, 1999, 22(3):351—356
[23]Calado L L, Bertollo L A C, Cioffi M B,et al. Evolutionary dynamics of rDNA genes on chromosomes of theEucinostomusfishes: cytotaxonomic and karyoevolutive implications [J].Genetics and Molecular Research, 2014,13(4): 9951—9959
[24]Coen E S, Dover G A. Unequal exchanges and the coevolution of X and Y rDNA arrays inDrosophila melanogaster[J].Cell, 1983, 33(3): 849—855
[25]Merlo M A, Cross I, Manchado M,et al. The 5S rDNA high dynamism inDiplodus sargusis a transposon-mediated mechanism. Comparison with other multigene families and Sparidae species [J].Journal of Molecular Evolution, 2013, 76(3): 83—97
[26]Mantovani M, Abel S, Moreira-Filho O. Conserved 5S and variable 45S rDNA chromosomal localisation revealed by FISH inAstyanax scabripinnis(Pisces, Characidae) [J].Genetica, 2005, 123(3): 211—216
[27]Fujiwara A, Fujiwara M, Nishida-Umehara C,et al. Characterization of Japanese flounder karyotype by chromosome bandings and fluorescencein situhybridization with DNA markers [J].Genetica, 2007, 131(3): 267—274
[28]Mandrioli M, Colomba M S, Vitturi R. Chromosomal analysis of repeated DNAs in the rainbow wrasseCoris julis(Pisces, Labridae) [J].Genetica, 2000, 108(2):191—195
[29]Lan T Y, Liu B, Dong F P,et al. Multicolor FISH analysis of rDNA and telomere on spinach [J].Frontiers of Agriculture in China, 2007, 29(11): 1405—1408 [蘭添穎,劉博, 董鳳平, 等. 菠菜rDNA及端粒多色熒光原位雜交分析. 遺傳, 2007, 29(11): 1405—1408]
[30]Ocalewicz K. Telomeres in fishes [J].Cytogenetic and Genome Research, 2013, 141(2—3): 114—125
[31]Lansdorp P M, Verwoerd N P, Fm V D R,et al. Heterogeneity in telomere length of human chromosomes [J].Human Molecular Genetics, 1996, 5(5): 685—691
LOCATION OF REPETITIVE DNA SEQUENCES ON THE CHROMOSOME OF SCIAENOPS OCELLATUS
ZHU Qi-Chun, ZHENG Jiao, ZHANG Jing, LIU Xian-De, WANG Zhi-Yong and CAI Ming-Yi
(The Key Laboratory of Helthy Mariculture for the East China Sea, Ministry of Agriculture, Fisheries College, Jimei University,Xiamen 361021, China)
The chromosome characteristics of red drum (Sciaenops ocellatus) were analyzed to enhance the cytogenetic markers inSciaenops ocellatusby fluorescencein situhybridization (FISH) with 18S rDNA, 5S rDNA and telomeric repetitive DNA as probes. The karyotype of red drum consists of 48 telomeric chromosomes. FISH with 18S rDNA probes showed a single pair of signals at the secondary constriction sites of Chromosome 1. Whereas, two pairs of signal with different intensity were detected with the 5S rDNA probes: the strong signals were on the proximal of Chromosome 8, and the weak ones were on the interstitial of Chromosome 3. The signals of telomeric repetitive DNA appeared at both ends of all chromosomes with uneven intensity, and signals at the centromeric ended stronger than those at the distal ends, which facilitated determining the direction of the chromosome. These results, together with the available cytogenetic data of other sciaenids, suggested that the karyotype of 2n=48t and single 18S rDNA locus were the symplesiomorphic cytogenetic features of sciaenids, and the active and cryptic chromosomal rearrangements may have occurred along the evolution of sciaenids. The distribution patterns of these three kinds of repetitive DNAs would be useful to assist identifying the chromosomes of red drum, and be the basic data for studying chromosome evolution in Sciaenidae.
Sciaenops ocellatus; Karyotype; Chromosome; FISH; Ribosomal DNA; Telomere
Q343.2
A
1000-3207(2017)06-1218-07
2016-12-28;
2017-04-26
國家自然科學基金(41706157和31272653); 福建省自然科學基金(2017J01449)資助 [Supported by the National Natural Science Foundation of China (41706157, 31272653); the Natural Science Foundation of Fujian Province (2017J01449)]
朱齊春(1991—), 男, 安徽安慶人; 碩士研究生; 主要從事水產生物遺傳育種研究。E-mail: 17859736800@163.com
蔡明夷(1973—), 女, 教授, 博士; 主要從事水產生物遺傳育種研究。E-mail: myicai@jmu.edu.cn

