肝癌細胞HepG2中增強子的識別及生物信息學分析
丁若凡,李宇鵬,張一鳴,朱小冬,胡海碧,劉文榮,李玲,郭志云
1.西南交通大學 生命科學與工程學院,四川 成都 610031;2.成都市第三人民醫(yī)院 病理科,四川 成都 610031
肝癌細胞HepG2中增強子的識別及生物信息學分析
丁若凡1,李宇鵬1,張一鳴1,朱小冬1,胡海碧1,劉文榮1,李玲2,郭志云2
1.西南交通大學 生命科學與工程學院,四川 成都 610031;2.成都市第三人民醫(yī)院 病理科,四川 成都 610031
目的:整合增強子特征識別肝癌細胞HepG2增強子,并對其保守性、GC含量、轉錄因子調控、靶基因功能等進行分析,以期解析肝癌細胞增強子參與的調控網絡。方法:通過整合H3K27ac、H3K4me1和H3K4me3組蛋白修飾及DNaseⅠ高敏位點的Chip-seq數(shù)據(jù)預測HepG2中的增強子,計算每個增強子的平均PhastCons分數(shù)和GC含量,評估整體增強子的保守性與GC含量,整合ENCODE轉錄因子結合位點數(shù)據(jù)尋找轉錄因子-增強子調控,使用GREAT和DAVID分別對增強子和增強子的靶基因進行GO與KEGG通路功能富集分析。結果:共識別2254個肝細胞癌增強子,1432個增強子靶基因,135個轉錄因子的9983個增強子結合位點;比較隨機位點靶基因,發(fā)現(xiàn)增強子顯著正調控靶基因的表達;保守性與GC含量分析表明增強子具有顯著高的保守性與GC含量,并存在C-T/C-T/C-T-G模式的motif;增強子功能分析顯示增強子顯著富集于蛋白結合、酶結合、轉錄因子結合、RNA聚合酶Ⅱ結合等已知增強子功能,增強子GO與KEGG通路功能富集分析表明增強子靶基因顯著參與細胞增殖、細胞凋亡、細胞周期調控和細胞遷移等腫瘤相關的生物進程與信號通路。結論:識別的肝細胞癌增強子具有顯著高的保守性與GC含量,受多種轉錄因子調控,對其靶基因起正調控作用并且顯著富集于腫瘤相關生物學進程與信號通路中。
增強子;肝細胞癌;Chip-seq;組蛋白修飾
肝細胞癌是死亡率最高的癌癥之一,其發(fā)生往往與基因表達失調相關,因此解析參與肝細胞癌發(fā)生發(fā)展的調控關鍵因子及調控網絡對于肝細胞癌研究具有重要意義[1]。增強子一般是幾百堿基對長度的DNA片段,并能被多個轉錄因子占據(jù),在基因調控中通過順式調控原件對靶基因起到正調控作用。已有研究表明肝細胞癌中的增強子突變會導致增強子失活,進而影響靶基因的表達[2],因此探尋增強子在肝細胞癌中的調控網絡,對于從轉錄水平了解肝細胞癌發(fā)生發(fā)展機制具有重要作用。增強子依據(jù)其活性分為活性增強子與失活增強子,而往往活性增強子是結合轉錄因子并行使下游調控功能的主要增強子類型。活性增強子一般處于能夠被DNaseⅠ所切割的開放染色質區(qū)域DHS(DNaseⅠhypersensitive sites)中,開放染色質結構對于轉錄因子結合是必需的,所以DHS也被認為是一種識別增強子區(qū)域傳統(tǒng)和有效的方法[3]。增強子相關的核小體的組蛋白末端共價修飾對于轉錄因子的招募具有重要作用[4],以往研究表明活性增強子存在顯著的組蛋白修飾H3K27ac信號富集及高的H3K4me1/H3K4me3信號比值,并且H3K27ac修飾可以作為區(qū)分活性增強子和非活性增強子的標志[5-6]。我們通過增強子特征識別了肝癌細胞HepG2中的增強子,并對增強子序列功能進行了分析。通過整合來自ENCODE[7]的HepG2細胞系的3種組蛋白修飾(H3K27ac、H3K4me1、H3K4me3)數(shù)據(jù)及DHS數(shù)據(jù),我們共識別了2254條肝癌細胞的增強子及1432個增強子的靶基因,分析了這些增強子的保守性、GC含量及結合轉錄因子富集情況,并對增強子及增強子的靶基因進行了GO與KEGG通路功能富集分析。結果發(fā)現(xiàn)HepG2中的增強子具有顯著高的保守性與GC含量,并有大量轉錄因子富集,增強子功能分析顯示增強子顯著與蛋白結合、轉錄因子結合等增強子功能相關,而增強子靶基因GO與KEGG通路富集分析表明增強子靶基因顯著參與細胞增殖、細胞凋亡、細胞周期調控、細胞遷移等腫瘤相關生物進程與信號通路。這些研究有望為進一步探討肝細胞癌的基因調控及功能分析提供理論依據(jù)。
1 材料與方法
1.1 材料
組蛋白修飾和DHS的Chip-seq[8]數(shù)據(jù)來自ENCODE,包括peak文件和signal文件:H3K4me1(ENCFF635NAK、ENCFF159AXA),H3K4me3(ENCFF822KCS),H3K27ac(ENCFF558QIW、ENCFF542IPN) , DHS (ENCFF673GQN、ENCFF776TKB);基因組序列保守性數(shù)據(jù)來自UC?SC cons46way track;基因組GC含量數(shù)據(jù)來自UC?SC GC Percent track;轉錄因子結合位點數(shù)據(jù)來自 ENCODE Txn Factor(V3);肝臟基因表達譜的RNA-seq數(shù)據(jù)(RPKM)自GTEx[9]獲得。
1.2 方法
1.2.1 HepG2中增強子的識別 首先用bwtool[10]和deeptools[11]對3種組蛋白修飾及DHS信號進行歸 一 化 ,篩 選 2kb內 有 DHS、H3K27ac和H3K4me1 peaks,且H3K4me1的平均歸一化信號高于H3K4me3的區(qū)域作為候選增強子區(qū)域,與參考基因組(GENCODE.V19)的蛋白編碼基因比較,去除與蛋白編碼基因區(qū)域及其蛋白編碼基因轉錄起始位點上游1kb區(qū)域內有交集的增強子后,剩余的增強子作為最終識別的增強子。
1.2.2 保守性分析及GC含量 通過bwtool獲得每個增強子的PhastCons分值,并計算每個增強子中心上下游100bp的DHS信號均值來表示該增強子的活性,將各個組織活性最高的前1000條增強子序列輸入MEME,獲得每個組織增強子的motif,參數(shù)如下:-revcomp-nmotifs 3-minw 10-evt 0.01-mod oops-dna。每個增強子中心及附近位點的GC含量通過bwtool獲得。
1.2.3 增強子上轉錄因子結合位點識別 結合在增強子中心2kb以內的轉錄因子結合位點被認為對增強子有調控作用,每個增強子周圍的轉錄因子結合位點數(shù)目被定義為該增強子上的轉錄因子富集程度。
1.2.4 增強子GO分析和靶基因定義及GO和KEGG信號通路分析 距離增強子100kb內的鄰近蛋白編碼基因被定義為該增強子的靶基因,并根據(jù)從GTEx獲得的基因表達量數(shù)據(jù)得到每個增強子的靶基因表達量。用GREAT[12]預測每個組織增強子的功能(P<0.01),用 DAVID[13]對增強子靶基因進行GO和KEGG信號通路分析,以P<0.05為顯著性閾值。
2 結果與討論
2.1 增強子的識別
根據(jù)3種組蛋白修飾特征及DHS,我們共識別了2254條增強子。正如預期,增強子整體呈現(xiàn)高的H3K27ac信號,以及高的H3K4me1/H3K4me3信號比,且3種組蛋白修飾信號都呈雙峰模式,即文獻報道的peak-valley-peak模式[14]。組蛋白修飾信號在開放染色體區(qū)域附近顯著增高,且DHS信號峰值位于增強子中心,這些結果說明我們找到的增強子符合活性增強子特征(圖1)。
2.2 增強子保守性及GC含量分析
增強子做為一種順式調控原件,在基因表達調控上起重要作用,因此,增強子序列往往較為保守。我們采用PhastCons計算了增強子的保守性分值,通過與隨機位點相比較,發(fā)現(xiàn)增強子中心附近500bp內呈現(xiàn)顯著高的保守性分值(圖2A),且保守性相對增強子中心呈對稱分布。相比于增強子來說,基因的保守性則在轉錄始位點(transcription start sites,TSS)下游呈現(xiàn)顯著高的保守性,這與先前的研究結果一致。另外,為了確定這些增強子所具有的保守功能模塊,我們用MEME工具對增強子進行了模體(motif)分析,結果顯示其具有典型的C-T/C-T/C-T-G模式的motif(圖2B)。先前研究表明,活性增強子普遍存在低甲基化現(xiàn)象[15],而甲基化與GC含量存在顯著的相關性[16],為此,我們分析了增強子的GC含量(圖2C)。正如預期,相比于人類基因組41.6%的背景GC含量,我們得到的HepG2增強子的GC含量(50.4%)顯著高于人基因組平均GC含量(圖2C),且GC含量在增強子中心附近200bp內富集最為顯著。與之相反,基因的GC含量在TSS上游區(qū)域較低而在TSS附近區(qū)域呈現(xiàn)顯著高的GC含量。這一結果說明我們獲取的增強子存在潛在的甲基化傾向,并且已有研究表明肝細胞癌中DNA甲基化受抑制會導致參與腫瘤的生理調控及能量代謝平衡的AMPK信號通路發(fā)生改變[17]。

圖1 增強子上下游2000bp內的3種組蛋白修飾及DHS歸一化平均信號
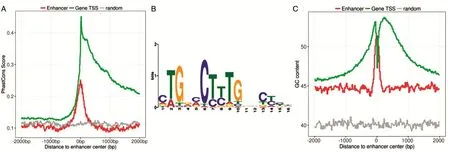
圖2 增強子保守性、motif及GC含量分析結果
2.3 轉錄因子富集程度分析
活性增強子往往通過募集大量轉錄因子從而增強下游基因的表達,為此識別增強子上的轉錄因子結合情況對于解析增強子參與的轉錄調控具有重要意義。因此,我們將ENCODE收錄的HepG2中的轉錄因子結合位點數(shù)據(jù)與本研究得到的增強子進行整合,發(fā)現(xiàn)有135個轉錄因子的9983個轉錄因子結合位點結合到了我們識別的增強子上,并且增強子的轉錄因子富集程度顯著高于隨機位點(P<2.2×10-16)(圖3A)。其中,轉錄因子FOXA1在增強子中富集最明顯,共有961轉錄因子結合位點。FOXA1能顯著抑制癌細胞特異的基因表達[18],并有研究表明增強子上的FOXA1結合位點突變會導致增強子失活進而影響其靶基因的表達[2],這些暗示著肝細胞癌增強子上的FOXA1結合位點對于肝癌細胞的代謝及生物進程可能發(fā)揮著重要作用。

圖3 增強子的轉錄因子富集程度與靶基因的表達量。
2.4 HepG2活性增強子靶基因識別及表達量分析
增強子作為一種順式調控原件,其最主要的功能是正調控鄰近基因的表達。為了驗證我們識別的增強子的這一功能,通過與鄰近已經注釋的蛋白編碼基因位置信息比較,共獲得1432個增強子的靶基因。為了驗證這些靶基因是否由于增強子的存在引起其表達上調,與隨機基因的表達進行了比較,發(fā)現(xiàn)識別的增強子靶基因表達量顯著高于隨機基因(P<0.05)(圖3B),這暗示本研究識別的肝細胞癌增強子對其靶基因的表達起到了明顯的增強作用。
2.5 增強子GO分析及靶基因的GO和KEGG信號通路分析
通過使用GREAT獲得增強子作為順勢調控元件的功能,可以看出增強子的分子功能顯著與增強子功能相一致,如蛋白結合(4.10E-16)、酶結合(2.48×10-15)、轉錄因子結合(8.82×10-8)和RNA聚合酶Ⅱ結合(1.70×10-8)。值得注意的是,RNA聚合酶Ⅱ在增強子上的結合意味著肝細胞癌增強子同樣可以招募RNA聚合酶Ⅱ從而轉錄出轉錄本,即增強子 RNA(enhancer RNA,eRNA),這類RNA的功能目前尚不清楚,有研究報道eRNA與增強子靶基因調控相關[19]。另外,通過對增強子靶基因的GO分析,發(fā)現(xiàn)增強子調控的靶基因顯著參與細胞增殖、細胞凋亡、細胞周期調控、細胞遷移等腫瘤相關的生物進程(表1)。并且,在增強子靶基因的KEGG分析中發(fā)現(xiàn)其靶基因顯著參與腫瘤相關的信號通路,如MAPK信號通路(在腫瘤細胞增殖、凋亡中發(fā)揮調控作用)、TNF信號通路(調控腫瘤內皮細胞因子活性)和癌癥中的通路等(表2)。
2.6 結論
本研究整合了3種組蛋白修飾(H3K27ac、H3K4me1和H3K4me3)數(shù)據(jù)及DNaseⅠ高敏位點DHS信號,預測了2254個增強子,并獲得1432個增強子的靶基因,結合轉錄因子結合位點數(shù)據(jù),獲得可能調控增強子的135個轉錄因子及9983個轉錄因子結合位點。在對增強子的保守性分析中獲得了HepG2中的增強子可能發(fā)揮功能元件作用的C-T/C-T/C-T-G模式的motif。通過靶基因的表達量分析,表明預測得到的增強子能顯著正調控基因表達。增強子的高GC含量意味著增強子存在潛在的甲基化調控模式,這與腫瘤的發(fā)生發(fā)展高度相關。對增強子的功能分析表明增強子發(fā)揮增強子已知的普遍功能,如蛋白結合、轉錄因子結合、RNA聚合酶Ⅱ結合等,其靶基因顯著參與腫瘤相關的生物進程及信號通路。這些結果將為進一步研究肝細胞癌的基因調控及功能提供理論依據(jù)。

表1 增強子靶基因參與的腫瘤相關生物進程

表2 增強子靶基因參與的腫瘤相關KEGG信號通路
[1] Costentin C.Hepatocellular carcinoma surveillance[M].Paris:La Presse Médicale,2017.
[2] Huang D,Ovcharenko I.Identifying causal regulatory SNPs in ChIP-seq enhancers[J].Nucleic Acids Res,2015,43(1):225-236.
[3] Wang Y M,Zhou P,Wang L Y,et al.Correlation be?tween DNaseⅠhypersensitive site distribution and gene expression in HeLa S3 cells[J].PLoS One,2012,7(8):e42414.
[4] Heinz S,Romanoski C E,Benner C,et al.The selec?tion and function of cell type-specific enhancers[J].Nat Rev Mol Cell Biol,2015,16(3):144-154.
[5] Creyghton M P,Cheng A W,Welstead G G,et al.Histone H3K27ac separates active from poised enhanc?ers and predicts developmentalstate[J].Proc Natl Acad Sci USA,2010,107(50):21931-21936.
[6] Guenther M G,Levine S S,Boyer L A,et al.A chro?matin landmark and transcription initiation atmost promoters in human cells[J].Cell,2007,130(1):77-88.
[7] Raney B J,Cline M S,Rosenbloom K R,et al.EN?CODE whole-genome data in the UCSC genome brows?er(2011 update)[J].Nucleic Acids Res,2011,39(Data?base issue):D871-875.
[8] Jothi R,Cuddapah S,Barski A,et al.Genome-wide identification ofin vivo protein-DNA binding sites from ChIP-Seq data[J].Nucleic Acids Res,2008,36(16):5221-5231.
[9] Consortium G T. The genotype-tissue expression(GTEx)project[J].Nat Genet,2013,45(6):580-585.
[10]Pohl A,Beato M.bwtool:a tool for bigWig files[J].Bioinformatics,2014,30(11):1618-1619.
[11]Ramirez F,Dundar F,Diehl S,et al.deepTools:a flexible platform for exploring deep-sequencing data[J].Nucleic AcidsRes,2014,42(Web Serverissue):W187-191.
[12]McLean C Y,Bristor D,Hiller M,et al.GREAT im?proves functional interpretation of cis-regulatory regions[J].Nat Biotechnol,2010,28(5):495-501.
[13]Jiao X,Sherman B T,Huang da W,et al.DAVIDWS:a stateful web service to facilitate gene/protein list analysis[J].Bioinformatics,2012,28(13):1805-1806.
[14]Pundhir S,Bagger F O,Lauridsen F B,et al.Peakvalley-peak pattern of histone modifications delineates active regulatory elementsand theirdirectionality[J].Nucleic Acids Res,2016,44(9):4037-4051.
[15]Stadler M B,Murr R,Burger L,et al.DNA-binding factors shape the mouse methylome at distal regulato?ry regions[J].Nature,2011,480(7378):490-495.
[16]EhrlichM,Gama-SosaM A,HuangL H,etal.Amount and distribution of 5-methylcytosine in hu?man DNA from different types of tissues of cells[J].Nucleic Acids Res,1982,10(8):2709-2721.
[17]Sun Q,Xie Y,Wang G,et al.Identification of genes in HepG2 cells that respond to DNA methylation and histone deacetylation inhibitor treatment[J].Exp Ther Med,2014,8(3):813-817.
[18]Zhang G,Zhao Y,Liu Y,et al.FOXA1 defines can?cer cell specificity[J].Sci Adv,2016,2(3):e1501473.
[19]Melamed P,Yosefzon Y,Rudnizky S,et al.Transcrip?tional enhancers:Transcription,function and flexibility[J].Transcription,2016,7(1):26-31.
Identification and Bioinformatics Analysis of Enhancers in Hepatocellular Carcinoma HepG2
DING Ruo-Fan1,LI Yu-Peng1,ZHANG Yi-Ming1,ZHU Xiao-Dong1,HU Hai-Bi1,LIU Wen-Rong1,LI Ling2,GUO Zhi-Yun2*
1.School of Life Science and Engineering,Southwest Jiaotong University,Chengdu 610031;2.Department of Pathology,Third People's Hospital,Chengdu 610031;China
*Corresponding author,E-mail:zhiyunguo@gmail.com
Objective:To resolve the enhancer regulation network of hepatoma cell,the enhancers were identi?fied by integrating the features of the enhancers in the hepatoma cell HepG2,and GC content,regulation of tran?scription factors,identification of target genes and functional enrichment were analyzed.Methods:Enhancers in HepG2 were predicted by integrating Chip-seq data of histone modifications H3K27ac,H3K4me1 and H3K4me3 and of DNaseⅠ hyper-sensitivity sites.The average PhastCons score and GC content of each enhancer were calcu?lated to assess the conservation and GC content of the overall enhancers.ENCODE transcription factor bindingsites data were integrated to search for transcription factor-enhancer regulation.The enrichment analysis of GO and KEGG pathway was performed by using GREAT and DAVID on enhancers and the target genes of enhancers respectively.Results:A total of 2254 enhancers in HepG2 were predicted,and 1432 target genes of enhancers,135 transcription factors and 9983 transcription factor binding sites of enhancers were obtained.The enhancers in HepG2 significantly promoted the expression of target genes by comparing with random regions.The analysis of conservation and GC content showed that the enhancers were significantly conserved and had a remarkably high GC content,and the motif of enhancer was C-T/C-T/C-T-G.The analysis of the function enrichment of GO and KEGG pathway of enhancers showed that the target genes of enhancers were involved in cell proliferation,cell apoptosis,regulation of cell cycle and cell migration and other tumor related biological processes and signaling pathways.Conclusion:Enhancers in HepG2 were significantly conserved and had a remarkable high GC content enrichment,and they were regulated by a variety of transcription factors and played a positive role in regulation on their target genes,and were significantly enriched in tumor-related biological processes and signaling pathways.
enhancer;hepatocellular carcinoma;Chip-seq;histone modification
Q751;Q811.4
A
1009-0002(2017)04-0455-05
2017-02-10
中央高校基本科研業(yè)務費專項(2682016YXZT04);國家大學生創(chuàng)新性實驗計劃(201610613066);四川省大學生創(chuàng)新創(chuàng)業(yè)訓練計劃(2016095)
丁若凡(1991- ),男,碩士研究生,(E-mail)dingruofan1@foxmail.com
郭志云,(E-mail)zhiyunguo@gmail.com
10.3969/j.issn.1009-0002.2017.04.010

