基于microRNA-mRNA配對表達譜進行聯合分析的方法學進展
趙路陽,張康,顧成磊,3,葉明俠,范文生,韓為東,孟元光
1.解放軍總醫院 a.婦產科;b.基礎研究所,北京 100853;2.北京華信醫院 婦產科,北京 100016;3.解放軍第309醫院 婦產科,北京 100091
基于microRNA-mRNA配對表達譜進行聯合分析的方法學進展
趙路陽1a,張康2,顧成磊1a,3,葉明俠1a,范文生1a,韓為東1b,孟元光1a
1.解放軍總醫院 a.婦產科;b.基礎研究所,北京 100853;2.北京華信醫院 婦產科,北京 100016;3.解放軍第309醫院 婦產科,北京 100091
microRNA(miRNA)是一類內源性非編碼小RNA,主要通過降解靶基因或抑制靶基因的翻譯而調控表達。由于其作用機制復雜,目前尚未發現高效而低成本的靶標定位方法。近年來,基于堿基互補配對原理的計算機預測法被廣為應用,但此方法假陽性高,不同算法所得結果差異大,會誤導和干擾下游的功能學實驗。因此,有研究者提出結合樣本配對mRNA表達量來進一步定位靶基因,明確miRNA-mRNA相互作用方式,這種聯合分析的方法受到了普遍認可。本文回顧了近年來基于miRNA-mRNA配對表達譜進行聯合分析的方法學進展,并簡要分析各類方法的應用范圍和優缺點,為后續研究者選擇方法提供參考。
microRNA;microRNA靶標預測;microRNA-mRNA相互作用;聯合分析
microRNA(miRNA)是一類長19~22 nt的內源性非編碼小RNA,主要在翻譯水平負調控靶基因的表達[1]。絕大多數miRNA的編碼基因首先在RNA聚合酶Ⅱ的作用下形成初級莖環結構,隨后在Drosha的作用下形成發夾狀的前體miRNA,再由Dicer剪切成為雙鏈miRNA并整合入RNA誘導沉默復合體中。miRNA的5'端含有一段種子序列,可與mRNA的3'非翻譯區(UTR)的調控元件相互作用,如果二者完全匹配,則靶mRNA被復合體降解,若為部分匹配則抑制靶mRNA的翻譯。既往研究發現miRNA參與調控機體多種病理生理學過程,如細胞發育、分化、凋亡,能量代謝,腫瘤發生,心血管疾病,糖尿病等[2]。因此,全面準確地認識miRNA的作用機制并了解相關生物學功能,將有助于揭示疾病的病因,發現診斷、預后標志物,并為靶向治療提供參考和依據。
目前,使用基于堿基互補配對原理的計算機軟件對miRNA靶基因進行預測的方法因成本低,對先驗知識要求少而廣為應用[3]。盡管通過軟件預測可以迅速獲得結果,但該方法的假陽性率和假陰性率高,不同軟件的結果差異較大,對下游功能學實驗造成了干擾和誤導[4]。究其原因,主要是miRNA與靶基因的結合位點短,特異性低,不同算法間極小的差異都會引起結果的巨大變化,同時軟件預測不能考慮某些情形下特異性miRNA和mRNA表達的情況[5]。在此背景下,通過計算機輔助算法整合miRNA和mRNA配對表達譜來確定miRNA-mRNA相互作用(miRNA-mRNA interactions,MMIs)的方法應運而生。由于miRNA表達發生改變時,其所調控的mRNA表達譜也隨之出現相應的表達改變,所以利用miRNA與mRNA的配對表達譜可以高精度地分辯出功能性miRNA的靶向關系。在此,我們回顧了近年來miRNA-mRNA聯合分析的方法學進展,并簡要分析各類方法的應用范圍和優缺點,希望能為后續MMIs研究者選擇分析方法提供參考。
1 MMIs分析流程
基于表達譜的MMIs分析主要包括數據的預處理、計算機分析和結果驗證三部分(圖1)。從數據庫或高通量等方法獲得的miRNA和mRNA表達量經過數據標準化、差異表達分析等預處理后,再根據實驗目的、標本數量等條件選擇合適的計算機分析方法進行統計學計算,獲得陽性的MMIs后采用RT-qPCR、干擾試驗、螢光素酶等實驗進一步加以驗證。本文重點對計算機分析這一步驟進行綜述。
2 MMIs常見分析方法
基于配對表達譜的MMIs聯合分析根據統計方法的差異可分為以下6類:相關性分析法、回歸分析法、貝葉斯推理法、因果推論法、其他方法和綜合分析法(表1)。由于每種方法的側重點不同,各有優勢,所以研究人員應根據數據類型和研究目選擇最佳的分析方法。
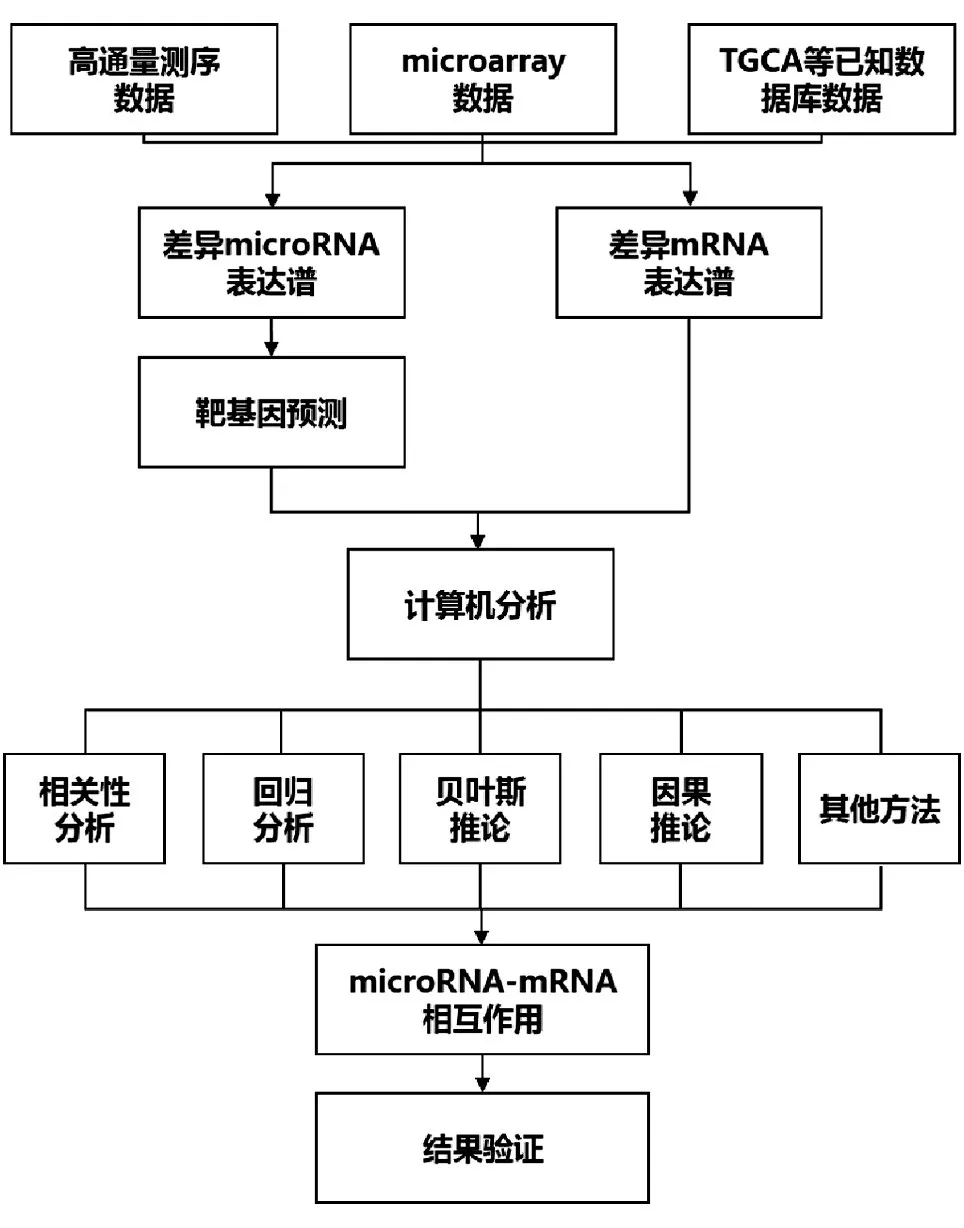
圖1 MMIs分析流程圖

表1 miRNA-mRNA常見分析方法
2.1 相關性分析
相關性分析是對2個或多個變量進行分析,以衡量變量間關系密切程度的一類統計方法。在MMIs中,miRNA主要對mRNA起負向調控作用(圖2A),因此重點關注負相關系數的結果,其絕對值越大說明相關性越強。Pearson相關系數是最常用來反映2個正態分布的變量間線性關系的統計量[6]。在計算MMIs時,須提供5例以上樣本的配對數據進行分析;當樣本量較少(3~5例)時,可采用Spearman相關系數方法進行分析。Pear?son和Spearman方法的優勢是計算簡便、結果明確,但只能反映變量間的線性關系,而實際數據中變量間存在大量非線性關系,為此,有研究者提出使用互信息(mutual information,MI)和最大信息數(maximal information coefficient,MIC)等模型來衡量變量間的非線性強度。MI是信息論中的一種度量方法,關注2個隨機變量之間的相互性,可以看成是一個隨機變量中包含的關于另一個隨機變量的信息量,適用于大樣本量的分析[7],在MMIs分析中需要提供20例以上的配對表達譜(n>20)。MIC是在MI的基礎上優化發展起來的一種分析方法,具有普適性、公平性、對稱性等優勢,當樣本量足夠時,可以捕獲多種函數關系,被稱為大數據時代相關性分析的最佳算法[8]。
2.2 回歸分析
回歸分析是判別因變量和自變量間關系的重要方法之一,它可以估計2個或2個以上變量間的關系,明確多個自變量對因變量的影響強度,構建預測模型。在實際MMIs中,1個miRNA可能調控多個靶基因,反之1個基因也可能被多個miRNA調控(圖2B)。因此,單純的相關性分析不能全面反映某種特定情況下miRNA-mRNA的實際關系,而回歸分析則彌補了這種不足,更符合miRNA-mRNA調控的真實情況。Jayaswal等首次用最小二乘法回歸對miRNA和mRNA表達值進行了MMI分析[9],隨后Li等完善了部分算法并建立了結腸癌特異性的miRNA-mRNA調控網絡[10]。近來,因為Lasso回歸、Ridge回歸和Elasticnet等3種分析方法能應對多重共線性數據,而被廣泛應用于高通量數據相關性的預測中[11-13]。

圖2 不同分析方法預測miRNA-Target相關性的示意圖
2.3 貝葉斯推理
傳統的預測方法在預測過程中大多只利用模型和樣本數據2種信息,而貝葉斯推理應用了決策者的主觀信息,是對基于假設的先驗概率進行修正的一種統計學方法。在MMIs分析中應用的預測假設是:①miRNA對mRNA起負調控作用;②當多個miRNA作用于同一個靶標時,對mRNA表達水平的影響是線性疊加。2007年,Huang等報道了一種運算法則——GenMiR++,他們用這種算法獲得了一張包含104個miRNA和1597個靶標的網絡圖,并得出結論:與以序列為基礎的預測相比,GenMiR++預測與基因注解有更高的一致性,是更精確的預測方法[14]。之后,他們在原有版本基礎上完善了評估特異性序列(如高AU含量、雜交能量等)的功能,升級為GenMiR3模型[15]。由于GenMir++引入的不同推論的最大期望算法耗時很長,Su等引入了馬爾可夫鏈蒙特卡洛算法,開發了HCtarget模型,增加了計算的時效性[16]。
2.4 因果推論
因果推論在相關性的基礎上強調原因先于結果的特性,其應用于MMI分析的理論依據是:相關分析、回歸分析和貝葉斯推理等研究方法關注miRNA-mRNA的相關性,而miRNA和mRNA之間的強相關性可能是由于mRNA對miRNA的調控作用或其他轉錄因子對二者共同的調控而產生的協同變化,不能說明二者之間的因果關系。經典的因果推論方法是在對照研究中通過敲除miRNA后觀察靶mRNA表達發生的變化,但該方法耗時、耗力、耗錢。2000年Pearl等首次提出應用do-calculus方法對觀測數據進行因果預測[17]。這種方法基于有向無環圖(directed absent graph,DAG)的因果框架,但在實際計算中有時難以獲得DAG,因此該方法不具有普適性。Maathuis等提出了一種名為IDA的方法,彌補了必須提供DAG結構的缺陷[18]。Le等認為miRNA與mRNA互作方式復雜,miRNA在調控mRNA的同時也會調控某些非編碼RNA,包括其他miRNA。因此,他們在IDA的基礎上增加了多對多的因果關系算法,并在miRNA敲除試驗中得到了驗證[19]。
2.5 其他方法
Z-score是用來預測基因敲除試驗結果的一種網絡方法,在敲除特定的miRNA后,可以計算出發生改變的基因偏離標準值的情況[20]。在分析MMI時,基于假設錄入的miRNA的表達量為最低值,以此模擬miRNA被敲除的情況,再對應已知mRNA的表達量計算得分。Li等認為,盡管回歸分析考慮了多個miRNA對同一個mRNA的競爭調控作用,但未考慮多個mRNA對同一個miRNA的競爭作用。當一個miRNA具有大量靶基因時,因為需要競爭靶結合位點,對每個靶標的調控作用將會被“稀釋”。于是,他們開發了ProMISe模型,側重預測mRNA與miRNA間的競爭性調節關系[21]。近來,Zoh等提出用典型相關分析方法很可能忽略二代測序中低表達(1~2個counts)數據間的強相關性,于是在Pearson相關系數和貝葉斯算法的基礎上設計出PCAN模型,用于估算低表達量間的相關性[22]。
2.6 綜合分析法
為了完整準確地預測miRNA-mRNA互作網絡,Le等將多種方法組合后進行綜合分析,采用波達計數法排列統計,最終提供研究者最為關注的Top k靶標列表(k值由研究者決定)。他們選用NCI-60數據庫中的EMT(Epithelial to Mesen?chymal Transition)數據、MCC(Multi-class cancer)數據庫中的多種癌癥數據及BR51(51 human breast cancer cell lines)數據庫中不同乳腺癌亞型的數據來綜合比較不同綜合分析方法的優缺點 ,利 用 Tarbase、miRecords、miRWalk 和 miRTar?Base等4個數據庫交集的結果驗證預測結果,最后得出Pearson+IDA+Lasso的組合是最優選的綜合分析法的結論[23]。但由于綜合分析法應用少,尚無足夠證據證明綜合分析法優于單一分析法。
3 MMIs分析常用軟件
上述分析方法均可在相應的參考文獻中找到對應的算法公式或開發的軟件包供研究人員下載使用,以下重點對3個常用軟件進行介紹。
MAGIA是由Bisognin等開發設計的一個MMI統計分析的網絡軟件,目前已升級到MAGIA2版本(http://gencomp.bio.unipd.it/magia2)[24-25]。使用者上傳miRNA和對應的mRNA表達譜后,可以在8個miRNA靶標預測數據庫(Microcosm、microrna.org、DIANA-microT、miRDB、PicTar、PITA、RNA22、TargetScan)中選擇交集或并集的靶基因,再根據數據量、研究方向選擇Spearman correlation、Pear?son correlation、MI(適用于20例以上的大樣本)和Meta-analysis(適用于非配對樣本)等4種統計方法中的一種進行關聯分析,結果提供交互網絡圖和表格下載。MAGIA軟件具有操作簡單、使用方便、可直接提供調控網絡圖的輸出等優點,但統計方法相對單一。
TaLasso是Muniategui等基于Lasso模型建立的網絡分析軟件(http://talasso.cnb.csic.es/)[26],需要研究者提供配對的miRNA和mRNA表達譜。在靶基因預測環節,提供 miRGen、miRBase、miRan?da、TarBase、miRecords和miRWalk等6個數據庫可供選擇。在聯合分析時,除Lasso回歸外,還可以選擇GenMiR++或Pearson系數2種分析方法,結果提供得分和P值,并以表格形式輸出。如果繪制網絡圖片,則需要使用Cytospace等繪圖軟件對結果進行二次制作。
miRLAB是Le等設計的一款基于R語言的軟件包(http://bioconductor.org/packages/release/bioc/ht?ml/miRLAB.html)[27],下載安裝后可在本地進行全部分析內容。該軟件提供了3個基礎數據庫包,也可直接通過模塊中TCGA數據庫中的鏈接進行下載,或上傳自備數據。獲得原始數據后還可進行標準化、差異分析等預處理。在數據分析模塊中,提供了12種MMI分析方式(Pearson相關、Spearman相關、Kendall等級相關、距離相關、Hoeffding相關、隨機相關系數、MI、Lasso回歸、Elastic-net回歸、IDA、Z-score和 ProMISe)以供選擇。在數據整合環節,在常規靶基因預測基礎上增加了HITS-CLIP、PAR-CLIP和iCLIP的分析方式,可以直接揭示miRNA分子與其結合蛋白的相互作用。在結果驗證環節,提供了既往驗證的miRNA數據庫和干擾試驗。此外,還提供GO和KEGG等下游分析模塊。與MAGIA和TaLasso相比,miRLAB的優勢在于MMI分析方式多,使用范圍廣,但須掌握一定的R語言基礎知識方能進行操作。
4 結語
目前人們對miRNA的研究仍處于早期探索階段,大部分研究還局限于通過生物信息學軟件結合miRNA的部分特征對其靶標和功能進行預測。但這種方法假陽性率高,不具有普遍適用性,預測效果并不樂觀。隨著高通量技術的廣泛應用,利用miRNA和mRNA表達譜進行聯合分析越來越受到人們的認可并逐漸普及。盡管現有模型預測的結果仍然不夠準確,且需要下游實驗驗證,但隨著對miRNA作用機制的深入研究,基于配對表達譜進行聯合預測的方法也在日漸豐富和完善。相信在不久的將來,在多學科研究人員的共同努力下,MMIs將會出現更加便捷、全面、準確的研究方法。
[1] Rana T M.Illuminating the silence:understanding the structure and function of small RNAs[J].Nat Rev Mol Cell Biol,2007,8(1):23-36.
[2] Hobert O.Gene regulation by transcription factors and miRNAs[J].Science,2008,319(5871):1785-1786.
[3] Enright A J,John B,Gaul U,et al.microRNA tar?gets in Drosophila[J].Genome Biol,2003,5(1):R1.
[4] Rajewsky N.miRNA target predictions in animals[J].Nat Genet,2006,38(Suppl):S8-13.
[5] Farazi T A,Spitzer J I,Morozov P,et al.miRNAs in human cancer[J].J Pathol,2011,223(2):102-115.
[6] Speed T.Mathematics.A correlation for the 21st cen?tury[J].Science,2011,334(6062):1502-1503.
[7] Moon Y I,Rajagopalan B,Lall U.Estimation of mutu?al information using kernel density estimators[J].Phys Rev E Stat Phys Plasmas Fluids Relat Interdiscip Top?ics,1995,52(3):2318-21.
[8] Reshef D N,Reshef Y A,Finucane H K,et al.De?tectingnovel associationsin largedatasets[J].Sci?ence,2011,334(6062):1518-1524.
[9] Jayaswal V,Lutherborrow M,Ma D D,et al.Identifi?cation ofmiRNAswith regulatorypotentialusinga matched miRNA-mRNA time-course data[J].Nucleic Acids Res,2009,37(8):e60.
[10]Li X,Gill R,Cooper N G,et al.Modeling miRNA-mRNA interactions using PLS regression in human co?lon cancer[J].BMC Med Genomics,2011,4:44.
[11]Ragan C,Zuker M,Ragan M A.Quantitative predic?tion of miRNA-mRNA interaction based on equilibri?um concentrations[J].PLoS ComputBiol,2011,7(2):e1001090.
[12]Nogales-Cadenas R,Carmona-Saez P,Vazquez M,et al.GeneCodis:interpreting gene lists through enrich?ment analysis and integration of diverse biological in?formation[J].Nucleic Acids Res,2009,37(Web Server issue):W317-322.
[13]Muniategui A,Pey J,Planes F J,et al.Joint analysis of miRNA and mRNA expression data[J].Brief Bioin?form,2013,14(3):263-278.
[14]Huang J C,Babak T,Corson T W,et al.Using ex?pression profiling data to identify human miRNA tar?gets[J].Nat Methods,2007,4(12):1045-1049.
[15]Huang J C,Frey B J,Morris Q D.Comparing se?quence and expression for predicting miRNA targets using GenMiR3[C]//Proceedings of the Pacific Sympo?sium.Biocomputing 2008.2008:52-63.
[16]Su N Wang Y,Qian M,et al.Predicting microRNA targets by integrating sequence and expression data in cancer[JC]//IEEE Int Conf Syst Biol.2011:219-224.
[17]PearlJ.Causality:models,reasoning,and inference[M].Cambridge University Press,2000:384.
[18]Maathuis M H,Colombo D,Kalisch M,et al.Predict?ing causal effects in large-scale systems from observa?tional data[J].Nat Methods,2010,7(4):247-248.
[19]Le T D,Liu L,Tsykin A,et al.Inferring miRNA-mRNA causal regulatory relationships from expression data[J].Bioinformatics,2013,29(6):765-771.
[20]Prill R J,Marbach D,Saez-Rodriguez J,et al.To?wards a rigorous assessment of systems biology mod?els:the DREAM3 challenges[J].PLoS One,2010,5(2):e9202.
[21]Li Y,Liang C,Wong K C,et al.Inferring probabilis?tic miRNA-mRNA interaction signatures in cancers:a role-switch approach[J].Nucleic Acids Res,2014,42(9):e76.
[22]Zoh R S,Mallick B,Ivanov I,et al.PCAN:probabilis?tic correlation analysis of two non-normal data sets[J].Biometrics,2016,72(4):1358-1368.
[23]Le T D,Zhang J,Liu L,et al.Ensemble methods for miRNA target prediction from expression data[J].PLoS One,2015,10(6):e0131627.
[24]Bisognin A,Sales G,Coppe A,et al.MAGIA(2):from miRNA and genes expression data integrative analysis to miRNA-transcription factor mixed regulatory circuits[J].Nucleic AcidsRes,2012,40(Web Serverissue):W13-21.
[25]Sales G,Coppe A,Bisognin A,et al.MAGIA,a webbased tool for miRNA and genes integrated analysis[J].Nucleic AcidsRes,2010,38(Web Serverissue):W352-359.
[26]Muniategui A,Nogales-Cadenas R,Vazquez M,et al.Quantification of miRNA-mRNA interactions[J].PLoS One,2012,7(2):e30766.
[27]Le T D,Zhang J,Liu L,et al.miRLAB:an R based dry lab for exploring miRNA-mRNA regulatory rela?tionships[J].PLoS One,2015,10(12):e0145386.
Progressin the Integrative AnalysisofmicroRNA and mRNA Expression Data
ZHAO Lu-Yang1a,ZHANG Kang2,GU Cheng-Lei1a,3,YE Ming-Xia1a,FAN Wen-Sheng1a,HAN Wei-Dong1b,MENG Yuan-Guang1a*
1.a.Department of Gynecology and Obstetrics;b.Institute of Basic Medicine;Chinese PLA General Hopsital,Bei?jing 100853;2.Department of Gynecology and Obstetrics,Beijing Huaxin Hospital,Beijing 10016;3.Department of Gynecology and Obstetrics,PLA 309 Hospital,Beijing 100091;China
*Corresponding author,E-mail:meng6512@vip.sina.com
microRNAs(miRNAs) are endogenous non-coding small RNAs that interact with their mRNAs by de?grading or inhibiting translation of the targets.Up to date,there is still no low-cost and effective miRNA target screening method because the regulatory mechanisms are complex.In recent years,some investigators built several computational methods based on sequence complementarity of the miRNA and the mRNAs.However,the results are various out of different algorithms and have huge rate of false positives,which caused great troubles in down?stream experiments.Therefore,methods of using the expression values of miRNAs and mRNAs to refine the re?sults has been proposed,which have shown to effectively identifying the most prominent interactions.Here,we summarized these methods that combining both miRNA expression values and mRNA expression values and to pre?dict miRNA targets,outlined the advantages and disadvantages of different methods,and provided directions for fu?ture investigation.
microRNA;microRNA target prediction;microRNA-mRNA integration;integrative analysis
Q78;Q811.4
A
1009-0002(2017)04-0545-06
2016-12-22
國家自然科學基金面上項目(81571411)
趙路陽(1988- ),女,博士研究生,(E-mail)zlynfyd@163.com
孟元光,(E-mail)meng6512@vip.sina.com
10.3969/j.issn.1009-0002.2017.04.029

