若爾蓋高原濕地泥炭沼澤土亞硝酸鹽還原酶(nirK)反硝化細菌群落結構分析
王鎣燕, 盧圣鄂, 陳小敏, 李躍飛, 辜運富
四川農業大學資源學院,成都 611130
若爾蓋高原濕地泥炭沼澤土亞硝酸鹽還原酶(nirK)反硝化細菌群落結構分析
王鎣燕, 盧圣鄂, 陳小敏, 李躍飛, 辜運富*
四川農業大學資源學院,成都 611130
若爾蓋泥炭濕地是世界少有的低緯度永久凍土濕地,具有高海拔、高紫外輻射、高有機質的特點。該區域N2O的排放量對全球氣候變暖有重要影響。對若爾蓋高原濕地泥炭沼澤土中的亞硝酸鹽還原酶(nirK)反硝化細菌群落結構多樣性進行分析,以期揭示該區域N2O釋放的微生物調控機制。基于若爾蓋高原濕地泥炭沼澤土的理化性質和反硝化活性(PDA),結合限制性酶切片段長度多態性(Restriction fragment length polymorphism, RFLP)技術、克隆文庫及分子測序對該生態系統中的nirK反硝化細菌群落結構及多樣性進行分析。反硝化活性測定結果顯示:阿西地區>麥溪地區>分區地區,反硝化活性與土壤有機碳、總氮和豐富度呈顯著正相關(P<0.05)。Shannon-wiener多樣性指數以阿西最高、分區最低。3個樣品中共測序15條nirK基因代表序列,系統發育表明若爾蓋高原濕地優勢nirK反硝化菌群為變形門菌群。其中,阿西地區主要為α-變形菌門,麥溪地區主要為β-變形菌門,分區地區無法確定優勢種群。冗余分析(Redundancy analysis, RDA)顯示:有效鉀和有效磷是影響nirK反硝化細菌群落結構的關鍵環境因子。本論文顯示,若爾蓋高原濕地存在著明顯的反硝化作用,調控這些反硝化作用的nirK反硝化細菌多樣性較高,且與土壤有效鉀和有效磷密切相關。
若爾蓋高原濕地;泥炭沼澤土;反硝化活性;nirK;多樣性
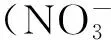
濕地,被稱作為“地球之腎”,在調控全球元素的生物地球化學循環中起著重要作用。若爾蓋高原濕地位于青藏高原東北隅,是世界上為數不多的低緯度永久凍土濕地,具有高海拔、高紫外輻射、高有機質含量等特點[10]。同時,若爾蓋高原濕地也是我國乃至全球面積最大的高原泥炭沼澤群[11],是中國生物多樣性研究關鍵地區,也是全球氣候變化的熱點地區[12]。目前,研究人員針對該區域的碳循環特征[13- 14]及相關微生物調控機制[15]進行了較系統地研究,而對該區域溫室氣體N2O排放的微生物調控機制研究卻鮮見報道。
本文結合限制性酶切片段長度多態性(RFLP)技術、克隆文庫及測序等手段對若爾蓋高原濕地泥炭沼澤土中nirK反硝化細菌的群落結構和多樣性進行分析,并通過冗余分析(RDA)來探索影響nirK反硝化細菌的關鍵環境因子。以期揭示若爾蓋高原濕地生態系統反硝化活性與反硝化菌群間的內在聯系,為深入認識該生境的反硝化作用微生物調控機制提供基礎理論依據。
1 材料與方法
1.1 研究去概況
若爾蓋高原濕地位于青海-西藏高原東部邊緣的四川、甘肅和青海省的三省交界地區(32°20′—34°00′N,101°30′—103°30′E;面積3.94×104hm2,平均海拔高度3500 m)。在青藏高原影響下該地區特殊的大陸性高原氣候環境,四季不分,長冬無夏,降水多,濕度大,霜凍期極長。年平均溫度為1.1 ℃ (年最高溫度為10.9 ℃,年最低溫度-10.3 ℃),年平均降水量為560—860 mm[16]。沼澤土全年被水和植被覆蓋,主要植被為水生植物和莎草科的水生-中生植物 (木里苔草、毛果苔草和烏拉苔草),土壤母質為均質粉砂和粘土[10]。相關采樣點信息見表1。
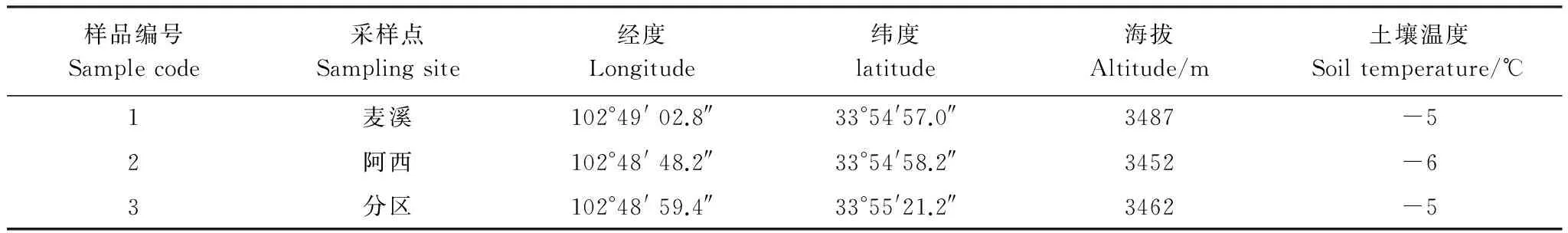
表1 采樣點信息
1.2 采樣方法
2012年8月在查閱有關資料的基礎上,選取阿西、麥溪和分區3個面積最大的典型牧區為研究區域,采集這3個區域被烏拉苔草覆蓋的泥炭沼澤土為研究樣品,按照“梅花型”原則進行采樣,樣地面積約0.15 hm2,布設3個采樣點,用土鉆取10—20 cm的土樣,按四分法進行混勻,分別取1 kg混后土樣,得到三等份樣品,用無菌PET樹脂袋封裝,放于冰盒中帶回實驗室。土樣于-20 ℃冰箱保存以備后續分析。
1.3 土壤理化性質與反硝化活性
土壤含水量(WC)在105 ℃烘干48 h后檢測[17]。有效氮(AN)通過堿解擴散法進行分析[18]。土壤有機質(SOM) 、pH值、總氮(TN)、總磷(TP)、有效磷(AP)、總鉀(TK)、有效鉀(AK)采用Liu等人[19]的方法進行分析。土壤理化性質見表2。
土壤反硝化活性測定采用乙炔抑制法[20]:稱取新鮮土壤樣品25 g (以干土計) 放入250 mL 三角瓶,添加25 mL溶液(包含1 mmol/L 葡萄糖和1 mmol/L KNO3)。用惰性氣體氦氣沖洗三角瓶,抽取瓶中土樣上方10%的氣體,添加抽取氣體同體積的無丙酮乙炔氣體,同時以不加乙炔氣體的樣品為對照。在20 ℃、225 r/min條件下振蕩土壤泥漿,在1 h和5 h用注射器抽取氣樣,用氣相測譜儀分析氣樣。
N2O濃度測定:N2O濃度用美國安捷倫公司生產的Agilent- 7890A氣相色譜儀測定,檢測器為電子捕獲器,分離柱內填充是Porapak Q,計算機程序控制定量管(1 mL)和通氣閥自動進樣,將空氣樣品中水分在進入檢測器前反吹掉,載氣為甲烷-Ar混合氣,流速為60 mL/min。分離柱溫度、進樣口溫度和檢測器溫度分別為60 ℃、100 ℃和300 ℃。
1.4 土壤總DNA提取
利用Fast DNA spin kit for soil 試劑盒(MP BIO, Inc., Irvine, CA, USA)根據制造商提供的說明進行提取。洗脫后總DNA體積為50 μL。利用1%的瓊脂糖凝膠電泳確認總DNA的條帶的單一性,同時用Nano- 200 核酸檢測儀(AoSheng, Hangzhou, China)檢測濃度。
1.5 nirK基因的擴增
所用引物為:nirK517F (5′-TTYGTSTAYCACTGCGCVCC- 3′)和nirK1055R (5′-GCYTC- GATCAGRTTR TGGTT- 3′)[21]。PCR擴增程序采用TOUCHDOWN程序。選用50 μL反應體系,分別是PCR Mix 25L,DNA模板取6L,引物(10mol/L)各1 μL,超純水補足50L。擴增程序:95 ℃預變性5 min,94 ℃ 30 s,57— 47℃ 45 s(每個3循環溫度降低2 ℃),72 ℃ 1 min,15個循環;其它條件不變,在47 ℃退火溫度下繼續擴增25個循環,終延伸72 ℃ 7 min。取產物2.0 μL在含EB的1%瓊脂糖凝膠上水平電泳檢測,120 V電泳30 min。用凝膠成像系統(Bio-Rad, USA)拍照檢測。PCR產物置于4 ℃冰箱保存。
1.6 克隆與RFLP分析
利用PCR Clean-UpTM試劑盒(MO BIO Labs, Solana Beach, CA, USA)對擴增PCR產物進行純化,純化產物通過pGEM-T載體(Promega)進行克隆。根據 “藍白斑”(抑制劑:X-Gal、Ampr和IPTG)對克隆子進行篩選。3個樣品平板培養基共挑取270個白色克隆子。利用nirK基因引物(如1.5所示)進行擴增,獲得250個nirK基因陽性克隆子。
將PCR產物用MspI和AvaII限制性內切酶(Promega)進行雙酶切。酶切體系總體積為10L,包括:2 U的限制性內切酶、8L的PCR產物和限制緩沖劑。酶切產物通過瓊脂糖凝膠電泳80V 60min進行分離,所用瓊脂糖凝膠濃度為2%。
根據限制性片段多樣性條帶的類型,選取15個代表克隆子送上海生工生物有限公司進行序列測定并將獲得的基因序列提交NCBI以獲取基因登錄號。序列登錄號為:KX018494-KX018508。
1.7 數據處理
基礎數據處理利用Excel 2010進行,單因素方差分析和皮爾遜相關分析利用SPSS 21.0 (SPSS Inc. Chicago)完成。nirK反硝化細菌群落結構多樣性通過Shannon多樣性指數(H),豐富度(S)、均勻度(Eh)[22]和覆蓋率(C)[23]進行計算。利用MEGA 5.0 軟件,通過最大相鄰法構建nirK基因序列系統發育樹。利用CANOCO5.0 軟件對土壤理化性質和nirK基因反硝化群落結構進行冗余分析(RDA)。
2 結果與分析
2.1 土壤理化性質與反硝化活性
3個土壤樣品的理化性質見表2。其中,阿西地區樣品的水含量、反硝化活性、土壤有機質、總氮和有效氮最高;而麥溪地區樣品pH值、總磷、總鉀、有效磷和有效鉀是最高的。相較于以上兩個地區樣品,分區地區樣品的理化性質值相對較低。
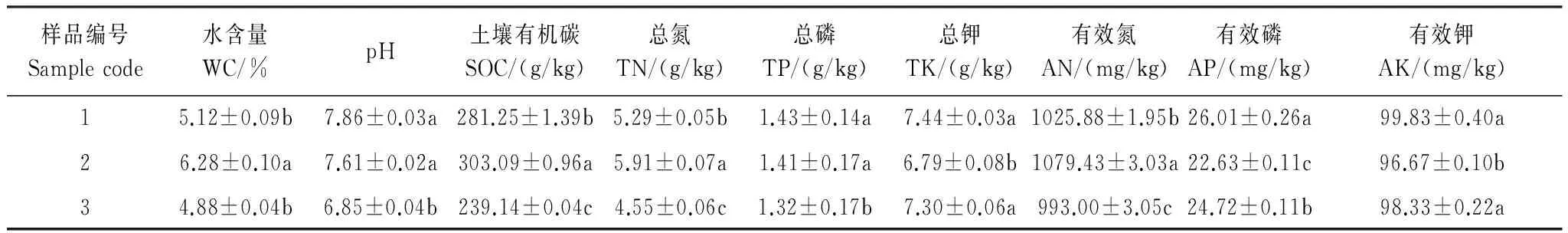
表2 若爾蓋高原泥炭沼澤土理化性質
WC:Water content;SOC:Soil organic carbon;TN:Total nitrogen;TP:Total phosphorus;TK:Total potassium;AN:Available nitrogen;AP:Available phosphorus;AK:Available potassium;結果:平均值±標準誤;每欄數據在P< 0.05水平下的顯著差異
反硝化活性范圍為:33.52—52.77 ng N2O-N g-1ds h-1,最高值出現于阿西地區,最低值位于分區地區(圖1)。皮爾遜相關分析顯示:反硝化活性與土壤有機碳、總氮和豐富度呈顯著正相關(表3)。
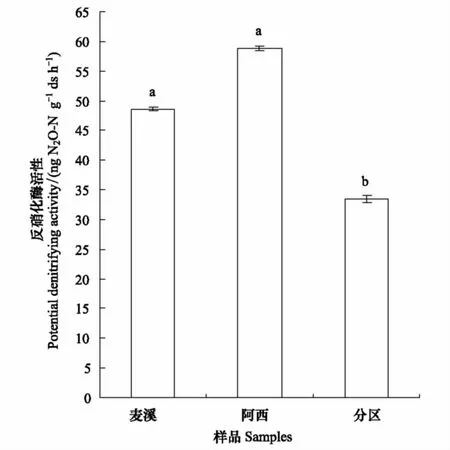
圖1 若爾蓋高原濕地泥炭沼澤土的反硝化活性 Fig.1 Potential denitrifying activity of the peat marsh soil in Zoige plateau wetland圖中小寫字母表示反硝化活性在P < 0.05水平上的顯著差異
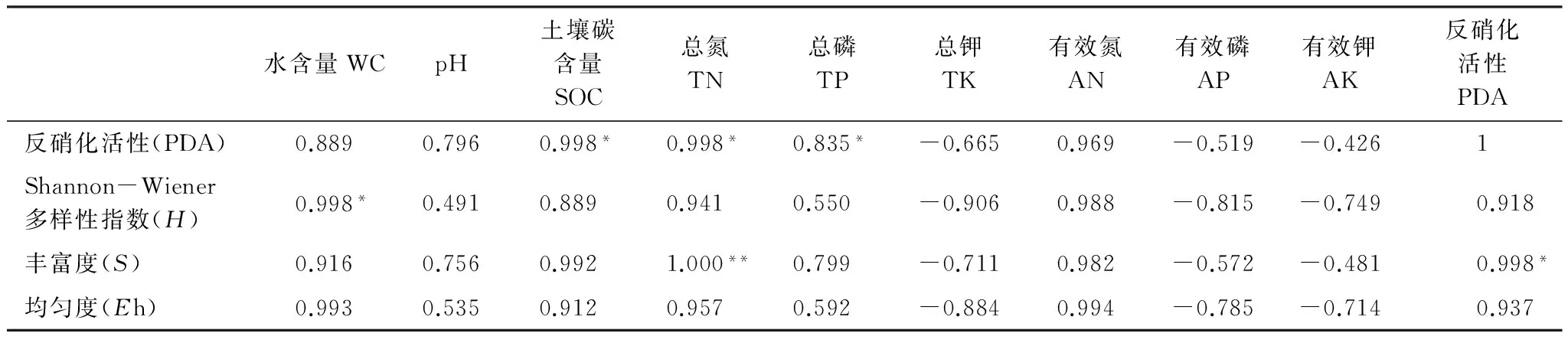
水含量WCpH土壤碳含量SOC總氮TN總磷TP總鉀TK有效氮AN有效磷AP有效鉀AK反硝化活性PDA反硝化活性(PDA)0.8890.7960.998*0.998*0.835*-0.6650.969-0.519-0.4261Shannon-Wiener多樣性指數(H)0.998*0.4910.8890.9410.550-0.9060.988-0.815-0.7490.918豐富度(S)0.9160.7560.9921.000**0.799-0.7110.982-0.572-0.4810.998*均勻度(Eh)0.9930.5350.9120.9570.592-0.8840.994-0.785-0.7140.937
WC:Water content;SOC:Soil organic carbon;TN:Total nitrogen;TP:Total phosphorus;TK:Total potassium;AN:Available nitrogen;AP:Available phosphorus;AK:Available potassium;PDA:Potential denitrifying activity;表中數據為:相關系數(r);*P< 0.05; **P< 0.01
2.2 nirK基因群落文庫和限制性片段長度多態性分析
限制性片段多態性分析顯示(表4),3個地區共包括15個OTUs,不同地區所包含的OTU類型存在一定差異。其中阿西地區所包含OTU類型最多,麥溪次之,分區最少。其中OTU1、2、5、8、11、12為3個地區共有,OTU13、OTU14為阿西地區特有;OTU7為麥溪地區特有;OTU15為分區地區特有。
克隆文庫的覆蓋率用于分析土壤微生物群落的多樣性,文庫中的特有OTU類型越多則多樣性的覆蓋率越低[24]。本文3個地區各自的克隆文庫的覆蓋率都高于91%(表4),充分說明所構建的克隆文庫幾乎全部覆蓋地區里的nirK基因種類。同時,克隆文庫也可以代表若爾蓋高原濕地泥炭沼澤土中nirK反硝化群落的特征。
基于RFLP數據計算nirK基因群落的Shannon多樣性指數(H)、豐富度(S)以及均勻度(Eh)(表4)。nirK基因反硝化細菌群落多樣性在3個采樣點的差異不明顯。Shannon-Wiener多樣性指數的變化范圍為:2.91—2.49,其中阿西采樣點多樣性指數、豐富度和均勻度最高,麥溪次之,分區最小。通過表3皮爾遜相關分析顯示:香儂-威爾多樣性指數與水含量顯著相關(P<0.05)。豐富度與總氮極顯著相關(P<0.05)。而均勻度與各理化性質的相關性不明顯。
2.3 系統發育地位分析
基于RFLP圖譜,同一RFLP類型下選取一個克隆序列作為一個OTU。已測序的15個陽性克隆序列長度均約為530 bp。根據Palmer的方法[25],將82%作為序列相似性標準,可以將nirK序列定位到屬的水平。系統發育分析顯示:15條序列中,10條與變形門細菌相似(圖 2),包括 Alpha-Proteobacteria和Beta-Proteobacteria兩個綱。還有5條序列無法確定其分類地位。
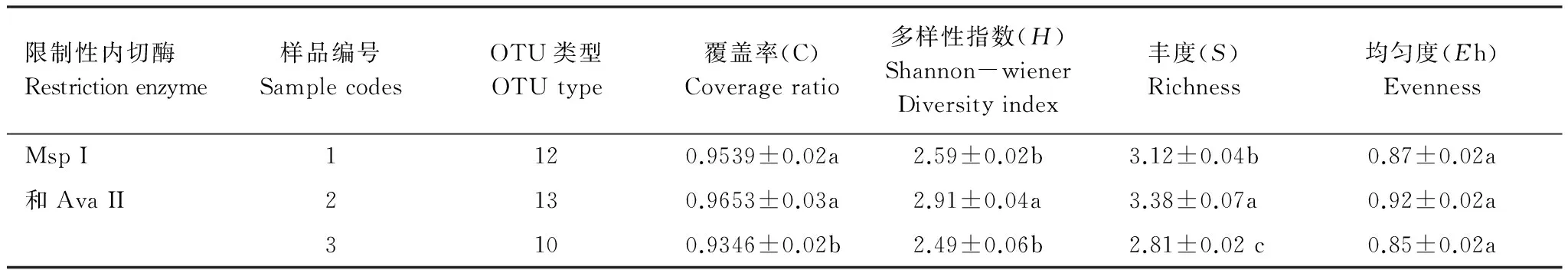
表4 若爾蓋高原濕地泥炭沼澤土nirK反硝化細菌多樣性
結果為平均值±標準誤;每欄數據在P< 0.05的水平下顯著差異
Alphaproteobacteria綱是若爾蓋泥炭沼澤土最優勢的nirK反硝化菌群,占克隆子總數的 51.0%,有5個OTU屬于這個類群(Cluster3、Cluster4)。其中,豐度最高的OTU12,在250個正確插入片段的克隆子中占有43個,與GenBank可培養反硝化細菌Rhodopseudomonassp. 2—8 (GU332847)在核酸水平上具有85%的序列相似性。此代表序列來自不同的生境,如沼澤土、農田土壤、酸性土壤、水稻土等。豐度第二的OTU14包括27個克隆子,占克隆子總數的10.8%,多來源于海底底泥、農業土壤等。OTU13的源于城市污水處理廠。此外,屬于OTU12、OTU13和OTU14的克隆子主要來自阿西地區,顯示阿西采樣點中的優勢種群屬于Alphaproteobacteria綱。
Betaproteobacteria綱包含5個OTU,占克隆子總數的38%。OTU8最豐富,該OTU占克隆子總數的14.4%,與可培養反硝化細菌Achromobacterxylosoxidan(AB969826)具有84%的序列相似性。其相似序列來源于草甸土和農田土壤,相似度在 82%—89%之間。此外, OTU6—9的克隆子大部分來自麥溪地區,顯示麥溪采樣點中的優勢種群屬于Betaproteobacteria綱。還有部分OTU無法確定其分類地位(Cluster 1),占克隆子總數的11%。分區采樣點的克隆序列大多屬于此類。以上OTU相似的序列大多來自于冰川,海灘、高山貧營養湖、農業土壤、河口、內陸湖底泥等,序列相似度在 85%—89%之間。
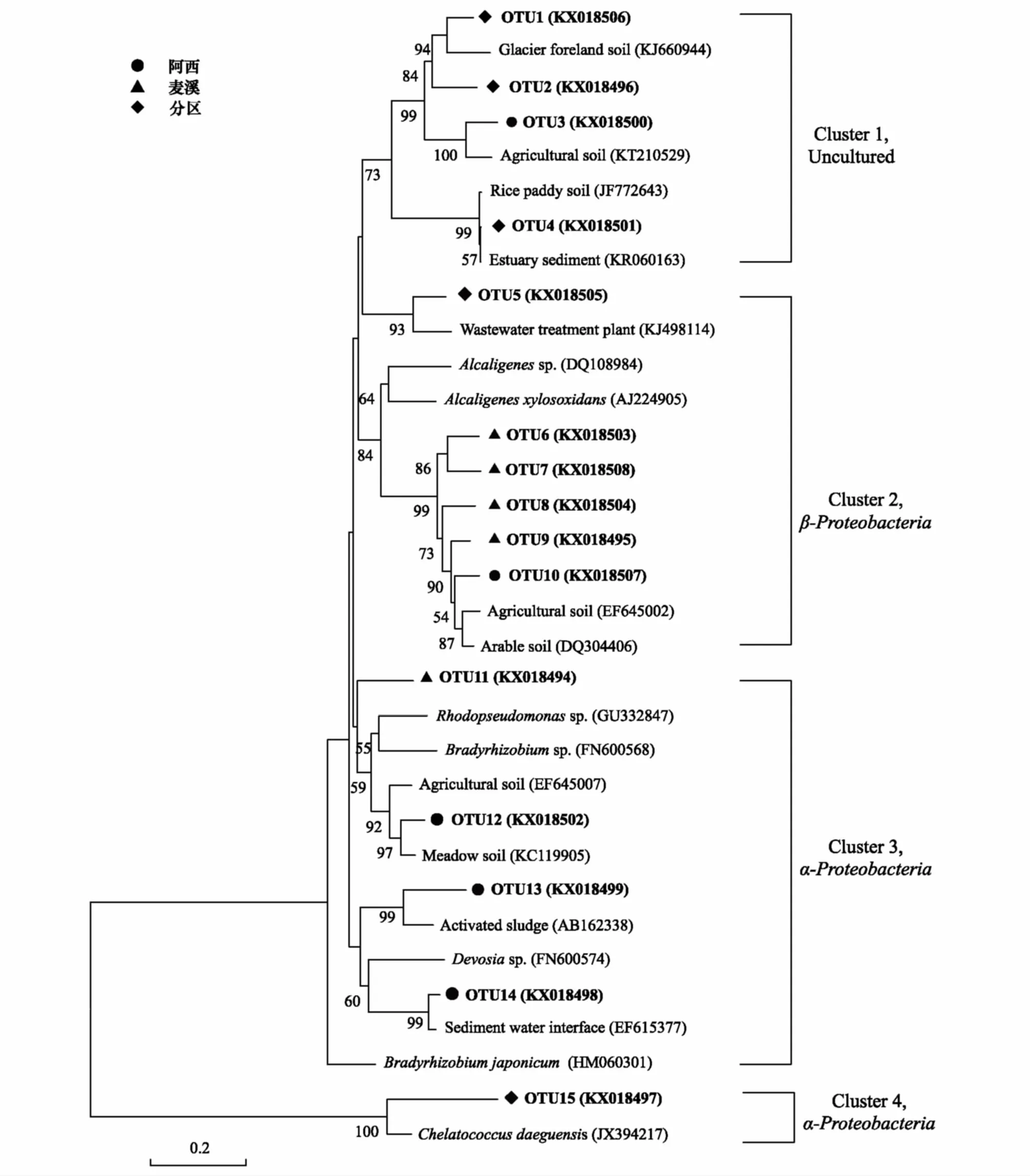
圖2 若爾蓋高原濕地泥炭沼澤土nirK反硝化細菌的系統發育分析Fig.2 Phylogeny analysis of the nirK denitrifiers of the peat marsh soil in Zoige plateau wetland在樹節上的數字代表 1 000取樣的百分比,僅顯示大于50%的情況
2.4 nirK反硝化細菌群落結構與土壤理化性質的冗余分析
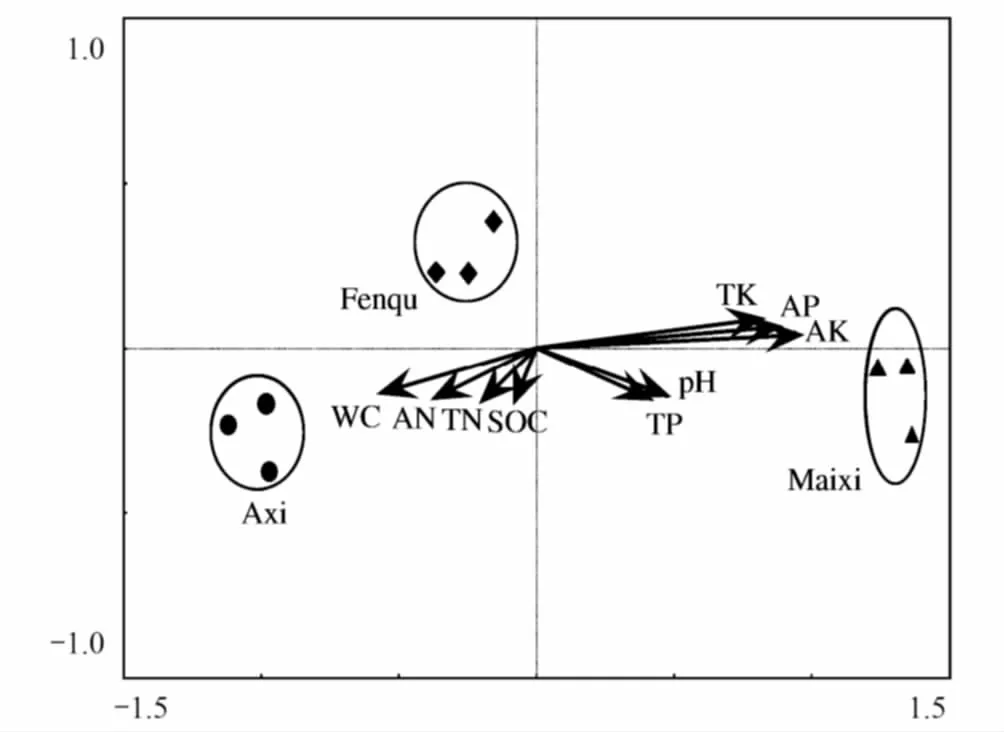
圖3 若爾蓋高原濕地泥炭沼澤土nirK反硝化細菌群落結構的RDA排序圖Fig.3 RDA ordination analysis of the nirK denitrifier communities in the peat marsh soil in Zoige plateau wetlandWC:水分含量 Water content;SOC:土壤有機碳 Soil organic carbon;TN:總氮 Total nitrogen;TP:總磷 Total phosphorus;TK:總鉀Total potassium;AN:有效氮 Available nitrogen;AP:有效磷 Available phosphorus;AK:有效鉀 Available potassium
若爾蓋高原濕地泥炭沼澤土nirK反硝化細菌群落與土壤理化性質之間的RDA相關性見圖3。分析結果顯示:第一排序軸的特征值為0.97,第二排序軸特征值為0.02。由圖可知,第一排序軸與有效磷、有效鉀關系最為密切,均達到極顯著水平(P<0.01)。第二軸與土壤有機碳、總氮、總磷和有效氮較為密切,均達到顯著水平(P<0.05)。總體而言,本實驗中有效鉀、有效磷對nirK基因反硝化群落結構的影響最顯著。此外,由采樣點投射到各理化性質上的投影點可知,阿西地區含高有機質,麥溪地區含高有效鉀、有效磷,分區地區各理化性質居中。而各采樣點的連線段長度表示各采樣點nirK反硝化細菌群落的相似程度,即麥溪地區與其他兩個地區的連接距離較長,則其nirK反硝化菌群有明顯的差異。
3 討論
環境因子如土壤含水量,pH和營養成分等是影響反硝化作用和反硝化細菌種群組成和多樣性的重要因素,本文皮爾遜相關分析顯示:反硝化活性與土壤有機質、總氮和總磷都呈極顯著相關(表3)。已有研究表明[26],反硝化活性的高低取決于提供給細菌生長的有機質的質量與數量。在污水灌溉的農田里檢測反硝化活性結果顯示,影響反硝化活性的主要環境因子是總氮含量[27]。White[28]等人在研究被水覆蓋40a的泥炭沼澤土反硝化活性時發現:總磷含量與反硝化活性呈極顯著相關性。上述研究結果與本文結果相似,顯示若爾高高原濕地的反硝化活性受土壤理化性質特別是有機質、氮素和磷素的有效性的明顯影響。
系統發育分析顯示nirK反硝化菌群主要屬于變形門。此外,若爾蓋高原濕地不同地區泥炭沼澤土中的nirK反硝化菌群存在明顯差異。其中,阿西地區分離的克隆子序列主要屬于α-變形門(Cluster 3- 4),少部分屬于β-變形門。在α-變形門中,以慢生型大豆根瘤菌屬(Bradyrhizobiumjaponicum)、紅假單胞菌屬(Rhodopseudomonas)、蒼白桿菌屬(Ochrobactrum)還有德沃斯氏菌屬(Devosia)為主。與本文結果相似,Priemé等[5]研究土耳其沼澤化潮土中亞硝酸鹽微生物群落得出慢生型大豆根瘤菌 (Bradyrhizobiumjaponicum, AJ002516)是該生境中的優勢反硝化微生物類群。Saito等研究也發現定向施無機肥的水稻土中nirK反硝化菌群除以上慢生型大豆根瘤菌屬外還有紅假單胞菌屬(Rhodopseudomonas, YP782977)以及蒼白桿菌屬(Ochrobactrum, YP00137298)[29]。但德沃斯氏菌屬(Devosia)未見相關報道。麥溪地區反硝化菌群主要為β-變形門,少部分為α-變形門,且主要為β-變形門的產堿桿菌屬(Alcaligenes)。與本文類似,Prieme等人研究土耳其沼澤土壤中的反硝化細菌類群時,也得出β-變形門細菌相對較少[5]。而分區地區的主要反硝化菌群不能確定其種屬地位。結合本文的反硝化活性和系統發育分析結果,若爾蓋高原濕地泥炭沼澤土的反硝化活性可能主要由α-變形門細菌調控。與本文研究相似,Katsuyama等人在研究不同森林土的反硝化活性和調控菌群時亦發現α-變形門的反硝化菌是調控該類生境反硝化活性的主要菌群[30]。綜上,若爾蓋高原濕地泥炭沼澤土中存在多樣化的反硝化菌群,原因可能是這些泥炭沼澤土微域生境條件如理化性質、含水量和含氧量及氧化還原電位存在差異而導致[3]。
環境因子是影響微生物群落結構變化的重要因子,研究土壤微生物群落結構與土壤理化性質間的相互關系對于深入認識土壤微生物在生態系統中扮演的角色和功能具有重要意義[31]。 RDA分析顯示有效鉀和有效磷是影響若爾蓋高原濕地泥炭沼澤土nirK反硝化細菌群落結構的主要因子。Zhang等[32]對青藏高原草甸土nirK反硝化細菌群落研究發現,溫度、水含量以及C/N比是影響nirK反硝化細菌群落結構的主要因子。而Xie等[33]研究青藏高原草甸土上放牧對反硝化細菌群落結構的影響發現土壤有機碳和硝態氮是影響nirK反硝化細菌群落結構的重要因子。而與本文結果相似,如Barta 等[34]研究發現森林土壤中有效磷對nirK反硝化細菌群落結構有顯著影響(r=0.83,P<0.001)。Xue等[35]研究表明有效鉀是影響有機農業土壤nirK反硝化細菌群落的重要因子。此外,相關研究也表明:當有效磷和有效鉀作為土壤限制營養因子時,會誘導溶磷溶鉀微生物分泌相關酶類釋放土壤中的磷、鉀元素,改善土壤微域生境,進而反過來調控這些微生物的種群和數量[36- 37]。綜上,磷、鉀元素作為生命活動的重要營養因子,它們與受微生物調控的氮素生物地球化學循環可能存在非常復雜的作用關系,這些關系的解釋尚須進一步研究證實。
本文分析了若爾蓋高原濕地泥炭沼澤土反硝化活性和nirK反硝化細菌群落結構和多樣性及影響因子。研究顯示nirK基因反硝化菌群主要屬于變形門菌,該區域的反硝化活性主要受α-變形門反硝化菌群影響。土壤有效鉀和有效磷是影響nirK基因反硝化菌群落結構的主要環境因子。
[1] Conrad R. Soil microorganisms as controllers of atmospheric trace gases (H2, CO, CH4, OCS, N2O and NO). Microbiology Reviews, 1996, 60(4): 609- 640.
[2] Zumft W G. Cell biology and molecular basis of denitrification. Microbiology and Molecular Biology Reviews, 1997, 61(4): 533- 616.
[3] Wolsing M, Priemé A. Observation of high seasonal variation in community structure of denitrifying bacteria in arable soil receiving artificial fertilizer and cattle manure by determining T-RFLP ofnirgene fragments. FEMS Microbiology Ecology, 2004, 48(2): 261- 271.
[4] Braker G, Zhou J Z, Wu L, Devol A H, Tiedje J M. Nitrite reductase genes (nirKandnirS) as functional markers to investigate diversity of denitrifying bacteria in pacific northwest marine sediment communities. Applied and Environmental Microbiology, 2000, 66(5): 2096- 2104.
[5] Priemé A, Braker G, Tiedje J M. Diversity of nitrite reductase (nirKandnirS) gene fragments in forested upland and wetland soils. Applied and Environmental Microbiology, 2002, 68(4): 1893- 1900.
[6] Keil D, Meyer A, Berner D, Poll C, Shcützenmeister A, Piepho H P, Vlasenko A, Philippot L, Schloter M, Kandeler E, Marhan S. Influence of land-use intensity on the spatial distribution of N-cycling microorganisms in grassland soils. FEMS Microbiology Ecology, 2011, 77(1): 95- 106.
[7] Scala D J, Kerkhof L J. Nitrous oxide reductase (nosZ) gene-specific PCR primers for detection of denitrifiers and threenosZgenes from marine sediments. FEMS Microbiology Letter, 1998, 162(1): 61- 68.
[8] Yoshida M, Ishii S, Otsuka S, Senoo K. Temporal shifts in diversity and quantity ofnirSandnirKin a rice paddy field soil. Soil Biology and Biochemistry, 2009, 41(10): 2044- 2051.
[9] Robertson G P, Paul E A, Harwood R R. Greenhouse gases in intensive agriculture: contributions of individual gases to the radiative forcing of the atmosphere. Science, 2000, 289(5486): 1922- 1925.
[10] Huo L L, Chen Z K, Zou Y C, Lu X G, Guo J W, Tang X G. Effect of Zoige alpine wetland degradation on the density and fractions of soil organic carbon. Ecological Engineering, 2013, 51: 287- 295.
[11] 張曉云, 呂憲國, 顧海軍. 若爾蓋濕地面臨的威脅、保護現狀及對策分析. 濕地科學, 2005, 3(4): 292- 297.
[12] 羅清, 彭國照. 若爾蓋及其鄰近地區氣候變化對濕地生態環境的影響. 高原山地氣象研究, 2008, 28(3): 44- 48.
[13] 陳槐, 高永恒, 姚守平, 吳寧, 王艷芬, 羅鵬, 田建卿. 若爾蓋高原濕地甲烷排放的時空異質性. 生態學報, 2008, 28(7): 3425- 3437.
[14] 王德宣. 若爾蓋高原泥炭沼澤二氧化碳、甲烷和氧化亞氮排放通量研究. 濕地科學, 2010, 8(3): 220- 224.
[15] Zhang G S, Tian J Q, Jiang N A, Guo X P, Wang Y F, Dong X Z. Methanogen community in Zoige wetland of Tibetan plateau and phenotypic characterization of a dominant uncultured methanogen cluster ZC-I. Environmental Microbiology, 2008, 10(7): 1850- 1860.
[16] Zhou W J, Lu X F, Wu Z K, Deng L, Jull A J T, Donahue D, Beck W. Peat record reflecting Holocene climatic change in the Zoigê Plateau and AMS radiocarbon dating. Chinese Science Bulletin, 2002, 47(1): 66- 70.
[17] Hartemink A E, Janssen B H, Buresh R J, Jama B. Soil nitrate and water dynamics in Sesbania fallows, weed fallows, and maize. Soil Science Society of America Journal, 1996, 60(2): 568- 574.
[18] 魯如坤. 土壤農業化學分析方法. 北京: 中國農業科技出版社, 2000: 125- 510.
[19] Liu H, Dasgupta P K. Analytical chemistry in a drop. Solvent extraction in a microdrop. Analytical Chemistry, 1996, 68(11): 1817- 1821.
[20] Dambreville C, Hallet S, Nguyen C, Morvan T, Germon J C, Philippot L. Structure and activity of the denitrifying community in a maize-cropped field fertilized with composted pig manure or ammonium nitrate. FEMS Microbiology Ecology, 2006, 56(1): 119- 131.
[21] Chen Z, Luo X Q, Hu R G, Wu M N, Wu J S, Wei W X. Impact of long-term fertilization on the composition of denitrifier communities based on nitrite reductase analyses in a paddy soil. Microbial Ecology, 2010, 60(4): 850- 861.
[22] Hill T C J, Walsh K A, Harris J A, Moffett B F. Using ecological diversity measures with bacterial communities. FEMS Microbiology Ecology, 2003, 43(1): 1- 11.
[23] Good I J. The population frequencies of species and the estimation of population parameters. Biometrika, 1953, 40(3/4): 237- 264.
[24] Qiu X Y, Wu L Y, Huang S H, McDonel P E, Palumbo A V, Tiedje J M, Zhou J Z. Evaluation of PCR-generated chimeras, mutations, and heteroduplexes with 16S rRNA gene-based cloning. Applied and Environmental Microbiology, 2001, 67(2): 880- 887.
[25] Palmer K, Drake H L, Horn M A. Genome-derived criteria for assigning environmentalnarGandnosZsequences to operational taxonomic units of nitrate reducers. Applied and Environmental Microbiology, 2009, 75(15): 5170- 5174.
[26] Jahangir M M R, Khalil M I, Johnston P, Cardenas L M, Hatch D J, Butler M, Barrett M, O′flaherty V, Richards K G. Denitrification potential in subsoils: a mechanism to reduce nitrate leaching to groundwater. Agriculture, Ecosystems & Environment, 2012, 147: 13- 23.
[27] Guo G X, Deng H, Qiao M, Yao H Y, Zhu Y G. Effect of long-term wastewater irrigation on potential denitrification and denitrifying communities in soils at the watershed scale. Environmental Science and Technology, 2013, 47(7): 3105- 3113.
[28] White J R, Reddy K R. Nitrification and denitrification rates of Everglades wetland soils along a phosphorus-impacted gradient. Journal of Environmental Quality, 2003, 32(6): 2436- 2443.
[29] Saito T, Ishii S, Otsuka S, Nishiyama M, Senoo K. Identification of novel Betaproteobacteria in a succinate-assimilating population in denitrifying rice paddy soil by using stable isotope probing. Microbes and Environments, 2008, 23(3): 192- 200.
[30] Katsuyama C, Kondo N, Suwa Y, Yamagishi T, Itoh M, Ohte N, Kimura H, Nagaosa K, Kato K. Denitrification activity and relevant bacteria revealed by nitrite reductase gene fragments in soil of temperate mixed forest. Microbes and Environments, 2008, 23(4): 337- 345.
[31] Ruiz-Rueda R O, Trias M R, Garcia-Gil L J, Baeras L. Diversity of the nitrite reductase genenirSin the sediment of a free-water surface constructed wetland. International Microbiology, 2007, 10(4): 253- 260.
[32] Zhang Y G, Li D Q, Wang H M, Xiao Q M, Liu X D. The diversity of denitrifying bacteria in the alpine meadow soil of Sanjiangyuan natural reserve in Tibet Plateau. Chinese Science Bulletin, 2006, 51(10): 1245- 1254.
[33] Xie Z, Le Roux X, Wang C P, Gu Z K, An M, Nan H Y, Chen B Z, Li F, Liu Y J, Du G Z, Feng H Y, Ma X J. Identifying response groups of soil nitrifiers and denitrifiers to grazing and associated soil environmental drivers in Tibetan alpine meadows. Soil Biology and Biochemistry, 2014, 77: 89- 99.
[34] Bárta J, Melichová T, Vaněk D, Picek T,antrková H. Effect of pH and dissolved organic matter on the abundance ofnirKandnirSdenitrifiers in spruce forest soil. Biogeochemistry, 2010, 101(1/3): 123- 132.
[35] Xue K, Wu L Y, Deng Y, He Z L, Van Nostrand J, Robertson P G, Schmidt T M, Zhou J Z. Functional gene differences in soil microbial communities from conventional, low-input, and organic farmlands. Applied and Environmental Microbiology, 2013, 79(4): 1284- 1292.
[36] 王斐, 李菊梅, 戴建軍, 馬義兵. 有機結合態磷肥對土壤微生物及磷酸酶的影響. 化肥工業, 2013, 40(3): 3- 9.
[37] McGill W B, Cole C V. Comparative aspects of cycling of organic C, N, S and P through soil organic matter. Geoderma, 1981, 26(4): 267- 286.
Analyzingthenitratereductasegene(nirK)communityinthepeatsoiloftheZoigeWetlandoftheTibetanPlateau
WANG Yingyan, LU Sheng′e, CHEN Xiaomin, LI Yuefei, GU Yunfu*
CollegeofResource,SichuanAgriculturalUniversity,Chengdu611130,China
The Zoige Wetland, which is located in the northeast of the Tibetan Plateau, is a typical low latitude permanent permafrost wetland, with high altitude intensive ultraviolet radiation and high soil organic matter content. The nitrous oxide fluxes in this area are critical to global warming. The main objective of this study was to elucidate the diversity and structure of the nitrate reductase (nirK) gene denitrifier community, and to further explore the microbial mediated mechanism of N2O release in this climatically extreme area. Based on the soil physicochemical properties and denitrifying activity (PDA) analysis, restriction fragment length polymorphism (RFLP), clone library and sequencing were further used to analyze the diversity and structure of thenirKgene in the denitrifier community. The results showed that the highest activity of PDA was detected in the Axi soil, while the lowest activity was in the Fenqu soil. The PDA was significantly and positively correlated with soil organic carbon, total nitrogen andnirKgene richness (P< 0.05). The highest and lowest Shannon-Wiener diversity indices were detected in Axi and Fenqu soils, respectively. Based on the RFLP patterns, 15 differentnirKgene clones were selected for sequencing and further phylogenetic analysis. The phylogenetic analysis showed that the majority of thenirK-denitrifiers in the peat soil of the Zoige Wetland belonged toProteobacteria, and the dominant species in the Axi soil wereAlphaproteobacteria, whereasBetaproteobacteriaspecies dominated in the Maixi soil. The dominantnirKdenitrifiers remained unidentified in the Fenqu soil. Redundancy analysis (RDA) was used to explore the possible relationship between thenirKdenitrifiers community and soil physicochemical parameters, and the results showed that soil available potassium and available phosphorus were the two most important factors in shapingnirKdenitrifier communities. In conclusion, the current study revealed obvious denitrifying activities in the Zoige Plateau Wetland. ThenirKgene denitrifying community, which plays an important role in mediating the denitrifying process, was relatively diverse and positively influenced by soil available potassium and available phosphorus.
Zoige Plateau Wetland; peat marsh soil; potential denitrifying activity;nirKgene; diversity
國家自然科學基金(41201256)
2016- 07- 15; < class="emphasis_bold">網絡出版日期
日期:2017- 05- 27
*通訊作者Corresponding author.E-mail: guyf@sicau.edu.cn.
10.5846/stxb201607151442
王鎣燕, 盧圣鄂, 陳小敏, 李躍飛, 辜運富.若爾蓋高原濕地泥炭沼澤土亞硝酸鹽還原酶(nirK)反硝化細菌群落結構分析.生態學報,2017,37(19):6607- 6615.
Wang Y Y, Lu S E, Chen X M, Li Y F, Gu Y F.Analyzing the nitrate reductase gene (nirK) community in the peat soil of the Zoige Wetland of the Tibetan Plateau.Acta Ecologica Sinica,2017,37(19):6607- 6615.

