美洲大蠊基因組重復序列分析
牟必琴, 嚴超超, 李午佼, 李靜, 沈詠梅, 岳碧松*
(1.四川大學生命科學學院,生物資源與生態環境教育部重點實驗室,成都610064; 2. 藥用美洲大蠊四川省重點實驗室,成都610081)
美洲大蠊基因組重復序列分析
牟必琴1, 嚴超超1, 李午佼1, 李靜1, 沈詠梅2, 岳碧松1*
(1.四川大學生命科學學院,生物資源與生態環境教育部重點實驗室,成都610064; 2. 藥用美洲大蠊四川省重點實驗室,成都610081)
重復序列是真核生物基因組的重要組成部分。一些重復序列,如自主型的逆轉錄轉座子LINE,在昆蟲的系統進化和遺傳多樣性研究方面得到了廣泛的應用。denovo從頭預測和基于同源比對預測相結合的方法被用來搜索美洲大蠊Periplanetaamericana基因組,共鑒定出大約占全基因組62%的重復序列。研究發現,散在重復序列中,DNA轉座子占美洲大蠊基因組的16.18%;逆轉座元件中LINE最多,占基因組的13.64%,SINE和LTR分別占基因組的3.52%和1.32%。LINEs中的BovBs亞家族在所有轉座子亞家族中比例最高(約6.73%)。美洲大蠊與德國小蠊Blattellagermanica相比,除LTR外,其他類型的轉座子占基因組的比例均高于德國小蠊。通過分析逆轉錄轉座子反轉錄酶完整度、氨基酸序列相似度及遺傳距離,從美洲大蠊基因組中鑒定出一類BovBs:RTE-1_PAm。BovBs的反轉錄酶氨基酸序列的系統樹表明,美洲大蠊與內華達古白蟻Zootermopsisnevadensis的進化關系比與其同屬蜚蠊科Blattidae的德國小蠊的關系更近。昆蟲中BovBs的進化關系與傳統核基因進化關系的不同,表明轉座子的進化相對宿主基因的進化具有一定的獨立性。
美洲大蠊; 基因組; 重復序列; 轉座子; BovBs; 系統進化
Abstract: Repetitive sequences constitute a large fraction of a eukaryote genome. Some classes of repetitive sequences, such as LINEs, which are a member of autonomous transposons, have been widely applied in molecular phylogenetic and genetic diversity studies of insects. In this study,denovoprediction and homology alignment were used to search the repetitive sequences inPeriplanetaamericanagenome, and the results showed that the screened repetitive sequences accounted for approximately 62% of the genome. Among interspersed repetitive sequences, the DNA transposons constituted 16.18% of the whole genome. In retrotransposons, LINEs accounted for 13.64% of the genome followed by SINE (3.52%) and LTR (1.32%), respectively. The BovBs subfamily in LINEs was the most abundant (6.73%) compared to other classes of transposons. The proportion of all classes of transposable elements inP.americanawere higher than that ofBlattellagermanicaexcept the LTRs. According to the integrality of the reverse transcriptase of retrotransposons, similarities and genetic distance of the amino acid sequences, a kind of BovB retrotransposons were identified inP.americanagenome and named RTE-1_PAm. Phylogenetic analyses based on the amino acid sequences of BovBs reverse transcriptase indicated thatP.americanashared closer relationship withZootermopsisnevadensisthan withB.germanicawhich are both Blattidae. The difference of phylogenetic analyses results among these insects inferred by using retrotransposons and karyogene suggested an independent evolutional history of TEs relative to the host karyogene.
Keywords:Periplanetaamericana; genome; repetitive sequences; transposable element; BovB retrotransposon; phylogeny
重復序列是真核基因組的重要組成部分,它分為串聯重復序列和散在重復序列,其中轉座子是散在重復序列的主要組成部分。轉座子又稱為轉座元件(transposable element,TE),是廣泛分布于真核生物基因組中的一種可移動的DNA序列(Kidwell & Lisch,2002),是宿主進化的重要動力之一(Kazazian,2004)。按轉座機制和序列特征,散在重復序列可分為DNA轉座子和逆轉錄轉座子兩大類。逆轉錄轉座子編碼逆轉錄酶,該酶由DNA轉錄形成的RNA經逆轉錄轉換成cDNA,最后將形成的cDNA整合到基因組完成轉座,引起穩定突變(Eickbushetal.,2002)。根據序列兩端是否含有長末端重復序列,逆轉錄轉座子又分為長末端重復序列(long terminal repeat, LTR)和非長末端重復序列。非自主的非長末端重復序列稱為短散在重復序列(short interspersed nuclear element, SINE)。非自主重復序列不能獨立轉座,自身不編碼有功能的逆轉錄轉座酶,只能依賴于相關的自主元件進行轉座。相反,自主的非長末端重復序列稱為長散在重復序列(long interspersed nuclear element, LINE),它本身含有有功能的反轉錄酶而完成自身的轉座。DNA轉座子編碼轉座酶,一般直接通過“剪切和粘貼”機制發生轉座,引起不穩定突變(Ohshimaetal.,1996)。
關于昆蟲BovB逆轉錄轉座子(BovB retrotransposons, BovBs)的進化研究迄今較少。BovBs屬于LINE,全長約3.2 kb,在蜚蠊目Blattodea物種的基因組中均占很高比例。完整的BovBs中含有一個長約1 000個氨基酸的開放閱讀框,用于編碼逆轉錄轉座子轉座活動過程中需要的4種酶:反轉錄酶、核酸內切酶、核酸外切酶及磷酸酶(Malik & Eickbush,1998)。BovBs在各類真核生物的基因組中均有報道,比如在短尾負鼠Monodelphisdomestica、牛、蜥形類和猛犸象Mammuthusprimigenius等物種的基因組中被發現占很高比例(Gentlesetal.,2007;Adelsonetal.,2009;Kordis,2009;Zhao & Qi,2009)。近年來,昆蟲中的BovBs也陸續被發現,如家蠶Bombyxmori(Mortonetal.,2012)和肩突硬蜱Ixodesscapularis(Tayetal.,2010)。此外,一些研究對蠅蛹金小峰Nasoniavitripennis、紅帶袖蝶Heliconiusmelpomene、果蠅屬Drosophila、埃及伊蚊Aedesaegypti、致倦庫蚊Culexquinquefasciatus和按蚊屬Anopheles基因組中BovBs進行了結構和活性的相關研究(Clark & Eisen,2007;Sinkins,2007;Arensburgeretal.,2010;Webbetal.,2011;Consortiumetal.,2012)。
隨著二代測序技術的飛速發展,越來越多物種的全基因組得以揭示,在物種的全基因組中鑒定重復序列的研究也越來越多。美洲大蠊Periplanetaamericana屬于蜚蠊目蜚蠊科Blattidae大蠊屬Periplaneta,具有較強的抗逆和適應能力,是世界性的衛生害蟲,同時在中國也是重要的藥用昆蟲。蜚蠊目的德國小蠊Blattellagermanica為其同目同科物種。近年來,原等翅目Blattaria的內華達古白蟻Zootermopsisnevadensis被歸類于蜚蠊目(Terraponetal.,2014)。為了了解美洲大蠊的遺傳背景,本研究利用denovo預測和同源性搜索相結合的方法首次對美洲大蠊基因組重復序列進行鑒定,并比較分析了重復序列在美洲大蠊、德國小蠊和內華達古白蟻3種蜚蠊目昆蟲基因組中的分布規律,最后以BovBs內反轉錄酶編碼序列作為分子標記,初步研究了BovBs在16種昆蟲中的進化關系。
1 基因組來源與方法
1.1基因組來源
美洲大蠊樣品來自四川好醫生攀西藥業有限公司西昌飼養基地。使用二代Illumina和三代Pacbio測序相結合的方法,分別得到了460×和10×的原始數據。通過DISCOVAR (https://www.broad-institute.org/software/discovar/blog)(Loveetal.,2016)和BESST(Sahlinetal.,2014,2016)組裝得到美洲大蠊全基因組。德國小蠊和內華達古白蟻的基因組均下載自NCBI (PRJNA203136和PRJNA203242)。
1.2方法
1.2.1重復序列的鑒定使用3種方案搜集預測基因組中的重復序列:(1)從頭預測轉座子:使用RepeatModeler(http://www.repeatmasker.org,version 1.0.8)掃描基因組序列,對不同重復序列的特性進行計算機建模,得到非冗余的重復序列數據庫(Smit & Hubley,2008-2015),然后使用RepeatMasker(http://www.repeatmasker.org,version 4.0.5)進行重復序列的搜索和識別(Smitetal.,2013-2015);(2)基于同源比對注釋重復序列:使用RepeatMasker和RepBase重復序列數據庫(version 19.09)進行比對并注釋(Jurkaetal.,2005;Baoetal.,2015);(3)使用RepeatProtein(version 4.0.5)和它的默認參數搜索已知轉座子內的蛋白質序列相關的重復序列(Huetal.,2012)。
1.2.2重復序列的統計重復序列統計的原始文件為RepeatMasker的輸出文件(“.out”)和RepeatProtein的輸出文件(“.annot”)。在對重復序列統計之前,首先對每種類型的重復序列過濾,去掉核苷酸序列長度小于10 bp和與重復序列庫相似度小于70%的重復元件。這一過程使用shell和python編寫的本地腳本完成。過濾得到的文件使用R語言統計每個物種基因組中重復序列的種類、占基因組的比例并作圖。
1.2.3BovBs進化分析使用LINE保守的反轉錄酶區域的氨基酸序列構建昆蟲的系統進化樹。首先,將參考基因組的BovBs序列作為質詢序列,使用Blastx搜索出與NCBI的蛋白保守序列庫(CDD)中LINE中的反轉錄酶氨基酸保守序列(cd01650)同源性較高的蛋白質序列(evalue值不大于1e-10,氨基酸長度大于100 aa)(Sun & Mueller,2014);然后將所得的蛋白質序列進行修整(如去除低質量的插入缺失位點),保留CDD庫內的一致性序列,然后使用CD-HIT去除冗余序列(Huangetal.,2010),得到氨基酸序列;最后使用RepeatMasker中的COSEG(Lavoieetal.,2013)合并得到BovBs中氨基酸的一致性序列。使用MEGA 5.2(Tamuraetal.,2011;Sormachevaetal.,2012)計算氨基酸序列之間的遺傳距離。此外,從RepBase重復序列蛋白質序列庫中下載了其他13種昆蟲的19條BovBs蛋白質序列(表1)。使用ClustalW (Thompsonetal.,1999)和prank v.140603 (L?ytynoja & Goldman,2010)將氨基酸序列集進行對齊。最后使用MEGA 5.2(Tamuraetal.,2011)構建最大似然樹,Bootstrap設置為1 000以檢驗分子系統樹各分支的置信度,并使用MrBayes 3.2(Ronquist & Huelsenbeck,2003)構建貝葉斯樹。
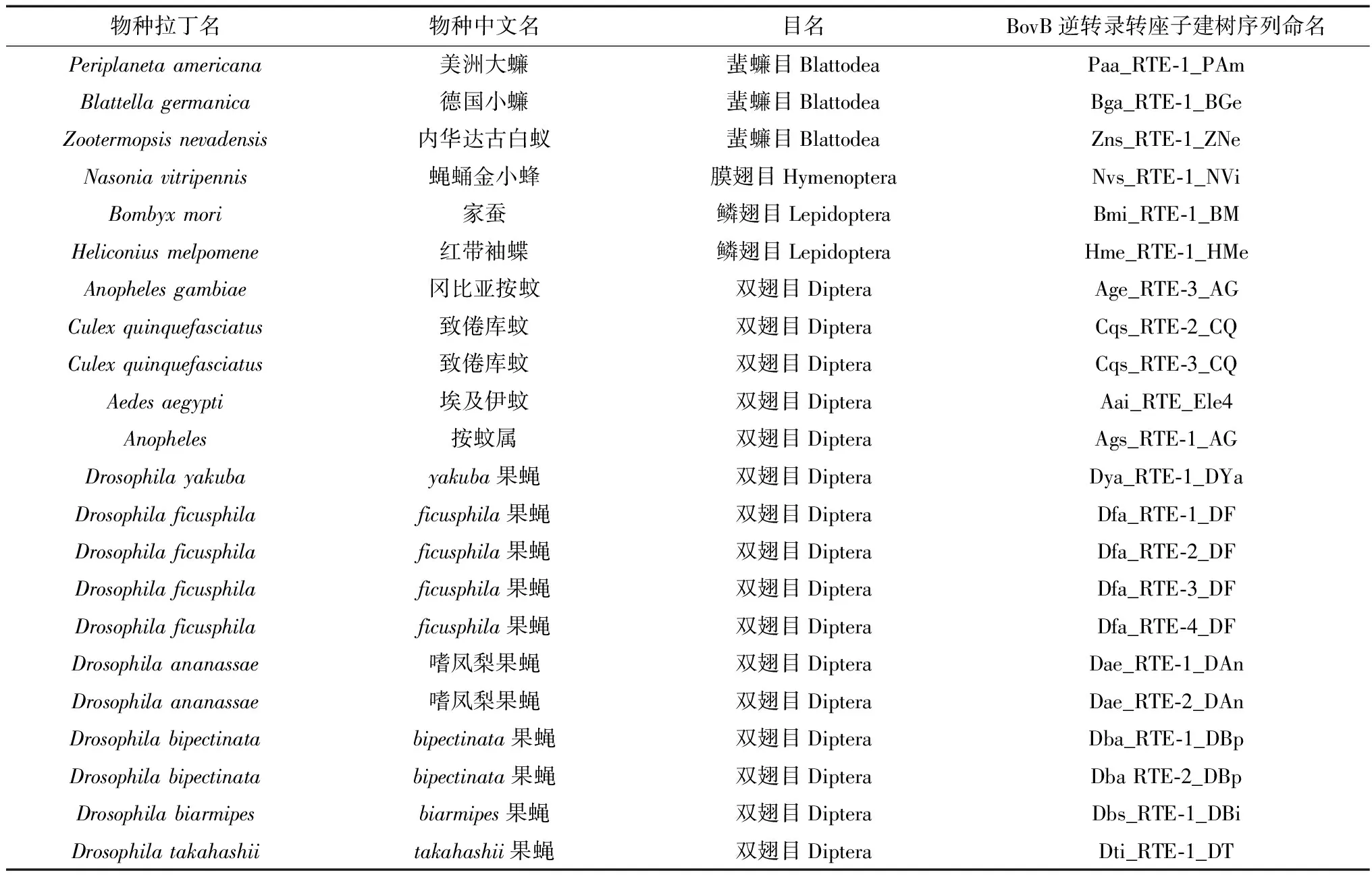
表1 BovB逆轉錄轉座子氨基酸序列信息Table 1 The amino acid sequence information of BovB retrotransposons
注: BovBs序列名稱的命名規則為: 物種拉丁名屬名的首字母和種名首尾字母+BovB逆轉錄轉座子的亞家族名。
Notes: The sequence names consists of the first letter of genus name, the first and the end letters of species’ name, as well as the subfamily name of BovB retrotransposons.
2 結果
2.1主要重復序列和比例
在美洲大蠊基因組中,使用RepeatModelerdenovo預測方法貢獻最大,得到了1 862.3 Mp的重復序列,占基因組的58.27%;使用RepBase同源比對注釋得到了355.3 Mp的重復序列,占基因組的11.12%;使用RepeatProtein識別的轉座子蛋白序列庫得到了417.5 Mp的重復序列,占基因組的13.06%。綜合以上3種不同的方法,去除冗余后(不同方法可能在同一個位置預測出轉座子),共得到了1 993.9 Mp的非冗余重復序列,占基因組的62.38%(圖1,表2),這和人類基因組的重復序列占比相似(International Human Genome Sequencing Consortium,2001)。在美洲大蠊基因組重復序列中,未知類型的重復序列(Unknown)占總重復序列的1/3左右,占基因組的25.75%。已知類型轉座子中,DNA轉座子總長度占基因組的16.18%,相對于所有的逆轉錄轉座子(占基因組的18.75%)較小。其中,LINE為13.64%。此外,SINE和LTR比例均較少,只占1.80%左右。其他類型的重復序列,如衛星重復序列、核糖體重復序列及其他簡單重復序列占基因組的3.70%(圖1,表2)。

圖1 美洲大蠊基因組中重復序列分布圖Fig. 1 The pie distribution diagram of the repetitive sequences in the Periplaneta americana genome

重復序列種類ClassesofrepeatsRepeatModeler注釋RepeatModelerRepBase庫注釋RepBaseRepeatProtein庫注釋RepeatProtein重復序列總和Total長度Length/Mp占基因組比例Percentageofgenome/%長度Length/Mp占基因組比例Percentageofgenome/%長度Length/Mp占基因組比例Percentageofgenome/%長度Length/Mp占基因組比例Percentageofgenome/%DNA442413841129353141944451701618LINE327110231238387268584043601364SINE112335295030001139356LTR42213213304257018570178RC/Helitron5761804101318006591185Other868272998312001182370Unknown82222572090030082312575Total1862358273553111241751306199396238
蜚蠊目物種不同類型的重復序列比較分析結果見表3。美洲大蠊基因組的重復序列含量(62.38%)略高于德國小蠊(61.94%),遠高于內華達古白蟻(35.84%)。這一結果與脊椎動物中隨基因組的增加重復序列占基因組比例增大的趨勢一致(Chalopinetal.,2014;Chalopinetal.,2015)。DNA型重復序列是美洲大蠊和德國小蠊基因組中最豐富的重復序列類型,其中美洲大蠊的DNA型重復序列含量最高,達16.18%,德國小蠊次之(13.07%),均顯著高于內華達古白蟻(6.20%)。同屬于DNA重復序列的RC/Helitron型重復序列與這一現象有所不同:美洲大蠊最高,德國小蠊最低。而LINE是內華達古白蟻基因組中最豐富的重復序列類型,約占基因組10%,但這一比例仍然低于美洲大蠊(13.64%)和德國小蠊(12.13%)。SINE在美洲大蠊和德國小蠊中的含量大致相當(大約3%),均高于內華達古白蟻(2.24%)。雖然大多數轉座子類型在美洲大蠊基因組中都超過德國小蠊,但德國小蠊基因組中LTR轉座子(5.02%)的比例卻顯著高于美洲大蠊(1.78%)。
2.2主要重復序列亞家族比較分析
DNA轉座子亞家族(TcMar、hAT)和LINE逆轉錄轉座子亞家族(BovB、L2)為3個物種基因組中重復序列的最主要組成部分(圖2),尤其是BovBs在3個近緣物種中均占有較高比例,美洲大蠊的BovBs也高于其他2個物種。而LTR逆轉錄轉座子亞家族中,德國小蠊的Copia、Gypsy和Pao亞家族比例高于美洲大蠊。相反,美洲大蠊的SINE類型的ID亞家族(0.85%)比例明顯高于德國小蠊(0.00%)。除此之外,內華達古白蟻的BovBs在其基因組中的比例最高(5.92%),且高于德國小蠊(4.50%),這歸功于BovBs屬于新型轉座子類型的LINE逆轉座子。
2.3美洲大蠊BovBs的序列分析
由于BovBs在美洲大蠊、德國小蠊及內華達古白蟻中占基因組比例均很高,可被用于轉座子系統進化研究。將BovBs通過Blastx比對到反轉錄酶氨基酸序列庫,分別得到3個物種具有90%以上同源性的一系列氨基酸序列。使用COSEG得到它們的一致性序列并分別命名為RTE-1_PAm,RTE-1_BGe和RTE-1_ZNe。將它們與其他13種昆蟲的19條BovBs氨基酸序列一起進行比較分析。本研究的蜚蠊目昆蟲和部分其他目昆蟲的遺傳距離及相似性結果見表4,蜚蠊目和與蜚蠊目相關的其他昆蟲BovBs反轉錄酶的遺傳距離為0.29~1.14,序列的相似度34.30%~75.20%。其中美洲大蠊與內華達古白蟻的反轉錄酶具有最高的相似度,為75.20%,與之對應,兩者遺傳距離最小,為0.29。美洲大蠊與德國小蠊的反轉錄酶的相似度為65.20%,它們的遺傳距離值為0.43,明顯大于美洲大蠊與內華達古白蟻的遺傳距離。美洲大蠊與紅帶袖蝶的BovBs反轉錄酶同源性最低,相似度為34.30%,遺傳距離值為1.07。

表3 不同物種重復序列的比較Table 3 Comparison of repetitive sequences in different species

圖2 3種蜚蠊目昆蟲基因組主要重復序列亞家族比較分析Fig. 2 Comparative analysis of the major repetitive sequence subfamilies in 3 Blattodea insect genomes

表4 部分昆蟲BovB逆轉錄轉座子氨基酸序列的遺傳距離及相似性(%)Table 4 The genetic distance and similarity (%) ofBovB retrotransposons in some insect genomes
注: 對角線上半部分數據為相似度, 下半部分數據為遺傳距離。
Notes: The data above the diagonal are the similarities between BovB retrotransposons, while those below the diagonal are the genetic distance.
2.4BovBs系統進化分析
將RTE-1_PAm,RTE-1_BGe和RTE-1_ZNe與其他昆蟲的19條BovBs家族的氨基酸進行序列比對,采用貝葉斯法和最大似然法構建分子系統發育樹。來自4個目16個物種的22條氨基酸序列被分為4大類群(圖3)。類群Ⅰ包含鱗翅目的家蠶和紅帶袖蝶。類群Ⅱ和Ⅲ均屬于雙翅目,類群Ⅱ含有雙翅目的果蠅屬物種,與鱗翅目聚在一起。類群Ⅲ既含有果蠅類,又含有蚊類,其中按蚊屬與果蠅屬聚在一個小支上。其他3種蚊類聚在另外一個小支上,包括1種埃及伊蚊、2條致倦庫蚊和1種岡比亞按蚊。類群Ⅳ中包括膜翅目的蠅蛹金小蜂和蜚蠊目的美洲大蠊、德國小蠊以及內華達古白蟻,類群Ⅳ位于系統進化樹內部,證明本研究鑒定的蜚蠊目的BovBs非常可靠。美洲大蠊和內華達古白蟻的BovBs聚在同一個進化小支上,且兩者的進化距離較德國小蠊與美洲大蠊的距離更近(圖3,表4)。
3 討論
在DNA轉座子類型中,2種蜚蠊科昆蟲的DNA轉座子占基因組的比例均遠高于內華達古白蟻,這種差異主要表現在2種蜚蠊科昆蟲DNA轉座子中的2個亞家族hAT和TcMar占基因組的比例明顯高于內華達古白蟻。在已知的反轉錄轉座子類型中,美洲大蠊LTR的類型和占基因組的比例明顯低于德國小蠊,但其他轉座子類型均是美洲大蠊略高于德國小蠊。從總體上看,美洲大蠊、德國小蠊和內華達古白蟻重復序列占基因組比例呈遞減趨勢,這與它們基因組大小依次遞減一致,符合隨著基因組的增大,重復序列占基因組比例也增大的規律(Chalopinetal.,2014,2015)。美洲大蠊重復序列最多的類型為Unknown(25.75%),說明其基因組中未知的重復序列類型還有很多,在這些未知類型的重復序列中除一部分簡單重復序列外,還可能存在一些物種特異的新類型重復序列,有待進一步挖掘分析。
采用存在于昆蟲基因組中且某些物種含量豐富的BovBs進行比較研究和系統進化分析,為理解其在昆蟲基因組的組成和進化歷程奠定了一定的基礎。美洲大蠊、德國小蠊和內華達古白蟻3個物種基因組中的一類BovBs序列,它們的氨基酸同源性高達90%。比對發現,RTE-1_PAm與RTE-1_BGe相似度為65.20%,而與RTE-1_ZNe聚在同一支上且其相似度為75.20%,與傳統的分類和系統進化關系(德國小蠊與美洲大蠊關系較近)不同(Misofetal.,2014)。形成這種差異的原因可能是轉座子的進化速率與基因組不一致,即轉座子的同義替換速率要低于或高于宿主基因的同義替換速率(Sánchezgraciaetal.,2005)。除此之外,選擇壓力和修復機制的不同也有可能使轉座子與宿主基因的進化速率出現差異。要解決這一問題,需要對更多昆蟲基因組的特性和轉座子的進化歷程進行更深入的研究。
轉座子在基因組中隨意跳動,導致基因組序列的刪除、擴增、移位、斷裂和重組等現象(Langleyetal.,1988;Charlesworthetal.,1994),從而影響物種的正常生命活動(Le & Capy,2006;Oliver & Greene,2009)。近年來,昆蟲中重復序列的研究較少,已有的研究主要集中于鱗翅目和雙翅目的果蠅屬中。例如家蠶基因組中的MITEs、Chapaev、Tcl/Mariner等轉座子的轉座活動使基因組發生重組(Zhangetal.,2014)。果蠅屬的P轉座子的擴增和水平轉移影響了基因組的變化(Clark & Kidwell,1997;Arcà & Savakis,2000)。本研究首次對美洲大蠊基因組的重復序列進行了系統的統計描述,并結合近緣物種的重復序列進行比較分析,這為美洲大蠊基因組研究提供了重要支持,對昆蟲重復序列的研究具有積極作用。

圖3 BovB逆轉錄轉座子反轉錄酶氨基酸序列系統進化樹Fig. 3 Phylogenetic relationship among BovB retrotransposons based on the amino acid sequence of reverse transcriptase
Adelson DL, Raison JM, Edgar RC. 2009. Characterization and distribution of retrotransposons and simple sequence repeats in the bovine genome[J]. Proceedings of the National Academy of Sciences, 106(31): 12855-12860.
Arcà B, Savakis C. 2000. Distribution of the transposable element Minos in the genusDrosophila[J]. Genetica, 108(3): 263.
Arensburger P, Megy K, Waterhouse RM,etal. 2010. Sequencing ofCulexquinquefasciatusestablishes a platform for mosquito comparative genomics[J]. Science, 330(6000): 86-88.Bao W, Kojima KK, Kohany O. 2015.Repbase update, a database of repetitive elements in eukaryotic genomes[J]. Mobile DNA, 6(1): 11.
Chalopin D, Fan S, Simakov O,etal. 2014. Evolutionary active transposable elements in the genome of the coelacanth[J]. Journal of Experimental Zoology Part B Molecular & Developmental Evolution, 322(6): 322-333.
Chalopin D, Naville M, Plard F,etal. 2015. Comparative analysis of transposable elements highlights mobilome diversity and evolution in vertebrates[J]. Genome Biology Evolution, 7(2): 567-580.
Charlesworth B, Sniegowski P, Stephan W. 1994. The evolutionary dynamics of repetitive DNA in eukaryotes[J]. Nature, 371(6494): 215-220.
Clark AG, Eisen MB. 2007. Evolution of genes and genomes on theDrosophilaphylogeny[J]. Nature, 450(7167): 203-218.
Clark JB, Kidwell MG. 1997. A phylogenetic perspective on P transposable element evolution inDrosophila[J]. Proceedings of the National Academy of Sciences of the United States of America, 94(21): 11428.
Consortium THG, Dasmahapatra KK, Walters C,etal. 2012. Butterfly genome reveals promiscuous exchange of mimicry adaptations among species[J]. Nature, 487(7405): 94-98.
Eickbush TH, Malik HS, Eickbush TH. 2002. Origins and evolution of retrotransposons[J]. Mobile DNA: 1111-1144.
Gentles AJ, Wakefield MJ, Kohany O,etal. 2007. Evolutionary dynamics of transposable elements in the short-tailed opossumMonodelphisdomestica[J]. Genome Research, 17(7): 992-1004.
Hu Q, Ma T, Wang K,etal. 2012. The yak genome database an integrative database for studying yak biology and high-altitude adaption[J]. BMC Genomics, 13(2): 600.
Huang Y, Niu B, Gao Y,etal. 2010. CD-HIT suite: a web server for clustering and comparing biological sequences[J]. Bioinformatics, 26(5): 680-682.
International Human Genome Sequencing Consortium. 2001. Initial sequencing and analysis of the human genome[J]. Nature, 409: 860-921.
Jurka J, Kapitonov VV, Pavlicek A,etal. 2005. Repbase update, a database of eukaryotic repetitive elements[J]. Cytogenetic & Genome Research, 110(1-4): 462.
Kazazian HH. 2004. Mobile elements: drivers of genome evolution[J]. Science, 303(5664): 1626-1632.
Kidwell M, Lisch D. 2002.Transposable elements as sources of genomic variation[M]// Craig NL, Cralgie R, Gellert M,etal. Mobile DNA II. Washington DC: ASM Press: 59-90.Kordis D. 2009. Transposable elements in reptilian and avian (sauropsida) genomes[J]. Cytogenetic & Genome Research, 127(2-4): 94-111.
Langley CH, Montgomery E, Hudson R,etal. 1988. On the role of unequal exchange in the containment of transposable element copy number[J]. Genetics Research, 52(3): 223-235.
Lavoie CA, Platt RN, Novick PA,etal. 2013. Transposable element evolution inHeliconius, suggests genome diversity within Lepidoptera[J]. Mobile DNA, 4(1): 21.
Le RA, Capy P. 2006. Population genetics models of competition between transposable element subfamilies[J]. Genetics, 174(2): 785-793.
Love RR, Weisenfeld NI, Jaffe DB,etal. 2016. Evaluation of DISCOVARdenovo, using a mosquito sample for cost-effective short-read genome assembly[J]. BMC Genomics, 17(1): 1-10.
L?ytynoja A, Goldman N. 2010. webPRANK: a phylogeny-aware multiple sequence aligner with interactive alignment browser[J]. BMC Bioinformatics, 11(1): 1-6.
Malik HS, Eickbush TH. 1998. The RTE class of non-LTR retrotransposons is widely distributed in animals and is the origin of many SINEs[J]. Molecular Biology and Evolution, 15(9): 1123-1134.
Misof B, Liu S, Meusemann K,etal. 2014. Phylogenomics resolves the timing and pattern of insect evolution[J]. Science, 346(346): 763-767.
Morton WA, Kortschak RD, Gardner MG,etal. 2012. Widespread horizontal transfer of retrotransposons[J]. Proceedings of the National Academy of Sciences of the United States of America, 110(3): 1012-1016.
Ohshima K, Hamada M, Terai Y,etal. 1996. The 3’ ends of tRNA-derived short interspersed repetitive elements are derived from the 3’ ends of long interspersed repetitive elements[J]. Molecular & Cellular Biology, 16(7): 3756-3764.
Oliver KR, Greene WK. 2009. Transposable elements: powerful facilitators of evolution[J]. Bioessays, 31(7): 703-714.
Ronquist F, Huelsenbeck JP. 2003. MrBayes 3: Bayesian phylogenetic inference under mixed models[J]. Bioinformatics, 19(12): 1572-1574.
Sahlin K, Chikhi R, Arvestad L. 2016. Assembly scaffolding with PE-contaminated mate-pair libraries[J]. Bioinformatics, 32(13): btw064.Sahlin K, Vezzi F, Nystedt B,etal. 2014. BESST-efficient scaffolding of large fragmented assemblies[J]. BMC Bioinformatics, 15(1): 281.
Sánchezgracia A, Maside X, Charlesworth B. 2005.High rate of horizontal transfer of transposable elements inDrosophila[J]. Trends in Genetics, 21(4): 200.
Sinkins S. 2007. Genome sequence ofAedesaegypti, a major arbovirus vector[J]. Science, 316(5832): 1718-1723.
Smit A, Hubley R. 2008-2015. RepeatModeler open-1.0[EB/OL]. URL: http: //www. repeatmasker. org.
Smit A, Hubley R, Green P. 2013-2015. RepeatMasker open-4.0[EB/OL]. URL: http://www. repeatmasker. org.
Sormacheva I, Smyshlyaev G, Mayorov V,etal. 2012. Vertical evolution and horizontal transfer of CR1 non-LTR retrotransposons and Tc1/mariner DNA transposons in Lepidoptera species[J]. Molecular Biology & Evolution, 29(12): 3685-3702.
Sun C, Mueller RL. 2014. Hellbender genome sequences shed light on genomic expansion at the base of crown salamanders[J]. Genome Biology & Evolution, 6(7): 1818-1829.
Tamura K, Peterson D, Peterson N,etal. 2011. MEGA5: molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods[J]. Molecular Biology & Evolution, 28(10): 2731-2739.
Tay WT, Behere GT, Batterham P,etal. 2010. Generation of microsatellite repeat families by RTE retrotransposons in lepidopteran genomes[J]. BMC Evolutionary Biology, 10(1): 144.
Terrapon N, Li C, Robertson HM,etal. 2014. Molecular traces of alternative social organization in a termite genome[J]. Nature Communications, 5(6183): 3636.
Thompson JD. 1999.CLUSTAL W: improving the sensitivity of progressive multiple sequence alignment through sequence weighting, positions-specific gap penalties and weight matrix choice[J]. Nucleic Acids Research, 22(22): 4673-4680.
Webb CHT, Riccitelli NJ, Ruminski DJ,etal. 2011. Widespread occurrence of self-cleaving ribozymes[J]. Science, 326(5955): 953.
Zhang HH, Feschotte C, Han MJ,etal.2014. Recurrent horizontal transfers of Chapaev transposons in diverse invertebrate and vertebrate animals[J]. Genome Biology & Evolution, 6(6): 1375.
Zhao FQ, Qi J, Schuster SC. 2009. Tracking the past: interspersed repeats in an extinct Afrotherian mammal,Mammuthusprimigenius[J]. Genome Research, 19(19): 1384-1392.
AnalysisofRepetitiveSequencesinPeriplanetaamericananaGenome
MOU Biqin1, YAN Chaochao1, LI Wujiao1, LI Jing1, SHEN Yongmei2, YUE Bisong1*
(1. Key Laboratory of Bio-resources and Eco-environment, Ministry of Education, College of Life Sciences, Sichuan University, Chengdu 610064, China; 2. Sichuan Key Laboratory of MedicinalPeriplanetaamericana, Chengdu 610081, China)
2017-02-22接受日期2017-05-09
牟必琴, 女, 碩士研究生, 研究方向:基因和遺傳信息分析, E-mail:435077636@qq.com
*通信作者Corresponding author, E-mail:bsyue@scu.edu.cn
10.11984/j.issn.1000-7083.20170051
Q78
A
1000-7083(2017)05-0540-08

