2014—2016年廣東省肇慶市人感染H7N9禽流感病毒基因組特征分析
譚翰清,程潔萍,譚海芳,朱穎梅,林 鳳,梁曉晴,陳 捷,麥 煒
2014—2016年廣東省肇慶市人感染H7N9禽流感病毒基因組特征分析
譚翰清1,程潔萍1,譚海芳1,朱穎梅1,林 鳳1,梁曉晴2,陳 捷2,麥 煒1
目的 了解廣東省肇慶市2014-2016年人感染H7N9禽流感病毒基因進化特征。方法 對肇慶市17例H7N9患者咽拭核酸直接進行8節段基因序列擴增與測定,用BioEdit5.0、MEGA6.0進行同源性和重要氨基酸位點分析,采用Neighbor-Joining法構建進化樹,參比序列從GenBank下載。結果 7份H7N9病例標本獲得8節段全基因序列,HA與A/chicken/Dongguan/695/2014(H7N9)相似度最高,NA與A/chicken/Dongguan/1075/2014(H7N9)相似度最高;內部基因與2013—2014年廣東東莞、深圳禽源H7N9及H9N2相似度較高。HA和NA基因與廣東省東莞、廣州、深圳等地市的H7N9序列同處于珠三角進化分支,與安徽、浙江、江蘇等長三角分支的序列相似性低。HA蛋白受體結合位點發生G186V、Q226L 變異,裂解位點序列均為PEIPKGR↓GLF,含有2個堿性氨基酸;M2蛋白上發現耐藥位點S31N,PB2蛋白上發現 E627K突變,NA蛋白上未發現耐神經氨酸酶的位點變異。結論 肇慶市人感染H7N9病毒株可能來源于2013—2014年廣東省珠三角地區禽源H7N9和H9N2,G186V、Q226L和E627K位點變異增強了對人的易感性。
人感染H7N9禽流感病毒;血凝素基因;神經氨酸酶基因;序列分析
甲型流感病毒屬于正黏病毒科,可感染人和禽鳥類、豬、馬等多種動物;其基因組為單負鏈分節段的RNA,不同宿主來源的病毒的基因組易重組產生新的亞型[1];根據血凝素(Hemagglutinin,HA)和神經氨基酸酶(Neuraminidase,NA)抗原分為不同的亞型,目前已發現18種HA、11種NA[2-4]。當流感病毒基因變異突破了物種的適應性,并對人具有明顯選擇優勢時,可對人類產生嚴重性疾病或導致人類流感大流行[5]。自1997年首次發現可感染人的H5N1禽流感以來,還發現H7N7、H9N2等亞型[6-7]。2013年2月上海報告了全球首個H7N9禽流感新亞型感染病例,緊接著在蘇、浙、徽等地相繼報告類似病例,并迅速在我國大部分省份蔓延,其導致嚴重的臨床癥狀和高病死率,引起了全球公共衛生的關注[8]。截至2016 年12月31日,我國內地累計報告H7N9 確診病例889例(死亡321例),廣東省累計報告211例(死亡82例),肇慶市(2014年1月報告首例)累計報告17例(死亡5例)。本研究對2014-2016年從廣東省肇慶市人感染H7N9禽流感病例標本中提取到的毒株進行序列分析,掌握其基因進化和關鍵氨基酸位點變異特征,為科學防控人感染H7N9禽流感疫情提供病原學依據。
1 材料與方法
1.1 標本來源 基于肇慶市國家級流感監測網絡系統,收集2014-2016年流感樣病例和不明原因肺炎病例咽拭標本,應用實時熒光RT-PCR試劑(上海之江生物科技有限公司)進行H7N9檢測,共發現17例人感染H7N9病例,樣本經國家疾病預防控制中心和廣東省流感參比實驗室復核確認,置-75 ℃保存備用。
1.2 主要試劑與儀器 MagMAXTM-96 Viral RNA Isolation Kit(Life Technologies,貨號:AM1836);PathAmpTMFluA Reagents(Life Technologies ,貨號:4401512);ExoSAP-IT試劑盒(USB,貨號:78201);Takara PCR Kit(Takara,貨號:DR011);8個節段基因擴增所用的22對引物參考文獻[9]合成。
1.3 基因擴增與序列測定 1)取400 μL咽拭標本,應用MagMAXTM-96 Viral RNA Isolation Kit進行病毒RNA提取,最后用20 μL去離子水洗脫;2)應用PathAmpTMFluA Reagents進行甲型流感8個節段序列擴增,使用ExoSAP-IT對產物進行快速純化與回收;3)分別應用22對引物進行H7N9病毒8個節段PCR擴增;4)PCR產物電泳、純化后在ABI3700測序儀上進行雙向序列測定。具體操作參照試劑盒說明書和參考文獻[9-10]。
1.4 同源性與進化樹分析 利用DNAStar5.0的SeqMan程序進行序列拼接,采用BioEdit5.0、NCBI的BLAST進行核苷酸同源性比對,應用MEGA6.0構建HA、NA基因進化發生樹(Neighbor Joining法,bootstrap = 1 000)和關鍵氨基酸位點分析[11]。參比序列從GenBank下載,HA和NA序列主要包括了上海、浙江、安徽、江蘇、廣東等地的病人、環境和禽源H7N9序列44條,其中2016年2條、2015年5條、2014年12條、2013年25條序列;此外,還包括了2005-2011年江西、浙江等地的4條H7N3序列,2005-2012年韓國、江西等地的6條H7N7序列,2008-2011年韓國、西班牙的H7N9序列3條,2005-2006年廣東、江西等地的5條H11N9序列。
2 結 果
2.1 疫情概括 2014-2016年肇慶市共發現17例H7N9病例,重癥11例(死亡5例)、輕癥6例(無死亡);發病年齡4-78歲,男女比11∶6;分布在懷集縣、四會市、鼎湖區;首例于2014年1月30日報告,末例于2016年12月28日報告,其中2014年10例、2015年4例、2016年3例,集中分布在每年12月至次年2月發病;病例2和3為家庭聚集病例,病例16有病死禽類接觸史;對病例樣本核酸直接進行擴增和測序,7例樣本獲得8個節段全序列,分別編號為ZQ01~07(表1)。
2.2 同源性分析結果 7份毒株的HA核苷酸同源性98.3%~100.0%,與A/chicken/Dongguan/695/2014(H7N9)相似度較高(98.3%~99.8%);7份毒株的NA核苷酸同源性99.0%~99.9%,與A/chicken/Dongguan/1075/2014(H7N9)相似度較高(99.1%~99.9%)。7份毒株的PB2、PB1、PA、NP、MP、NS核苷酸同源性分別為94.1%~100.0%、95.5%~99.8%、95.7%~100.0%、94.1%~100.0%、96.4%~99.9%、95.4%~100.0%,PB2、NP、M與2013-2014年廣東東莞的禽源H7N9相應基因相似度較高(95.1%~100.0%),PB1、PA、NS與2013-2014年廣東東莞、深圳禽源H9N2相應基因相似度較高(96.2%~99.8%)(表2)。
表1 2014-2016年肇慶市人感染H7N9病例信息概況
Tab.1 Information profile for cases of human infection with avian influenza A (H7N9) virus in Zhaoqing, China, 2014-2016

病例性別年齡地區發病日期診斷日期病例分類愈后發病前10d內是否與疑似或確診病例接觸過是否接觸過病、死禽類呼吸道樣本實時熒光PCR檢測的Ct值直接測序結果1男28懷集縣2014/1/192014/1/30重癥治愈否否33/2男5懷集縣2014/1/282014/2/1輕癥治愈否否21ZQ013女4懷集縣2014/1/262014/2/4輕癥治愈是否22ZQ024女5懷集縣2014/1/312014/2/5輕癥治愈否否32/5男42懷集縣2014/1/262014/2/5重癥治愈否否34/6男62懷集縣2014/2/12014/2/7重癥治愈否否33/7男49懷集縣2014/1/272014/2/7重癥治愈否否33/8男11懷集縣2014/2/42014/2/9輕癥治愈否否21ZQ039男8懷集縣2014/2/62014/2/12輕癥治愈否否23ZQ0410女45四會市2014/2/132014/2/21重癥死亡否否34/11男54四會市2014/12/252015/1/7重癥死亡否否20ZQ0512男5四會市2015/1/192015/1/23輕癥治愈否否22ZQ0613男59四會市2015/2/52015/2/23重癥死亡否否35/14男78四會市2015/2/182015/2/26重癥治愈否否34/15女76四會市2016/2/92016/2/16重癥治愈否否23ZQ0716女45懷集縣2016/12/172016/12/23重癥死亡否是33/17女50鼎湖區2016/12/212016/12/28重癥死亡否否32/
注:“/”表示未成功獲得序列信息
表2 2014-2016年肇慶市人感染H7N9各基因片段相似度分析結果
Tab.2 Nucleotide identity and similarity analysis of avian influenza A(H7N9) virus in Zhaoqing, China, 2014-2016
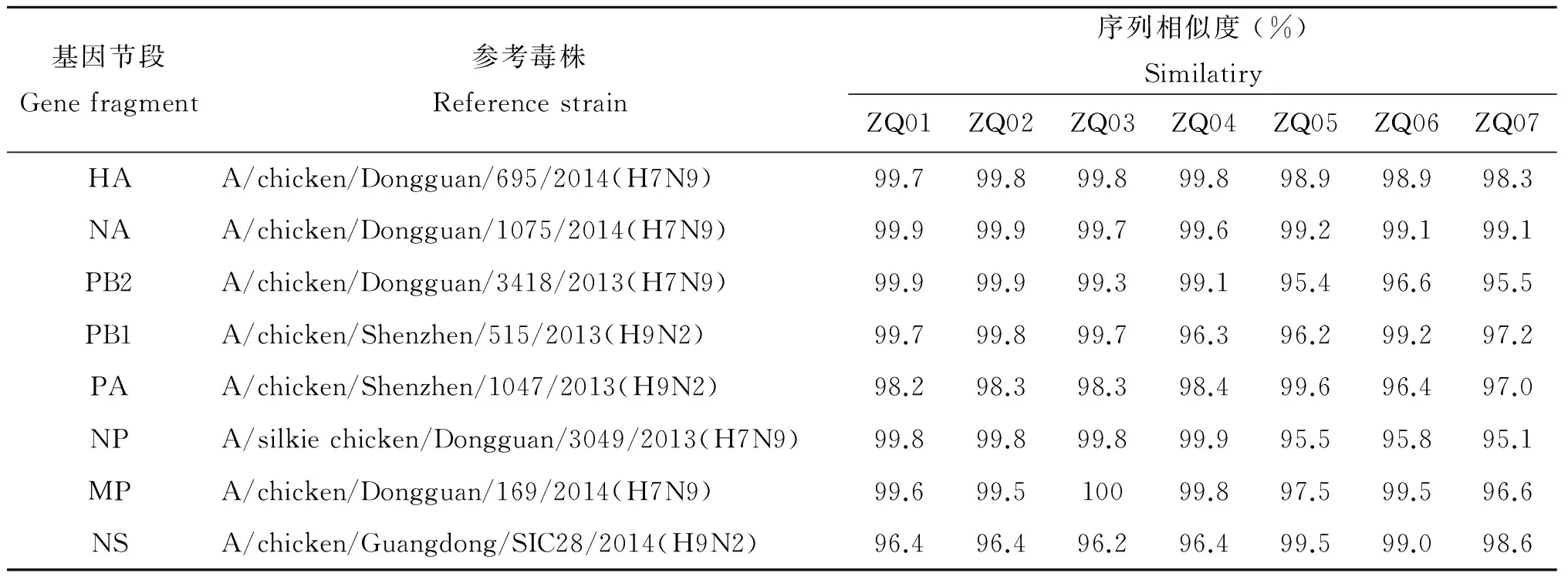
基因節段Genefragment參考毒株Referencestrain序列相似度(%)SimilatiryZQ01ZQ02ZQ03ZQ04ZQ05ZQ06ZQ07HAA/chicken/Dongguan/695/2014(H7N9)99.799.899.899.898.998.998.3NAA/chicken/Dongguan/1075/2014(H7N9)99.999.999.799.699.299.199.1PB2A/chicken/Dongguan/3418/2013(H7N9)99.999.999.399.195.496.695.5PB1A/chicken/Shenzhen/515/2013(H9N2)99.799.899.796.396.299.297.2PAA/chicken/Shenzhen/1047/2013(H9N2)98.298.398.398.499.696.497.0NPA/silkiechicken/Dongguan/3049/2013(H7N9)99.899.899.899.995.595.895.1MPA/chicken/Dongguan/169/2014(H7N9)99.699.510099.897.599.596.6NSA/chicken/Guangdong/SIC28/2014(H9N2)96.496.496.296.499.599.098.6
2.3 HA與NA基因進化特征分析 目前分析我國發現的H7N9病毒的HA和NA基因序列,在進化樹上明顯的分為兩大分支,一支是以廣東珠三角地區為代表的珠三角分支,一支是以上海、浙江為代表的長三角分支[11-12]。本文中研究的這7例從肇慶人感染H7N9病例分離的病毒HA和NA基因進化樹分析結果顯示,ZQ01-07病毒的HA和NA基因在進化樹上與廣東省其他地市,如東莞、廣州、深圳等地的禽、市場環境及人中分離的H7N9病毒序列相似性高,位于同一分支——珠三角分支;而與在進化樹上位于長三角分支的序列,如安徽、浙江、江蘇等地分離的H7N9病毒基因序列相似性低。其中ZQ01-07序列在進化樹中又集中在一簇,與廣東東莞雞中分離的H7N9病毒相似性最高(圖1,2)。
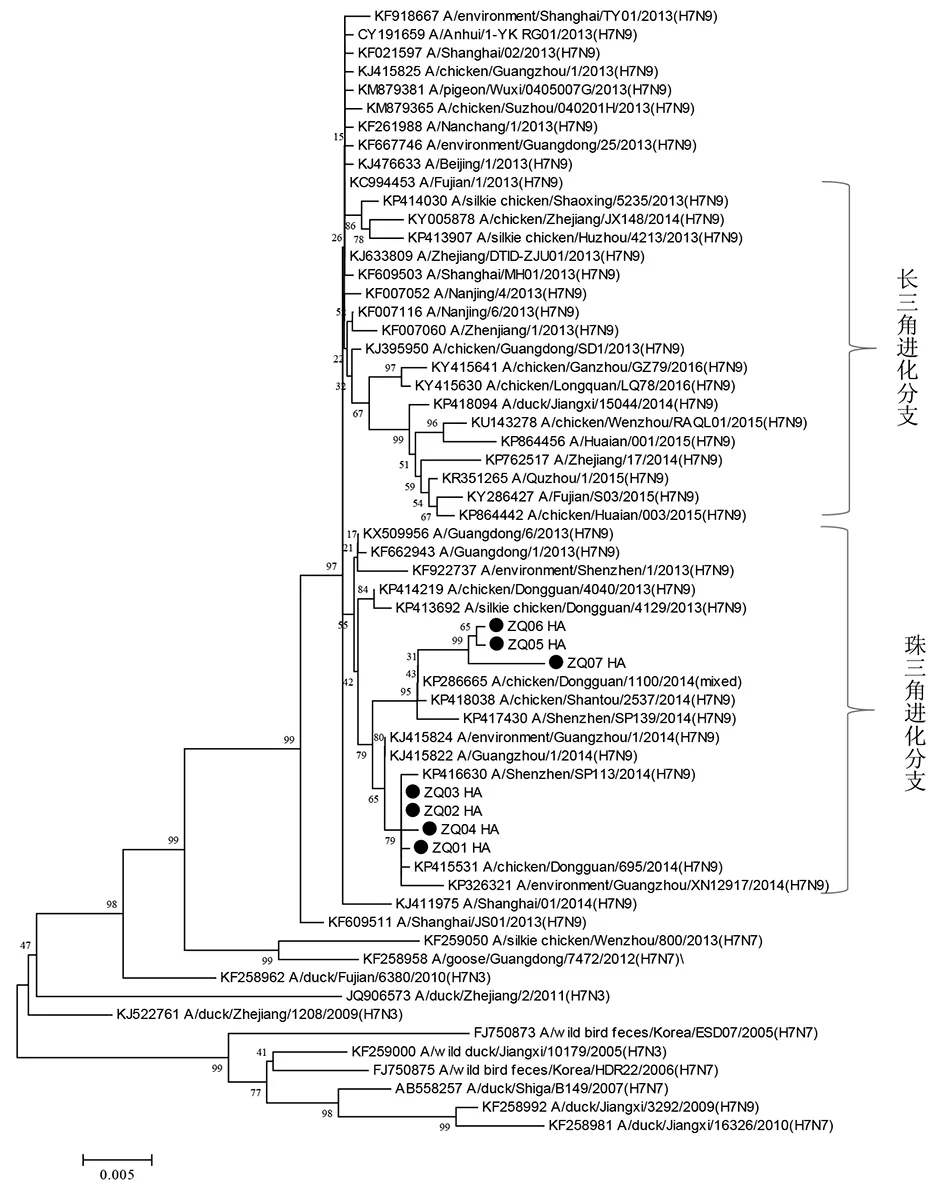
圖1 2014-2016年肇慶市人感染H7N9病毒株HA基因進化樹Fig.1 Phylogenetic tree of the HA gene of human infection with avian influenza A (H7N9) virus in Zhaoqing, China, 2014-2016
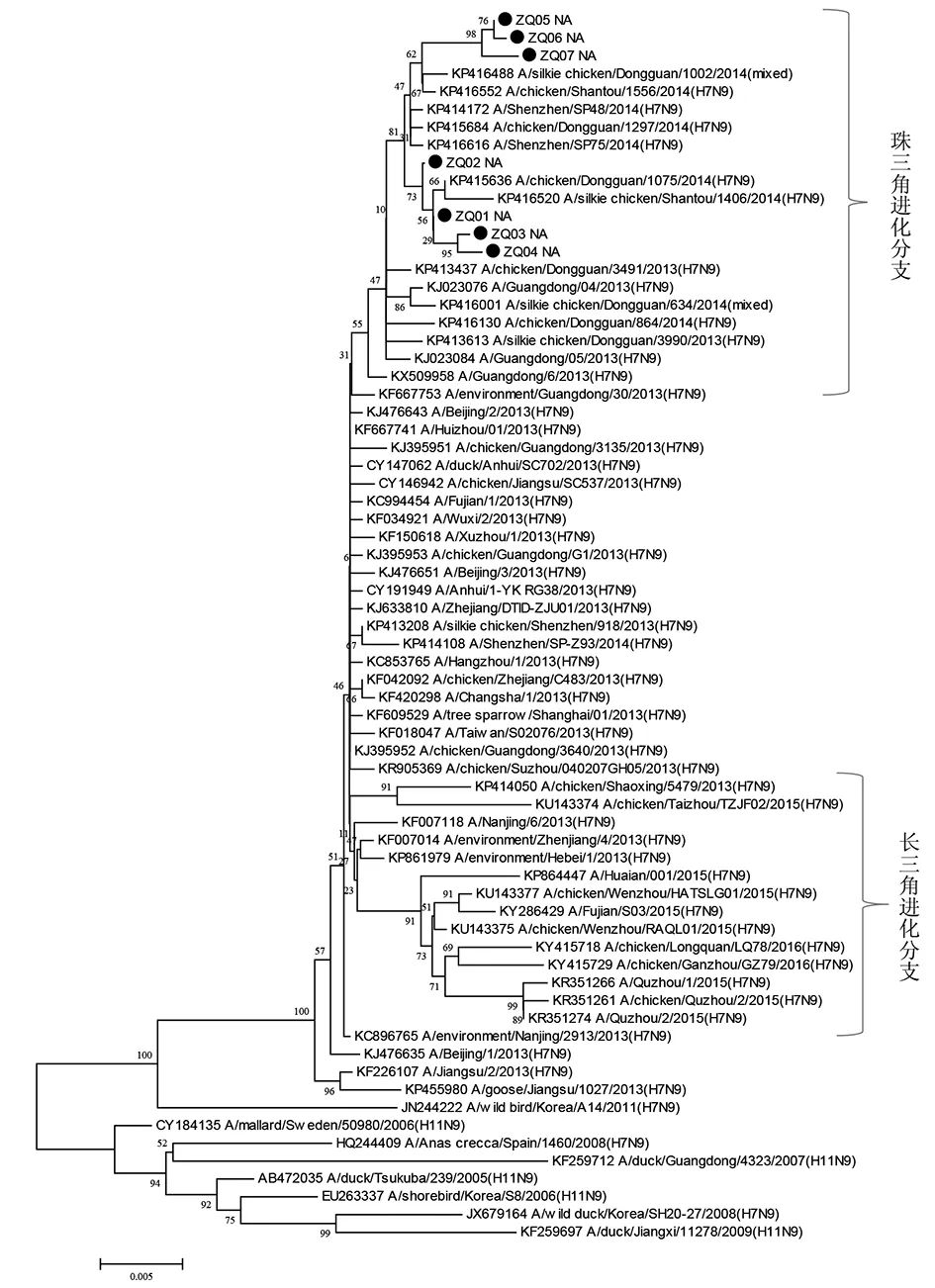
圖2 2014-2016年肇慶市人感染H7N9病毒株NA基因進化樹Fig.2 Phylogenetic tree of the NA gene of human infection with avian influenza A (H7N9) virus in Zhaoqing, China, 2014-2016
2.4 重要氨基酸位點分子特征 以H3N2(A/Aichi/2/1968)氨基酸序列為基準對H7N9的HA基因編碼的氨基酸位點進行分析[13],并以A/Guangdong/6/2013(H7N9)HA氨基酸序列為參照,ZQ01-07HA蛋白受體結合位點發生G186V、Q226L變異;HA蛋白裂解位點序列均為PEIPKGR↓GLF,含有2個堿性氨基酸(表3)。HA蛋白上第30、46、249、421、493位氨基酸上的糖基化位點分別為NGT、NAT、NDT、NWT、NNT,未發生糖基化位點缺失。NA蛋白柄部發生了69-73位的“QISNT”氨基酸刪除;NA蛋白上分別在42、52、63、66、87、147、202上有7個潛在糖基化位點,分別表現為NCS、NTS、NET、NIT、NLT、NGT、NAS;未發現NA蛋白對達菲耐藥的H274Y、R292K、N294S、E119V/G位點變異,未發現對扎那米韋耐藥的E119A/D/G、Q136K/ L、I222R/K、D151A /E /G /V位點突變,也未發現對帕拉米韋耐藥的R292K突變。M蛋白出現了對烷胺類耐藥的S31N的突變;PB2蛋白均發生了E627K突變,提示對哺乳動物細胞親和力增強。
表3 HA蛋白裂解位點和受體結合關鍵氨基酸位點
Tab.3 Amino acid sequence at the cleavage site and receptor-binding region of the HA protein
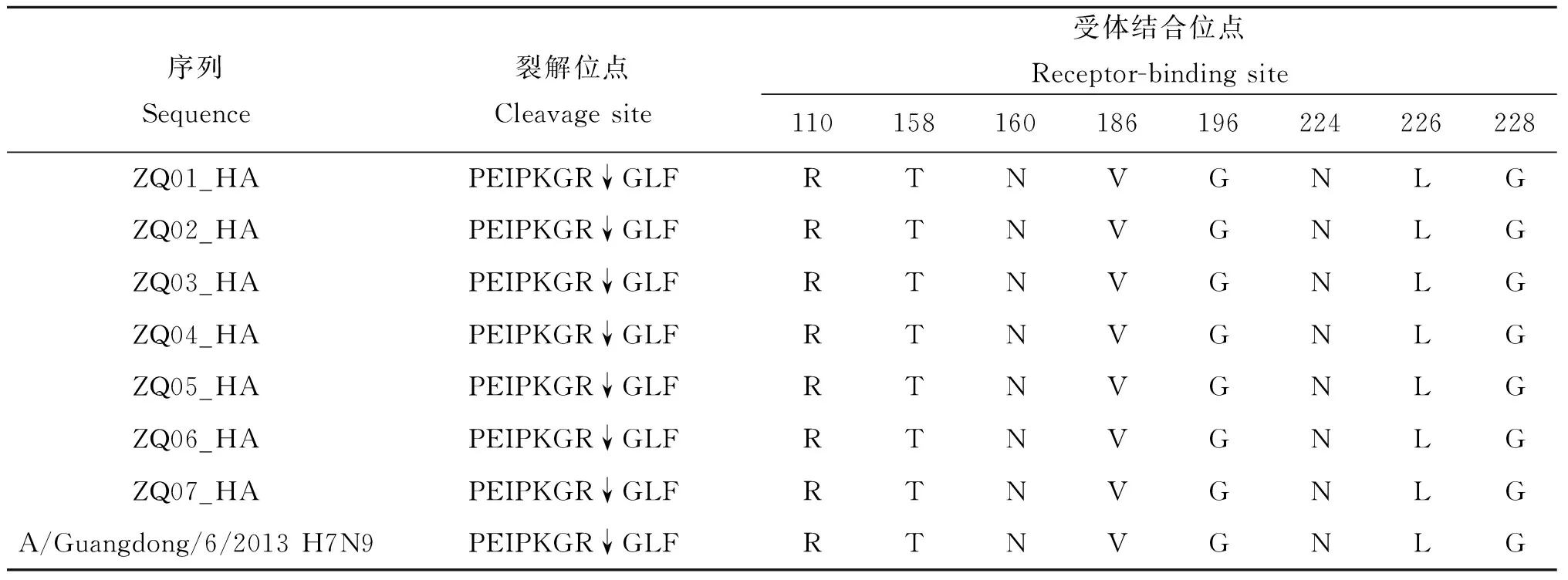
序列Sequence裂解位點Cleavagesite受體結合位點Receptor-bindingsite110158160186196224226228ZQ01_HAPEIPKGR↓GLFRTNVGNLGZQ02_HAPEIPKGR↓GLFRTNVGNLGZQ03_HAPEIPKGR↓GLFRTNVGNLGZQ04_HAPEIPKGR↓GLFRTNVGNLGZQ05_HAPEIPKGR↓GLFRTNVGNLGZQ06_HAPEIPKGR↓GLFRTNVGNLGZQ07_HAPEIPKGR↓GLFRTNVGNLGA/Guangdong/6/2013H7N9PEIPKGR↓GLFRTNVGNLG
3 討 論
獲取流感病毒基因組序列的傳統方法常用Sanger法[14],近年來,高通量測序技術迅速發展和應用,可對核酸濃度較低的H7N9樣本檢測,并在短時間內快速獲得大量有效數據[15]。本研究應用PathAmpTMFluA Reagents Kit對咽拭樣本核酸直接擴增和Sanger法測序,核酸濃度較高的7份樣本成功獲得8個節段基因全序列。研究表明新型H7N9禽流感病毒為三源重組病毒,HA和NA最初來自于2011年浙江鴨中流行的H7N3和韓國野鳥中流行的H7N9,內部基因來自于兩個不同源的H9N2[8,16]。在H7N9傳播與進化過程中,H9N2為內部基因的重排提供了基本的骨架[17],并與鄰近區域或本地的禽源H7N9病毒發生持續性重排,進化呈現區域差異[4]。本研究中的外部基因進化樹結果顯示,ZQ01-07病毒的HA和NA基因均在珠三角分支上進化,與廣東東莞雞中分離的H7N9病毒相似性最高,與長三角分支上安徽、浙江、江蘇等地分離的H7N9病毒基因序列相似性低。推測肇慶的該7例病例被同樣的傳染源傳染,可能是通過禽類貿易被廣東東莞的雞感染,也可能是和廣東東莞雞被同樣的傳染源傳染。
肇慶市是廣東省禽類養殖和消費大戶,首例H7N9病例于2014年1月在懷集縣發現時,當地活禽市場環境標本H7陽性率高達65. 0%,是人感染H7N9的高危場所。在之后2周的強化和應急監測中,該縣區域共發現9例H7N9病例。其中病例3為病例2的密接者,兩者共同生活并接觸家中飼養的雞,而其家庭環境及雞樣本檢測出H7病毒,流行病學調查判斷為家庭聚集疫情[18]。HA蛋白關鍵位點的變異與流感致病性、傳播能力密切相關[19-21]。研究表明,當HA受體結合位點發生Q226L和G186V突變時,H7N9病毒對SAα-2,6Gal 的結合力增強,導致人感染概率大大增加[8,22];當HA蛋白裂解位點處有4個以上連續的堿性氨基酸時,病毒株具有高致病性[23];當HA蛋白出現158D/224K/226L、110Y/160A/226L/228S、196R/226L/228S三種突變組合中的一種,就能使H5N1禽流感病毒在上呼吸道有效增殖并通過飛沫在雪貂間傳播[24]。ZQ01-07的HA受體結合位點均發生Q226L和G186V突變,HA蛋白裂解位點處氨基酸序列與廣東、浙江、安徽等[9,25-26]地序列一樣,均為PEIPKGR↓GLF,含有2個堿性氨基酸,對禽類是低致病性的,也未發現能使禽流感病毒在上呼吸道有效增殖并通過飛沫傳播突變組合,提示該病毒尚未獲得飛沫傳播的充分條件,人傳人可能性較小。ZQ01-02分別為病例2和3的毒株序列,HA序列分析結果進一步證明了病例2和3為共同暴露的家庭聚集疫情,而非人傳人導致的。
2016年12月以來江、浙、粵等地人感染H7N9病例數迅猛增加,報告的病例數和疫情波及范圍高于前幾年同期水平,疫情形勢嚴峻[27]。2017年1月,廣東省疾控中心對2例人感染H7N9病例分離到的病毒株中發現血凝素鏈接肽位置發生了基因插入性突變,提示該病毒突變為對禽高致病性的病毒,檢測結果已經國家流感中心復核確認[28]。值得注意的是,當中1例為本研究病例16的妹妹,43歲,曾照顧病例16,屬于密切接觸者,在其醫學觀察期間的2016年12月30日出現發熱癥狀,即到廣州市第八人民醫院就診,經廣東省疾控中心確診并發現其血凝素鏈接肽位置發生了基因插入性突變。而病例16曾接觸病死禽類,遺憾的是病例16及其家禽環境樣本的H7核酸濃度較弱,其血凝素鏈接肽位置是否發生了基因插入性突變,有待進一步研究。
[1] Jin Q. Molecular virology of medicine[M].Beijing: Science Press, 2001: 635-637. (in Chinese)
金奇. 醫學分子病毒學[M]. 北京: 科學出版社, 2001: 635-637.
[2] Tong S, Zhu X, Li Y, et al, New World Bats harbor diverse influenza A viruses[J]. PLoS Pathog, 2015, 9(10): 1078-1084.
[3] Tong S, Li Y, Rivailler P, et al, A distinct lineage of influenza A virus from bats[J]. Proc Nat Acad Sci, 2012, 109(11): 4269-4274.
[4] Qi X, Tang FY. Biological characteristics and evolutionary trend for H7N9 avian influenza virus[J]. Jiangsu J Preventive Med, 2014, 25(2): 47-50. (in Chinese)
祁賢, 湯奮揚. H7N9禽流感病毒的生物學特征及進化趨勢[J]. 江蘇預防醫學, 2014, 25(2): 47-50.
[5] Kawaoka Y, Krauss S, Webster RG. Avian-to-human transmission of the PB1 gene of influenza A viruses in the 1957 and 1968 pandemics[J]. J Virol, 1989, 63(11): 4603-4608.
[6] Vries ED, Guo H, Dai M, et al. Rapid emergence of highly pathogenic avian influenza subtypes from a subtype H5N1 hemagglutinin variant[J]. Emerg Infect Dis, 2015, 21(5): 842-846.
[7] Yu YF, Guo XL, Wang Y, et al. Analysis of molecular basis about pathogenicity of avian influenza A H7N9 virus to human[J]. J Sun Yat-sen Univ: Med Sci, 2013, 34(5): 657-665. (in Chinese)
于玉鳳, 郭曉蘭, 王穎, 等. H7N9禽流感病毒對人類致病的分子基礎分析[J]. 中山大學學報(醫學科學版), 2013, 34(5): 657-665.
[8] Gao R, Cao B, Hu Y, et al. Human infection with a novel avian-origin influenza A (H7N9) virus[J]. N Engl J Med, 2013, 368(20): 1888.
[9] Xie JF, Zhao L, Zhang YH, et al. Establishment of assay for amplifying and sequencing the complete genome of avian influenza virus A (H7N9)[J]. Chin J Zoonoses, 2015, 31(9): 795-799. (in Chinese)
謝劍鋒, 趙琳, 張炎華, 等. 人感染H7N9禽流感病毒全基因組擴增與測序方法的建立[J]. 中國人獸共患病學報, 2015, 31(9): 795-799.
[10] Chen Y, Mao HY, Li Z, et al. Genomic sequences of human infection of avian-origin influenza A (H7N9) virus in Zhejiang province[J]. Chin J Epidemiol, 2013, 34(6): 604-608. (in Chinese)
陳寅, 茅海燕, 李榛, 等. 浙江省人感染H7N9禽流感病毒的基因組序列分析[J]. 中華流行病學雜志, 2013, 34(6): 604-608.
[11] Wu J, Lu J, Faria NR, et al. Effect of live poultry market interventions on influenza A(H7N9) virus, Guangdong, China[J]. Emerg Infect Dis, 2016, 22(12): 2104-2112.
[12] Wu J, Lau EH, Xing Q, et al. Seasonality of avian influenza A(H7N9) activity and risk of human A(H7N9) infections from live poultry markets[J]. J Infect, 2015, 12(6): 690-693.
[13] Ha Y, Stevens DJ, Skehel JJ, et al. X-ray structure of the hemagglutinin of a potential H3 avian progenitor of the 1968 Hong Kong pandemic influenza virus[J]. Virology, 2003, 309(2): 209-218.
[14] Hoffmann E, Stech J, Guan Y, et al. Universal primer set for the full-length amplification of all influenza A viruses[J]. Arch Virol, 2001, 146(12): 2275-2289.
[15] Pei GQ, Fan H, An XP, et al. Detection of avian influenza A(H7N9) virus by high through put sequencing and genomic sequence analysis[J]. Acta Universitatis Medicinalis Anhui, 2014, 49(8): 1033-1038. (in Chinese)
裴廣倩, 范航, 安小平, 等. 利用高通量測序快速檢測H7N9禽流感病毒及基因組序列分析[J]. 安徽醫科大學學報, 2014, 49(8): 1033-1038.
[16] Lam TY, Wang J, Shen Y, et al, The genesis and source of the H7N9 influenza viruses causing human infections in China[J]. Nature, 2013, 502(7470): 241-244.
[17] Zou LR, Mao XX, Wu J, et al. Investigation on the environmental pollution status of avian influenza virus in live poultry market in Guangdong province during 2012-2015[J]. Acta Laser Biology Sinica, 2016, 25(3): 277-282. (in Chinese)
鄒麗容, 毛小曉, 武婕, 等. 2012-2015年廣東省活禽市場外環境禽流感病毒污染狀況研究[J]. 激光生物學報, 2016, 25(3): 276-282.
[18] Zhu BL, Mai W, Deng H, et al. Epidemiology of human infection with avian influenza A (H7N9) virus in Huaiji, Guangdong[J]. Dis Surveill, 2014, 29(9): 696-699. (in Chinese)
朱碧柳, 麥煒, 鄧鴻, 等. 廣東省肇慶市懷集縣人感染H7N9禽流感流行特征分析[J]. 疾病監測, 2014, 29(9): 696-699.
[19] Kaverin NV, Rudneva IA, Ilyushina NA, et al. Structure of antigenic sites on the haemagglutinin molecule of H5 avian influenza virus and phenotypic variation of escape mutants[J]. J General Virol, 2002, 83(Pt 10): 169-179.
[20] Sun S, Wang Q, Zhao F, et al. Glycosylation site alteration in the evolution of influenza A (H1N1) viruses[J]. PLoS One, 2010, 6(6): e22844.
[21] Guo XL, Si LL, Guan DW, et al. Whole genome sequencing and analysis of molecular characteristics of the two avian influenza A H7N9 viruses[J]. J Sun Yat-Sen Univ: Med Sci, 2015, 36(2): 167-175. (in Chinese)
郭曉蘭, 司露露, 管大偉, 等. 兩株人H7N9禽流感病毒的全基因組測序及分子特征分析[J]. 中山大學學報醫學科學版, 2015, 36(2): 167-175.
[22] Dortmans JCFM, Dekkers J, Wickramasinghe INA, et al. Adaptation of novel H7N9 influenza A virus to human receptors[J]. Scientific Reports, 2013, 3(10): 3058-3058.
[23] Jr SA, Matsuoka Y, Lau YF, et al. The multibasic cleavage site of the hemagglutinin of highly pathogenic A/Vietnam/1203/2004 (H5N1) avian influenza virus acts as a virulence factor in a host-specific manner in mammals[J]. J Virol, 2012, 86(5): 2706-2714.
[24] Russell CA, Fonville JM, Brown AE, et al. The potential for respiratory droplet-transmissible A/H5N1 influenza virus to evolve in a mammalian host[J]. Science, 2012, 336(6088): 1541-1547.
[25] Wang XG, Lei YL, Ye BF, et al. Sequencing of genome of avian influenza A (H7N9) virus isolated from human infection cases in Lishui, Zhejiang, 2014[J]. Dis Surveill, 2016, 31(2): 106-110. (in Chinese)
王曉光, 雷永良, 葉碧峰, 等. 2014年浙江省麗水市人感染H7N9禽流感病毒分離株基因特征分析[J]. 疾病監測, 2016, 31(2): 106-110.
[26] He J, Hu WF, Li FR, et al. Characterization of avian influenza virus A (H7N9) genome in Anhui Province, China[J]. Chin J Zoonoses, 2014, 30(9): 913-918. (in Chinese)
何軍, 胡萬富, 李芙蓉, 等. 安徽省人感染H7N9禽流感病毒基因組特征分析[J].中國人獸共患病學報, 2014, 30(9): 913-918.
[27] Chinese Center for Disease Control and Prevention. Chinese center for disease control and prevention held a national video conference for epidemic of human infection with avian influenza A (H7N9)[J]. Dis Surveill, 2017, 32(1): 56-65. (in Chinese)
中國疾病預防控制中心召開全國人感染H7N9禽流感疫情視頻會商會[J]. 疾病監測, 2017, 32(1): 56-65.
[28] Chinese Center for Disease Control and Prevention. H7N9 mutated virus strains were found in human cases from China[EB/OL]. (2017-02-19)[2017-03-01]. http: //www, chinacdc, cn/yw_9324/201702/t20170219_138185, html. (in Chinese)
中國疾病預防控制中心. 我國從人感染病例中發現H7N9病毒變異株 [EB/OL].(2017-02-19)[2017-03-01]. http: //www,chinacdc,cn/yw_9324/201702/t20170219_138185, html.
收稿日期:2017-01-25 編輯:林丹
Genomic characterization of avian influenza A(H7N9) virus in Zhaoqing, China, 2014-2016
TAN Han-qing1, CHENG Jie-ping1, TAN Hai-fang1, ZHU Ying-mei1, LIN Feng1, LIANG Xiao-qing2, CHEN Jie2, MAI Wei1
(1.ZhaoqingPrefectureCenterforDiseaseControlandPrevention,Zhaoqing526060,China;2.TheFirstPeople’sHospitalofZhaoqing,Zhaoqing526000,China)
We analyzed genetic evolution characteristics of avian influenza A (H7N9) virus isolated in Zhaoqing, China, 2014-2016. Nucleic acid were extracted and sequenced from 17 samples of H7N9 positive cases in Zhaoqing. Genetic characteristics of homology and important amino acid sites were analyzed by using BioEdit5.0 and MEGA6.0. The evolutionary trees were constructed by Neighbor-Joining and the referenced sequences were downloaded from GenBank. Eight nucleic acid fragments from 7 strains of H7N9 viruses were successfully generated. The highest homology was found in HA gene with A/chicken/Dongguan/695/2014(H7N9), and NA gene with A/chicken/Dongguan/1075/2014(H7N9). The internal genes were high homology with avian H7N9 and H9N2 virus from Dongguan and Shenzhen in Guangdong, China. The HA and NA genes were directly evolved in the Pearl River Delta evolution branch with the H7N9 sequences from the cities of Dongguan, Guangzhou and Shenzhen, while the sequences from the provinces of Anhui, Zhejiang, and Jiangsu were in the Yangtze River Delta evolution branch. There were 2 alkaline amino acids in cleavage site of HA, 2 mutations (G186V and Q226L) in the crucial sites related with the receptor of HA protein, 1 mutation (E627K) in PB2 protein, and 1 drug resistance mutation (S31N) in M2 protein. And no evidence of neuraminidase resistance in NA protein was found. In conclusion, the H7N9 virus for human infection in Zhaoqing may originate from avian H7N9 and H9N2 viruses, which circulated in the Pearl River Delta region of Guangdong from 2013 to 2014. The mutations of G186V, Q226L and E627K might be related with high susceptibility to human beings.
human infection with avian influenza A(H7N9) virus; hemagglutinin gene; neuraminidase gene; sequence analysis Funded by the Medical Science and Technology Research Foundation of Guangdong Province (No. A2015573) and the Zhaoqing Science and Technology Innovation Project (No. 2015E1810)
2017-03-20 編輯:林丹
10.3969/j.issn.1002-2694.2017.03.003
1.廣東省肇慶市疾病預防控制中心,肇慶 526060; 2.廣東省肇慶市第一人民醫院,肇慶 526000
Email:tanhaqing2000@163.com
R373.1
A
1002-2694(2017)03-0202-06
廣東省醫學科學技術研究基金(No.A2015573)和肇慶市科技創新項目(No.2015E1810)聯合資助

