系統性硬化癥遺傳學研究進展
濮偉霖+郭士成+王久存
摘 要 系統性硬化癥又稱為硬皮病,是一種復雜的自身免疫性疾病,目前對于該疾病的病因了解尚不清晰。多項研究顯示,遺傳因素在硬皮病的發病過程中起重要作用。因此對于硬皮病遺傳因素的探索有助于我們發現硬皮病發病的相關通路和分子機制。本文介紹并綜述了硬皮病截止目前的遺傳學相關研究的前沿進展。
關鍵詞 硬皮病 遺傳 全基因組關聯研究(GWAS)
中圖分類號:R593.25 文獻標識碼:A 文章編號:1006-1533(2017)S1-0030-07
The review of the advancement of SSc genetic studies*
PU Weilin**, GUO Shicheng, WANG Jiucun***
(Department of Life Sciences, Fudan University, Shanghai 200438, China)
ABSTRACT Systemic sclerosis is a kind of complex autoimmune diseases with different manifestations. The pathogenesis of SSc is still unknown. Multiple studies have revealed the important role of genetic factors in the pathogenesis of SSc. As a result, the exploration of the genetic factors of SSc will be of great benefit to the discovery of the associated pathways and mechanisms of SSc pathogenesis. This paper summarizes the advances and leading edge of the SSc genetic studies till now.
KEY WORDS Scleroderma; Genetics; genome-wide association study (GWAS)
系統性硬化癥(systemic sclerosis,SSc,又稱硬皮病)是不明病因的復雜性自身免疫性疾病。多項研究已經發現系統性硬化癥的發病與遺傳因素密切相關。從早期的候選基因研究到近年來的全基因組關聯研究,大量的遺傳易感因素與硬皮病的臨床表型密切相關,提示遺傳因素對于硬皮病的發病具有重要作用。近年來,隨著基因芯片及高通量測序技術的進步和價格的不斷下降,使得對于復雜疾病的遺傳因素解析更為便捷。本文綜述了從早期至近期的硬皮病遺傳學研究進展。
1 系統性硬化癥的早期遺傳學研究
在SSc研究早期,美國德克薩斯大學學者Arnett對俄克拉荷馬州21 255名Choctaw 印第安人進行研究,發現Choctaw 印第安人系統性硬皮病(即系統性硬化癥,SSc)發病率遠高于正常人群,且未發現顯著環境因素差異,故提示可能存在遺傳因素影響[1]。早期研究已經發現SSc患者體內可產生自身抗體。而HLA II 型分子可以將處理后的抗原呈遞給T細胞,從而產生抗原特異性的反應,說明HLA區域和系統性硬皮病的發生密切相關。此外,當時在其他美國印第安人中也發現了HLA區域與其他自身免疫性疾病如風濕病和結締組織病相關。故Arnett等用12個患者樣本和48個對照樣本研究HLA區域與SSc的關聯,發現3個HLA區域的等位基因與SSc相關,分別為DRB1*1602、DQB1*0301和DQA1*0501。
這一研究成果確認了SSc與HLA區域的相關性,隨后針對HLA區域和系統性硬皮病(SSc)遺傳易感性之間的關聯研究逐漸增多,并陸續發現HLA區域的多個等位基因與SSc遺傳易感性相關(表1)[2]。
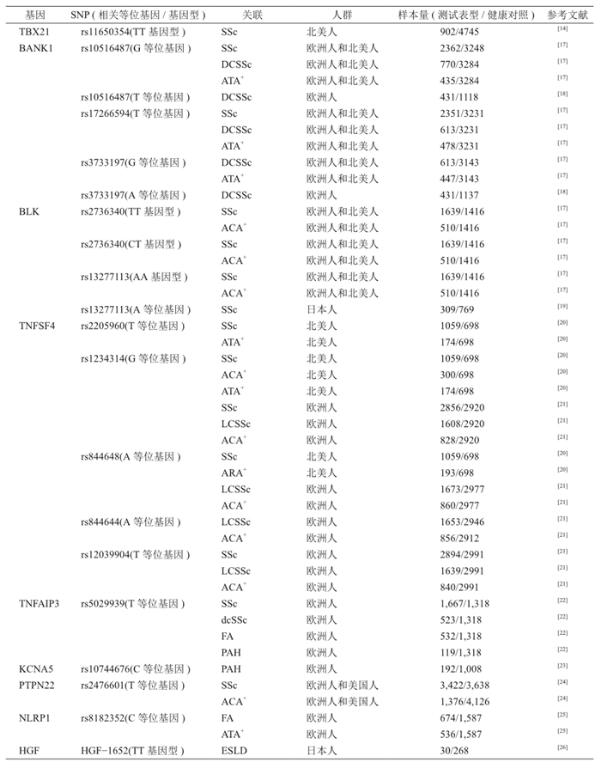
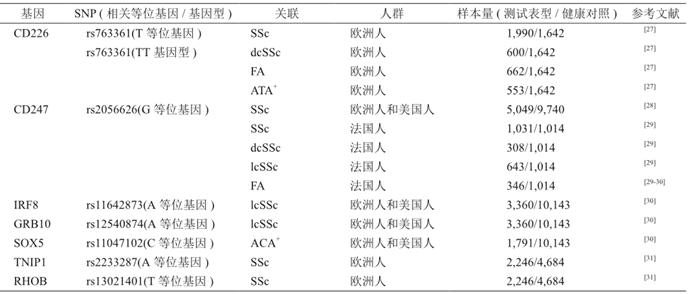
SSc作為一種復雜的自身免疫性疾病,除了自身免疫被激活外,其特征還包括微血管的損傷和皮膚及內臟的纖維化,因此SSc的發生除了與HLA區域的基因密切相關外,也與非HLA區域的基因密切相關。故在HLA基因與SSc的關系不斷被揭示時,也有許多研究發現SSc與非HLA區域的基因之間的關聯(表2)[9-10]。
1.1 IRF5
IRF5 (interferon regulatory factor 5,干擾素調節因子5) 是 I型干擾素(type I IFN) 信號通路中的重要調節因子。而干擾素(interferon, IFN)是固有免疫的重要組成部分,可以通過激活NK細胞、細胞毒性T細胞,促進單核細胞和漿細胞的成熟等方式來應對感染。一項法國的研究利用法國高加索人群(881 病例/760對照)發現IRF5 內含子區域rs2004640 T 等位基因與SSc相關聯[15]。隨后另一項日本的研究(281病例/477對照)確認IRF5了 rs2004640與SSc的關聯,并發現rs10954213和rs2280714這兩個SNP也與SSc關聯[16]。
1.2 STAT4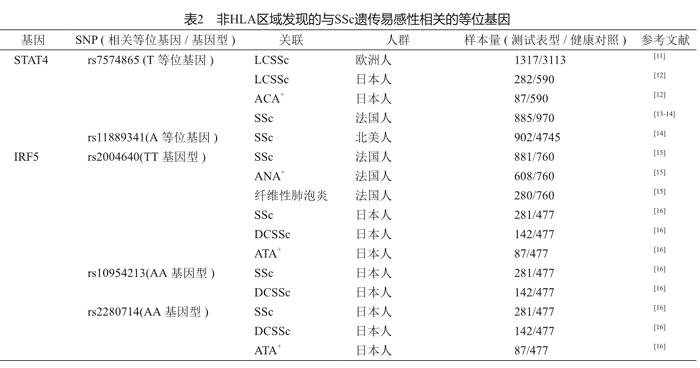
STAT4(signal transduction and activator of transcription-4,信號轉導和轉錄激活因子4)屬于轉錄因子家族,參與Th1和Th17細胞的分化過程。已有研究證明STAT4基因與SLE和RA均相關[32]。目前已發現STAT4基因的多個SNP和SSc相關。在一項包括了5個歐洲高加索人群的共計1 317名SSc病例和3 113名正常對照的研究中,發現STAT4 上的rs7574865 T 等位基因和lcSSc相關,但與dcSSc不相關[19]。隨后的兩個GWAS研究均證明了STAT4基因和SSc之間的關聯[28,31]。
1.3 CTGF
CTGF(connective tissue growth factor,結締組織生長因子)可以通過膠原和細胞外基質的生成來參與纖維化的過程。已發現在SSc病人血清中CTGF表達量上調[33]。一項英國的病例對照研究(500病例/500對照)首次發現CTGF上945G等位基因和SSc相關[34]。隨后,日本的一項SSc研究(395病例/269對照)驗證了CTGF和SSc的關聯[35]。但是,一項多中心的法國SSc研究(1 180病例/1 784對照)沒能驗證CTGF和SSc之間的關聯[11]。這些研究說明CTGF和SSc的關聯尚待進一步的研究確認。
1.4 BANK1
BANK1(B cell scaffold protein with ankyrin repeats,具有錨蛋白重復序列的B 細胞支架蛋白)基因編碼BANK1蛋白,BANK1蛋白是一種B細胞適配蛋白,可以與B細胞上受體和B細胞下游激酶結合。已有研究確認BANK1與RA和SLE相關,提示BANK1也可能與SSc相關[11,17,36]。在一項包括2 380名SSc患者和3 270名正常對照的多中心研究中,發現rs10516487G 和rs17266594T 等位基因和dcSSc關聯[17]。隨后的德國和法國的研究也確認了上述等位基因和dcSSc之間的關聯[18]。
2 SSc全基因組關聯研究進展
從2009年至今,已發表4篇SSc的全基因組關聯研究(GWAS),通過大樣本量研究,確認了早期發現的多個基因與SSc的關聯,同時也發現了多個新的與SSc相關的基因。
第一項針對SSc的GWAS研究利用137名韓國SSc患者和564名正常對照,發現HLA-DPB1和HLA-DPB2上5個SNP rs3128930A、 rs7764491C、rs7763822T、rs3128965A和rs3117230G與SSc亞型ATA(antitopoisomerase I,抗拓撲異構酶I)陽性相關。在驗證階段該研究采納了110名美國高加索裔SSc病人和2 747名正常對照,成功驗證兩對SNP rs7763822/rs7764491和rs3117230/rs3128965與ATA+或ACA+相關[37]。
2010年,Mayes等發表了迄今最大規模的SSc GWAS研究。這項研究包括了美國、西班牙、德國和荷蘭的共計2 296名SSc患者和5 171名正常對照,發現多個SNP在經過多重檢驗矯正后P值仍然達到了GWAS的顯著水平(P≤5×10-7)。其中,HLA-DQB1基因的SNP rs6457617 最為顯著。在MHC區域外,包括TNPO3/IRF5、STAT4、CD247、CDH7、EXOC2/IRF4在內的5個基因達到了GWAS顯著水平。隨后,在驗證階段,該研究納入了2 753名高加索SSc患者和4 569名正常對照,結果CDH7和EXOC2/IRF4未能通過驗證。其他通過驗證的三個基因中,STAT4和IRF5與SSc的關聯之前已經發現,而CD247是首次發現與SSc相關[28]。
2011年,Martin 等利用2010年SSc GWAS的數據,擴大了驗證階段的樣本量,合并樣本量達到5 471例SSc病人和10 143例正常對照。該研究合并分析后發現IRF8基因上的SNP rs11642873與lcSSc相關,且P值(2.32×10-12)達到了GWAS的顯著水平。而另外兩個基因GRB10和SOX5分別和lcSSc(P=1.27×10-6)和ATA(1.39×10-7)陽性相關,但是P值略低于GWAS的顯著水平,提示這兩個基因可能也與SSc相關,但尚需進一步確認[30]。
同在2011年,法國的Allanore等[31]也發表了一項GWAS研究,他們主要利用法國SSc樣本,并在驗證階段中包括了意大利、德國等多個種族的人群,合并樣本量達到1 682例病人和3 926例正常對照。該研究發現HLA區域的PSORS1C1(P=5.7×10-10)和HLA-DQB1(P=2.33×10-37)與SSc相關。而在HLA區域之外,還發現了RHOB和TNIP上的多個SNP可能與SSc相關。
2013年,Martin等對SSc和SLE進行了一項metaGWAS研究。他們利用Mayes[28]的SSc GWAS研究數據和Harley[38] 的SLE GWAS數據,將這兩篇GWAS研究合并,從中篩選20個共有的SNP進行驗證。該研究在驗證階段納入了1 578例SSc病例和3 446名正常對照以及1 727例SLE病例和3 447例正常對照,總計樣本量達到21 109例。該研究結果發現KIAA0319L基因(P=3.31×10-11)是SSc和SLE共同的新發現的關聯基因,并首次發現SLE的易感基因PXK(P=3.27×10-11)和JAZF(P=1.11×10-8)與SSc相關聯[39]。
除了傳統的GWAS之外,Mayes等于2014年首次完成了SSc的Immunochip的研究。他們利用Immunochip對1 833名SSc病例和3 466名正常對照進行檢測,在HLA區域發現了6個氨基酸多態位置和7個SNP與SSc相關。而在非HLA區域,該研究利用4 017名SSc病例和5 935名正常對照進行了驗證,合并樣本量達到5 835/9 401,發現了DNASE1L3、CHIPIL12A和ATG53個基因與SSc和SSc的亞型相關。其中DNASE1L3與SSc(P = 3.36×10-16),ATA+(4.25×10-31)和lcSSc(P = 1.28×10-20)均相關,SCHIP-IL12A也與SSc(P= 1.22×10-11),ATA+(2.40×10-8)和lcSSc(P = 1.53×10-11)均相關,而ATG5則與SSc相關(P = 3.75×10-8)[40]。
3 SSc多態位點在中國漢族人群中的驗證
不同種族人群存在著遺傳結構差異,硬皮病的流行病學特征在不同地區及種族中具異質性,因此有必要對不同種族人群進行比較研究,以發現影響硬皮病的關鍵基因或位點。在高加索人中發現及驗證過的多態位點是否也是漢族人群SSc的易感位點需要在漢族人群樣本中進行研究。
本實驗室及合作者于2013年利用453例漢族SSc患者和534例正常對照進行研究,發現中國漢族人群中 STAT4基因上的兩個SNP(rs7574865和rs10168266)與SSc相關。其中rs7574865的基因型GG(P = 4.1×10-3)和等位基因T(P = 4.1×10-4)在SSc病人中出現頻率與正常對照中頻率存在明顯差異,而rs10168266 的基因型GG(P = 0.005 1)和等位基因T(P = 0.005 1)在SSc病人中出現的頻率與正常對照中頻率也存在明顯差異[41]。本項目組利用424例漢族SSc患者和502例正常對照研究后發現IRF5 rs2004640 TT 基因型在纖維化的SSc患者和ATA陽性患者中頻率顯著上升。且ACA+和lcSSc這兩種亞型中IRF5 rs2004640 T 基因頻率顯著上升,成功驗證了IRF5基因與漢族人群SSc的關聯[42]。
本項目組利用213名漢族SSc患者和239名漢族正常對照在漢族人群中驗證了HLA-DQB1*0501(P = 1.6×10-5)是漢族人群SSc的易感位點。而DQB1*0601與 ATA+ SSc(P = 0.031)相關聯,DQB1*0303與antiU1RNP陽性(P = 0.02)關聯[43]。此外,在漢族人群(585 SSc患者/458正常對照)中驗證了HLA-DRB1的多個等位基因與SSc相關[44]。我們也在漢族人群中(338名SSc患者/480名正常對照)中驗證出HLA-DPB1上的多個等位基因與SSc和SSc的亞型相關[45]。雖然2010年的最大規模GWAS研究中發現CD247與SSc相關,但我們在漢族人群中(387病例/523對照)未能驗證出CD247基因與漢族人群SSc之間相關,說明CD247與SSc在漢族人群中的相關性尚需進一步確認[46]。
4 拷貝數變異
GWAS研究的原理基于常見疾病/常見變異的理論。雖然目前GWAS已經發現了一系列硬皮病易感基因及位點,但從根本上其只能發現頻率高于5%的疾病相關遺傳變異。因此目前GWAS發現的SSc易感位點只能解釋很小一部分的性狀遺傳或致病風險。頻率較低的單核苷酸多態性則無法通過現有GWAS方法進行研究,即目前的SSc的易感性研究中存在大量的遺傳力丟失[47]。研究發現,拷貝數變異(CNV)在復雜疾病的易感性上也占有重要的貢獻。最新的千人基因組發現CNV變異所涉及的基因組區域是SNP的10倍之多,因此,CNV可能對人類表型或疾病產生更加廣泛的影響。目前基于全基因組CNV關聯研究主要集中在腫瘤及心血管、神經系統疾病中,截至2015年,尚沒有基于CNV的全基因組關聯研究在SSc中開展,但已經有一些基于候選基因方法的CNV關聯研究。2010年Zorzetto博士基于意大利人群的研究結果顯示C4基因(6p21.3)的拷貝數變異與SSc的易感性顯著相關。2012年McKinney博士通過對777位歐洲高加索SSc患者和1 000正常人的研究發現FCGR3B拷貝數缺失是SSc的一個風險遺傳變異[48]。本實驗室2014年首次利用比較基因組雜交方法(CGH)開展了針對中國人群的全基因組CNV關聯研究,發現在中國漢族SSc人群中存在大量的候選易感性CNV位點,并通過大樣本人群實驗表明HLA-DQA1和APOBEC3A/3B基因的CNV對SSc的易感性具有顯著的貢獻[49]。
5 全外顯子組研究
隨著高通量測序技術價格的不斷下降,個人基因組測序的成本急劇下降,使得基于全基因組或全外顯子組測序的大樣本疾病遺傳學研究成為可能。Gao等于2016年首次利用全外顯子測序技術對78例歐洲高加索硬皮病患者進行測序。該研究利用公共數據庫中的3 179例正常樣本的全外顯子數據作為對照,發現ATP8B4基因頻率在硬皮病患者和正常對照中存在顯著差異(P= 2.77×10-7)。深入研究發現ATP8B4基因中的rs55687265位點的差異最為顯著(P=9.35×10-10)。隨后于第二階段利用415例硬皮病患者和2 848例正常對照,對第一階段的結果進行驗證。合并分析的結果也驗證了該位點與硬皮病的發病相關(P=1.92×10-7, OR=2.5)。此外,該研究還發現ASB10、CNGB3和HLA-DRB5和HSPB2等基因可能與硬皮病的發病存在可能的相關性,需要更多的研究加以確認[50]。
6 結語
截止目前,包括本實驗室在內,通過高通量的芯片技術和測序技術發現了大量與硬皮病發病相關的風險和保護位點,使我們對于硬皮病發病的遺傳機制有了深入的了解。但由于硬皮病患病率低,研究的樣本量較小,同時研究技術手段的局限也使得對于硬皮病遺傳背景的了解尚待深入。隨著高通量測序技術的開展,相信在近期可對硬皮病的遺傳風險因素有更加細致的了解,同時對其作用和功能進行更為精細的描述和認識。
參考文獻
[1] Arnett FC, Howard RF, Tan F, et al. Increased prevalence of systemic sclerosis in a Native American tribe in Oklahoma. Association with an Amerindian HLA haplotype[J]. Arthritis Rheum, 1996, 39(8): 1362-1370.
[2] Martin JE, Bossini-Castillo L, Martin J. Unraveling the genetic component of systemic sclerosis[J]. Hum Genet, 2012, 131(7): 1023-1037.
[3] Muller-Hilke B. HLA class II and autoimmunity: epitope selection vs differential expression[J]. Acta Histochem, 2009, 111(4): 379-381.
[4] Simeon CP, Fonollosa V, Tolosa C, et al. Association of HLA class II genes with systemic sclerosis in Spanish patients[J]. J Rheumatol, 2009, 36(12): 2733-2736.
[5] Gilchrist FC, Bunn C, Foley PJ, et al. Class II HLA associations with autoantibodies in scleroderma: a highly significant role for HLA-DP[J]. Genes Immun, 2001, 2(2): 76-81.
[6] Arnett FC, Gourh P, Shete S, et al. Major histocompatibility complex (MHC) class II alleles, haplotypes and epitopes which confer susceptibility or protection in systemic sclerosis: analyses in 1300 Caucasian, African-American and Hispanic cases and 1000 controls[J]. Ann Rheum Dis, 2010, 69(5): 822-827.
[7] Rands AL, Whyte J, Cox B, et al. MHC class II associations with autoantibody and T cell immune responses to the scleroderma autoantigen topoisomerase I[J]. J Autoimmun, 2000, 15(4): 451-458.
[8] Kuwana M, Inoko H, Kameda H, et al. Association of human leukocyte antigen class II genes with autoantibody profiles, but not with disease susceptibility in Japanese patients with systemic sclerosis[J]. Intern Med, 1999, 38(4): 336-344.
[9] Broen JC, Coenen MJ, Radstake TR. Deciphering the genetic background of systemic sclerosis[J]. Expert Rev Clin Immunol, 2011, 7(4): 449-462.
[10] Broen JC, Coenen MJ, Radstake TR. Genetics of systemic sclerosis: an update[J]. Curr Rheumatol Rep, 2012, 14(1): 11-21.
[11] Rueda B, Simeon C, Hesselstrand R, et al. A large multicentre analysis of CTGF -945 promoter polymorphism does not confirm association with systemic sclerosis susceptibility or phenotype[J]. Ann Rheum Dis, 2009, 68(10): 1618-1620.
[12] Tsuchiya N, Kawasaki A, Hasegawa M, et al. Association of STAT4 polymorphism with systemic sclerosis in a Japanese population[J]. Ann Rheum Dis, 2009, 68(8): 1375-1376.
[13] Dieude P, Guedj M, Wipff J, et al. STAT4 is a genetic risk factor for systemic sclerosis having additive effects with IRF5 on disease susceptibility and related pulmonary fibrosis[J]. Arthritis Rheum, 2009, 60(8): 2472-2479.
[14] Gourh P, Agarwal SK, Divecha D, et al. Polymorphisms in TBX21 and STAT4 increase the risk of systemic sclerosis: evidence of possible gene-gene interaction and alterations in Th1/Th2 cytokines[J]. Arthritis Rheum, 2009, 60(12): 3794-3806.
[15] Dieude P, Guedj M, Wipff J, et al. Association between the IRF5 rs2004640 functional polymorphism and systemic sclerosis: a new perspective for pulmonary fibrosis[J]. Arthritis Rheum, 2009, 60(1): 225-233.
[16] Ito I, Kawaguchi Y, Kawasaki A, et al. Association of a functional polymorphism in the IRF5 region with systemic sclerosis in a Japanese population[J]. Arthritis Rheum, 2009, 60(6): 1845-1850.
[17] Rueda B, Gourh P, Broen J, et al. BANK1 functional variants are associated with susceptibility to diffuse systemic sclerosis in Caucasians[J]. Ann Rheum Dis, 2010, 69(4): 700-705.
[18] Dieude P, Wipff J, Guedj M, et al. BANK1 is a genetic risk factor for diffuse cutaneous systemic sclerosis and has additive effects with IRF5 and STAT4[J]. Arthritis Rheum, 2009, 60(11): 3447-3454.
[19] Ito I, Kawaguchi Y, Kawasaki A, et al. Association of the FAM167A-BLK region with systemic sclerosis[J]. Arthritis Rheum, 2010, 62(3): 890-895.
[20] Gourh P, Arnett FC, Tan FK, et al. Association of TNFSF4 (OX40L) polymorphisms with susceptibility to systemic sclerosis[J]. Ann Rheum Dis, 2010, 69(3): 550-555.
[21] Bossini-Castillo L, Broen JC, Simeon CP, et al. A replication study confirms the association of TNFSF4 (OX40L) polymorphisms with systemic sclerosis in a large European cohort[J]. Ann Rheum Dis, 2011, 70(4): 638-641.
[22] Dieude P, Guedj M, Wipff J, et al. Association of the TNFAIP3 rs5029939 variant with systemic sclerosis in the European Caucasian population[J]. Ann Rheum Dis, 2010, 69(11): 1958-1964.
[23] Wipff J, Dieude P, Guedj M, et al. Association of a KCNA5 gene polymorphism with systemic sclerosis-associated pulmonary arterial hypertension in the European Caucasian population[J]. Arthritis Rheum, 2010, 62(10): 3093-3100.
[24] Diaz-Gallo LM, Gourh P, Broen J, et al. Analysis of the influence of PTPN22 gene polymorphisms in systemic sclerosis[J]. Ann Rheum Dis, 2011, 70(3): 454-462.
[25] Dieude P, Guedj M, Wipff J, et al. NLRP1 influences the systemic sclerosis phenotype: a new clue for the contribution of innate immunity in systemic sclerosis-related fibrosing alveolitis pathogenesis[J]. Ann Rheum Dis, 2011, 70(4): 668-674.
[26] Lakos G, Melichian D, Wu M, et al. Increased bleomycin-induced skin fibrosis in mice lacking the Th1-specific transcription factor T-bet[J]. Pathobiology, 2006, 73(5): 224-237.
[27] Dieude P, Guedj M, Truchetet ME, et al. Association of the CD226 Ser(307) variant with systemic sclerosis: evidence of a contribution of costimulation pathways in systemic sclerosis pathogenesis[J]. Arthritis Rheum, 2011, 63(4): 1097-1105.
[28] Radstake TR, Gorlova O, Rueda B, et al. Genome-wide association study of systemic sclerosis identifies CD247 as a new susceptibility locus[J]. Nat Genet, 2010, 42(5): 426-429.
[29] Dieude P, Boileau C, Guedj M, et al. Independent replication establishes the CD247 gene as a genetic systemic sclerosis susceptibility factor[J]. Ann Rheum Dis, 2011, 70(9): 1695-1696.
[30] Gorlova O, Martin JE, Rueda B, et al. Identification of novel genetic markers associated with clinical phenotypes of systemic sclerosis through a genomewide association strategy[J]. PLoS Genet, 2011, 7(7): e1002178.
[31] Allanore Y, Saad M, Dieude P, et al. Genome-wide scan identifies TNIP1, PSORS1C1, and RHOB as novel risk loci for systemic sclerosis[J]. PLoS Genet, 2011, 7(7): e1002091.
[32] Remmers EF, Plenge RM, Lee AT, et al. STAT4 and the risk of rheumatoid arthritis and systemic lupus erythematosus[J]. N Engl J Med, 2007, 357(10): 977-986.
[33] Igarashi A, Nashiro K, Kikuchi K, et al. Significant correlation between connective tissue growth factor gene expression and skin sclerosis in tissue sections from patients with systemic sclerosis[J]. J Invest Dermatol, 1995, 105(2): 280-284.
[34] Fonseca C, Lindahl GE, Ponticos M, et al. A polymorphism in the CTGF promoter region associated with systemic sclerosis[J]. N Engl J Med, 2007, 357(12): 1210-1220.
[35] Kawaguchi Y, Ota Y, Kawamoto M, et al. Association study of a polymorphism of the CTGF gene and susceptibility to systemic sclerosis in the Japanese population[J]. Ann Rheum Dis, 2009, 68(12): 1921-1924.
[36] Kozyrev SV, Abelson AK, Wojcik J, et al. Functional variants in the B-cell gene BANK1 are associated with systemic lupus erythematosus[J]. Nat Genet, 2008, 40(2): 211-216.
[37] Zhou X, Lee JE, Arnett FC, et al. HLA-DPB1 and DPB2 are genetic loci for systemic sclerosis: a genome-wide association study in Koreans with replication in North Americans[J]. Arthritis Rheum, 2009, 60(12): 3807-3814.
[38] International Consortium for Systemic Lupus Erythematosus G, Harley JB, Alarcon-Riquelme ME, et al. Genome-wide association scan in women with systemic lupus erythematosus identifies susceptibility variants in ITGAM, PXK, KIAA1542 and other loci[J]. Nat Genet, 2008, 40(2): 204-210.
[39] Martin JE, Assassi S, Diaz-Gallo LM, et al. A systemic sclerosis and systemic lupus erythematosus pan-meta-GWAS reveals new shared susceptibility loci[J]. Hum Mol Genet, 2013, 22(19): 4021-4029.
[40] Mayes MD, Bossini-Castillo L, Gorlova O, et al. Immunochip analysis identifies multiple susceptibility loci for systemic sclerosis[J]. Am J Hum Genet, 2014, 94(1): 47-61.
[41] Yi L, Wang JC, Guo XJ, et al. STAT4 is a genetic risk factor for systemic sclerosis in a Chinese population[J]. Int J Immunopathol Pharmacol, 2013, 26(2): 473-478.
[42] Wang J, Yi L, Guo X, et al. Association of the IRF5 SNP rs2004640 with systemic sclerosis in Han Chinese[J]. Int J Immunopathol Pharmacol, 2014, 27(4): 635-638.
[43] Zhou XD, Yi L, Guo XJ, et al. Association of HLA-DQB1*0501 with scleroderma and its clinical features in Chinese population[J]. Int J Immunopathol Pharmacol, 2013, 26(3): 747-751.
[44] He D, Wang J, Yi L, et al. Association of the HLA-DRB1 with scleroderma in Chinese population[J]. PLoS One, 2014, 9(9): e106939.
[45] Wang J, Guo X, Yi L, et al. Association of HLA-DPB1 with scleroderma and its clinical features in Chinese population[J]. PLoS One, 2014, 9(1): e87363.
[46] Wang J, Yi L, Guo X, et al. Lack of Association of the CD247 SNP rs2056626 with Systemic Sclerosis in Han Chinese[J]. Open Rheumatol J, 2014, 8: 43-45.
[47] Hemminki K, Forsti A, Houlston R, et al. Searching for the missing heritability of complex diseases[J]. Hum Mutat, 2011, 32(2): 259-262.
[48] McKinney C, Broen JC, Vonk MC, et al. Evidence that deletion at FCGR3B is a risk factor for systemic sclerosis[J]. Genes Immun, 2012, 13(6): 458-460.
[49] Guo S, Li Y, Wang Y, et al. Copy Number Variation of HLA-DQA1 and APOBEC3A/3B Contribute to the Susceptibility of Systemic Sclerosis in the Chinese Han Population[J]. J Rheumatol, 2016, 43(5): 880-886.
[50] Gao L, Emond MJ, Louie T, et al. Identification of Rare Variants in ATP8B4 as a Risk Factor for Systemic Sclerosis by Whole-Exome Sequencing[J]. Arthritis Rheumatol, 2016, 68(1): 191-200.

