油梨果肉DNA提取方法的比較
葛宇 林興娥 王甲水 臧小平 馬蔚紅

摘 要 為了獲取高質量的油梨果肉DNA,以油梨果肉為試材,比較改良SDS法、改良CTAB法和試劑盒法對油梨果肉DNA的提取效果。結果表明:上述3種方法均可從油梨果肉中提取DNA,但改良SDS法與改良CTAB法所獲得的DNA濃度都在600 μg/mL以上,純度(OD260nm/OD280nm)均在1.8左右,純度高且完整性較好,可用于未來分子標記分析。
關鍵詞 油梨 ;果肉 ;DNA提取 ;改良SDS法 ;改良CTAB法
中圖分類號 S667.9 文獻標識碼 A Doi:10.12008/j.issn.1009-2196.2016.12.007
油梨(Persea americana Mill.)為樟科油梨屬植物,又名鱷梨、牛油果,英文名為avocado。油梨起源于中美洲、墨西哥熱帶濕潤地區及海拔較高的山地森林或熱帶高原,是著名的熱帶、亞熱帶果樹,也是一種木本油料作物[1]。油梨于1918年被引種到中國臺灣省,在海南、廣東、廣西、貴州、云南、臺灣等地均有栽培[2-5]。油梨果實形狀一般為卵圓形、倒卵圓形、圓形及梨形,重量通常為50~2 000 g[6]。油梨果肉是由果皮發育而成,富含脂肪酸、膳食纖維、蛋白質、多酚、黃酮、葉酸等多種營養物質及活性成分,其中油梨脂肪酸含量根據品種及種植條件的差異可達到其果實重量的15%~30%[7-8]。但對于DNA提取,多酚、黃酮、脂肪酸等物質會使DNA氧化成褐色凝膠狀物或與DNA結合成黏稠膠狀物,阻礙其作為PCR模板進行分析[9-11]。這就需要對傳統的DNA提取方法加以改進,以提取出高質量的DNA。周海蘭等[12]以油梨葉片為試材,對比了傳統CTAB法、改良CTAB法和試劑盒法3種方法的提取效果。結果顯示,通過改良CTAB法所提取的DNA的濃度和純度要遠高于通過傳統CTAB法所提取的DNA。但周海蘭等[12]僅對傳統的CTAB法進行了改良,而未對另一種廣泛應用的SDS法進行改良并分析其效果。此外,油梨果肉中所含有的影響DNA提取的物質含量,如脂肪酸、多酚、黃酮等含量,要遠高于油梨葉片[6]。所以提取油梨果肉的難度要遠高于油梨葉片。提取到高質量的DNA或RNA是許多分子生物學實驗(PCR擴增、基因克隆、基因表達等)的初始第一步驟,而油梨作為熱帶水果,果肉是油梨研究的主要部位,對油梨果肉組織進行分子生物學實驗將有助于深入研究油梨果肉中營養物質及活性成分的生成、代謝等[1,13]。前人已經發表了有關油梨果肉RNA提取的優化方案,但對于相對較容易的油梨果肉DNA提取優化方案未見相關報道[13]。本實驗通過比較改良SDS法、改良CTAB法和試劑盒法對油梨果肉DNA的提取效果,以期獲得高效的油梨果肉DNA提取方法,為后續的分子生物學實驗提供參考。
1 材料與方法
1.1 材料
本試驗所用優良油梨品系‘RN-5果實樣品于2016年8月在海南省儋州市中國熱帶農業科學院果樹基地采集。每個DNA提取方法重復3次。
1.2 方法
1.2.1 改良SDS法和改良CTAB法
本實驗采用的改良SDS法和改良CTAB法參照朱威龍等[14]的SDS法和CTAB法,改良之處如下:(1)在樣品研磨步驟中加入0.03 g聚乙烯吡咯烷酮;(2)在SDS提取緩沖液或CTAB提取緩沖液中加入2%(m/V)的聚乙烯吡咯烷酮;(3)在溫水水浴前,加入20 μL β-巰基乙醇,并于SDS提取緩沖液或CTAB提取緩沖液混勻;(4)水浴冷卻后加入的提取液中加入酚,并且酚與氯仿和異戊醇的體積比為25∶24∶1;(5)延長異丙醇沉淀時間到過夜。
1.2.2 試劑盒法
稱取幼嫩葉片0.2 g,加入液氮充分研磨,使用天根生化科技(北京)有限公司生產的DNA secure plant Kit(DP320)新型植物基因組DNA提取試劑盒(離心柱型)進行提取,具體操作見產品說明書。
1.2.3 DNA質量檢測
取DNA樣品20 μL,用TE緩沖液稀釋至10倍,用紫外分光光度計測定其OD260nm和OD280nm。DNA濃度=OD260nm×10(稀釋倍數)×50(μg/mL);DNA純度=OD260nm/OD280nm。
取DNA樣品10 μL,加入5 μL溴酚藍緩沖液,用5%瓊脂糖凝膠電泳,15 min后拍照,分析DNA的質量和完整性。
1.2.4 EST-SSR分子標記驗證提取效果
以3種提取方法所獲得的DNA為模板,采用油梨EST-SSR引物SHRSPa003進行PCR擴增,以檢測3種DNA 提取方法的效果。油梨EST-SSR引物SHRSPa003序列信息來自Borrone等[15]的文獻,油梨EST-SSR引物PCR擴增反應體系參照Ge等[16]的方法。擴增反應結束后取10 μL擴增產物進行電泳、染色、拍照分析。
2 結果與分析
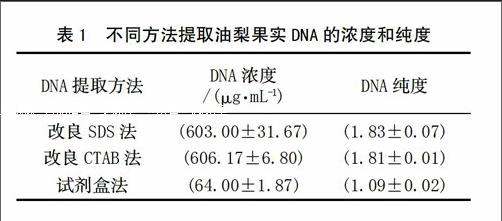
2.1 不同提取方法所得的DNA濃度和純度
由表1可知,改良SDS法與改良CTAB法提取出的DNA濃度均超過600 μg/mL,濃度較高。2種方法純度也均超過1.8,表明DNA樣品中的蛋白質、酚類和脂肪酸去除較為充分,雜質較少,其DNA質量滿足后續實驗的要求。試劑盒法提取的DNA濃度僅為其他2種方法的十分之一,DNA純度也較比其他2種方法低近1倍,其DNA質量很難滿足后續實驗的要求。
2.2 DNA瓊脂糖凝膠電泳
由圖1可知,改良SDS法、改良CTAB法和試劑盒法3種方法均能從油梨果肉中提取出DNA,其大小均在2.0 kb以上。改良SDS法與改良CTAB法提取的DNA量明顯高于試劑盒法,其電泳條帶也明顯比試劑盒法的亮,提取效果的穩定性好。但改良SDS法提取DNA的3次重復所得的效果并不相同,有些DNA條帶較亮,有些較暗,重復性較比改良CTAB法較差。
2.3 EST-SSR標記PCR擴增驗證
由圖2可知,改良SDS法與改良CTAB法提取的DNA模板均可擴增出比較清晰的EST-SSR標記條帶,且穩定性好;試劑盒法提取的DNA未成功擴增出清晰的EST-SSR標記條帶,這可能是含有較多的蛋白質、糖、色素等反應抑制物所導致的。綜上所述,改良SDS法與改良CTAB法提取的油梨果肉DNA純度高,雜質去除充分,可滿足EST-SSR等分子標記分析的需要。
3 討論與結論
3.1 討論
利用紫外分光光度法測定DNA純度時,如果1.9>OD260nm/OD280nm>1.8時,表明DNA樣品受雜質污染少,質量高;如果OD260nm/OD280nm>1.9時,表明DNA樣品存在少量RNA;如果OD260nm/OD280nm<1.8時,表明DNA樣品存在蛋白質等雜質[17]。由于油梨果肉含有大量脂肪酸,利用試劑盒法提取過程中脂肪酸等物質很容易形成粘稠的膠狀物質,難于溶解堵塞柱子造成所獲得的DNA濃度低,質量不高。
與寒溫帶水果相比,熱帶水果葉片、果實等各部位普遍含有更多的多糖、多酚、蛋白質等物質,其中果實內各種物質又比其它部位更多,這些物質對DNA的提取影響較大[11,18-19]。前人對熱帶水果DNA的提取,把聚乙烯吡咯烷酮和β-巰基乙醇加入到傳統的SDS法和CTAB法中,大幅提高了DNA的濃度與質量,取得了較好的結果[10-12,20-23]。本實驗中的改良SDS法和改良CTAB法,在前處理期加入了聚乙烯吡咯烷酮,其可有效防止多酚氧化,并可與酚類、單寧酸、脂肪酸等共沉淀用以去除雜質。在裂解液中加入了聚乙烯吡咯烷酮和β-巰基乙醇,可進一步抑制多酚氧化,并去除雜質。此外,延長異丙醇沉淀時間到過夜。前人研究結果表明,異丙醇沉淀時間越長,DNA濃度越高[24]。本實驗改良CTAB法與周海蘭等[12]的改良CTAB法略有區別,本實驗額外增加了2個步驟,即水浴冷卻后加入的提取液中加入了酚和延長異丙醇沉淀時間到過夜。這2個步驟前一個可更有效地抽提DNA,而第2個步驟可提高DNA濃度。雖然油梨果肉的雜質或抑制物質比葉片多,但本實驗改良CTAB法增加的2個步驟使得提取的DNA純度(1.81)與周海蘭等[12]的通過改良CTAB法提取的DNA純度(1.7~2.0)大致相同。
3.2 結論
本實驗采取改良SDS法、改良CTAB法和試劑盒法3種DNA提取方案從油梨果實中提取DNA,通過比較DNA的濃度和純度,進一步通過瓊脂糖凝膠電泳檢測DNA濃度和純度,確定了提取DNA質量較高的方法為改良SDS法和改良CTAB法。
參考文獻
[1] Kilaru A, Cao X, Dabbs P B, et al. Oil biosynthesis in a basal angiosperm:transcriptome analysis of Persea Americana mesocarp[J]. BMC Plant Biology, 2015, 15(1): 203.
[2] 李家興. 油梨在貴州亞熱帶地區的適應性及發展前景分析[J]. 貴州農業科學,2010,38(2):165-167.
[3] 何國祥. 廣西油梨科研生產的現狀及發展前景[J]. 廣西職業技術學院學報,2012,5(1):1-6.
[4] 張 良,張德生,劉康德. 海南省油梨產業發展的環境分析與對策[J]. 中國農業資源與區劃,2015,36(4):78-84.
[5] Papademetriou M K. Avocado production in Asia and the Pacific[M]. Bangkok: FAO Publisher, 2000.
[6] Schaffer B, Wolstenholme B N, Whiley A W. The avocado: botany, production and uses(2nd Edition) [M]. Croydon: CPI Group(UK)Ltd, 2012.
[7] Meyer M D, Terry L A. Development of a rapid method for the sequential extraction and subsequent quantification of fatty acids and sugars from avocado mesocarp tissue[J]. Journal of Agricultural Food Chemistry, 2008, 56(16): 7 439-7 445.
[8] Dreher M L, Davenport A J. Hass avocado composition and potential health effects[J]. Critical Reviews in Food Science and Nutrition, 2013, 53(7):738-750.
[9] 段婧婧,肖 琳,陳 軍. 用于轉基因檢測的水果果肉總DNA提取法[J]. 食品科學,2009,30(3):231-234.
[10] 林興娥,葛 宇,周兆禧,等. 一種高質量的紅毛丹基因組DNA提取方法[J]. 熱帶農業科學,2015,35(8):16-20.
[11] 齊玲倩,劉 秀,丁夢璇,等. 水果果肉中總DNA提取方法的比較研究[J]. 食品研究與開發,2016,37(5):55-58.
[12] 周海蘭,李紹鵬,李衛亮,等. 油梨基因組DNA提取、SSR-PCR反應體系優化及引物篩選[J]. 生物技術通報,2016,32(4):143-150.
[13] Djami-Tchatchou A T, Straker C J. The isolation of high quality RNA from the fruit of avocado (Pesea americana Mill.)[J]. South African Journal of Botany, 2012, 78(1): 44-46.
[14] 朱威龍,閆 碩,石冀哲,等. 棉花不同組織DNA的不同提取方法比較[J]. 安徽農業科學,2015,43(2):16-18.
[15] Borrone J W, Brown J S, Tondo C L, et al. An EST-SSR-based linkage map for Persea Americana Mill.(avocado)[J]. Tree Genetics & Genomes, 2009, 5(3):553-560.
[16] Ge Y, Ramchiary N, Wang T, et al. Development and linkage mapping of unigene-derived microsatellite markers in Brassica rapa L.[J]. Breeding Science, 2011, 61(2): 160-167.
[17] 貝盞臨,張 欣,杜 進,等.不同方法提取牛心樸子基因組DNA效果的比較研究[J]. 北方園藝,2011,18(1):135-137.
[18] 李 武,李艷君,楊瑞麗. 熱帶水果多酚提取物的抗氧化和抗增殖活性研究[J]. 現代食品科技,2013,29(10):2 383-2 387.
[19] Chen G L, Chen S G, Zhao Y Y, et al. Total phenolic contents of 33 fruits and their antioxidant capacitiesbefore and after in vitro digestion[J]. Industrial Crops and Products, 2014, 57(1): 150-157.
[20] 余志雄,歐高政,陳清西,等. 火龍果總DNA提取方法比較研究[J]. 中國農學通報,2010,26(4):300-303.
[21] 趙志常,陳業淵,高愛平,等. 改良CTAB法提取番石榴總DNA的初步研究[J]. 北方園藝,2013,9(1):123-125.
[22] 林方養,楊耀東,萬 婕,等. 棕櫚科植物DNA的提取及SRAP引物篩選[J]. 江西農業學報,2014,26(7):115-117.
[23] 羅 意,范海闊,黃綿佳,等. 3種椰子基因組DNA提取方法的比較[J]. 中國農學通報,2013,29(27):154-158.
[24] Ramos-Gómez S, Busto M D, Perez-Mateos M, et al. Development of a method to recovery and amplification DNA by real-time PCR from commercial vegetable oils[J]. Food Chemistry, 2014, 158(2): 374-383.

