MBR+蠕蟲反應器膜污染特征及微生物群落結構
劉 嘉, 左 薇, 張 軍, 李 慧, 李俐頻, 田 禹
(1.城市水資源與水環境國家重點實驗室(哈爾濱工業大學), 哈爾濱150090; 2.哈爾濱工業大學 市政環境工程學院,哈爾濱150090)
MBR+蠕蟲反應器膜污染特征及微生物群落結構
劉 嘉1,2, 左 薇2, 張 軍2, 李 慧2, 李俐頻2, 田 禹1,2
(1.城市水資源與水環境國家重點實驗室(哈爾濱工業大學), 哈爾濱150090; 2.哈爾濱工業大學 市政環境工程學院,哈爾濱150090)
為研究針對MBR+蠕蟲反應器工藝中蠕蟲捕食對膜污染的影響, 在常溫下分別平行運行MBR+蠕蟲反應器(R1)和作為對照系統的MBR+空白蠕蟲反應器(R2). 監測R1工藝中MBR(S-MBR)和R2工藝中MBR(C-MBR)的跨膜壓力(pTM), 檢測污泥混合液及泥餅層微生物代謝產物的變化. 利用變性梯度凝膠電泳(DGGE)技術分析S-MBR和C-MBR中微生物種類和分布. 結果表明: S-MBR的膜污染周期為90 d, C-MBR的膜污染周期為28~37 d, 蠕蟲捕食導致S-MBR中SMP和EPS的多糖和蛋白質減少. S-MBR膜絲表面是微生物菌群Alphaproteobacterium,Betaproteobacterium,Deltaproteobacterium和Geobacter, 而C-MBR膜絲表面是微生物菌群Azorhizobium,Rhodobacter,Gammaproteobacterium和Flavobacteria, 對MBR膜污染進程起主要作用.Caldilinea可能與S-MBR膜污染減輕有關. 蠕蟲捕食可改變微生物群落結構, 減緩S-MBR膜污染.
膜生物反應器; 膜污染; 胞外聚合物; 溶解性微生物代謝產物; 微生物群落結構
污水處理過程中產生的大量污泥容易導致二次環境污染, 進一步的污泥處理和處置增加了污水處理的成本, 限制了污水處理工藝的大規模應用. 目前, 作為一項有效的生態技術, 利用微型動物捕食污泥越來越受到關注[1-2]. 研究表明, 與傳統MBR相比, MBR+蠕蟲反應器組合工藝具有同步廢水處理和污泥減量的特點[3]. 然而,關于蠕蟲捕食對MBR膜污染影響的研究較少. 隨著MBR膜污染程度的加深, 影響膜污染的微生物代謝產物質量濃度增高, 如溶解性代謝產物(SMP)以及胞外聚合物(EPS)[4].微生物群落結構對膜污染也具有一定影響, 膜污染發展變化過程中, 微生物群落不斷演替并最終形成分泌大量黏性物質的優勢種群, 此時膜污染程度最深[5-8]. 為研究蠕蟲捕食對膜污染的影響, 通過實驗比較了MBR+蠕蟲反應器在接種和不接種蠕蟲的不同工藝條件下, 微生物代謝產物及微生物群落結構特征, 探索了微生物代謝產物、微生物種群結構與膜污染之間的關系. 進一步探討了微生物引起的膜污染機理.
1 實 驗
1.1 實驗材料
MBR接種污泥取自哈爾濱太平污水處理廠二沉池, 污泥經反應器馴化后, MLSS(混合液懸浮固體)質量濃度控制在9 000 ~10 000 mg·L-1. 蠕蟲反應器接種污泥為MBR產生的剩余活性污泥, 運行過程中MLSS質量濃度控制在3 000~4 000 mg·L-1. 進水為人工模擬生活污水, COD為(337.9±17.2)mg·L-1, NH3-N為(28.2±1.4)mg·L-1, TN(總氮)為(30.5±1.2) mg·L-1, pH為7.5±0.2.
1.2 反應器裝置
MBR+蠕蟲反應器(R1)和作為對照系統的MBR+空白蠕蟲反應器(R2)如圖1所示, R1工藝的蠕蟲反應器接種蠕蟲, R2工藝的空白蠕蟲反應器不接種蠕蟲. 反應器壁的材質為透明有機玻璃, 便于觀察反應狀態. S-MBR和C-MBR有效容積均為40 L, 都安裝一個中空纖維濾膜組件, 有效過濾面積為1 m2, 膜孔徑為0.2 μm. 蠕蟲反應器及空白蠕蟲反應器有效容積均為39 L, 兩個反應器內都均勻分布多層數個填充了聚乙烯多孔性填料的穿孔板筐, 作為蠕蟲的載體. 實驗所用蠕蟲主要為顫蚓類, 經馴化后接種到蠕蟲反應器中. 蠕蟲反應器中穿孔板的面積為0.36 m2, 蠕蟲總質量為0.096 kg, 反應器運行過程中蠕蟲密度控制在濕重0.27 kg/m2左右.
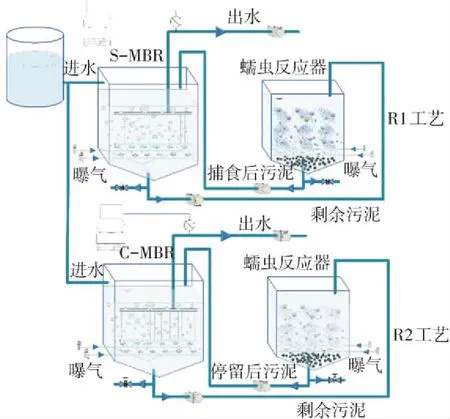
圖1 MBR+蠕蟲反應器(R1)和MBR+空白蠕蟲反應器(R2)
Fig.1 MBR+worm reactor (R1) and MBR+worm reactor without worms (R2)
1.3 反應器運行

表1 工藝運行條件Tab.1 Parameters of reactors
1.4 檢測分析方法
1.4.1 常規檢測方法
采用國家標準方法, 每隔3 d測定S-MBR和C-MBR進水和出水的COD、NH3-N、NO3-N、NO2-N. DO、pH和溫度,分別利用JCB-607便攜式溶解氧儀、PHSJ-3F實驗室pH計和溫度計測定. MBR運行過程中,利用膜組件上的真空壓力表測定pTM, 表征膜污染程度。
1.4.2 樣品處理
泥餅層分離方法: 選取膜污染周期結束時S-MBR和C-MBR反應器上的膜絲1~2根, 截取中間位置的等長段, 用等量的磷酸鹽緩沖溶液將膜絲上的泥餅層沖洗下來, 分別置于離心管中, 用于EPS和DNA提取.
膜絲表面凝膠層分離方法: 將沖洗后的膜絲放入離心管中, 加入PBS緩沖溶液進行超聲破碎. 微生物樣品經過超聲震動后從膜絲表面脫離到離心管的緩沖溶液中, 按照DNA試劑盒提取方法進行樣品中微生物DNA的提取.
1.4.3 SMP、EPS提取方法
樣品置于離心管中, 5 000 r/min離心5 min, 取上清液經0.45 μm濾膜過濾, 所得濾液即為SMP; 用蒸餾水將離心管中剩余污泥重新定容至原體積, 混合均勻后水浴(80 ℃, 30 min), 離心(5 000 r/min, 5 min), 上清液經0.45 μm濾膜過濾后, 濾液用于EPS分析[9].
劉康寧 男.1992年3月出生,河北石家莊人.天津大學智能與計算學部碩士研究生,主要研究方向為無線(長距離)mesh網絡和無線ad hoc網絡.
1.4.4 微生物群落結構分析
利用PCR-DGGE分子生物學方法[10]分析S-MBR和C-MBR中微生物群落結構特征. PCR引物為GC夾的338F和518R[11].
2 結果與討論
2.1 工藝運行效果比較
S-MBR和C-MBR連續運行90 d, 出水COD平均分別為(26.5±5.4)和(25.9±7.3)mg·L-1, COD去除效率分別為(92.4±1.4)%和(92.6±2.0)%; NH3-N平均質量濃度分別為(2.1±0.5)和(2.2±0.8)mg·L-1, NH3-N去除效率分別為(92.6±2.1)%和(92.1±2.8)%. 實驗結果表明盡管蠕蟲捕食釋放大量的營養物質(COD和NH3-N)[3], S-MBR仍保持較高的COD和NH3-N去除效率(見表2).
表2 S-MBR和C-MBR廢水處理效果
Tab.2 Performance of the S-MBR and the C-MBR treating the synthetic wastewater

檢測指標進水平均值/(mg·L-1)S-MBRC-MBR出水平均值/(mg·L-1)平均去除效率/%出水平均值/(mg·L-1)平均去除效率/%COD347.2±17.426.5±5.492.4±1.425.9±7.392.6±2.0NH3-N28.5±1.32.1±0.592.6±2.12.2±0.892.1±2.8TN29.5±3.223.5±1.420.3±3.123.2±2.121.4±2.3
2.2 膜污染分析
S-MBR和C-MBR連續運行90 d, 分別監測S-MBR和C-MBR的pTM變化, 如圖2所示. 當pTM達30 kPa時,一個膜污染周期結束.C-MBR的膜污染周期較短為28~37 d, 平均pTM增長速率為1.0 kPa/d. S-MBR的膜污染周期較長為90 d, 平均pTM增長速率為0.3 kPa/d, 較C-MBR降低了70%. 由圖2可看出, 蠕蟲捕食可有效地減緩S-MBR的膜污染速率.

圖2 S-MBR和C-MBR反應器pTM隨運行時間變化Fig.2 Changes ofpTMin the S-MBR and C-MBR
工藝運行至90 d時, S-MBR和C-MBR反應器pTM均達30 kPa, 分別對各MBR反應器污泥混合液、生物膜表面泥餅層中微生物代謝產物的成分及其質量濃度進行分析, 結果如表3所示.可以看出, S-MBR污泥混合液中SMP的糖類和蛋白質類物質質量濃度分別為8.1和7.9 mg·L-1, 較C-MBR低14.7%和18.6%; EPS的糖類和蛋白類物質較C-MBR低2.5%和23.4%. S-MBR中膜表面泥餅層SMP的糖類和蛋白質類物質質量濃度分別為13.5和11.7 mg·L-1, 較C-MBR低24.2%和22.5%; EPS的糖類和蛋白質類物質較C-MBR低3.1%和13.4%. 實驗結果表明, 與污泥暫時停留在空白蠕蟲反應器中相比, 蠕蟲捕食污泥可有效地減少S-MBR中污泥混合液及膜表面泥餅層上SMP和EPS的糖類和蛋白質類物質質量濃度.
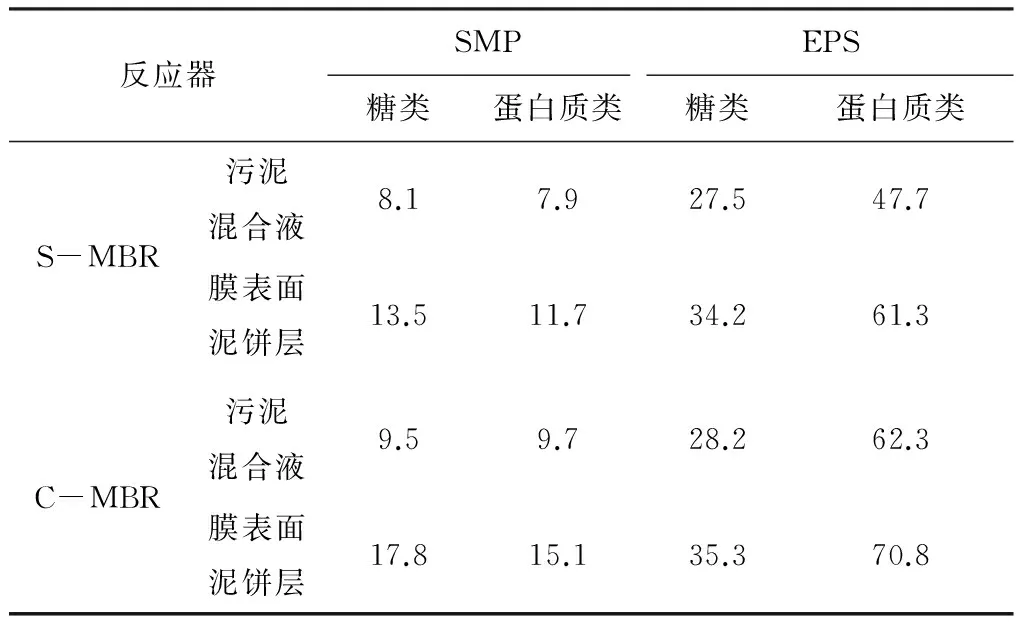
表3 S-MBR和C-MBR反應器微生物代謝產物Tab.3 Microbial metabolites of the S-MBR and the C-MBR mg·L-1
有研究表明, SMP和EPS在生物污染物和膜表面泥餅層形成過程中起到重要作用[12-13], 其中蛋白質和糖類物質可導致膜污染[14]. 在MBRs膜污染過程中, 膜絲上的微生物代謝產物及微生物菌群的黏附使膜孔阻塞, 膜通量降低. 由此推斷, S-MBR中污泥混合液及膜表面泥餅層中較低質量濃度的糖類和蛋白質類物質與S-MBR的膜污染減輕有關; 可以通過生態技術手段改變微生物群落結構, 降低產生胞外聚合物的微生物數量, 從而達到延緩膜污染的目的.
2.3 微生物群落與膜污染關系
反應器的核心組成主要是活性污泥中的微生物種群[15], 微生物群落結構的特征決定了反應器的功能和運行效果。反應器運行結束時, 即第90天, S-MBR和C-MBR的膜孔被完全堵塞,膜污染周期終止. 此時, 分別在S-MBR和C-MBR反應器內對污泥混合液、膜上泥餅層以及膜絲表面凝膠層取樣, 分析其微生物群落結構, 結果見圖3.
根據圖3, 對DGGE圖譜中主要條帶進行測序分析, 結果如表4所示. 由圖3和表4可以看出, 無論是S-MBR還是C-MBR, 不同的生態環境微生物種群結構也不相同. 每種微生物種群都有其自身所適應的生態位, 并且隨著周圍環境的變化而進行演替. 通過對比兩個MBRs相同生態位上的微生物種群發現, S-MBR的微生物群落結構與C-MBR中不同, 這可能與蠕蟲捕食有關.
另外, S-MBR和C-MBR中污泥混合液的微生物種群結構與其泥餅層的相似, 而與膜絲上的微生物種群基本不同. MBRs反應器運行過程中, 由于曝氣強度較高造成水力和氣流在膜的泥餅層上不斷攪動, 導致膜上泥餅層與污泥混合液不斷融合, 形成相似的微生物群落結構; 而膜絲表面上的微生物由于受污泥混合液的沖擊較少, 其種群結構與污泥混合液及泥餅層的差別較大.

圖3 基于16S rDNA PCR產物的DGGE圖譜Fig.3 DGGE profile based on the 16S rDNA PCR products
膜污染周期結束, 膜壓30 kPa時, S-MBR膜絲表面的主要微生物為Proteobacteria(變形菌門)的Alphaproteobacterium、Betaproteobacterium、Deltaproteobacterium(變形菌屬)、Geobacter(地桿菌屬), Chloroflexi(綠彎菌門)的Caldilinea(暖繩菌屬)和Bacteria; C-MBR膜絲表面的主要微生物為Proteobacteria(變形菌門)的Azorhizobium(生絲微菌屬)、Rhodobacter(紅細菌屬)、Gammaproteobacterium(變形菌屬), Bacteroidetes(擬桿菌門)的Flavobacteria(黃桿菌屬), Firmicutes(厚壁菌門)的Thermoanaerobacteriales(熱厭氧桿菌目)、Uncultured Firmicutes bacterium和Bacteria. 其中, Proteobacteria(變形菌門)和Bacteroidetes(擬桿菌門)易在膜組件形成優勢菌群, 同時使膜組件容易發生污染[8]. 因此推斷, S-MBR和C-MBR膜絲表面上檢測到的Proteobacteria(變形菌門)和Bacteroidetes(擬桿菌門)對反應器的膜污染具有重要作用.
另外, Proteobacteria(變形菌門)的Paracoccus(副球菌屬)為具有反硝化作用的菌屬, 表面可產生黏液層, 利于細胞間的黏連, 對膜污染的發展貢獻較大[4]. 蠕蟲反應器中存在同步硝化反硝化過程[16], 由于捕食后污泥的回流, S-MBR的污泥混合液、 膜上泥餅層和膜絲表面上都發現了Paracoccus(副球菌屬), 負面影響了反應器的膜污染進程. 與C-MBR相比, Chloroflexi(綠彎菌門)的Caldilinea(暖繩菌屬)存在S-MBR的污泥混合液、 膜上泥餅層和膜絲表面上不同的生態環境中. Chloroflexi(綠彎菌門)可降解SMP中的碳水化合物及細胞物質, 減少其膜污染傾向, 對MBR處理城市廢水具有重要的生態意義[17]. 因此推斷, S-MBR中存在Chloroflexi(綠彎菌門)的Caldilinea(暖繩菌屬)可能與反應器膜污染減輕有關.

表4 16S rDNA序列比對結果Tab.4 Results of the 16S rDNA sequences
3 結 論
1)MBR+蠕蟲反應器工藝能顯著降低膜污染速率, 延長膜污染周期. S-MBR膜污染周期為90 d, 膜污染速率為0.3 kPa/d; C-MBR為28~37 d, 1.0 kPa/d.
2)蠕蟲捕食污泥可有效地減少MBR污泥混合液及膜表面泥餅層中SMP和EPS的糖類和蛋白質類物質質量濃度.
3)S-MBR膜絲表面的主要微生物為Proteobacteria(變形菌門)的Alphaproteobacterium、Betaproteobacterium、Deltaproteobacterium(變形菌屬)、Geobacter(地桿菌屬); 而C-MBR膜絲表面主要是Proteobacteria(變形菌門)的Azorhizobium(生絲微菌屬)、Rhodobacter(紅細菌屬)、Gammaproteobacterium(變形菌屬), Bacteroidetes(擬桿菌門)的Flavobacteria(黃桿菌屬). 二者的微生物群落結構明顯不同.
4) S-MBR污泥混合液、 泥餅層和膜絲上均檢測到Chloroflexi(綠彎菌門)的Caldilinea(暖繩菌屬), 可能與反應器的膜污染減輕有關.
[1] HENDRICKX T L G, ELISSEN H J H, TEMMINK H, et al. Operation of an aquatic worm reactor suitable for sludge reduction at large scale [J]. Water Research, 2011, 45(16): 4923-4929. DOI:10.1016/j.watres.2011.06.031.
[2] TAMIS J, SCHOUWENBURG VAN G, KLEEREBEZEM R, et al. A full scale worm reactor for efficient sludge reduction by predation in a wastewater treatment plant[J]. Water Research, 2011, 45(18): 5916-5924. DOI:10.1016/j.watres.2011.08.046.
[3] TIAN Y, LU Y B, LI Z P. Performance analysis of a combined system of membrane bioreactor and worm reactor: Wastewater treatment, sludge reduction and membrane fouling [J]. Bioresource Technology, 2012, 121: 176-182. DOI:10.1016/j.biortech.2012.06.071.
[4] 高大文, 辛曉東. MBR 膜污染過程中微生物群落結構與代謝產物分析[J]. 哈爾濱工業大學學報, 2014, 46(2): 26-32. GAO Dawen, XIN Xiaodong. Analysis of microbial community structure and metabolites during the MBR membrane fouling process[J].Journal of Harbin Institute of Technology, 2014, 46(2): 26-32.
[5] 張斌, 孫寶盛, 劉慧娜, 等. 處理不同廢水 MBR 系統中微生物群落結構的比較[J]. 環境科學, 2008, 29(10): 2944-2949. ZHANG Bin,SUN Baosheng,LIU Huina,et al. Comparison of microbial community structure in MBRs treating different wastewater[J].Environmental Science, 2008, 29(10): 2944-2949.
[6] LIM S Y, KIM S, YEON K M, et al. Correlation between microbial community structure and biofouling in a laboratory scale membrane bioreactor with synthetic wastewater[J]. Desalination, 2012, 287: 209-215. DOI:10.1016/j.desal.2011.09.030.
[7] GAO W J, LIN H J, LEUNG K T, et al. Structure of cake layer in a submerged anaerobic membrane bioreactor[J]. Journal of Membrane Science, 2011, 374(1): 110-120. DOI:10.1016/j.memsci.2011.03.019.
[8] HUANG L N, WEVER H D, DIELS L. Diverse and distinct bacterial communities induced biofilm fouling in membrane bioreactors operated under different conditions[J]. Environmental Science & Technology, 2008, 42(22): 8360-8366. DOI: 10.1021/es801283q.
[9] 盧耀斌. MBR+ 蠕蟲床污泥減量效能及膜污染控制機制 [D]. 哈爾濱:哈爾濱工業大學,2014. LU Yaobin. Research on a combined system of membrane bioreactor and worm reactor for sludge reduction and membrane fouling mitigation[D].Harbin: Harbin Institute of Technology,2014.
[10]高瓊. 聚合酶鏈式反應 (PCR) 技術在環境監測中的應用[J]. 安徽農業科學, 2014, 42(36): 12825-12828. GAO Qiong.Application of polymerase chain reaction(PCR) technology in environmental monitoring[J].Journal of Anhui Agri Sci, 2014, 42(36): 12825-12828.
[11]LAPARA T M, NAKATSU C H, PANTEA L M, et al. Stability of the bacterial communities supported by a seven-stage biological process treating pharmaceutical wastewater as revealed by PCR-DGGE[J]. Water Research, 2002, 36(3): 638-646. DOI:10.1016/S0043-1354(01)00277-9.
[12]FLEMMING H C, SCHAULE G, GRIEBE T, et al. Biofouling—the achilles heel of membrane processes[J]. Desalination, 1997, 113(2): 215-225. DOI:10.1016/S0011-9164(97)00132-X.
[13]RAMESH A, LEE D J, LAI J Y. Membrane biofouling by extracellular polymeric substances or soluble microbial products from membrane bioreactor sludge[J]. Applied Microbiology and Biotechnology, 2007, 74(3): 699-707. DOI: 10.1007/s00253-006-0706-x.
[14]GüELL C, DAVIS R H. Membrane fouling during microfiltration of protein mixtures[J]. Journal of Membrane Science, 1996, 119(2): 269-284. DOI:10.1016/0376-7388(96)80001-J.
[15]WAN C Y, WEVER H D, DIELS L, et al. Biodiversity and population dynamics of microorganisms in a full-scale membrane bioreactor for municipal wastewater treatment[J]. Water Research, 2011, 45(3): 1129-1138. DOI:10.1016/j.watres.2010.11.008.
[16]TIAN Y, LU Y B. Simultaneous nitrification and denitrification process in a new tubificidae-reactor for minimizing nutrient release during sludge reduction[J]. Water Research, 2010, 44(20): 6031-6040. DOI:10.1016/j.watres.2010.07.069.
[17]MIURA Y, WATANABE Y, OKABE S. Significance of chloroflexi in performance of submerged membrane bioreactors (MBR) treating municipal wastewater[J]. Environmental Science & Technology,2007,41(22): 7787-7794. DOI:10.1021/es071263x.
Analysis of membrane fouling and microbial community structure in an MBR+worm reactor
LIU Jia1,2,ZUO Wei2,ZHANG Jun2, LI Hui2, LI Lipin2, TIAN Yu1,2
(1.State Key Laboratory of Urban Water Resource and Environment (Harbin Institute of Technology), Harbin 150090, China;2.School of Municipal and Environmental Engineering, Harbin Institute of Technology, Harbin 150090, China)
To study the effects of worm predation on membrane fouling in an MBR+worm reactor, an MBR+worm reactor with worm (R1) and an MBR+worm reactor without worm (R2) were operated in parallel. Variation of transmembrane pressure (pTM) and microbial metabolites were studied in the MBR (S-MBR) of the R1 and the MBR (C-MBR) of the R2. Denatured gradient gel electrophoresis (DGGE) was used for analyzing the composition and distribution of microbial community in the S-MBR and the C-MBR. The results showed that the membrane fouling cycles of the S-MBR and the C-MBR were 90 d and 28-37 d, respectively. Worm predation decreased the polysaccharide and proteins in the soluble microbial products (SMP) and extracellular polymeric substances (EPS) of the S-MBR.Alphaproteobacterium,Betaproteobacterium,Deltaproteobacterium,Geobacteron the membrane wire surface of the S-MBR andAzorhizobium,Rhodobacter,Gammaproteobacterium,Flavobacteriaon the membrane wire surface of the C-MBR played an important role in the membrane fouling.Caldilineawas suggested to be related to the membrane fouling alleviation of the S-MBR. Worm predation changed the microbial community structure of the S-MBR, resulting in membrane fouling alleviation.
MBR; membrane fouling; EPS; SMP; microbial community structure
10.11918/j.issn.0367-6234.2017.02.006
2016-06-19
水體污染控制與治理科技重大專項(2013ZX07201007);黑龍江省杰出青年科學基金(JC201303);哈爾濱工業大學城市水資源與水環境國家重點實驗室項目(2014DX03);哈爾濱工業大學城市水資源與水環境國家重點實驗室開放基金(QA201207)
劉 嘉 (1983—),女,博士研究生; 田 禹 (1968—),女,博士生導師,長江學者特聘教授
左 薇,zuoweistar@163.com; 田 禹,hit_tianyu@163.com
X172
A
0367-6234(2017)02-0032-05

