基于SRAP分子標記的黃顙魚屬遺傳多樣性分析
龔瑞玥++方春林++萬正義++舒漢鼎++胡成鈺++毛慧玲
doi:10.15889/j.issn.1002-1302.2016.10.021
摘要:選用64對SRAP引物對黃顙魚屬進行遺傳多樣性研究。結果顯示,有46對引物能成功擴增,其中19對條帶清晰、顯示較高多態性。檢測其PCR產物共獲得179條,每對引物擴增條帶5~15條不等,擴增片段介于90~510 bp 之間,物種間遺傳相似系數范圍為0.500~1.000,黃顙魚屬各物種間具有較大的遺傳差異性。SRAP聚類結果顯示,瓦氏黃顙魚與其他物種差異最大,普通黃顙魚、長須黃顙魚和中間黃顙魚親緣關系較近。遺傳多樣性分析結果顯示,5種黃顙魚遺傳多樣性處于較高水平,有助于后續深入對黃顙魚屬各物種親緣關系和系統演化進行分析。
關鍵詞:黃顙魚屬;SRAP;遺傳多樣性;遺傳相似系數
中圖分類號: S961.2文獻標志碼: A文章編號:1002-1302(2016)10-0092-03
收稿日期:2015-08-15
基金項目:江西省科技支撐資助項目(編號:20112BBF60011);江西省教育廳科技資助項目 (編號:GJJ14152)。
作者簡介:龔瑞玥(1990—),女,江西南昌人,碩士研究生,主要從事生物化學與分子生物學研究。 E-mail:enidpace713@gmail.com。
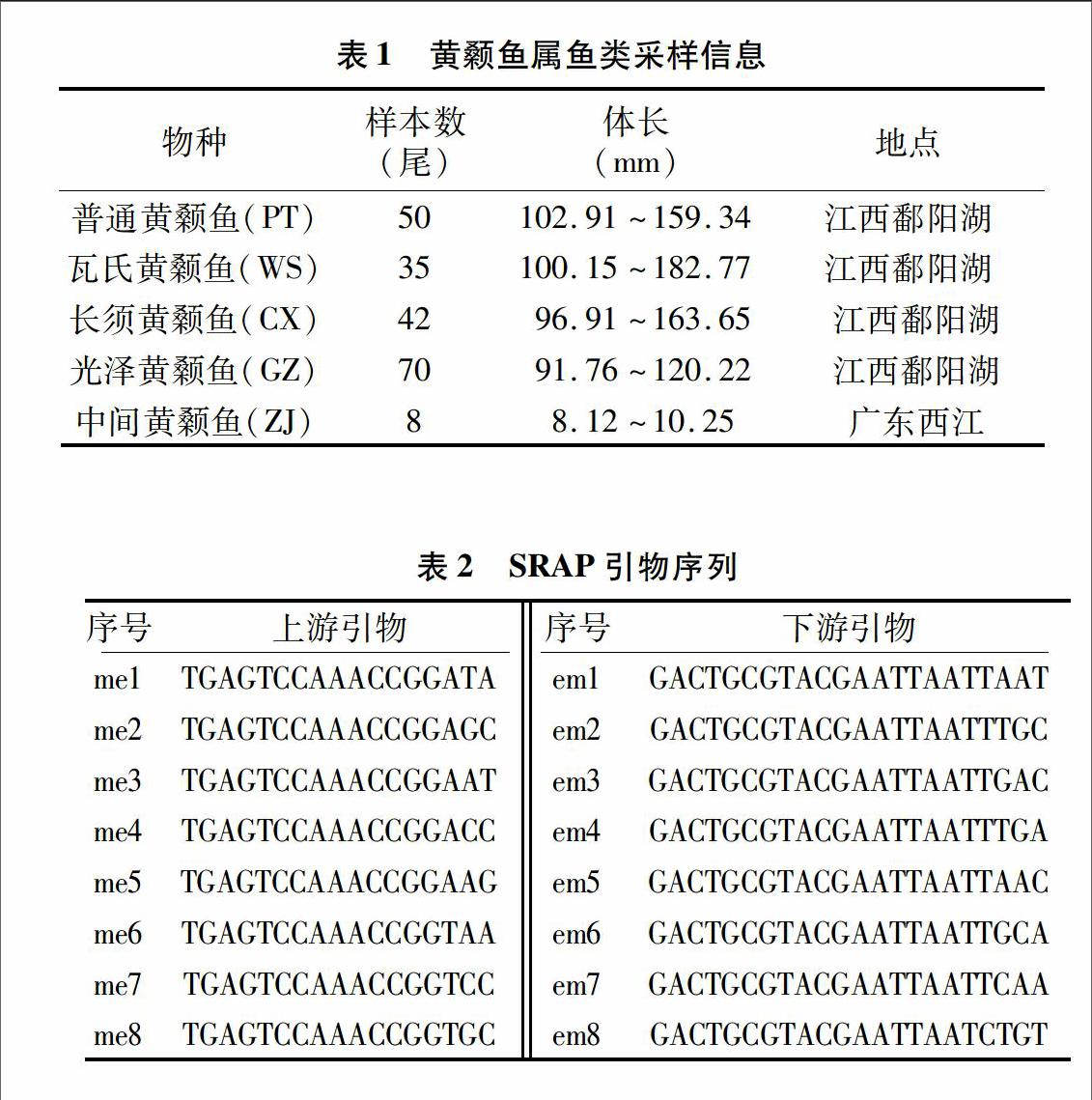
通信作者:毛慧玲,碩士,教授,主要從事魚類遺傳與育種研究。E-mail:huilinm6@163.com。黃顙魚俗稱黃丫頭,是備受我國老百姓喜愛的主要經濟魚類之一。據中國動物志中報道,黃顙魚屬共有普通黃顙魚(P.eupogon)、長須黃顙魚(P.fulvidraco)、瓦氏黃顙魚(P.vachelli)、光澤黃顙魚(P.nitidus)和中間黃顙魚(P.intermedius)等5種,中間黃顙魚僅分布于珠江水系及海南島,其他4種均廣泛分布于長江水系[1-2]。隨著市場對黃顙魚需求的增大,其養殖規模不斷擴大,隨之出現魚種混雜,品種退化現象,主要表現在生長速度變慢、抗病力及抗逆力降低等方面。從良種選育、養殖和種質資源保護角度考慮,亟須提供一種有效的方法來鑒別這些物種,并研究其遺傳多樣性。
序列相關擴增多態性(SRAP,sequence-related amplified polymorphism)分子標記是由美國加州大學蔬菜系Li等于2001年提出[3],該標記具有簡單、高效、高共顯、高重復、易測序等優點[4-5],廣泛運用于物種遺傳多樣性、品種鑒定和親緣關系的研究當中。本研究從分子水平角度,應用SRAP分子標記技術來鑒別黃顙魚屬5個種,并探討它們的遺傳多樣性及其親緣關系,為黃顙魚屬種質資源保護和開發利用提供理論資料[6-7],對黃顙魚漁業資源的可持續發展具有十分重要的科學指導意義。
1材料與方法
1.1樣品采集
2011年6月至2012年9月,從江西鄱陽湖和廣東西江野外采得魚類(表1),并應用傳統形態學方法將它們逐一進行分類。
1.2基因組DNA
每個物種隨機選取4尾,共20尾樣品。取背部肌肉,采用苯酚-氯仿法提取基因組DNA[8]。
1.3SRAP引物來源
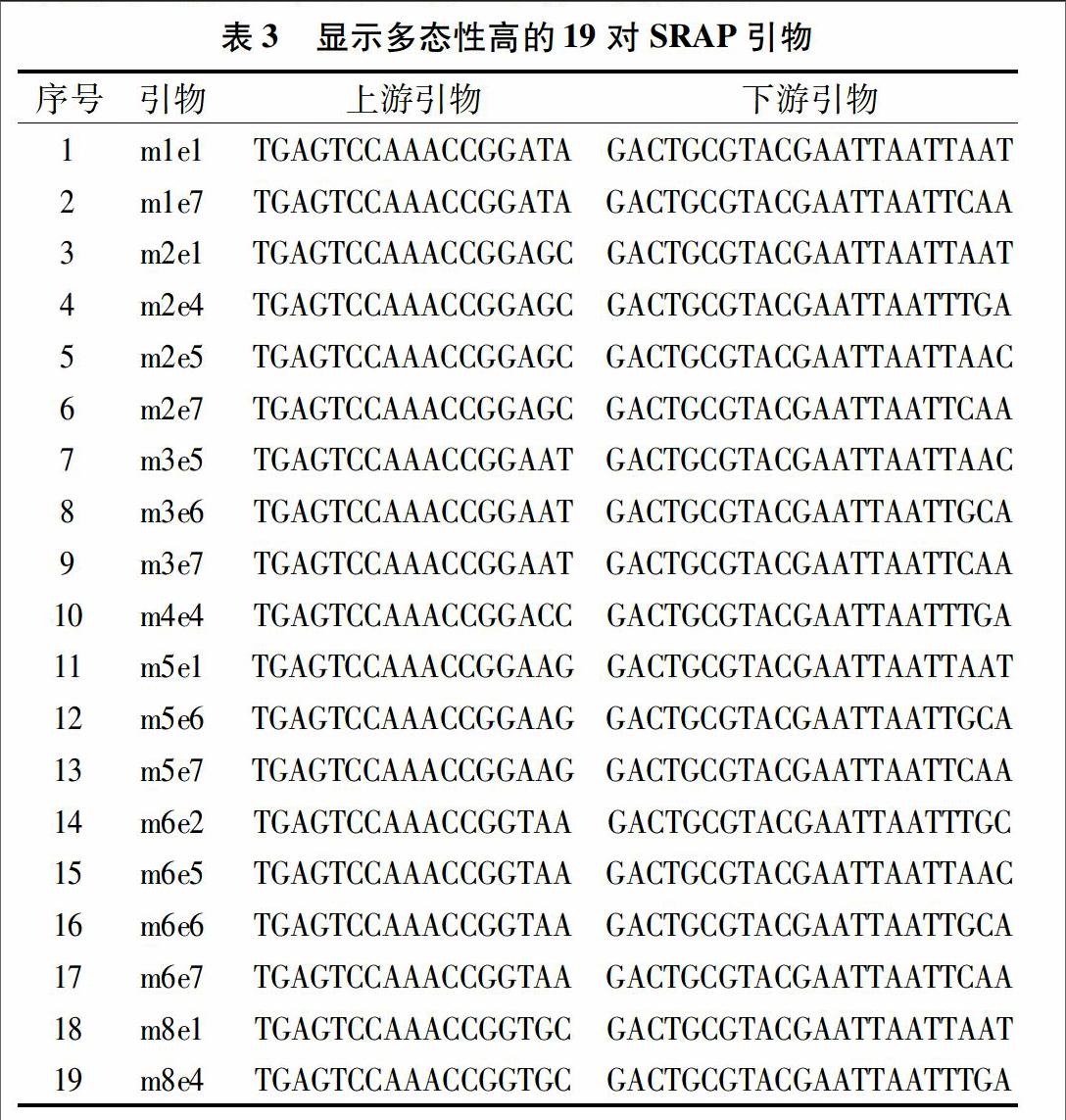
由表2可知,SRAP引物序列,將上游引物與下游引物交叉組合得到64對SRAP引物,按照簡寫上下游序號原則進行命名,如上游2號引物me與下游3號引物em組合而成的SRAP引物命為m2e3。
1.4PCR擴增和電泳檢測
20 μL SRAP的PCR擴增體系含:100 ng/μL模板DNA 1.0 μL,50 μmol/L上下游引物各0.06 μL,10×PCR buffer (Mg2+) 2.0 μL,200 μmol/L dNTPs 1.6 μL,5.0 U/μL Taq DNA聚合酶0.2 μL,ddH2O 15.08 μL;PCR反應條件為:94 ℃ 預變性5 min;94 ℃變性1 min,35 ℃退火1 min,72 ℃延伸2 min,5個循環;94 ℃變性1 min,53 ℃退火1 min,72 ℃延伸2 min,35個循環;72 ℃延伸7 min。采用6%的聚丙烯酰胺凝膠電泳檢測PCR產物多態性。
1.5數據統計分析
對著SRAP的PCR擴增圖譜,自下而上按遷移率不同排列計數,無帶則賦值為“0”,有帶則賦值為“1”,建立不同SRAP引物“0-1”矩陣表。應用軟件包括POPGENE32、POWERMARKER V3.25等不同軟件處理、分析“0-1”矩陣表數據,獲得等位基因數(Na)、有效等位基因數(Ne)、等位基因有效率(A)、多態信息含量(PIC)等遺傳多樣性指標。分別應用NTSYS-pc2.0軟件、UPGMA程序計算遺傳相似系數[9]、聚類分析[10]。
2結果與分析
2.1SRAP擴增結果
由64對SRAP引物擴增結果獲悉,46對引物能成功擴增,占引物總數的71.9%。檢測引物擴增的PCR產物,共獲得179條,每對引物擴增條帶5~15條不等,擴增片段介于 90~510 bp之間。由此可確定46對引物是黃顙魚屬SRAP標記的有效引物。
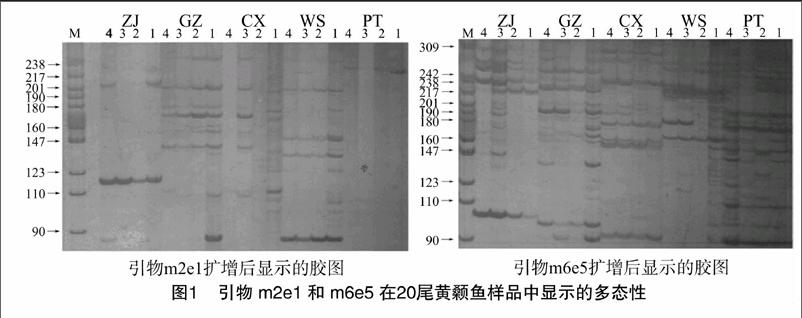
選取其中條帶清晰、多態性較高的19對引物(表3)用于分析。DNA圖譜(圖1)清楚地顯示了引物m2e1、m6e5在20尾黃顙魚樣品特異性擴增產物所具有的多態性,相同引物下擴增出的同種不同個體均能顯示明顯的相同特征性條帶,體現出種內差異較小;而同屬不同種間條帶差異明顯,多態性高,清晰地體現出黃顙魚屬5個種之間的差異。
2.2遺傳多樣性指標分析
19對SRAP引物對黃顙魚屬5個物種20尾樣品進行擴增,檢測到118個等位基因,每個位點可擴增出的等位基因數為4~8個不等,平均數為6.2個,大小在90~510 bp之間,計算出有效等位基因數。由表4可知,有效等位基因數等位基因有效率、多態信息含量等平均數值分別為5.348 6、0.834 2、0.766 7。由表5可見,除中間黃顙魚外的4個物種Shannon信息指數均較高,特別是長須黃顙魚高達0.683 9。上述數據都是反映遺傳多樣性的指標,其數值越大,說明基因豐富度越高。
2.3黃顙魚屬各種聚類分析
由圖2可知,根據PCR產物的多態性和聚類分析結果,5種黃顙魚間遺傳相似系數范圍為0.500~1.000,這說明黃顙魚屬各物種間具有較大的遺傳差異性。其中,不同物種間遺傳相似系數低,同一物種間遺傳相似系數高,說明親緣關系近。根據材料間的遺傳相似系數對黃顙魚屬進行聚類分析,在遺傳相似系數為0.50水平時,可將瓦氏黃顙魚與其他物種區分開;在遺傳相似系數為0.56水平時,可明確地將光澤黃顙魚與其他3個物種區分開來;在遺傳相似系數為 0.64 的水平下,可將普通黃顙魚與長須黃顙魚、中間黃顙魚分開;隨后在0.72水平時,長須黃顙魚和光澤黃顙魚分為2類。綜合來說,供試的黃顙魚屬5個物種相似系數不高,遺傳距離相對較遠,分屬于不同種。19對SRAP引物所構成的各物種的特異性譜系,能夠為黃顙魚屬各物種提供更準確的分子水平上的鑒定。
3討論
SRAP作為一種新型的分子標記技術,不需要像RAPD、SSR標記那樣花費大量人力和時間進行引物設計開發,重復試驗結果穩定,PCR擴增所需求的DNA質量要求低,最為重要的一點是SRAP標記穩定且多態性可與AFLP標記相媲美[11-12]。本試驗利用了19對SRAP引物對黃顙魚屬進行遺傳多樣性研究,結果顯示5種黃顙魚遺傳多樣性處于較高水平。相對于其他幾個物種而言,中間黃顙魚的多樣性指數偏低,原因可能是廣州西江采樣點為經濟高度發達區域,環境污染、過度捕撈等人為干擾比較嚴重,而其余4個物種都采于生態環境較好的鄱陽湖,物種受到破壞程度相對較小[13]。
對196個擴增條帶構建的各物種特異性譜系,雖然能夠用于對黃顙魚屬幼苗和幼魚的鑒別,但是由于SRAP引物擴增出的條帶較多,需通過多對引物擴增檢測才能對黃顙魚屬進行區分,這將浪費大量人力、物力,如果將SRAP轉化為SCAR標記能夠克服這一缺點[14-17]。丁煒東等通過SRAP方法對3種草魚基因組進行分析,設計了3對SCAR引物,其中SCAR1能夠區分人工養殖和野生草魚種群[18]。由此可見,將SRAP標記轉化為SCAR標記后,其特異性和穩定性均大幅度提高,可以更方便快捷地應用于相似物種的鑒定。
鲿科魚類的分類系統一直比較混亂,在屬的劃分上還存在許多爭議,黃顙魚屬亦如此。肖調義等對洞庭湖4種黃顙魚的生態學特征和遺傳多樣性的RAPD分析結果表明,長須黃顙魚與瓦氏黃顙魚具有較近的親緣關系,而光澤黃顙魚與普通黃顙魚關系較近[19],與本研究結果相反。SRAP聚類結
果表明普通黃顙魚和長須黃顙魚關系相近,光澤黃顙魚與瓦氏黃顙魚關系相近,這與丁言偉對黃顙魚屬分子系統發育研究結果[20]相近;與趙哲霞等的SSR分析結果[9]略有不同,主要原因可能在于普通黃顙魚和瓦氏黃顙魚之間的位置調換了,各物種種內遺傳差異與SSR分析相比較小。其可能的原因在于SSR引物設計是根據普通黃顙魚相關序列設計的,擴增出來的多態性條帶相對于其他物種較多;而SRAP分子標記是通過獨特的引物設計對ORFs進行擴增。
基因組DNA不同區域進化速率相差很大,而分子標記是根據特定序列設計引物進行擴增檢測來解決所研究物種的親緣關系和系統發生,結果肯定存在差別。至于選取那種方法得出的結論最準確、具有更高的可信性都還沒有定論,需要之后進一步研究比較。
參考文獻:
[1]褚新洛,鄭葆珊,戴定遠,等. 中國動物志:硬骨魚綱鲇形目[M]. 北京:科學出版社,1999:1-43.
[2]劉世平. 鄱陽湖黃顙魚生物學研究[J]. 動物學雜志,1997,32(4):10-15.
[3]Li G,Quiros C F. Genetic analysis,expression and molecular characterization of BoGSL-ELONG,a major gene involved in the aliphatic glucosinolate pathway of brassica species[J]. Genetics,2002,162(4):1937-1943.
[4]Budak H,Shearman R C,Parmaksiz I,et al. Comparative analysis of seeded and vegetative biotype buffalograsses based on phylogenetic relationship using ISSRs,SSRs,RAPDs,and SRAPs[J]. Theoretical and Applied Genetics,2004,109(2):280-288.
[5]Budak H,Shearman R C,Parmaksiz I,et al. Molecular characterization of buffalograss germplasm using sequence-ralated amplified polymorphism markers[J]. Theoretical and Applied Genetics,2004,108(2):328-334.
[6]Sekino M,Hara M. Inheritance characteristics of microsatellite DNA loci in experimental families of Japanese flounder Paralichthys olivaceus[J]. Marine Biotechnology,2001,3(4):310-315.
[7]Nakabo T. The third international symposium on the marine sciences
of the yellow sea[C]. Qingdao:Ocean Press,1994:81.
[8]趙哲霞,蔣珊,王濱花,等. 黃顙魚屬SSR分子鑒定及其遺傳多樣性[J]. 南昌大學學報:理科版,2014,38(5):498-501.
[9]Yu H T,Lee Y J,Huang S W,et al. Genetic analysis of the populations of Japanese anchovy (Engraulidae:Engraulis japonicus) using microsatellite DNA[J]. Marine Biotechnology,2002,4(5):471-479.
[10]何琳,王群. 基于PCR的SSR標記分離方法綜述基因組學與應用生物學[J]. 遺傳組學與應用生物學,2010,29(4):775-782.
[11]Ferriol M,Belén Picó M,Nuez F. Genetic diversity of some accessions of Cucurbita maxima from Spain using RAPD and SBAP markers[J]. Genetic Resources and Crop Evolution,2003,50(3):227-238.
[12]Lin X Y,Kaul S,Rounsley S,et al. Sequence and analysis of chromosome 2 of the plant Arabidopsis thaliana[J]. Nature,1999,402:761-768.
[13]冉瑋,張桂蓉,王衛民,等. 利用SRAP標記分析3個團頭魴群體的遺傳多樣性[J]. 華中農業大學學報,2010,29(5):601-606.
[14]謝傳曉,朱蘇文,李培金,等. 玉米對生性狀兩個顯性基因SCAR分子標記[J]. 高技術通訊,2002,12(8):38-41.
[15]張增艷,辛志勇,陳孝,等. 源于L1的小麥抗黃矮病基因的特異PCR標記及輔助育種的研究[J]. 作物學報,2002,28(4):486-491.
[16]石金鋒,賈建航,金德敏,等. 紫菜無性系特異SCAR標記的獲得[J]. 海洋學報,2003,25(1):128-131.
[17]La Rosa R,Angiolillo A,Guerrero C,et al. A first linkage map of olive (Olea europaea L.) cultivars using RAPD,AFLP,RFLP and SSR markers[J]. Theoretical and Applied Genetics,2003,106(7):1273-1282.
[18]丁煒東,曹麗萍,曹哲明. 草魚種質相關SRAP及SCAR的分子標記[J]. 動物學報,2008,54(3):475-481.
[19]肖調義,張學文,章懷云,等. 洞庭湖四種黃顙魚基因組DNA遺傳多樣性的RAPD分析[J]. 中國生物工程雜志,2004,24(3):84-89.
[20]丁言偉. 黃顙魚屬(硬骨魚綱,鲿科)魚類分子系統發育及種群遺傳結構的研究[D]. 武漢:華中農業大學,2005:1-58.張仕林,許玉超,帥強,等. 洋蔥不同組織RNA提取方法比較分析[J]. 江蘇農業科學,2016,44(10):95-97.

