珠江口3種鯨豚的MHC-I基因多態性研究
余新建, 張西陽, 林文治, 周蕊蓮, 吳玉萍
?
珠江口3種鯨豚的基因多態性研究
余新建1, 2, 3, 4, 張西陽1, 2, 3, 4, 林文治1, 2, 3, 4, 周蕊蓮1, 2, 3, 4, 吳玉萍1, 2, 3, 4
(1. 南海生物資源開發與利用協同創新中心, 廣東珠海 519082; 2. 廣東省海洋資源與近岸工程重點實驗室, 廣東珠海 519082; 3. 珠海市海洋生物資源與環境重點實驗室, 廣東珠海 519082; 4. 中山大學海洋科學學院, 廣東珠海 519082)
旨在了解珠江口棲息的中華白海豚()、寬脊江豚()和點斑原海豚()免疫相關基因()的多態性及其表達情況, 以期為這3種鯨豚的保育工作提供基礎資料。通過克隆測序的方法, 首次證實基因在這3種鯨豚體內表達。選擇壓力分析, 表明基因在這3種鯨豚中均受到強烈的正選擇作用, 提示其具有重要的免疫功能。3種鯨豚的基因在系統發育樹上相互混雜在一起, 表明基因存在跨物種多態性。結合選擇壓力分析和跨物種多態性, 發現基因受到平衡選擇作用。珠江口3種鯨豚基因多態性可能由平衡選擇維持。
鯨豚;(); 表達; 平衡選擇
主要組織相容性復合體(,)是存在于大部分脊椎動物基因組中與免疫功能密切相關的一個基因家族。基因通過編碼不同的細胞表面受體, 識別并結合抗原, 并將其遞呈到效應細胞, 從而激發一系列的免疫應答反應。根據其結構組成、組織表達類型及進化史的差異,基因家族又可分為Ⅰ類、Ⅱ類和Ⅲ類3個亞家族。其中,類分子多表達在有核細胞表面, 在對細胞內病原體(主要是病毒)的免疫防御中扮演著重要角色。基因是脊椎動物中最具多態性的功能基因, 其中病原體介導的平衡選擇被認為是維持基因家族的等位基因多樣性的主要原因[1]。基因多樣性水平的降低會削弱種群對突發性傳染性病原體的抵抗力和生存力[2]。因此, 對野生動物尤其是保護物種的基因多樣性研究已成為近年來的熱點。
目前, 國內有關脊椎動物基因的研究比較多, 如應用在半滑舌鰨()的輔助育種研究中[3]。哺乳動物基因的研究主要集中在陸生動物, 如大熊貓()、恒河猴()等[4-5]。然而有關海洋哺乳動物, 如鯨豚類基因的研究則較少。目前中國有關中華白海豚()、寬脊江豚()、點斑原海豚()類基因的研究涉及多個水域[6-8]。張西陽等[9]對珠江口中華白海豚基因的研究發現其基因多樣性較低, 推測原因可能是該種群歷史上經歷了長期的瓶頸效應。但尚未發現有關珠江口水域這3種鯨豚基因研究的報道。
近年來, 珠江口鯨豚感染病原體的情況時有報道。香港的中華白海豚、寬脊江豚和寬吻海豚()中, 64頭鯨豚中有29%的個體發現病原體感染的情況[10]。另有研究發現2頭中華白海豚受病原體感染[11]。遺傳多樣性降低會造成中華白海豚對環境的適應力下降[12], 可能導致其易感染病原體。病原體具有引起動物大規模死亡的風險[13-14]。因此, 對瀕危物種的基因多態性的研究, 對于探討環境中的病原體對該物種的威脅及該物種在抵抗疾病和適應環境變化等方面的能力具有重要意義。
本研究通過對珠江口水域的中華白海豚、寬脊江豚和點斑原海豚的基因進行表達分析和克隆測序, 以探究這3種常見鯨豚的基因是否表達及基因多態性, 并通過選擇壓力分析和系統發育樹分析, 初步探討形成這種基因多態性特征的機制。
1 材料和方法
1.1 實驗材料
本研究選用擱淺死亡于珠江口的中華白海豚、寬脊江豚、點斑原海豚新鮮肌肉樣本。樣本取得后立即保存于Trizol中及–80℃中。
1.2 實驗方法
總RNA的提取參照RNAisoTMPlus(TaKaRa), 經瓊脂糖凝膠電泳和微量紫外分光光度計檢測其完整性和濃度, 用PrimeScript RT-PCR Kit(TaKaRa)將提取的RNA用Oligo(dT)引物逆轉錄為互補鏈DNA (Complementary DNA, cDNA)。采用經典酚-氯仿法提取基因組DNA(Genomic DNA, gDNA)。
1.3 PCR擴增、產物的純化、克隆及測序
使用2對引物DoLA-F/R和I2F/R分別擴增3種鯨豚的基因表達序列及其外顯子2序列, 產物經1.2%的瓊脂糖凝膠電泳檢驗目的片段。引物信息見表1。PCR反應體系包含6.88 μL ddH2O, 2×GC buffer I 12.5 μL, dNTP(10 mmol/L)2 μL, BSA(200 m g/L) 1.50 μL, 正、反引物(20 μmol/L)各0.5 μL, LA Taq 0.12 μL, 模板DNA(約100 mg/L) 1.00 μL。PCR反應程序為: 95℃預變性5 min; 95℃變性1 min, 55/50℃退火1 min, 72℃延伸4 min, 共35個循環; 然后72℃延伸10 min。PCR產物經1.2%瓊脂糖電泳檢測后, 用Agarose Gel DNA Purification Kit試劑盒(TaKaRa)純化回收。

表1 MHC-I表達片段及外顯子2片段的擴增所用引物
*: 引文的引物為簡并引物, 本研究根據表達序列對引物有所修改
純化后的DNA連接上pMD-19載體(TaKaRa), 轉化感受態大腸桿菌DH5α 菌株。待平板上長出菌落, 利用藍白斑篩選的方法挑出至少15個白色陽性菌落, 送至北京六合華大基因科技股份有限公司廣州測序部進行雙向測序。
1.4 數據分析
采用BioEdit軟件[17]進行序列比對, 以確定變異位點及等位基因。等位基因的命名參照家貓的命名原則: 一個新的等位基因的確定, 需要在同一個體的至少兩個克隆中出現, 或者在不同個體中出現[18]。為了分析物種間基因的系統發育關系, 使用Modeltest 3.7確定最合適的核酸替代模型[19]; 以家牛()作為外群, 使用MrBayes3.2軟件構建系統發育樹[20]。使用MEGA6計算平均核苷酸差異。使用DNAsp軟件和MEGA 6軟件對基因外顯子2及其肽結合區(Peptide Binding Region, PBR)進行選擇壓力分析[21-22]。
2 結果與分析
2.1基因的表達和序列變異
本研究從中華白海豚(1例)、寬脊江豚(2例)和點斑原海豚(1例)肌肉組織中均成功提取高質量的RNA和DNA。通過使用特異性引物DoLA-F和DoLA-R對cDNA進行擴增, 證實基因在3種鯨豚肌肉組織中的表達。中華白海豚、寬脊江豚和點斑原海豚肌肉組織中分別檢測到5、4和6條cDNA序列(822 bp) (GenBank: KU757454-KU757468),均包含完整的外顯子2(1~271 bp)、外顯子3(272~546 bp)和外顯子4(547~ 822 bp)。所有序列均未檢測到插入、缺失和終止子, 表明所有的序列都來源于基因組中的功能性分子。使用I2F和I2R引物從相同個體基因組DNA中擴增基因外顯子2序列。這些cDNA序列與gDNA序列均完全配對。
從中華白海豚、寬脊江豚和點斑原海豚基因組中分別擴增得到6、5和6個等位基因(147 bp), 變異位點數分別為32(21.8%)、23(15.6%)和28個(19.0%), 相應的氨基酸序列變異位點數為18(36.7%)、16(32.7%)和17個(34.7%), 大部分變異位點集中在基因的功能區, 如PBR區(表2)。珠江口中華白海豚有4個等位基因與其他水域中華白海豚一致[8](GenBank: EF375575, EF375576, EF375579, EF375580)。珠江口寬脊江豚中有2個等位基因與其他水域寬脊江豚一致(GenBank: DQ843624, DQ843625)[7]。新發現的基因命名如下: 中華白海豚為(GenBank: KU759499、KU759500); 寬脊江豚為(GenBank: KU759501- KU759503); 點斑原海豚為(GenBank: KU759504- KU759509)。
2.2系統發育
對基因核苷酸替代模型進行分析后, 采用AIC(Akaike Information Criterion)分析結果中的TVM+I+G模型構建系統發育樹。珠江口3種鯨豚與灰鯨()、印度-太平洋瓶鼻海豚()、條紋原海豚()、赫氏海豚()等序列相似度都在90%以上。灰鯨作為須鯨, 其基因單獨聚為1支(Mysticeti)(圖1)。其他齒鯨基因主要聚為5支(Odontoceti Ⅰ~Ⅴ), 但并沒有明顯的種屬聚類趨勢, 不同物種的等位基因分散聚合, 表現出明顯的跨物種多態性。例如其中1支為寬脊江豚(Ⅳ), 另外4支中均有寬脊江豚基因存在(圖1)。分支Ⅰ中均發現中華白海豚、寬脊江豚、點斑原海豚和赫氏海豚基因相互交叉(如與分別聚合后形成1支); 分支Ⅱ和Ⅴ中也與此類似(圖1)。
: DQ190936;: EU024810~EU024816;: AF149216;: AF149219;: AF149220;: AF188615;: DG843624;: DG843625;: DQ843630;: DQ843634; Neph-I*17: DQ843640;: DQ843653;: DQ843655;: DQ843656;: EF375575 ~EF375580;: EU698989;: EU698990;: EU698992 ~EU698996
2.3 選擇作用
使用DNAsp軟件對中華白海豚、寬脊江豚和點斑原海豚的基因外顯子2分析Tagima’值, 發現中華白海豚(0.114 14,>0.10)、寬脊江豚(0.376 64,>0.10)和點斑原海豚(1.06748,>0.10)的值均大于0(表3), 但是統計學差異并不顯著, 不能排除無正選擇作用存在的零假設。而使用MEGA 6.0軟件的檢驗對3種鯨豚的外顯子2及其肽結合區(PBR)進行選擇壓力分析(表4), 發現中華白海豚、寬脊江豚和點斑原海豚基因外顯子2及其PBR區的非同義替代率(N)顯著高于同義替代率(S), 均達到顯著水平(<0.05)甚至極顯著水平(<0.01), 提示正選擇作用的存在[23]。而點斑原海豚基因外顯子2非PBR區也顯示出較高的值(N/S), 且達到極顯著水平(<0.01), 提示其整個外顯子2區都受到正選擇作用。
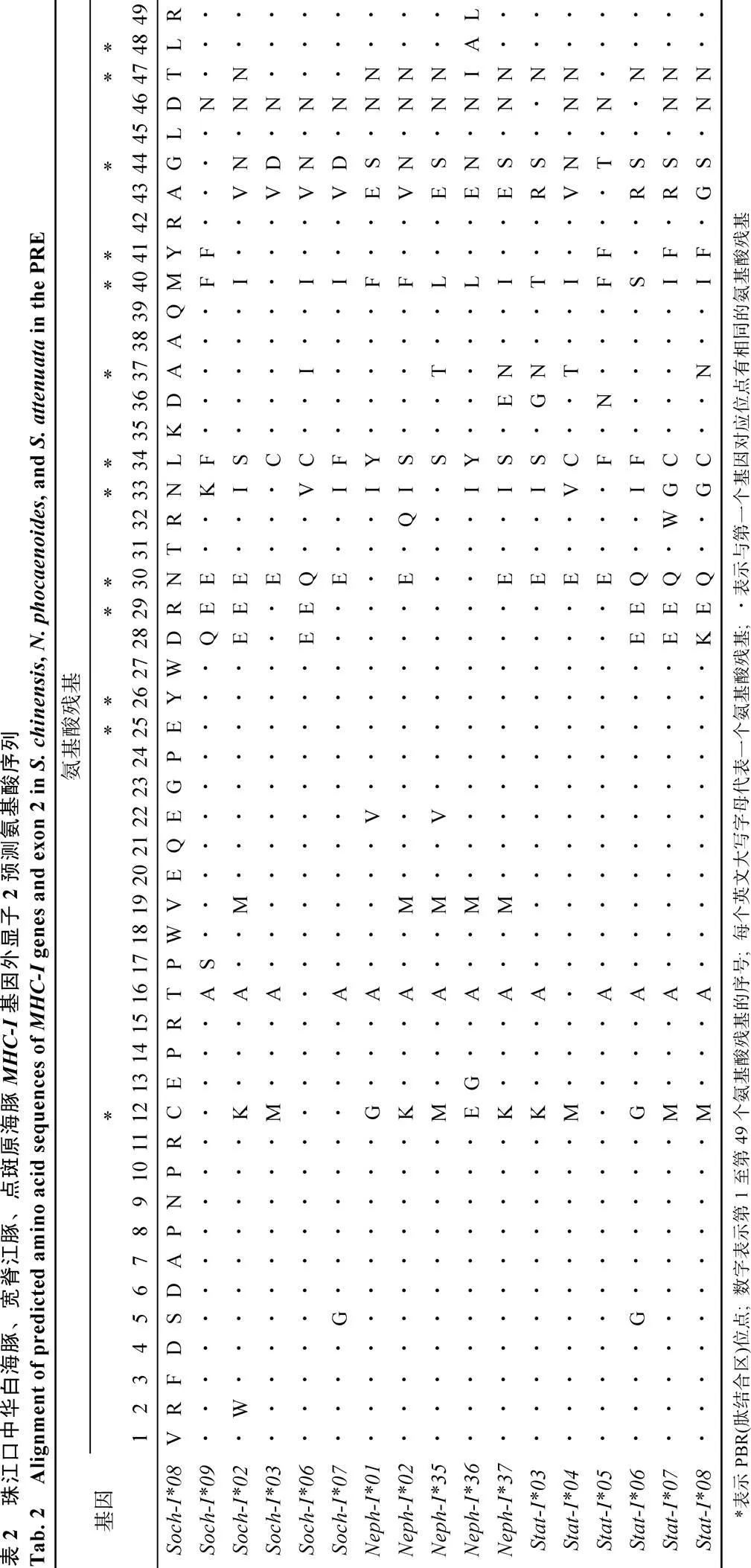
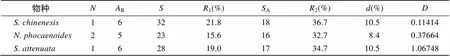
表3 珠江口中華白海豚、寬脊江豚、點斑原海豚MHC-I基因的多態性和選擇作用參數
: 個體數量;R: 等位基因豐度;: 差異的堿基位點數;1:占總堿基數的比例;A: 差異的氨基酸位點數;2:A占總氨基酸殘基數的比例;: 平均核苷酸差異;: Tajima檢測統計值
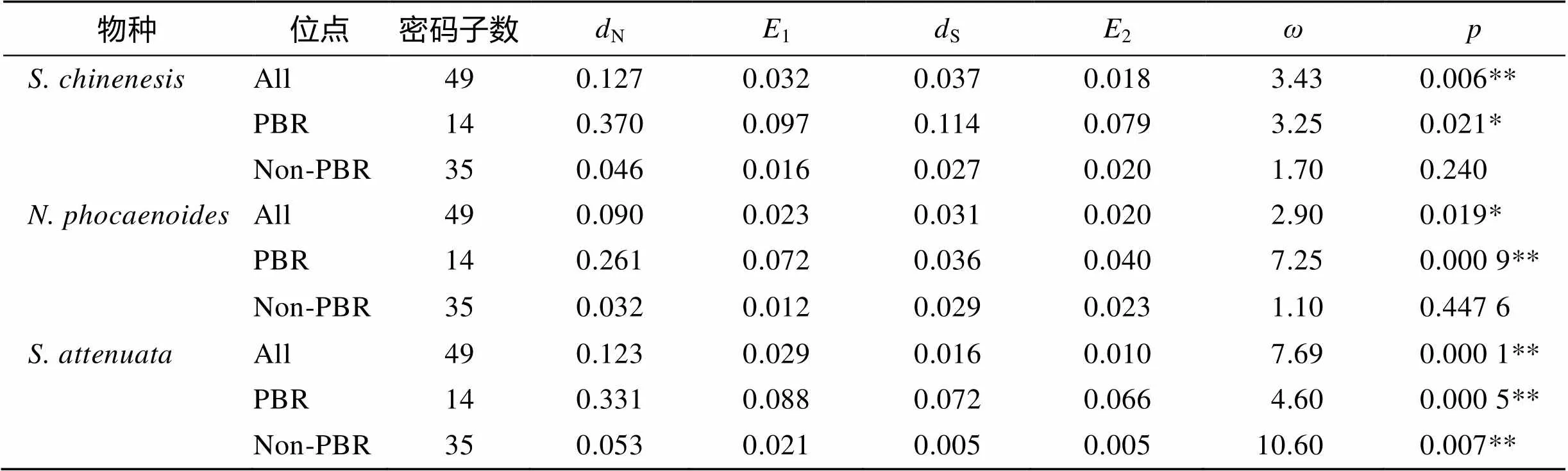
表4 珠江口3種鯨豚MHC-I基因、PBR區和非PBR區非同義替代與同義替代的估計值
N、S均根據Nei-Gojobori方法計算;N: 非同義替代率的估計值;S: 同義替代率的估計值;1、2: 1 000次重復計算N、S的標準誤差;為N/S;為使用檢驗時接受零假設的可能性; *:<0.05, 顯著水平; **:<0.01, 極顯著水平
3 討論
3.1 基因表達
本研究通過從新鮮樣本肌肉中獲取cDNA和gDNA, 擴增測序后發現這些cDNA序列與對應的gDNA外顯子序列完全匹配, 排除假基因的可能性, 確認基因在珠江口3種常見鯨豚體內表達。一般來說, 脊椎動物均表達基因。目前, 關于鯨豚基因表達的研究還比較少。例如赫氏海豚[24], 在2個個體中僅發現1個共同的基因cDNA。有趣的是多種海豚中僅發現1~2條cDNA[15, 24], 而本研究中3種鯨豚均表達4條以上基因cDNA。這可能與珠江口生存的海豚接觸病原體機會較高有關[25]。
3.2 基因位點數及基因變異
本研究的中華白海豚、點斑原海豚每個個體含有6個等位基因, 估計中華白海豚、點斑原海豚均至少有3個基因座位。這與Xu等人對中華白海豚基因座位估計的數目一致[8]。目前鯨豚中只有窄脊江豚()基因座位數被確定為4個, 然而各基因座位序列之間相似度較高[26]。
將珠江口中華白海豚種群與廈門等水域種群相比, 本研究發現有2個獨有的等位基因, 但未發現廈門等水域共有的1個等位基因(GenBank: EF375574)[8]。與其他等位基因差異較大。
3.3 平衡選擇
平衡選擇作用于珠江口3種鯨豚基因主要有以下兩方面的依據[27]。一方面是本研究的中華白海豚、寬脊江豚和點斑原海豚的基因外顯子2和PBR區均檢測到正選擇作用。另一方面是本研究中的3種鯨豚基因存在跨物種多態性。一般來說平衡選擇趨向于提高種群雜合度和在維持基因較高多樣性中扮演重要角色[28-29]。中華白海豚、寬脊江豚和點斑原海豚均檢測到多個等位基因, 結果與之吻合。PBR區具有重要功能是其檢測到強烈平衡選擇作用的原因, 而處于其毗連區的非PBR區的突變位點也可能更容易被保留[30]。這可能是導致在點斑原海豚的基因的非PBR區也檢測到選擇作用的原因。
3.4 跨物種多態性
系統發育樹顯示中華白海豚、寬脊江豚和點斑原海豚的基因外顯子2序列并不按照物種分開, 而是與其他的鯨豚混雜在一起, 提示跨物種多態性的存在。跨物種多態性是指一些相同的或相似的等位基因在不同物種中同時存在。一般有3種可能的機制來解釋這個現象。第一種機制是親緣關系較近的物種具有相似的等位基因被認為是來自于共同祖先, 這種跨物種等位基因在物種形成之前就存在不同物種世系中[31]。本研究的3種鯨豚都隸屬于齒鯨亞目, 它們親緣關系較近, 所以跨物種多態性有可能是共同祖先造成。同樣的機制也在其他鯨豚中發現[7]。第二種機制是趨同進化, 生物在應對相同的環境壓力而產生相同的適應性變化[32]。盡管本研究的中華白海豚、寬脊江豚、點斑原海豚均為近岸型鯨豚, 但它們生存環境的特性并不相同, 因而面臨不同的病原體壓力。然而赫氏海豚、條紋原海豚為遠洋性齒鯨, 其生存環境與近岸環境可能有較大差別。因而趨同進化并不適用于解釋其基因跨物種多態性。第三種機制是滲透雜交。海豚較其他哺乳動物容易產生可育后代[33], 例如寬吻海豚和長吻真海豚()[34]。但通過多年對珠江口中華白海豚的監測, 我們并沒有發現任何潛在的雜交后代。
綜上所述, 珠江口水域的中華白海豚、寬脊江豚和點斑原海豚均生活在具有較大的病原體壓力的近海環境中,基因的表達對于其抵抗病原體感染具有重要作用。珠江口的中華白海豚、寬脊江豚和點斑原海豚基因受到病原體介導的平衡選擇作用, 對于維持基因多態性具有重要意義。3種鯨豚之間的基因存在跨物種多態性, 可能來源于它們的共同祖先。考慮到日益嚴峻的環境趨勢[35-36],珠江口水域的中華白海豚、寬脊江豚和點斑原海豚的保育工作仍然任重道遠。
致謝: 感謝香港海洋公園保育基金(OPCFHK)和廣東珠江口中華白海豚國家級自然保護區管理局的大力支持。
[1] Prugnolle F, Manica A, Charpentier M, et al. Pathogen-driven selection and worldwideclass I diversity[J]. Current Biology, 2005, 15(11): 1022-1027.
[2] Radwan J, Biedrzycka A, Babik W. Does reduceddiversity decrease viability of vertebrate populations?[J] Biological Conservation, 2010, 143(3): 537-544.
[3] 牛寶珍, 杜民, 陳松林. 10 個半滑舌鰨家系基因多態性初步研究[J]. 海洋科學, 2015, 39(12): 70-76.Niu Baozhen, Du Min, Chen Songlin. Polymorphisms and balancing selection in the half-smooth tongue sole,[J]. Marine Sciences, 2015, 39(12): 70-76.
[4] 萬玉玲, 季芳, 饒軍華, 等. 中國不同地域恒河猴型部分等位基因的調查[J]. 動物學雜志, 2007, 42(2): 1-5. Wan Yuling, Ji Fang, Rao Junhua, et al. Typing of severalalleles of rhesus monkeys derived from different regions of China[J]. Chinese Journal of Zoology, 2007, 42(2): 1-5.
[5] Zhu Ying, Sun Dandan, Ge Yunfa, et al. Isolation and characterization of class Igenes in the giant panda ()[J]. Chinese Science Bulletin, 2013, 58(18): 2140-2147.
[6] Xu Shixia, Sun Peng, Zhou Kaiya, et al. Sequence variability at threeloci of finless porpoises ()[J]. Immunogenetics, 2007, 59(7): 581- 592.
[7] Xu Shixia, Ren Wenhua, Li Shuzhen, et al. Sequence polymorphism and evolution of three cetaceangenes[J]. Journal of Molecular Evolution, 2009, 69(3): 260-275.
[8] Xu Shixia, Zhang Peng, Li Shuzhen, et al. A preliminary analysis of genetic variation at threeloci of the Indo-Pacific humpback dolphin ()[J]. Acta Theriologica Sinica, 2009b, 29(4): 372.
[9] Zhang Xiyang, Lin Wenzhi, Zhou Ruilian, et al. Low major histocompatibility complex class II variation in the endangered Indo-pacific humpback dolphin (): inferences about the role of balancing selection[J]. Journal of Heredity, 2016, 107(2): 143-152.
[10] Parsons E, Jefferson T. Post-mortem investigations on stranded dolphins and porpoises from Hong Kong waters[J]. Journal of Wildlife Diseases, 2000, 36(2): 342- 356.
[11] Jefferson T, Hung S, Lam P. Strandings, mortality and morbidity of Indo-Pacific humpback dolphins in Hong Kong, with emphasis on the role of organochlorine contaminants[J]. Journal of Cetacean Research and Management, 2006, 8(2): 181-193.
[12] Lin Wenzhi, Chang Lihong, Frère C H, et al. Differentiated or not? An assessment of current knowledge of genetic structure ofin China[J]. Journal of Experimental Marine Biology and Ecology, 2012, 416: 17-20.
[13] 宋微波, 王崇明, 王秀華, 等. 櫛孔扇貝大規模死亡的病原研究新進展[J]. 海洋科學, 2001, 25(12): 23-26. Song Weibo, Wang Congming, Wang Xiuhua, et al. New research progress on massive mortality of cultured scallop[J]. Marine Sciences, 2001, 25(12): 23-26.
[14] Van Bressem M F, Duignan P J, Banyard A, et al. Cetacean morbillivirus: current knowledge and future directions[J]. Viruses, 2014, 6(12): 5145-5181.
[15] Shirai K, Sakai T, Oike T. Molecular cloning of bottle-nosed dolphin ()class I cDNA[J]. Journal of Veterinary Medical Science, 1998, 60(10): 1093-1096.
[16] Floresramirez S, Urbanramirez J, Miller R. Major histocompatibility complex class I loci from the gray whale ()[J]. Journal of Heredity, 2000, 91(4): 279-282.
[17] Hall T A. BioEdit: a user-friendly biological sequence alignment editor and analysis program for Windows 95/98/NT[J]. Nucleic Acids Symposium Series, 1999, 41: 95-98.
[18] Kennedy L J, Ryvar R, Gaskell R M, et al. Sequence analysis ofalleles indomestic cats from the United Kingdom[J]. Immunogenetics, 2002, 54(5): 348-352.
[19] Posada D, Crandall K. MODELTEST: testing the model of DNA substitution[J]. Bioinformatics, 1998, 14(9): 817-818.
[20] Ronquist F, Teslenko M, Van d M P, et al. MrBayes 3.2: efficient Bayesian phylogenetic inference and model choice across a large model space[J]. Systematic Biology, 2012, 61(3): 539-542.
[21] Bjorkman P J, Saper M A, Samraoui B, et al. Structure of the human class I histocompatibility antigen,-[J]. Nature, 1987, 329(6139): 506-512.
[22] Tamura K, Stecher G, Peterson D, et al. MEGA6: molecular evolutionary genetics analysis version 6.0[J]. Molecular Biology & Evolution, 2013, 30(4): 576-577.
[23] Figueroa F, Gúnther E, Klein J.polymorphism pre-dating speciation[J]. Nature, 1988, 335(6187): 265- 267.
[24] Heimeier D, Baker C S, Russell K, et al. Confirmed expression ofclass I and class II genes in the New Zealand endemic hector's dolphin ()[J]. Marine Mammal Science, 2008, 25(1): 68-90.
[25] 閆冬, 陳加林, 鄭銳強, 等. 珠江口中華白海豚主要棲息地的細菌菌群結構[J]. 海洋環境科學, 2013, 32(1): 49-53. Yan Dong, Chen Jialin, Zheng Ruiqiang, et al. Bacterial community structure of main habitat forin the Zhujiang Estuary[J]. Marine EnvironmentalScience, 2013, 32(1): 49-53.
[26] Ruan Rui, Wan Xiaoling, Zheng Yang, et al. Assembly and characterization of theclass I region of the Yangtze finless porpoise ()[J]. Immunogenetics, 2016, 68(1): 77-82.
[27] Hughes A L, Nei M. Pattern of nucleotide substitution at major histocompatibility complex class I loci reveals overdominant selection[J]. Nature, 1988, 335(6186): 167-170.
[28] Hedrick P W. Balancing selection and[J]. Genetica, 1998, 104(3): 207-214.
[29] Muirhead C A. Consequences of population structure on genes under balancing selection[J]. Evolution, 2001, 55(8): 1532-1541.
[30] Charlesworth D. Balancing selection and its effects on sequences in nearby genome regions[J]. PLoS Genet, 2006, 2(4): e64.
[31] Klein J. Origin of major histocompatibility complex polymorphism: the trans-species hypothesis[J]. Human Immunology, 1987, 19(3): 155-162.
[32] Kriener K, O'hUigin C, Tichy H, et al. Convergent evolution of major histocompatibility complex molecules in humans and New World monkeys[J]. Immunogenetics, 2000, 51(3): 169-178.
[33] Amaral A, Sequeira M, Martínez-Cedeira J, et al. New insights on population genetic structure offrom the northeast Atlantic and phylogenetic relationships within the genus inferred from two mitochondrial markers[J]. Marine Biology, 2007, 151(5): 1967-1976.
[34] Zornetzer H R, Duffield D A. Captive-born bottlenose dolphin × common dolphin (×) intergeneric hybrids[J]. Canadian Journal of Zoology, 2003, 81(10): 1755-1762.
[35] 周濤, 韓彬, 劉新民, 等. 南中國海海水中多環芳烴的分布特征及源分析[J]. 海洋科學, 2014, 38(8): 39- 45. Zhou Tao, Han Bin, Liu Xinmin, et al. Distribution and origin of polycyclic aromatic hydrocarbons in the sea water of the South Sea[J]. Marine Sciences, 2014, 38(8): 39-45.
[36] Gui Duan, Yu Riqing, He Xuan, et al. Bioaccumulation and biomagnification of persistent organic pollutants in Indo-Pacific humpback dolphins () from the Pearl River Estuary, China[J]. Chemosphere, 2014, 114(22): 106-113.
Study ofpolymorphism in three cetaceans from the Pearl River Estuary, China
YU Xin-jian1, 2, 3, 4, ZHANG Xi-yang1, 2, 3, 4, LIN Wen-zhi1, 2, 3, 4, ZHOU Rui-lian1, 2, 3, 4, WU Yu-ping1, 2, 3, 4
(1. South China Sea Bio-Resource Exploitation and Utilization Collaborative Innovation Center, Zhuhai 519082, China; 2. Guangdong Provincial Key Laboratory of Marine Resources and Coastal Engineering, Zhuhai 519082, China; 3. Zhuhai Key Laboratory of Marine Bioresources and Environment, Zhuhai 519082, China; 4. School of Marine Sciences, Sun Yat-Sen University, Zhuhai 519082, China)
To gain the basic knowledge about cetacean conservation, we assessed the polymorphism and expression of the immunologic gene() in three cetaceans (,, and) around the Pearl River Estuary. We first confirmed the expression ofgenes in the three cetaceans by cloning and sequencing. After selection pressure analysis, we found that relatively strong positive selections have acted ongenes in these three cetaceans, suggesting their important immunologic function. Phylogenetic analysis showed that thegenes of the three cetaceans did not cluster to three branches according to species but were mixed up, which implies the existence of trans-species polymorphism (TSP). Together with the results of selection pressure analysis and TSP, we suggest that intense balancing selection has acted ongenes in these three cetaceans, which further maintained the polymorphism ofgenes.
cetaceans;(); expression; balancing selection
(本文編輯: 劉珊珊)
Apr. 24, 2016
[National Natural Science Foundation of China, No.41276147, No.41576128; Chinese White Dolphin Conservation Action Plan of the Ministry of Agriculture, No.2015; Ocean Park Conservation Fund Hong Kong, No.2013]
Q953
A
1000-3096(2016)10-0126-08
10.11759/hykx20160424001
2016-04-24;
2016-08-30
國家自然科學基金項目(41276147, 41576128); 農業部中華白海豚保護行動計劃(2015); 香港海洋公園保育基金(2013)
余新建(1991-), 男, 江西撫州人, 碩士研究生, 主要從事海洋鯨豚保護遺傳學研究, 電話: 15820590518, E-mail: earthclean@ 163.com; 張西陽, 與第一作者同等貢獻, 博士研究生, 主要從事海洋鯨豚保護遺傳學研究; 吳玉萍, 通信作者, 教授, E-mail: exwyp@ mail.sysu.edu.cn

