基因多態性與藥品不良反應的相關性Δ
孫雪林,胡 欣,徐巧玲(.北京醫院藥學部/國家老年醫學研究中心/藥物臨床風險與個體化應用評價北京市重點實驗室,北京 00730;2.解放軍第305醫院藥局,北京 0007)
·精準醫療·
基因多態性與藥品不良反應的相關性Δ
孫雪林1*,胡欣1,徐巧玲2#(1.北京醫院藥學部/國家老年醫學研究中心/藥物臨床風險與個體化應用評價北京市重點實驗室,北京100730;2.解放軍第305醫院藥局,北京100017)
目的:為臨床精準用藥、預防和減少藥品不良反應(ADR)的發生提供參考。方法:收集近年來國內外有關藥物基因組學與ADR的研究文獻,對基因多態性與ADR的相關性進行歸納和總結。結果與結論:目前已發現多種ADR相關基因。人類白細胞抗原基因多態性與雙氯芬酸、阿莫西林克拉維酸鉀等藥物致肝損傷,卡馬西平、阿巴卡韋等藥物致皮膚過敏反應有關;肝臟藥物轉運體1B1基因多態性與他汀類藥物致肌病有關;N-乙酰基轉移酶2基因多態性與異煙肼致肝損傷有關;一氧化氮合酶1調酶基因、心臟鈉離子通道5A基因和鉀離子通道E1基因多態性與藥物致QT間期延長有關等。基因多態性研究可為預防ADR提供新的參考依據,臨床可在藥物治療前對患者進行基因型檢測,以降低ADR的發生率。
藥品不良反應;藥物基因組學;基因多態性;精準用藥
藥品不良反應(Adverse drug reaction,ADR)增加了患者在用藥過程中非疾病因素導致的發病率和病死率,輕者影響藥物治療、延長病程、增加患者經濟負擔,重者則對患者生命安全造成威脅,目前已成為全球高度關注的公共衛生問題[1-2]。2015年全國藥品ADR監測網絡共收到ADR/不良事件報告表139.8萬份,較2014年增長5.3%,新的和嚴重ADR/不良事件報告數量占同期報告總數的28.2%[3]。ADR可影響機體肝臟、心臟、皮膚和肌肉等多個器官和組織,有的藥品甚至還可引發更廣泛的過敏反應。嚴重的ADR主要表現為肝臟毒性和心臟毒性。其中,藥物引起的肝臟毒性表現為肝細胞損傷和膽管細胞膜損傷所致的膽汁淤積,約10%肝毒性ADR可進展為肝衰竭,對患者生命安全造成嚴重威脅[4];而藥物引起的心臟毒性則主要表現為心臟復極延遲,心電圖檢測示QT間期延長,引起室顫,最終導致患者死亡[5]。
藥物基因組學(Pharmacogenomics,PGx)是研究基因多態性如何影響藥物治療的反應性和治療過程中遺傳變異與ADR風險的關聯性的一門學科。PGx可確定藥物的作用和遺傳因素間的相關性,并以此來減少ADR的發生。例如,服用伯氨喹導致的溶血反應是由于患者缺乏葡萄糖-6-磷酸脫氫酶(G6PD)[6],而卡馬西平引起的中毒性表皮壞死溶解癥(TEN)和Steven-Johnson綜合征(SJS)則與人類白細胞抗原(Human leukocyte antigen,HLA)-B*1502等位基因密切相關[7]。
本文通過收集近年來國內外有關PGx與ADR的研究文獻,對基因多態性與ADR的相關性進行了歸納和總結,為提高藥物治療效果、減少患者開支、預防嚴重ADR/不良事件的發生提供借鑒,為臨床精準用藥提供參考。
1 HLA與藥物性肝損傷、過敏反應和皮疹
HLA是人類白細胞抗原的簡稱,編碼基因是人類主要組織相容性復合體,與人類免疫系統的功能密切相關。HLA基因位于第6號染色體短臂6p21.3區域,長度為3.6×106bp。根據其在染色體上的排列可分為3個區域:Ⅰ類基因區包括HLA-A、HLA-B和HLA-C;Ⅱ類基因區包含HLA-DR、HLADP和HLA-DQ;Ⅲ類基因區位于上述2個基因區之間,為補體系統,包括C2和C4。HLA基因多態性與藥物性肝損傷、SJS和TEN等相關,可作為某些ADR的風險預測因子。
1.1HLA與藥物性肝損傷
目前臨床應用的許多藥物均會引起藥物性肝損傷,發生概率約為1/10 000[8],發生機制可能與藥物對肝臟細胞的直接毒性有關,包括氧化應激和細胞損傷等,給肝細胞帶來直接傷害[9]。日本首先報道了血清Ⅱ型HLA-DR2與藥物性肝損傷的關系。在發生黃疸的患者中,DR2血清分型的檢出率達58.3%;在藥物性肝損傷引發黃疸的患者中,DR2單倍體的頻率為0.236,與對照組比較差異有統計學意義(P<0.004 2)[10]。此外,血清Ⅰ型HLA-A11與三環類抗抑郁藥和雙氯芬酸相關,其引發肝炎的概率分別為50%和75%;血清Ⅱ型HLA-DR6與氯丙嗪相關,在氯丙嗪所致肝炎患者中HLA-DR6的檢出率高達80%[11]。
目前,研究HLA與藥物性肝損傷的相關性主要是利用患者的基因分型。第一個被發現HLA基因分型與藥物性肝損傷相關的藥物是阿莫西林克拉維酸鉀。這種損傷和經典的免疫反應不同,其與HLA-DR B1*1501編碼的血清HLA-DR2蛋白有關[12]。相關性最強的是抗菌藥物氟氯西林與Ⅰ型HLA-B* 5701基因,同時該基因也與阿巴卡韋引起的過敏反應高度相關[13-14]。HLA與ADR的相關性見表1。
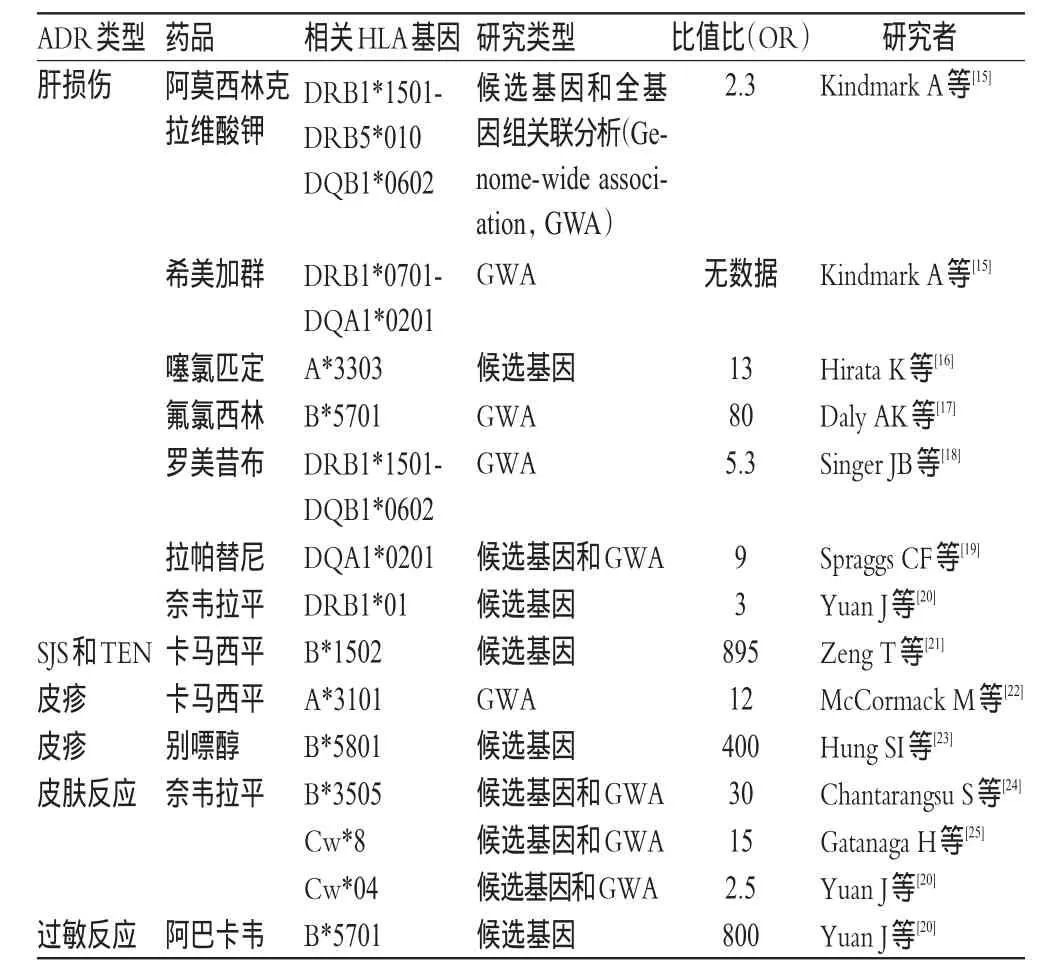
表1 HLA與ADR的相關性Tab 1 Correlation between HLAandADR
1.2HLA與皮膚過敏反應
ADR引起的皮膚過敏反應可分為速發型和遲發型反應。速發型反應主要由免疫球蛋白(Ig)E介導,涉及的遺傳因素尚不明確。遲發型過敏反應的皮膚表現個體差異較大,停藥后皮膚癥狀可立即改善。該類過敏反應還可涉及其他組織和器官,表現為發熱和嗜酸性粒細胞增多,甚至可引發嚴重的皮疹(包括SJS和TEN)。相關研究表明,T細胞與藥物引起的遲發型皮膚過敏反應有關[26]。由HLA基因編碼的人類白細胞抗原提呈抗原給T細胞,故其基因多態性可能與遲發型過敏反應相關。
1.2.1卡馬西平與皮膚過敏卡馬西平致ADR的相關基因多態性首次在臺灣人群中被報道,該藥引發的SJS和HLA-B* 1502高度相關[27]。HLA-B*1502位點的突變在我國漢族及泰國、馬來西亞、印度尼西亞、菲律賓和印度南部等種族人群中普遍存在,建議以上人群在服用卡馬西平之前檢測HLA-B* 1502基因位點是否存在變異,以減少皮膚過敏反應的發生[7];由于HLA-B*1502基因在其他人群中突變頻率較低,暫不推薦其他人群檢測。
1.2.2阿巴卡韋與皮膚過敏抗人類免疫缺陷病毒(Human immunodef iciency virus,HIV)藥阿巴卡韋引起的ADR主要表現為皮疹、呼吸道和胃腸道癥狀,其癥狀一般較輕,且停藥后即可緩解,但若再次服藥則會引發嚴重癥狀,有些甚至是致命的。HLA-B*5701和阿巴卡韋致過敏反應的相關性在19個國家1 956例患者的雙盲、隨機、對照試驗中得到證實,HLA-B* 5701在試驗人群中有5.6%的攜帶率,94%的患者服用阿巴卡韋發生過敏反應的風險較低;但在發生過敏反應的患者中,47.9%的患者攜帶HLA-B*5701[28]。表明HLA-B*5701可能與阿巴卡韋致過敏反應有關,該基因型的檢測可能有助于降低阿巴卡韋致過敏反應的發生率,因此建議在服用阿巴卡韋前應對患者進行HLA基因型檢測。
1.2.3致皮膚過敏的其他ADR別嘌呤醇引起的SJS、TEN或其他輕度的皮膚過敏反應和HLA-B*5801基因位點相關,該位點首先在我國臺灣人群中被發現,且在我國漢族人群中的關聯性也較強[23,29]。在別嘌醇所致的皮疹患者中,有95%的患者攜帶HLA-B*5801,而未發生皮疹的患者中攜帶該基因的比例僅為12%[OR=127.6,95%置信區間(CI)(40.8,398.6)][29],表明HLA-B*5801可能與別嘌醇致皮膚過敏有關,建議在服用該藥前進行HLA-B*5801多態性位點檢測。在歐洲人群中同樣發現了該位點與ADR的相關性[30]。抗HIV病毒藥奈韋拉平常見的ADR也是皮膚反應,嚴重程度因人而異。在薩丁和日本人群中,奈韋拉平致皮疹與HLA Cw*8相關[25,31];在泰國人群中,與HLA-B*3505相關[32];在歐洲、亞洲和非裔美國人群中,與HLACw*04相關[20]。
2 引起ADR的其他基因多態性
除HLA基因多態性與ADR相關外,還有一些其他遺傳因素與ADR有關,包括影響藥物體內代謝過程、先天免疫系統和心臟離子通道的相關基因及其多態性。
2.1藥物體內代謝相關基因及其多態性
目前已發現肝臟藥物轉運體1B1基因(SLCO1B1)多態性和他汀類藥物所致肌病、N-乙酰基轉移酶2基因(NAT2)多態性和異煙肼所致藥物性肝損傷有關,其他與ADR相關的基因還包括三磷酸腺苷結合盒轉運體(ABC)和糖基轉移酶(UGT)等基因,詳見表2。
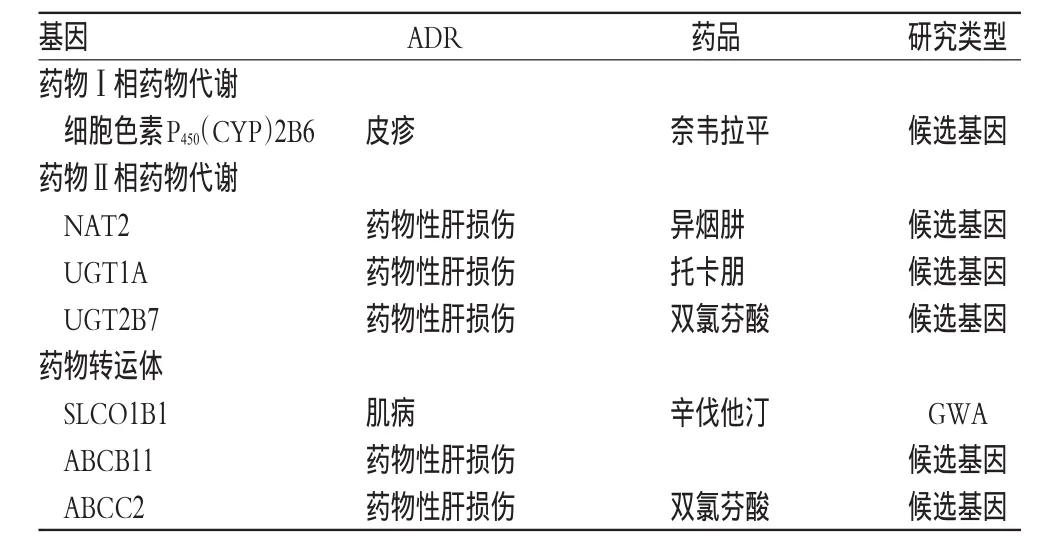
表2 藥物體內代謝相關基因及其多態性與ADRTab 2 Drug metabolism-related gene and its polymorphisms andADR
他汀類藥物是目前最有效的調脂藥,主要ADR為肌肉毒性,通常表現為無癥狀的血清肌酸激酶水平升高,嚴重的可引發橫紋肌溶解癥,甚至導致死亡[33]。有文獻報道,辛伐他汀引起的肌肉酸痛等ADR與SLCO1B1基因多態性有關,SLCO1B1在肝臟細胞中編碼轉運蛋白[34],后者將外周血中的藥物轉運到肝臟細胞中進行代謝。SLCO1B1*5/*15基因突變可延長藥-時曲線下面積(AUC),使轉運蛋白合成受損,導致他汀類藥物代謝受阻,血藥濃度升高,ADR發生率增加。但SCLO1B1*5/*15基因多態性在預測不同種類他汀類藥物的ADR時有較大差異,影響最大的為辛伐他汀,其次為匹伐他汀和阿托伐他汀[35]。
CYP2B6是代謝奈韋拉平的酶類,516G>T的突變可降低該酶催化奈韋拉平及其代謝物的能力,增加皮疹發生的風險[20]。NAT2基因編碼N-乙酰基轉移酶2,在異煙肼代謝過程中發揮著重要的作用。異煙肼經過代謝后生成乙酰肼,可進一步被CYP或NAT2代謝為毒性更低的二乙酰基肼。若NAT2酶活性正常,異煙肼可被代謝為二乙酰基肼,發生ADR的概率將大大降低;若NAT2等位基因發生純合突變,其酶活性將明顯降低,則異煙肼無法被代謝,發生藥物性肝損傷等ADR的概率將明顯增加[36]。
此外,與藥物性肝損傷相關的還有UGT基因多態性。UGT1A6基因與托卡朋引起的轉氨酶升高相關[37];UGT2B7基因多態性位點與雙氯芬酸引起的肝毒性有關[38]。ABC家族除了轉運藥物之外還負責膽汁的轉運,因此ABCB4(編碼MDR3蛋白,磷脂輸出泵)和ABCB11(編碼BSEP蛋白,膽鹽輸出泵)基因突變后會導致膽汁淤積[39]。ABCC2(編碼MRP2蛋白,多藥耐藥相關蛋白)在膽汁分泌葡糖醛酸結合物中具有重要作用,ABCC2(24C>T)基因多態性與肝毒性的發生密切相關:24C>T突變可導致MRP2蛋白合成減少,葡糖醛酸易在細胞內堆積,發生雙氯芬酸酰基葡糖醛酸反應從而引發肝毒性[38,40]。
2.2先天免疫系統相關基因及其多態性
基因篩查發現,免疫系統某些細胞因子的多態性可影響患者的先天免疫系統,并可預測藥物性肝損傷的發生。例如,在服用雙氯芬酸所致肝損傷的患者中發現,白細胞介素(IL)-10基因-627位點和IL-4基因-590位點存在突變,可引起IL-10的低轉錄和IL-4的高轉錄,有助于T細胞介導的疾病易感基因相關的抗體反應[41]。
2.3心臟離子通道相關基因及其多態性
心臟毒性是藥品撤市最常見的原因[42]。非藥物引起的QT間期延長主要與患者心臟離子通道相關基因的突變有關。目前已確認與之有關的基因包括:一氧化氮合酶1調節酶基因(NOS1AP)、心臟鈉離子通道(SCN)5A基因和鉀離子通道(KCN)J2基因[43-44]。
對藥物致QT間期延長的患者進行離子通道基因測序,結果顯示,在32例患者中,有4例患者存在突變,其中有2例是KCNE1 253G>A(D85N,rs1805128)發生突變[45]。對307例藥物致QT間期延長的患者進行基因型分析,也證實了D85N和KCNE1基因多態性與藥物心臟毒性相關[46]。NOS1AP(rs10494366)突變與維拉帕米引起的QT間期延長有關,且在前瞻性研究(n=7 000)中得到證實:各患者服用維拉帕米后,GG基因型[QT間期為25.4 ms,95%CI(5.9,44.9)]與TT基因型[QT間期為19.8 ms,95%CI(1.9,37.7)]患者比較,其QT間期明顯延長[47]。最近英國學者又發現,NOS1AP的另外一個單核苷酸多態性位點(rs10800397)與藥物引起的QT間期延長有關[OR=3.3,99.95%CI(1.0,10.8)][48]。
3 結語
目前,我國ADR監測主要依靠醫療機構、藥品經營企業、藥品生產企業以及個人上報的數據,最后以ADR信息通報的形式向社會發布,起到警示、宣傳和促進合理用藥的作用,但要精準或個體化地預防ADR的發生,就必須在用藥前對高危人群進行相關的基因型檢測,若患者帶有某ADR相關位點的突變基因,建議降低藥物劑量或直接換用其他藥物。
雖然現有基因多態性可解釋部分ADR發生的原因,但與大多數ADR之間的對應關系還有待于進一步研究,特別是對我國人群的深入研究。人體對藥物反應的差異受多種因素的影響,除了基因多態性外,還包括其健康狀況、飲食、性別、年齡等因素;臨床上常見的聯合用藥也使ADR的發生機制更為復雜,給預防和判斷增加了難度。因此,如何將PGx知識轉化為臨床應用,是目前臨床面臨的一個重大課題。此外,從分子水平深入研究導致ADR(尤其是嚴重ADR)發生的確切原因,利用基因差異篩選技術,對特定人群推薦或開發“量身定做”的新藥,提高藥物療效、降低用藥風險,也是未來醫藥發展的方向。
[1]Daly AK.Pharmacogenomics of adverse drug reactions[J]. Genome Med,2013,5(1):5.
[2]Bienfait KL,Shaw PM,Murthy G,et al.Mobilizing pharmacogenomic analyses during clinical trials in drug development[J].Pharmacogenomics,2013,14(10):1 227.
[3]國家食品藥品監督管理總局.國家藥品不良反應監測年度報告:2015年[S].2016-07-13.
[4]Assis DN,Navarro VJ.Human drug hepatotoxicity:a contemporaryclinicalperspective[J].ExpertOpinDrug Metab Toxicol,2009,5(5):463.
[5]Service JA,Waring WS.QT Prolongation and delayed atrioventricular conduction caused by acute ingestion of trazodone[J].Clin Toxicol:Phila,2008,46(1):71.
[6]Beutler E.The hemolytic effect of primaquine and related compounds:a review[J].Blood,1959,14(2):103.
[7]Chen Z,Liew D,Kwan P.Effects of a HLA-B*15:02 screening policy on antiepileptic drug use and severe skin reactions[J].Neurology,2014,83(22):2 077.
[8]Mengoli M,Parmeggiani D,Mengoli MC,et al.Drug-induced hepatotoxicity:clinical and biochemical features of 26 patients and a review of the literature[J].Recenti Prog Med,2011,102(6):253.
[9]Leung L,Kalgutkar AS,Obach RS.Metabolic activation in drug-induced liver injury[J].Drug Metab Rev,2012,44(1):18.
[10]Otsuka S,Yamamoto M,Kasuya S,et al.HLA antigens in patients with unexplained hepatitis following halothane anesthesia[J].Acta Anaesthesiol Scand,1985,29(5):497.
[11]Berson A,Fréneaux E,Larrey D,et al.Possible role of HLA in hepatotoxicity:an exploratory study in 71 patients with drug-induced idiosyncratic hepatitis[J].J Hepatol,1994,20(3):336.
[12]Hautekeete ML,Horsmans Y,van Waeyenberge C,et al. HLA association of amoxicillin-clavulanate-induced hepatitis[J].Gastroenterology,1999,117(5):1 181.
[13]Mallal S,Nolan D,Witt C,et al.Association between presence of HLA-B*5701,HLA-DR7,and HLA-DQ3 and hypersensitivity to HIV-1 reverse-transcriptase inhibitor abacavir[J].Lancet,2002,359(9 308):727.
[14]Hetherington S,Hughes AR,Mosteller M,et al.Genetic variations in HLA-B region and hypersensitivity reactions to abacavir[J].Lancet,2002,359(9 312):1 121.
[15]Kindmark A,Jawaid A,Harbron CG,et al.Genome-wide pharmacogenetic investigation of a hepatic adverse event without clinical signs of immunopathology suggests an underlying immune pathogenesis[J].Pharmacogenomics J,2008,8(3):186.
[16]Hirata K,Takagi H,Yamamoto M,et al.Ticlopidine-induced hepatotoxicity is associated with specific human leukocyte antigen genomic subtypes in Japanese patients:a preliminary case-control study[J].Pharmacogenomics J,2008,8(1):29.
[17]Daly AK,Donaldson PT,Bhatnagar P,et al.HLA-B*5701 genotype is a major determinant of drug-induced liver injury due to flucloxacillin[J].Nat Genet,2009,41(7):816.
[18]Singer JB,Lewitzky S,Leroy E,et al.A genome-wide study identifies HLA alleles associated with lumiracoxibrelated liver injury[J].Nat Genet,2010,42(8):711.
[19]Spraggs CF,Budde LR,Briley LP,et al.HLA-DQA1*02∶01 is a major risk factor for lapatinib-induced hepatotoxicity in women with advanced breast cancer[J].J Clin Oncol,2011,29(6):667.
[20]Yuan J,Guo S,Hall D,et al.Toxicogenomics of nevirapine-associated cutaneous and hepatic adverse events among populations of African,Asian,and European descent[J].Aids,2011,25(10):1 271.
[21]Zeng T,Long YS,Min FL,et al.Association of HLA-B* 1 502 allele with lamotrigine-induced Stevens-Johnson syndrome and toxic epidermal necrolysis in Han Chinese subjects:a meta-analysis[J].Int J Dermatol,2015,54(4):488.
[22]McCormack M,Alfirevic A,Bourgeois S,et al.HLA-A* 3101 and carbamazepine-induced hypersensitivity reactions in Europeans[J].N Engl J Med,2011,364(12):1 134.
[23]Hung SI,Chung WH,Liou LB,et al.HLA-B*5801 allele as a genetic marker for severe cutaneous adverse reactions caused by allopurinol[J].Proc Natl Acad Sci USA,2005,102(11):4 134.
[24]Chantarangsu S,Mushiroda T,Mahasirimongkol S,et al. Genome-wide association study identifies variations in 6p21.3 associated with nevirapine-induced rash[J].Clin Infect Dis,2011,53(4):341.
[25]Gatanaga H,Yazaki H,Tanuma J,et al.HLA-Cw8 primarily associated with hypersensitivity to nevirapine[J].Aids,2007,21(2):264.
[26]Svensson CK,Cowen EW,Gaspari AA.Cutaneous drug reactions[J].Pharmacol Rev,2001,53(3):357.
[27]Chung WH,Hung SI,Hong HS,et al.Medical genetics:a marker for Stevens-Johnson syndrome[J].Nature,2004,428(6 982):486.
[28]Mallal S,Phillips E,Carosi G,et al.HLA-B*5701 screening for hypersensitivity to abacavir[J].N Engl J Med,2008,358(6):568.
[29]Cheng L,Xiong Y,Qin CZ,et al.HLA-B*58∶01 is strongly associated with allopurinol-induced severe cutaneous adverse reactions in Han Chinese patients:a multicentre retrospective case-control clinical study[J].Br J Dermatol,2015,173(2):555.
[30]Lonjou C,Borot N,Sekula P,et al.A European study of HLA-B in Stevens-Johnson syndrome and toxic epidermal necrolysis related to five high-risk drugs[J].Pharmacogenet Genomics,2008,18(2):99.
[31]Littera R,Carcassi C,Masala A,et al.HLA-dependent hypersensitivity to nevirapine in Sardinian HIV patients[J]. Aids,2006,20(12):1 621.
[32]Chantarangsu S,Mushiroda T,Mahasirimongkol S,et al. HLA-B*3505 allele is a strong predictor for nevirapine-induced skin adverse drug reactions in HIV-infected Thai patients[J].Pharmacogenet Genomics,2009,19(2):139.
[33]Dalakas MC.Toxic and drug-induced myopathies[J].J Neurol Neurosurg Psychiatry,2009,80(8):832.
[34]Search Collaboraive Group,Link E,Parish S,et al.SLCO1B1 variants and statin-induced myopathy:a genomewide study[J].N Engl J Med,2008,359(8):789.
[35]Niemi M.Transporter pharmacogenetics and statin toxicity [J].Clin Pharmacol Ther,2010,87(1):130.
[36]Metushi IG,Cai P,Zhu X,et al.A fresh look at the mechanism of isoniazid-induced hepatotoxicity[J].Clin Pharmacol Ther,2011,89(6):911.
[37]Acu?a G,Foernzler D,Leong D,et al.Pharmacogenetic analysis of adverse drug effect reveals genetic variant for susceptibility to liver toxicity[J].Pharmacogenomics J,2002,2(5):327.
[38]Daly AK,Aithal GP,Leathart JB,et al.Genetic susceptibility to diclofenac-induced hepatotoxicity:contribution of UGT2B7,CYP2C8,and ABCC2 genotypes[J].Gastroenterology,2007,132(1):272.
[39]Noe J,Kullak-Ublick GA,Jochum W,et al.Impaired expression and function of the bile salt export pump due to three novel ABCB11 mutations in intrahepatic cholestasis [J].J Hepatol,2005,43(3):536.
[40]Choi JH,Ahn BM,Yi J,et al.MRP2 haplotypes confer differential susceptibility to toxic liver injury[J].Pharmacogenet Genomics,2007,17(6):403.
[41]Aithal GP,Ramsay L,DalyAK,et al.Hepatic adducts,circulating antibodies,and cytokine polymorphisms in patients with diclofenac hepatotoxicity[J].Hepatology,2004,39(5):1 430.
[42]Kannankeril P,Roden DM,Darbar D.Drug-induced long QT syndrome[J].Pharmacol Rev,2010,62(4):760.
[43]Arking DE,Pfeufer A,Post W,et al.A common genetic variant in the NOS1 regulator NOS1AP modulates cardiac repolarization[J].Nat Genet,2006,38(6):644.
[44]Newton-Cheh C,Eijgelsheim M,Rice KM,et al.Common variants at ten loci influence QT interval duration in the QTGEN Study[J].Nat Genet,2009,41(4):399.
[45]Nishio Y,Makiyama T,Itoh H,et al.D85N,a KCNE1 polymorphism,is a disease-causing gene variant in long QT syndrome[J].J Am Coll Cardiol,2009,54(9):812.
[46]K??b S,Crawford DC,Sinner MF,et al.A large candidate gene survey identifies the KCNE1 D85N polymorphism as a possible modulator of drug-induced torsades de pointes[J].Circ Cardiovasc Genet,2012,5(1):91.
[47]van Noord C,Aarnoudse AJ,Eijgelsheim M,et al.Calcium channel blockers,NOS1AP,and heart-rate-corrected QT prolongation[J].Pharmacogenet Genomics,2009,19(4):260.
[48]Jamshidi Y,Nolte IM,Dalageorgou C,et al.Common variation in the NOS1AP gene is associated with drug-induced QT prolongation and ventricular arrhythmia[J].J Am Coll Cardiol,2012,60(9):841.
(編輯:張元媛)
Relationship between Genetic Polymorphisms andAdverse Drug Reaction
SUN Xuelin1,HU Xin1,XU Qiaoling2(1.Dept.of Pharmacy,Beijing Hospital/National Center of Gerontology/ Beijing Key Laboratory for Assessment of Clinical Drugs Risk and Individual Application,Beijing 100730,China;2.Dept.of Pharmacy No.305 Hospital of PLA,Beijing 100730,China)
OBJECTIVE:To provide reference for precision medication in the clinic,preventing and reducing the occurrence of adverse drug reaction(ADR).METHODS:Foreign and domestic literatures about pharmacogenomics and ADR were collected to summarize the relationship of genetic polymorphisms with ADR.RESULTS&CONCLUSIONS:At present,many ADR-related genes are identified.The polymorphisms of human leukocyte antigen genes are associated with liver injury induced by diclofenac,amoxicillin clavulanate potassium and other drugs,and allergic reaction induced by carbamazepine,abacavir and other drugs.The polymorphisms of liver drug transporter 1B1 genes are associated with myopathy induced by statins.The polymorphisms of N-acetylase 2 genes are associated with liver injury induced by isoniazid.The polymorphisms of NOS1AP,SCN5A and KCNE1 genes are associated with drug-induced QT prolongation.Gene polymorphism study can provide a new reference for the prevention of ADR. Gene detection can be conducted before drug treatment to reduce the incidence of ADR.
Adverse drug reaction;Pharmacogenomics;Gene polymorphisms;Precise medication
R969.3
A
1001-0408(2016)32-4469-04
10.6039/j.issn.1001-0408.2016.32.02
衛生計生委保健局保健重點科研項目(No.W2016ZD01)
*主管藥師,博士。研究方向:臨床藥學。電話:010-85133632。E-mail:sunxuelin4543@bjhmoh.cn
主任藥師,博士。研究方向:醫院藥學。電話:010-66799176。E-mail:xuqiaoling305@163.com
(2016-07-23
2016-09-05)

