4 株E. coli O157:H7毒力基因檢測及其冷應激損傷
水新云,王虎虎,高 峰,江 蕓,(.南京師范大學金陵女子學院食品科學系,江蘇 南京 0097;.南京農業大學 國家肉品質量安全控制工程技術研究中心,江蘇 南京 0095)
4 株E. coli O157:H7毒力基因檢測及其冷應激損傷
水新云1,王虎虎2,高 峰2,江 蕓1,*
(1.南京師范大學金陵女子學院食品科學系,江蘇 南京 210097;2.南京農業大學 國家肉品質量安全控制工程技術研究中心,江蘇 南京 210095)
采用多重聚合酶鏈式反應對4 株大腸埃希氏菌O157:H7(Escherichia coli O157:H7)進行毒力特性評價,研究3 種常用選擇性生長基質對E.coli O157:H7的精確定量對比,篩選出的適宜選擇性培養基用于冷應激時菌體損傷的研究。結果顯示,菌株CICC 21530的stx1、stx2、eae基因均為陽性,NCTC 12900和牛肉分離菌1的eae呈現陽性,牛肉分離菌2三種毒力基因均為陰性,表明4 株菌的致病性不同;4 株測試菌在改良山梨醇麥康凱瓊脂(cefiximetellurite sorbitol macconkey agar,CT-SMAC)上計數均顯著低于胰蛋白胨大豆瓊脂(tryptose soya agar,TSA)上計數(P<0.05),而SMAC和改良伊紅美藍瓊脂(modified eosin methylene blue agar,mEMB)上的計數與TSA相比無顯著性差異,表明改良SMAC對正常菌體既有較強抑制作用,不適合用于E. coli O157:H7的精確計數,可以選用SMAC或mEMB;進一步以SMAC和mEMB作為選擇性培養基研究菌體在4 ℃冷應激時的損傷情況,結果表明冷藏過程中SMAC、mEMB及TSA上的菌數均逐漸下降,第10天時4 株菌均發生了一定程度的損傷或死亡。本方法可為食品安全中E. coli O157:H7的定量評估和風險控制提供科學依據。
大腸埃希氏菌O157:H7;毒力基因;半致死損傷;冷應激;選擇性培養基
大腸埃希氏菌O157:H7(Escherichia coli O157:H7)是一種重要的食源性人畜共患病原菌,已成為世界性的重要公共衛生問題之一[1-2]。E. coli O157:H7是腸出血性大腸埃希氏菌(enterohemorrhagic E. coli,EHEC)的一個主要血清型,可引起腹瀉、出血性腸炎,極易繼發溶血性尿毒綜合癥和血栓性血小板減少性紫癜兩種嚴重的并發癥[3]。該菌株最主要的毒力因子是志賀毒素(Stx),包括Stx1和Stx2。由eae基因編碼的分子質量在94~97 kD的緊密素對E. coli O157:H7在腸道定居起重要作用,其黏附作用導致特征性黏附-抹去損傷(attaching and effacing lesion,A/E)病變的發生。
食品在加工貯藏過程中,食品中的微生物可受到不利于其生長的多種脅迫環境,如加熱、冷凍、酸漬、鹽漬、防腐劑等。經這些脅迫條件處理后微生物存在3 種狀態:死亡細胞(致死的或不可逆的損傷)、未損傷細胞(正常細胞)和損傷細胞(應激的、亞致死的或可逆的損傷)[4]。研究表明,常見的食品加工處理工藝均會導致E. coli O157:H7等微生物產生亞致死損傷[5-7]。微生物的亞致死損傷狀態被認為是介于微生物死亡和正常生長的中間狀態。研究表明,4 ℃冷藏條件也會對細菌造成冷應激,從而可能導致菌體發生應激損傷[8]。損傷菌失去了一些內在特性,如致病特性、在選擇性培養基上的生長能力,進而在食品安全的常規檢測中被低估或忽視。然而,已有研究表明,處于亞致死損傷狀態的食源性致病菌在一定條件下可以修復,最終恢復正常生理特性,包括致病性[9]。因而,這部分亞致死損傷微生物的存在給食品的安全性以及貨架期造成極大隱患。
亞致死損傷菌在非選擇性培養基上可形成菌落而在選擇性培養基上不能形成菌落,因此非選擇性培養基上菌落數與選擇性培養基上菌落數的差值即可反映亞致死損傷菌的數量[10]。對食源性致病菌進行準確計數是食品安全性評估的重要前提。關于E. coli O157:H7亞致死損傷菌計數的選擇性培養基,目前報道并不一致。有的應用山梨醇麥康凱瓊脂(sorbitol macconkey agar,SMAC)[11-13],或應用改良山梨醇麥康凱瓊脂,即山梨醇麥康凱瓊脂添加頭孢克肟-亞碲酸鉀(cefiximetellurite SMAC,CT-SMAC)[8,14-15],也有的應用改良伊紅美藍瓊脂(modified eosin methylene blue agar,mEMB)[16]。此外,還有研究應用大腸菌群顯色培養基、乳糖膽鹽瓊脂,或胰蛋白胨大豆瓊脂(tr yptose soya agar,TSA)中添加一定濃度NaCl作為選擇性培養基來檢測E. coli O157:H7損傷情況[17-18]。
因此本實驗首先應用多重聚合酶鏈式反應(polymerase chain reaction,PCR)方法對實驗用4 株E. coli O157:H7進行stx1、stx2、eae毒力基因的檢測,以了解其致病性;進一步選用常見的3 種選擇性培養基SMAC、CT-SMAC、mEMB對其新鮮菌液進行培養計數,以非選擇性培養基TSA為對照,擬篩選出合適的選擇性培養基;在此基礎上研究冷應激條件下E. coli O157:H7的損傷情況。本實驗將為E. coli O157:H7的精確計數提供合理手段,為食品安全危險性評估提供科學依據。
1 材料與方法
1.1 菌種與試劑
本實驗所用E. coli O157:H7共4 株,1 株(編號CICC 21530)由南京農業大學國家肉品質量安全控制工程技術研究中心惠贈,菌種原購自中國工業微生物菌種保藏管理中心;1 株(編號NCTC12900) 廣東環凱微生物科技有限公司;2 株由南京農業大學江蘇省動物源食品生產與安全保障重點實驗室惠贈,均分離自牛肉。
胰蛋白胨大豆肉湯(tryptic soy broth,TSB)、TSA、SMAC、亞碲酸鉀、P-09噻孢霉素 北京路橋技術有限公司;mEMB 山東青島捷世康生物科技有限公司;TaqTM(with Mg2+free buffer)、100 bp DNA Marker 大連寶生物(TaKaRa)工程有限公司;瓊脂糖 北京天根生化科技有限公司;4S Red染料 上海生工生物科技有限公司;所有引物均由上海英俊生物技術有限公司合成。
1.2 儀器與設備
Mastercycler ep personal PCR儀 德國Enperndorf公司;GelDoc 2000 system凝膠成像儀 美國BioRad公司;TCL-16G高速離心機 上海安亭儀器廠;DYY-2C瓊脂糖凝膠電泳儀 上海天能科技有限公司;SZX超凈工作臺 上海浦東躍欣儀器廠;DHP-9052電熱恒溫培養箱 上海一恒科技公司。
1.3 方法
1.3.1 菌株的培養
將4 株E. coli O157:H7分別經TSA劃線培養,37 ℃培養24 h,挑取單菌落分別接種于3 mL TSB中,37 ℃培養18 h,再轉接至100 mL TSB中,37 ℃培養18 h。得到菌液置于4 ℃,不同時間取樣進行傳統培養計數。
1.3.2 細菌基因組DNA提取
將4 株實驗用E. coli O157:H7、E. coli Sakai(陽性對照菌)和DH5α(陰性對照菌)共6株菌分別用TSA劃線培養24 h。挑一環單菌落于150 μL雙蒸水,放于滅菌1.5 mL離心管中,徹底渦旋,使其充分混勻。99 ℃水浴處理10 min,冰浴2 min,12 000×g離心2 min。吸取上清液至另一離心管中即為細菌基因組DNA,-20 ℃凍存備用。
1.3.3 多重PCR
采用多重PCR方法對4 株實驗用E. coli O157:H7進行stx1、stx2、eae毒力基因檢測[19-20]。對應stx1、stx2、eae毒力基因的3 對引物分別是:stx1F(5’-ACA CTG GAT GAT CTC AGT GG-3’),stx1R(5’-CTG AAT CCC CCT CCA TTA TG-3’);stx2F(5’-GGC ACT GTC TGA AAC TGC TCC-3’),stx2R(5’-TCG CCA GTT ATC TGA CAT TCT G-3’);eaeF(5’-GTG GCG AAT GGC ACT GAG ACT-3’),eaeR(5’-CCC CAT TCT TTT TCA CCG TCG-3’)。PCR反應體系(20 μL):10×PCR buffer 2 μL,dNTP 1.4 μL,MgCl21.2 μL,6條引物(10 μmol/L)各0.2 μL,Taq polymerase 0.1 μL,DNA模板1 μL。PCR反應程序:94 ℃預變性4 min;然后90 ℃變性45 s,55 ℃退火30 s,72 ℃退火50 s,進行30 個循環;最后72 ℃延伸8 min。PCR產物經1.4%瓊脂糖凝膠電泳檢測。
1.3.4 傳統培養計數
取新鮮TSB菌液及4 ℃放置一定時間的菌液,經過系列10 倍梯度稀釋,選擇3 個合適稀釋梯度,取100 μL菌液至非選擇性培養基TSA和選擇性培養基CTSMAC、SMAC、mEMB上,采用平板涂布法,37 ℃培養24 h,對E. coli O157:H7進行菌落計數,并進一步計算損傷率:
損傷率/%=[(非選擇性培養基上的平均菌落數-選擇性培養基上的平均菌落數)/非選擇性培養基上的平均菌落數]×100
式中:非選擇培養基TSA平板上的菌落數為未損傷、損傷和已繁殖的菌數,選擇性培養基上的菌落數為未損傷、已修復、已繁殖的菌落數。
1.4 統計分析
數據采用SPSS V 17.0進行數據分析處理,采用方差分析進行單因素方差分析中的Duncan’s多重比較檢驗(P<0.05),數據均以
2 結果與分析
2.1 菌株毒力基因的檢測
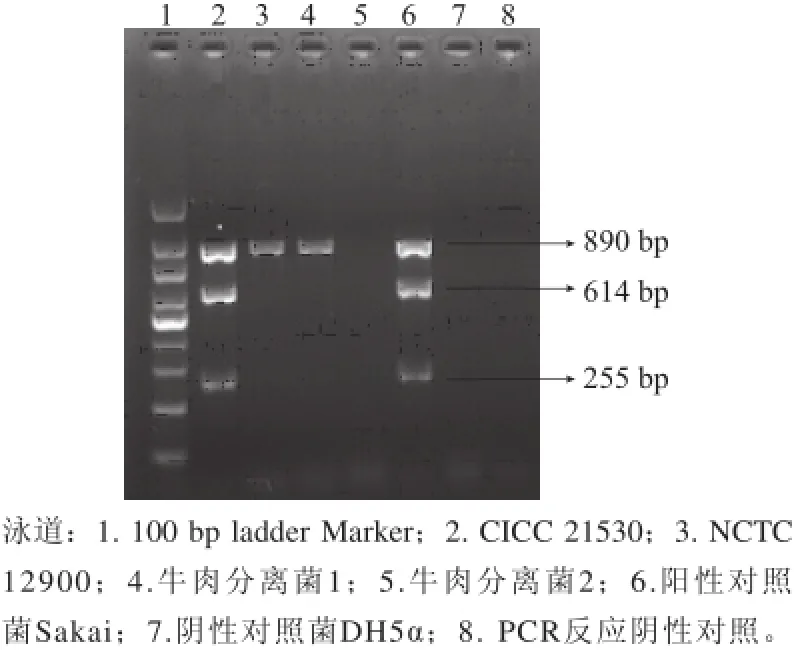
圖1 E. coli三種毒力基因多重PCR電泳圖Fig.1 Electrophoresis profile for three virulence genes of E. coli amplified by multiplex PCR
如圖1所示,陽性對照菌Sakai成功擴增出3 條陽性條帶,分別是eae基因條帶(890 bp)、stx1基因條帶(614 bp)、stx2基因條帶(255 bp)。本實驗所用菌株結果顯示,CICC 21530三種毒力基因均陽性,NCTC 12900只有eae基因陽性,牛肉分離菌1 eae基因陽性,牛肉分離菌2三種毒力基因均為陰性。結果表明,4 株E. coli O157:H7所帶毒力基因不盡相同,表明它們的致病性是不同的。
2.2 選擇性培養基的篩選
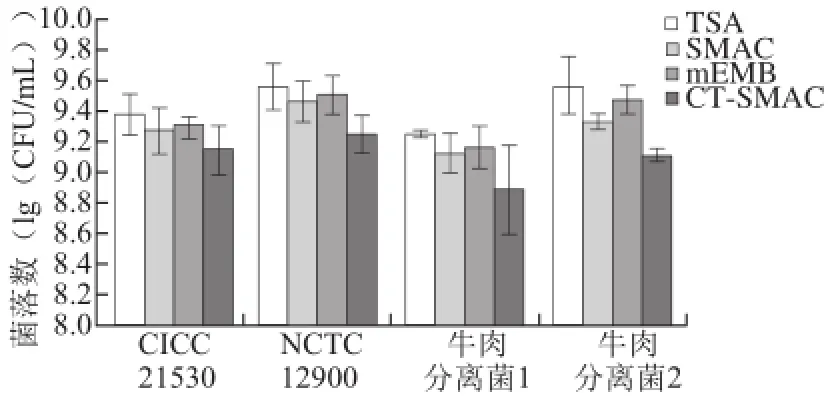
圖2 2 E. coli coli O157:H7新鮮菌液在不同培養基上的菌落數Fig.2 Counts of fresh E. coli culture on different medium plates
如圖2所示,本實驗應用非選擇性培養基TSA和選擇性培養基CT-SMAC、SMAC、mEMB對新鮮菌液進行菌落計數,與TSA計數相比,4 株菌在選擇性培養基CT-SMAC上的菌落數CICC 21530、NCTC 12900、牛肉分離菌1、牛肉分離菌2分別下降了0.26、0.32、0.36、0.45 (lg(CFU/mL)),進一步統計分析表明4 株菌在CT-SMAC上的計數均顯著低于TSA上的計數(P<0.05),表明CT-SMAC對正常菌可產生顯著的抑制作用,會導致計數結果偏差;4 株菌在選擇性培養基SMAC上的計數與TSA計數相比下降了約0.1~0.2(lg(CFU/mL)),而在mEMB上的計數與TSA計數相比下降數<0.1(lg(CFU/mL)),進一步統計分析表明4 株菌在TSA、SMAC、mEMB上的菌落數無顯著性差異(P>0.05),表明這兩種選擇性培養基對正常菌抑制較小。本實驗結果表明,可以選用SMAC或mEMB用于后續實驗中E. coli O157:H7損傷菌的選擇性培養計數,而CT-SMAC并不合適。
2.3 冷應激條件下E. coli O157:H7損傷的研究
本實驗進一步將菌液放置4 ℃不同時間,應用上述篩選的適宜選擇性培養基SMAC和mEMB進行培養計數來觀察冷應激條件下E. coli O157:H7的損傷情況,結果見圖3。冷藏過程中4株菌在SMAC、mEMB及TSA上的計數整體呈下降趨勢,在放置12 h時即低于初始菌數。放置10 d時與初始菌數相比,TSA上菌數降低了0.2~0.5(lg(CFU/mL))。統計分析發現,菌株CICC 21530、NCTC 12900以及牛肉分離菌2在0 d和10 d在TSA上的計數結果有顯著性差異(P<0.05),表明4 ℃放置10 d時有細菌發生了一定程度的死亡。
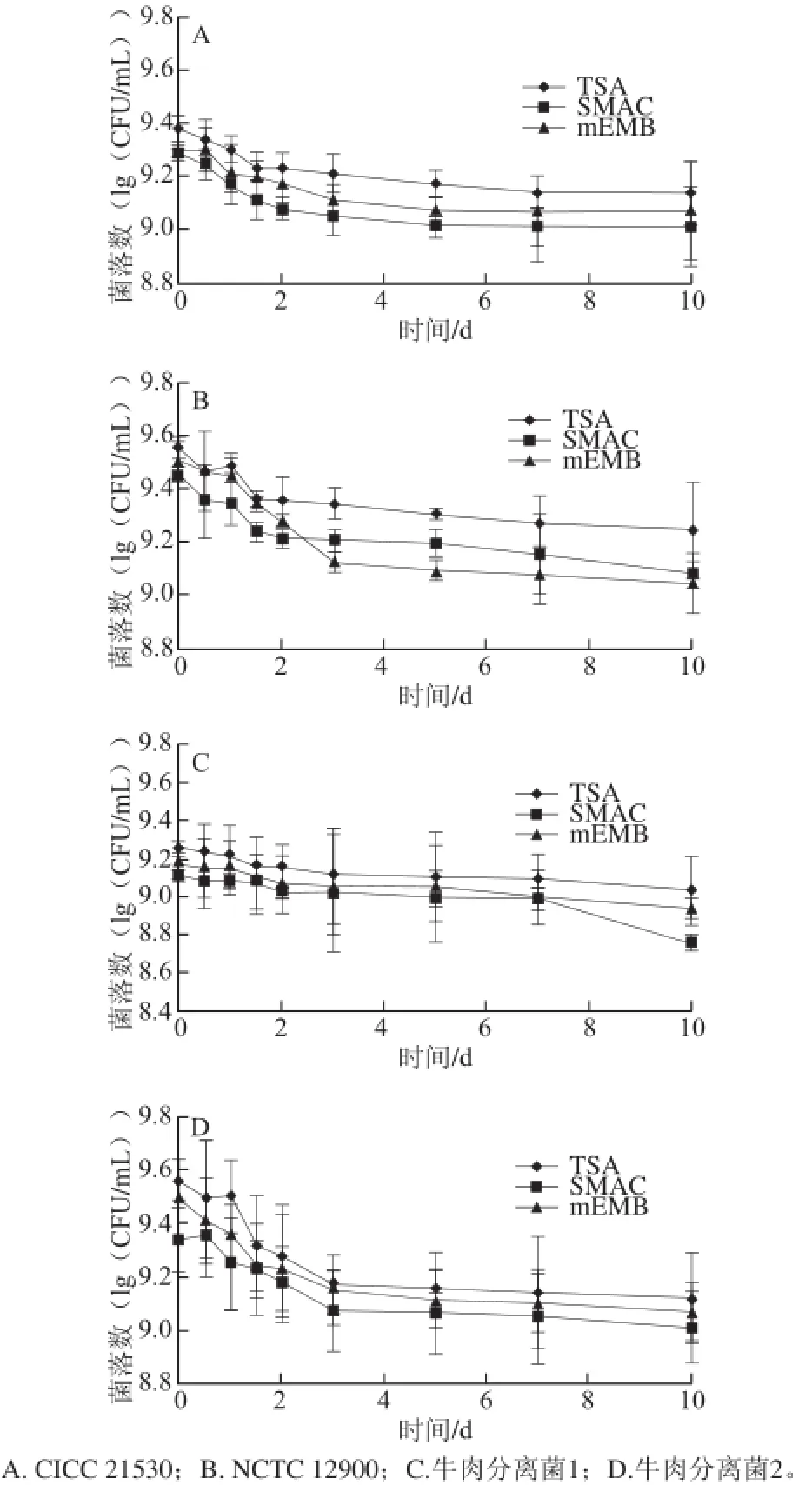
圖3 3 E. coli coli O157:H7菌液放置4 ℃條件下在不同培養基上的菌落數Fig.3 Counts of E. coli during storage in 4 ℃ cultured on different medium plates
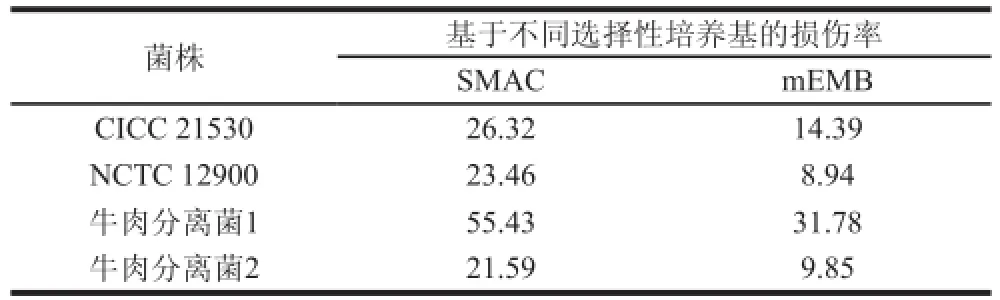
表1 4 株E. coli O157:H7 TSB培養液4 ℃放置10 d時的損傷率Table 1 Injury rates of four E. coli strains cultured in TSB stored in 4 ℃ for 10 days %
進一步計算10 d時的損傷率,結果如表1所示,基于SMAC或mEMB上的計數,各菌損傷率不盡相同,其中牛肉分離菌1在SMAC、mEMB上的損傷率均較高。實驗結果表明,新鮮菌液置于4 ℃冰箱放置一段時間后,各菌發生了不同程度的損傷,甚至死亡。
3 討 論
E. coli O157:H7的毒力基因主要有stx1、stx2、rfbE、flic、hly、ehxA等[21-22],E. coli O157:H7的致病機理并不僅僅因為單一的毒力因子,更是多種毒力因子共同作用的結果。其最主要的毒力因子是Stx,包括Stx1和Stx2,此外還有由eae基因編碼的緊密素。多重PCR是在常規PCR基礎上發展起來的一種PCR擴增技術,是在同一反應體系中加入多對引物同時擴增多條目的DNA片段的方法,利用擴增片段在瓊脂糖凝膠電泳上的位置差異來區分和鑒定,該方法節省模板DNA、節省時間和費用[23]。本實驗首先應用多重PCR方法對4 株實驗用E. coli O157:H7進行了stx1、stx2、eae毒力基因的檢測,結果表明,4 株E. coli O157:H7所帶毒力基因不盡相同,表明它們的致病性是不同的。
已有較多研究應用選擇性培養基與非選擇性培養基進行傳統培養來計數某特定食源性致病菌的半致死損傷菌,如單增李斯特菌、沙門氏菌、空腸彎曲菌、金黃色葡萄球菌以及E. coli O157:H7等。關于損傷性E. coli O157:H7培養計數方面,常見的報道有應用CTSMAC[8,14-15]、SMAC[11-13]、mEMB[16]等作為選擇性培養基。這些選擇性培養基中含有一些抑菌成分,使得E. coli O157:H7可以生長而抑制其他細菌的生長。我國食品衛生微生物學檢驗國家標準采用CT-SMAC來分離培養E. coli O157:H7[24]。但本實驗對E. coli O157:H7新鮮菌液進行菌落計數時發現,CT-SMAC對正常菌體產生了顯著的抑制作用,導致計數偏低,實驗結果表明,CT-SMAC可用于E. coli O157:H7的分離培養,但并不適合用于其精確計數,因而不適合用于后續實驗E. coli O157:H7損傷菌的計數。Jasson等[8]研究也發現CT-SMAC對E. coli O157:H7正常菌會產生一定的抑制作用。而選擇性培養基SMAC、mEMB上E. coli O157:H7計數結果亦低于TSA上的計數,但無顯著差異,表明這兩種選擇性培養基對正常菌抑制較小,可用于其精確計數。因此本實驗進一步應用SMAC和mEMB作為選擇性培養基研究了冷應激時E. coli O157:H7的損傷情況,結果發現4 ℃冷藏10 d時4 株菌均發生了一定程度的損傷。Jasson等[8]研究表明冷應激一段時間后E. coli O157:H7損傷率顯著增加,而空腸彎曲菌和單增李斯特菌的損傷率則無顯著性變化。本實驗還發現,4 株菌損傷程度不盡相同,其中牛肉分離菌1較為敏感,表明冷應激損傷與菌株有關。
4 結 論
本實驗采用多重P C R方法,確定4 株實驗用E. coli O157:H7攜帶毒力基因stx1、stx2、eae不盡相同,表明它們的致病性是不同的。E. coli O157:H7的選擇性培養基,CT-SMAC對正常菌有明顯的抑制作用,不適合用于E. coli O157:H7的精確計數,而SMAC和mEMB抑制小,可以使用。冷應激損傷實驗發現,4 ℃冷藏10 d時4 株菌均發生了不同程度的損傷,甚至死亡。本實驗確定了4 株不同毒力基因的E. coli O157精確計數的選擇性培養基,可為食品安全中E. coli O157的定量評估和風險控制提供科學依據。
[1] ATKINSON R, JOHNSON G, ROOT T, et al. Importance of culture confirmation of Shiga toxin-producing Escherichia coli infection as illustrated by outbreaks of gastroenteritis: New York and North Carolina, 2005[J]. Morbidity and Mortality Weekly Report, 2006, 55(38): 1042-1045. DOI:10.1037/e550322006-003.
[2] MAGWEDERE K, DANG H A, MILLS E W, et al. Incidence of Shiga toxin-producing Escherichia coli strains in beef, pork, chicken, deer, boar, bison, and rabbit retail meat[J]. Journal of Veterinary Diagnostic Investigation, 2013, 25(2): 254-258. DOI:10.1177/1040638713477407.
[3] PAGE A V, LILES W C. Enterohemorrhagic Escherichia coli infections and the hemolytic-uremic syndrome[J]. The Medical Clinics of North America, 2013, 97(4): 681-695. DOI:10.1016/ j.mcna.2013.04.001.
[4] NORIEGA E, VELLIOU E, van DERLINDEN E, et al. Effect of cell immobilization on heat-induced sublethal injury of Escherichia coli, Salmonella typhimurium and Listeria innocua[J]. Food Microbiology, 2013, 36(2): 355-364. DOI:10.1016/j.fm.2013.06.015.
[5] PILAVTEPE-?ELIK M, BUZRUL S, ALPAS H, et al. Multi-pulsed high hydrostatic pressure treatment for inactivation and injury of Escherichia coli[J]. Journal of Consumer Protection and Food Safety, 2011, 6(3): 343-348. DOI:10.1007/s00003-010-0647-2.
[6] GUYOT S, POTTIER L, FERRET E, et al. Physiological responses of Escherichia coli exposed to different heat-stress kinetics[J]. Archives of Microbiology, 2010, 192(8): 651-661. DOI:10.1007/s00203-010-0597-1.
[7] BACCUS-TAYLOR G S, FALLOON O C, HENRY N. Pressure resistance of cold shocked Escherichia coli O157:H7 in ground beef, beef gravy and peptone water[J]. Journal of Applied Microbiology, 2015, 118(6): 1521-1529. DOI:10.1111/jam.12794.
[8] JASSON V, UYTTENDAELE M, RAJKOVIC A, et al. Establishment of procedures provoking sub-lethal injury of Listeria monocytogenes, Campylobacter jejuni and Escherichia coli O157 to serve method performance testing[J]. International Journal of Food Microbiology, 2007, 118(3): 241-249. DOI:10.1016/j.ijfoodmicro.2007.07.016.
[9] RITZ M, PILET M F, JUGIAU F, et al. Inactivation of Salmonella typhimurium and Listeria monocytogenes using high-pressure treatments: destruction or sublethal stress[J]. Letters in Applied Microbiology, 2006, 42(4): 357-362. DOI:10.1111/j.1472-765X.2006.01868.x.
[10] BESSE N G. Influence of various environmental parameters and of detection procedures on the recovery of stressed L. monocytogenes: a review[J]. Food Microbiology, 2002, 19(2/3): 221-234. DOI:10.1006/ fmic.2001.0448.
[11] SMITH A R, ELLISON A L, ROBINSON A L, et al. Enumeration of sublethally injured Escherichia coli O157:H7 ATCC 43895 and Escherichia coli strain B-41560 using selective agar overlays versus commercial methods[J]. Journal of Food Protection, 2013, 76(4): 674-679. DOI:10.4315/0362-028X.JFP-12-363.
[12] PATEL J, MACARISIN D, SANGLAY G, et al. Inactivation and injury of pathogens on intact beef treated with hydrodynamic pressure[J]. Innovative Food Science and Emerging Technologies, 2012, 14(1): 38-45. DOI:10.1016/j.ifset.2011.08.002.
[13] HONG S, MENDON?A A F, DARABA A, et al. Radiation resistance and injury in starved Escherichia coli O157:H7 treated with electronbeam irradiation in 0.85% saline and in apple juice[J]. Foodborne Pathogens and Disease, 2014, 11(11): 900-906. DOI:10.1089/ fpd.2014.1782.
[14] RODRIGUEZ E, ARQUES J L, NU?EZ M, et al. Combined effect of high-pressure treatments and bacteriocin-producing lactic acid bacteria on inactivation of Escherichia coli O157:H7 in raw-milk cheese[J]. Applied and Environmental Microbiology, 2005, 71(7): 3399-3404. DOI:10.1128/AEM.71.7.3399-3404.2005.
[15] YOSHITOMI K J, ZAPATA R, JINNEMAN K C, et al. Recovery of E. coli O157 strains after exposure to acidification at pH 2[J]. Letters in Applied Microbiology, 2012, 54(6): 499-503. DOI:10.1111/j.1472-765X.2012.03250.x.
[16] YUSTE J, CAPELLAS M, DANIEL Y C, et al. Inactivation and sublethal injury of foodborne pathogens by high pressure processing: evaluation with conventional media and thin agar layer method[J]. Food Research International, 2004, 37(9): 861-866. DOI:10.1016/ j.foodres.2004.05.002.
[17] GABRIEL A A, NAKANO H. Influences of simultaneous physicochemical stress exposures on injury and subsequent responses of E. coli O157:H7 to resuscitative and inactivative challenges[J]. International Journal of Food Microbiology, 2010, 139(3): 182-192. DOI:10.1016/j.ijfoodmicro.2010.02.028.
[18] YANG Baowei, SHI Ying, XIA Xiaodong, et al. Inactivation of foodborne pathogens in raw milk using high hydrostatic pressure[J]. Food Control, 2012, 28(2): 273-278. DOI:10.1016/ j.foodcont.2012.04.030.
[19] PATON A W, PATON J C. Direct detection and characterization of Shiga toxigenic Escherichia coli by multiplex PCR assays for stx1, stx2, eae, ehxA, and saa[J]. Journal of Clinical Microbiology, 2002, 40(1): 271-274. DOI:10.1128/JCM.40.1.271-274.2002.
[20] SVOBODA A L, DUDLEY E G, DEBROY C, et al. Presence of Shiga toxin-producing Escherichia coli O-group in small and verysmall beef-processing plant and resulting ground beef detected by a multiplex polymerase chain reaction assay[J]. Foodborne Pathogens and Disease, 2013, 10(9): 789-795. DOI:10.1089/fpd.2012.1445.
[21] SON I, BINET R, LIN A, et al. Identification of five Shiga toxinproducing Escherichia coli genes by luminex microbead-based suspension array[J]. Journal of Microbiological Methods, 2015, 111: 108-110. DOI:10.1016/j.mimet.2015.02.008.
[22] BUGAREL M, BEUTIN L, MARTIN A, et al. Micro-array for the identification of Shiga toxin-producing Escherichia coli (STEC) seropathotypes associated with hemorrhagic colitis and hemolytic uremic syndrome in humans[J]. International Journal of Food Microbiology, 2010, 142(3): 318-329. DOI:10.1016/ j.ijfoodmicro.2010.07.010.
[23] S?NCHEZ S, LLORENTE M T, ECHEITA M A, et al. Development of three multiplex PCR assays targeting the 21 most clinically relevant serogroups associated with Shiga toxin-producing E. coli infection in humans[J]. PLoS One, 2015, 10(1): e0117660. DOI:10.1371/journal. pone.0117660.
[24] GB/T 4789.36—2008 食品衛生微生物學檢驗: 大腸埃希氏菌O157:H7/NM檢驗[S]. 北京: 中國標準出版社, 2008.
Detection of Virulence Genes of Four Escherichia coli O157:H7 Strains and Cold Stress Injury in the Bacteria
SHUI Xinyun1, WANG Huhu2, GAO Feng2, JIANG Yun1,*
(1. Department of Food Science, Ginling College, Nanjing Normal University, Nanjing 210097, China; 2. National Center of Meat Quality and Safety Control, Nanjing Agricultural University, Nanjing 210095, China)
In this study, the virulence genes of four Escherichia coli O157:H7 strains were detected by multiplex polymerase chain reaction (PCR). Then three selective media were compared for enumeration of E. coli O157:H7 and the injury of E. coli O157:H7 under cold stress was also investigated with the appropriate selective medium. The results showed that CICC 21530 strain was positive for all the three genes stx1, stx2 and eae, while NCTC 12900 and one beef isolate were eae-positive, and two other isolates were negative for all the three virulence genes, which indicated that the pathogenicity of four strains was different. The counts of all four strains on three selective media were lower than those on tryptose soya agar (TSA) (P < 0.05) but there was no significant difference between them except for cefixime-tellurite sorbitol macconkey agar (CT-SMAC), which indicated that CT-SMAC was not suitable for counting E. coli O157:H7 due to the greater inhibition on normal bacteria, while SMAC or mEMB could be used. Then the injury of E. coli O157:H7 stored at 4 ℃ (cold stress) was investigated with each of these two selective media. The results showed that the counts on SMAC, mEMB, or TSA were decreased during the whole storage and some bacteria were injured or killed on the 10thday. In the current study, the selective media for accurate enumeration of four E. coli O157:H7 strains with different virulence genes were finalized, which would provide the scientific basis for quantitative assessment and risk control of E. coli O157:H7 in food safety evaluation.
Escherichia coli O157:H7; virulence genes; sub-lethal injury; cold stress; selective media
10.7506/spkx1002-6630-201604031
TS201.3
A
1002-6630(2016)04-0176-05
水新云, 王虎虎, 高峰, 等. 4 株E. coli O157:H7毒力基因檢測及其冷應激損傷[J]. 食品科學, 2016, 37(4): 176-180.
DOI:10.7506/spkx1002-6630-201604031. http://www.spkx.net.cn
SHUI Xinyun, WANG Huhu, GAO Feng, et al. Detection of virulence genes of four Escherichia coli O157:H7 strains and cold stress injury in the bacteria[J]. Food Science, 2016, 37(4): 176-180. (in Chinese with English abstract) DOI:10.7506/ spkx1002-6630-201604031. http://www.spkx.net.cn
2015-04-05
國家自然科學基金面上項目(31371861)
水新云(1989—),女,碩士研究生,研究方向為肉品安全與質量控制。E-mail:951619906@qq.com
*通信作者:江蕓(1971—),女,教授,博士,研究方向為肉品安全與質量控制。E-mail:jiangyun@njnu.edu.cn

