巴西橡膠樹鈣調蛋白基因的克隆和表達分析
肖小虎 隋金蕾 戚繼艷 陽江華 秦云霞 唐朝榮

摘 要 以擬南芥和楊樹的鈣調蛋白氨基酸序列為探針,對橡膠樹轉錄組數據庫進行搜索,并設計引物進行PCR擴增,得到7個橡膠樹鈣調蛋白基因的cDNA序列,命名為HbCaM1-7。序列分析結果發現,HbCaM1-7的CDS序列均為450 bp,編碼149個氨基酸,分子量為16.8 ku,等電點為3.95。其中,HbCaM1-6間氨基酸同源性為100%,HbCaM7與其他成員之間的氨基酸同源性為98%。在基因結構組織方面,HbCaM1-7均為一個內含子,且內含子的插入位置相同,但內含子的長度明顯不同。從氨基酸序列同源性和進化關系分析結果可看出,鈣調蛋白基因家族在進化上非常保守。在表達方面,HbCaM1-6在各組織中均有表達,除了HbCaM3在根中表達豐度相對較高,其他成員均在膠乳中表達豐度相對較高,而HbCaM7在各組織中的表達均比較低;乙烯利處理后,HbCaM2-7的表達均有一定的上升趨勢,而在葉片發育過程中HbCaM1-5圴呈明顯下調表達。由此可見,橡膠樹鈣調蛋白與橡膠樹葉片發育、膠乳乙烯信號通路具有一定的相關性。
關鍵詞 巴西橡膠樹 ;鈣調蛋白 ;基因克隆 ;基因家族 ;表達分析
中圖分類號 S794.1 文獻標識碼 A Doi:10.12008/j.issn.1009-2196.2016.07.009
在植物生長發育的過程中,鈣離子作為第二信使扮演著至關重要的角色。鈣調蛋白(Calmodulin,CaM)是一類存在于動植物真核細胞內和鈣離子相結合的小分子蛋白[1],也是生物細胞內功能和分布最廣泛的高度保守的蛋白之一[2]。鈣調蛋白有4個Ca+結合域,多數CaM氨基酸序列的長度為148個,細胞內鈣離子濃度升高,使鈣調蛋白的鈣離子結合位點被占據,因而改變了其構象,并促進其與細胞內靶酶的結合,由此改變細胞內的代謝活動。由于鈣調蛋白重要的調控功能引起了人們的重視,鈣調蛋白基因在動物[3-4]、真菌[5]和原生動物[6]中都已被分離和克隆。在植物界,迄今為止已成功克隆了擬南芥[7]、水稻[8]、大麥[9]、苜蓿[10]、香蕉[11]等物種的鈣調蛋白基因,并對其進化和表達進行了分析。在橡膠樹中,僅有陳鑫等[12]利用RACE克隆了一個鈣調蛋白基因HbCAM1,并利用RT-PCR技術對該基因在不同組織和乙烯利處理膠乳中的表達進行了初步分析。隨著橡膠樹基因組測序的完成,使人們能夠對整個橡膠樹鈣調蛋白基因家族進行全面系統的分析。本研究從橡膠樹轉錄組和基因組數據庫中搜索并克隆得到7個鈣調蛋白基因,并從基因結構、系統進化和表達模式幾方面對這些家族成員進行了全面系統的分析。研究結果將有助于深入了解鈣調蛋白基因家族成員在橡膠樹生長發育和脅迫應答調控方面的重要功能。
1 材料與方法
1.1 材料
1.1.1 植物材料
本實驗室Solexa測序所用的材料是巴西橡膠樹(Hevea brasiliensis)熱研7-33-97,其中膠乳、樹皮、樹葉、種子、雌花和雄花均來自中國熱帶農業科學院試驗場三隊的正常割膠橡膠樹(開割2 a以上),根來自于中國熱帶農業科學院橡膠研究所種質資源圃華于偉老師贈送的熱研7-33-9 組培苗;不同發育時期的橡膠樹葉片來自于中國熱帶農業科學院橡膠所種質資源圃,為一年生的熱研7-33-97嫁接苗;乙烯利處理的材料來自于海南省儋州市中國熱帶農業科學院試驗場三隊,品系為熱研7-33-97,正常開割樹(3天1刀,不涂乙烯利刺激),用1.5%乙烯利在不同時間處理橡膠樹,相應4個時間點處理為0、3、12和24 h。
1.1.2 菌株與試劑
反轉錄試劑盒購自Fementas公司;pMD18-T載體和Taq DNA聚合酶均購自Takara公司;引物合成和測序由華大生物技術有限公司完成;DNA凝膠回收試劑盒購自Axygen公司;其它生化試劑和常規試劑均為進口或國產分析純試劑。
1.2 方法
1.2.1 不同組織RNA的提取與cDNA合成
橡膠樹膠乳RNA的提取參照Tang等[13]的方法;除膠乳以外其他組織的RNA提取方法參照Kiefer等[14]的方法;cDNA第一條鏈的合成采用反轉錄試劑盒,操作步驟按照Fementas公司產品說明書進行。
1.2.2 cDNA全長和基因組序列的克隆
利用擬南芥和楊樹的鈣調蛋白氨基酸序列,通過本地BLAST搜索本實驗室EST數據庫,得到7個鈣調蛋白基因的cDNA片段。然后設計特異性引物進行PCR擴增(表1),具體反應體系如下:模板(膠乳cDNA)0.5 μL、10×PCR Buffer 2 μL、Taq plus DNA Polymerase(5 U/L) 0.3 μL、dNTPs(2.5 mmol/L) 1.6 μL、上下游引物各1 μL、ddH2O 13.6 μL,共20 μL體系。反應程序:94℃預變性5 min;94℃變性30 s,60℃退火30 s,72℃延伸60 s,35個循環;72℃延伸10 min。擴增完成后,用1.2%的瓊脂糖凝膠電泳檢測,回收目的片段,并連接到pMD18-T載體上,最后送公司測序。
1.2.3 生物信息學分析
利用NCBI在線軟件ORF finder(http://www.ncbi.nlm.nih.gov/gorf/gorf.html)進行ORF閱讀框預測,利用生物軟件DNAMAN對橡膠樹和擬南芥的CaM氨基酸序列進行多序列比對。利用在線軟件GSDS2.0(http://gsds.cbi.pku.edu.cn/)分析鈣調蛋白基因的外顯子/內含子組織結構。利用MEGA 6.0軟件構建系統進化樹,采用Neighbor-Joining方法進行分子系統學分析,并進行1 000次bootstrap統計學檢驗。
1.2.4 基因的表達模式分析
利用高通量測序數據[15](Solexa轉錄組測序數據)對橡膠樹鈣調蛋白基因在不同組織、不同葉片發育時期和乙烯利處理下的表達進行分析。具體分析流程包括:對原始數據去除低質量序列,利用程序RSEM進行表達分析[16]和數據處理作圖3個部分,其中前2步在本地服務器完成。
2 結果與分析
2.1 橡膠樹鈣調蛋白基因的克隆與序列分析
本研究以擬南芥和楊樹鈣調蛋白氨基酸序列為探針,利用tblastn搜索本實驗室的EST數據庫,分離并克隆得到了7個橡膠樹鈣調蛋白基因的cDNA序列,命名為HbCaM1-7(對應序列的NCBI登陸號為:HbCaM1,AJJZ010938311.1;HbCaM2,AJJZ010269732.1;HbCaM3,AJJZ010929368.1;HbCaM4,AJJZ010665409.1;HbCaM5,AJJZ010127638.1;HbCaM6,AJJZ010231096.1;HbCaM7,AJJZ010372997.1)。通過NCBI在線軟件CDD分析,結果發現這7個基因均具有鈣調蛋白基因所特有的保守結構域(圖1)。經序列分析結果發現,HbCaM1-7的CDS序列均為450 bp,編碼149個氨基酸,分子量為16.8 ku,等電點為3.95。其中,HbCaM1-6之間氨基酸同源性為100%,HbCaM7與其他成員之間的氨基酸同源性為98%,各成員之間CDS序列同源性為80.4%~96.4%。
2.2 基因結構和進化分析
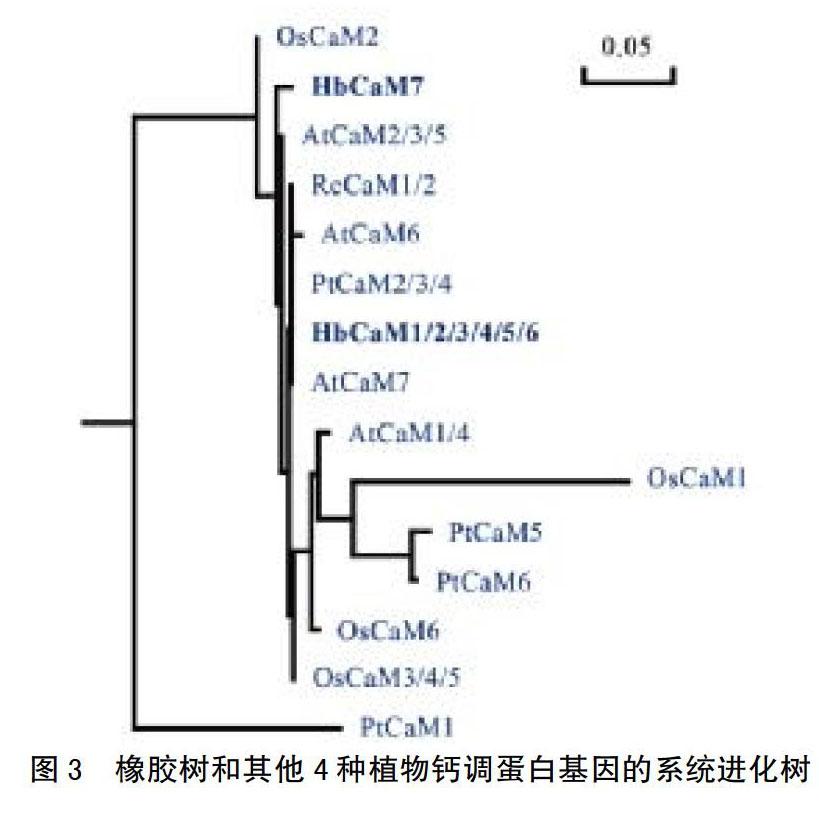
利用在線軟件GSDS 2.0,對橡膠樹鈣調蛋白基因的外顯子/內含子組織結構進行分析(圖2)。分析結果發現,各家族成員在內含子數目和相對位置都非常一致,而內含子的長度卻存在一定的差異。為了比較橡膠樹和其他植物鈣調蛋白在進化上的相互關系,選取了楊樹(Populus trichocarpa, Pt)、擬南芥(Arabidopsis thaliana,At)、水稻(Oryza sativa,Os)、蓖麻(Ricicus communis,Rc)及7條橡膠樹(Hevea brasiliensis,Hb)鈣調蛋白氨基酸序列,利用軟件MEGA 6.0采用Neighbor-Joining法構建進化樹,并進行1 000次bootstrap統計學檢驗(圖3)。從圖3中可看出,很多家族成員聚在一條直線上,說明這些成員之間的親緣關系比較近,這和植物鈣調蛋白氨基酸序列的高度保守性是一致的,例如橡膠樹中HbCaM1-6雖然核苷酸序列存在差異,但氨基酸序列卻是完全一致的,類似的還有擬南芥中AtCaM2-5、楊樹中的PtCaM2-4等。植物鈣調蛋白在進化上的保守性也說明其在功能上的重要性。
2.3 橡膠樹鈣調蛋白基因家族成員的表達分析
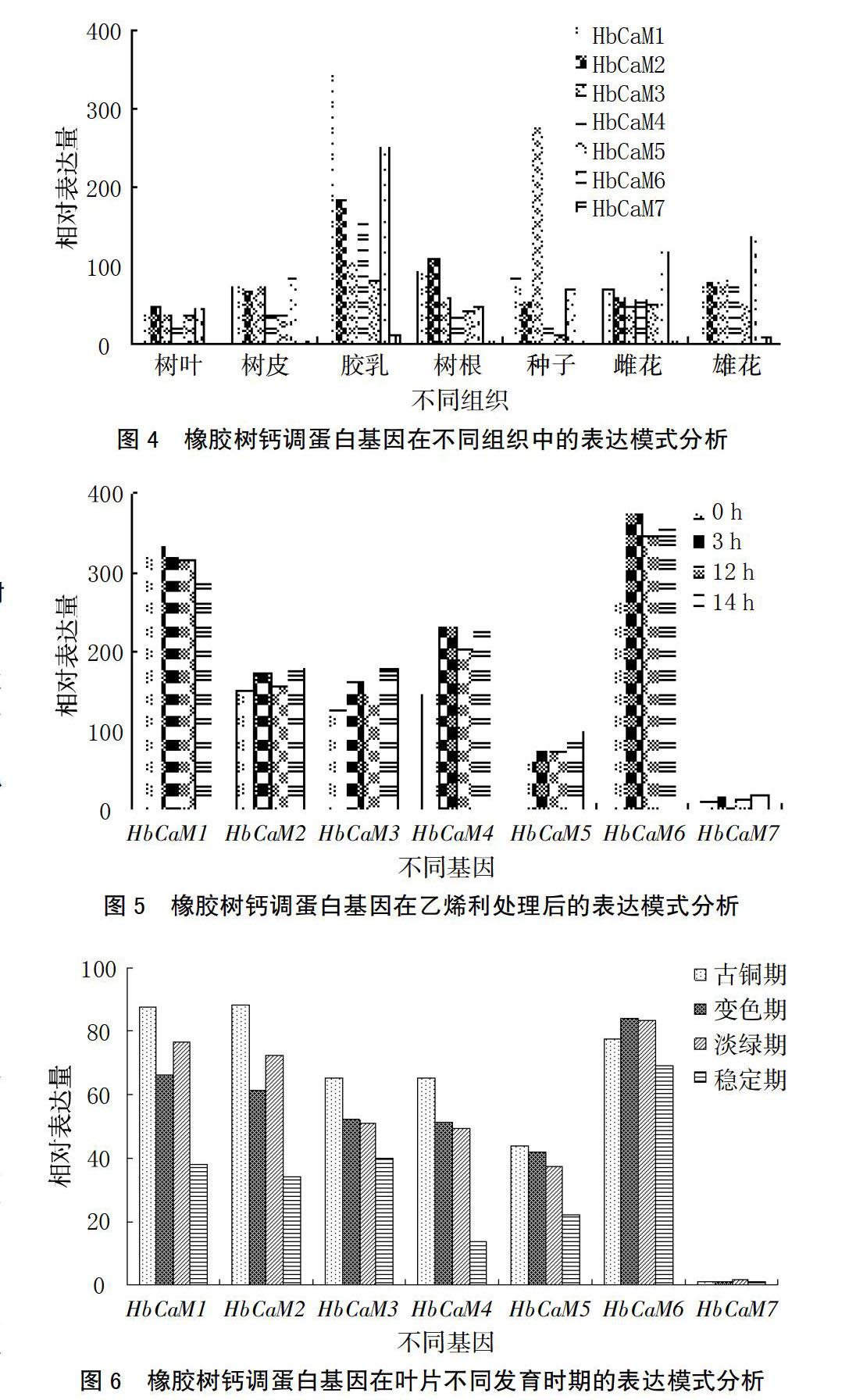
利用本實驗室Solexa高通量測序數據對7個橡膠樹鈣調蛋白基因在不同組織、不同葉片發育時期和乙烯利處理后不同時間點的表達情況進行分析。其中不同組織包括膠乳、樹皮、樹葉、根、種子、雌花和雄花。結果表明,雖然7個家族成員在序列上具有較高的同源性,但在表達方面卻存在著一定的差異(圖4)。各家族成員在所檢測的各組織中普遍表達,除了HbCaM3在種子中表達豐度最高,其他成員均是在膠乳中表達豐度最高,而HbCaM7在各組織中的表達豐度都很低,這說明橡膠樹鈣調蛋白基因在膠乳信號傳導方面可能發揮著重要作用。
乙烯刺激對橡膠樹膠乳中基因表達的影響結果見圖5。由圖5可知,乙烯利刺激3 h后,橡膠樹鈣調蛋白各家族成員的表達均有一定的上升趨勢,其中HbCaM4和HbCaM6的變化趨勢相對更加明顯。據此推測,橡膠樹鈣調蛋白基因參與了橡膠樹膠乳乙烯信號應答相關通路。橡膠樹鈣調蛋白基因家族成員在葉片發育過程中的表達情況見圖 6,除了HbCaM7基因外,其他各成員在葉片發育過程中的表達均呈下降趨勢,在穩定期的表達明顯低于其他各時期。這說明橡膠樹鈣調蛋白基因可能參與了橡膠樹葉片生長發育相關的調控。
3 討論與結論
鈣調蛋白在調控植物生長發育和參與植物逆境脅迫應答等方面具有重要作用。通過對擬南芥鈣調蛋白基因家族的研究發現,不同家族成員在擬南芥生長發育的不同階段具有不同的表達豐度,一葉期AtCAM4表達豐度最高,二葉期AtCAM2表達豐度最高,而到四葉期AtCAM1、AtCAM4、AtCAM5和AtCAM7具有較高的表達豐度[17]。在脅迫應答方面,研究結果發現,擬南芥大部分鈣調蛋白基因家族成員都參與了脅迫和激素刺激相關應答反應[18]。程曉培等[19]對逆境脅迫下MaCAM的表達進行分析,結果發現MaCAM明顯鹽脅迫、干旱和低溫誘導。在水稻中,通過分析基因家族成員在不同組織中的表達,結果發現,各家族成員在葉片和花中的表達明顯高于根和種子等其他組織[20]。本研究結果發現,除了HbCaM3在種子中表達豐度最高,其他各成員均在膠乳中表達豐度最高,并且在乙烯利刺激后大部分成員的表達都有一定的上升趨勢,這說明橡膠樹鈣調蛋白參與了橡膠樹乳管逆境脅迫應答相關信號通路。另外,通過研究橡膠樹鈣調蛋白各家族成員在葉片發育過程中的表達,結果發現,所有成員在葉片發育過程中的表達均呈下降趨勢,在穩定期葉片中的表達最低,這說明鈣調蛋白參與橡膠樹葉片生長發育相關的信號調控,并且更多在代謝較活躍的組織中表達。本研究從基因結構、系統進化和表達模式幾方面對橡膠樹鈣調蛋白各家族成員進行了系統的分析。研究結果將有助于深入了解鈣調蛋白基因家族成員在橡膠樹生長發育和脅迫應答等方面的重要功能。
參考文獻
[1] Cheung W Y. Cyclic 3′,5′-nucleotide phosphodiesterase: Pronounced stimulation by snake venom[J]. Biochemical & Biophysical Research Communications,1967, 29(4): 478-482.
[2] Defalco T A, David C, Kim M, et al. Characterization of GmCaMK1, a member of a soybean calmodulin-binding receptor-like kinase family[J]. Febs Letters, 2010, 584(23): 4 717-4 724.
[3] Watterson D M, Sharief F, Vanaman T C. The complete amino acid sequence of the Ca2+-dependent modulator protein(calmodulin)of bovine brain[J]. Journal of Biological Chemistry, 1980, 255(3):962-975.
[4] Floyd E E, Gong Z Y, Brandhorst B P, et al. Calmodulin gene expression during sea urchin development:persistence of a prevalent maternal protein[J]. Developmental Biology, 1986, 113(2): 501-511.
[5] Davis T N, Urdea M S, Masiarz F R, et al. Isolation of the yeast calmodulin gene: Calmodulin is an essential protein[J]. Cell, 1986, 47(3): 423-431.
[6] Tschudi C, Young A S, Ruben L, et al. Calmodulin genes in trypanosomes are tandemly repeated and produce multiple mRNAs with a common 5'leader sequence[J]. Proceedings of the National Academy of Sciences, 1985, 82(12): 3 998-4 002.
[7] Mccormack E, Braam J. Calmodulins and related potential calcium sensors of Arabidopsis[J]. New Phytologist, 2003, 159(3): 585-598.
[8] Boonburapong B, Buaboocha T. Genome-wide identification and analyses of the rice calmodulin and related potential calcium sensor proteins[J]. Bmc Plant Biology, 2007, 7(1): 1-17.
[9] Ling V, Zielinski R E. Cloning of cDNA sequences encoding the calcium-binding protein, calmodulin, from barley(Hordeum vulgare L.)[J]. Plant Physiology, 1989, 90(2): 714-719.
[10] Barnett M J, Long S R. Nucleotide sequence of an alfalfa calmodulin cDNA[J]. Nucleic Acids Research, 1990, 18(18): 3 395.
[11] 龔潔敏,金志強,孔德騫,等. 香蕉鈣調蛋白基因的克隆及序列分析[J]. 熱帶作物學報,1994,15(1):65-71.
[12] 陳 鑫,朱家紅,張治禮. 巴西橡膠樹鈣調蛋白基因的克隆及表達分析[J]. 中國農學通報,2011,27(22):46-50.
[13] Tang C R, Qi J Y , Li H P, et al. A convenient and efficient protocol for isolating high-quality RNA from latex of Hevea brasiliensis(para rubber tree)[J]. J Biochem Biophys Methods, 2007, 70(5): 749-754.
[14] Kiefer E, Heller W, Ernst D. A simple and efficient protocol for isolation of functional RNA from plant tissues rich in secondary metabolites[J]. Plant Mol Biol Rep, 2000, 18(1): 33-39.
[15] 肖小虎. 巴西橡膠樹蔗糖代謝相關基因家族的克隆、結構進化和表達分析[D]. 海口:海南大學, 2013.
[16] Li B, Dewey C N, Li B, Dewey C N. RSEM:accurate transcript quantification from RNA-Seq data with or without a reference genome [J]. BMC Bioinformatics, 2011, 12(1):93-99.
[17] McCormack E, Tsai Y C, Braam J. Handling calcium signaling:Arabidopsis CaMs and CMLs[J]. Trends inPlant Science, 2005, 10(8): 383-389.
[18] McCormack E, Braam J. Calmodulins and related potential calcium sensors of Arabidopsis[J]. New Phytologist, 2003, 159(3): 585-598.
[19] 程曉培,徐碧玉,劉菊華,等. 香蕉鈣調蛋白基因MaCAM在非生物脅迫下的表達分析[J]. 生命科學研究,2013,17(1):20-23.
[20] Boonburapong B, Buaboocha T. Genome-wide identification and analyses of the rice calmodulin and related potential calcium sensor proteins[J]. BMC Plant Biology, 2007, 7(1): 1-17.

