一種新ω-3脂肪酸脫飽和酶的克隆表達和活性鑒定
梅甜甜,陳海琴,郝光飛,顧震南,陳衛(wèi),陳永泉
(江南大學 食品學院,食品科學與技術國家重點實驗室,江蘇 無錫,214122)
?
一種新ω-3脂肪酸脫飽和酶的克隆表達和活性鑒定
梅甜甜,陳海琴*,郝光飛,顧震南,陳衛(wèi),陳永泉
(江南大學 食品學院,食品科學與技術國家重點實驗室,江蘇 無錫,214122)
ω-3脂肪酸脫飽和酶催化ω-6多不飽和脂肪酸(PUFAs)轉化為ω-3 PUFAs,對ω-3長鏈多不飽和脂肪酸(LC-PUFAs)的合成至關重要。為了實現(xiàn)在常溫下發(fā)酵生產(chǎn)ω-3 LC-PUFAs(主要是二十碳五烯酸,EPA),根據(jù)現(xiàn)有常溫下偏好催化20C PUFAs的ω-3脂肪酸脫飽和酶序列,從GenBank數(shù)據(jù)庫篩選出與之高度相似的序列并進行生物信息學分析。為了確定序列的生物活性,進一步在釀酒酵母系統(tǒng)中進行重組表達,通過外源添加不同碳鏈長度的脂肪酸底物,測定重組釀酒酵母轉化子在28 ℃和12 ℃下對不同脂肪酸的轉化率。結果顯示,新篩選序列編碼的蛋白oAiFADS17既能催化18C PUFAs,又能催化20C PUFAs,尤其偏好催化二十碳四烯酸(AA)轉化為EPA。oAiFADS17蛋白在28 ℃下對各種底物的轉化率均高于12 ℃下的轉化率,其中對AA的轉化率達到46.3%。該研究成功測定了oAiFADS17蛋白對不同脂肪酸底物的轉化率,得到了一種新的常溫偏好催化20C PUFAs的ω-3脂肪酸脫飽和酶,為構建高產(chǎn)EPA的基因工程菌株及EPA的工業(yè)化生產(chǎn)奠定了理論基礎。
ω-3脂肪酸脫飽和酶;轉化率;二十碳五烯酸(EPA)
ω-3長鏈多不飽和脂肪酸(LC-PUFAs)主要包括二十碳五烯酸(Eicosapentaenoic acid, EPA, C20∶5)和二十二碳六烯酸(Docosahexaenoic acid, DHA, C22∶ 6)。EPA和DHA可以促進哺乳動物大腦發(fā)育以及神經(jīng)組織的形成和修復[1],并對哮喘、癌癥、抑郁、肥胖、免疫紊亂以及心血管疾病具有十分顯著的預防作用[2-3]。但人體自身不能從源頭合成EPA和DHA,需要從富含ω-3 LC-PUFAs的食物(如深海魚)中攝取。然而由于大肆捕撈以及海洋環(huán)境的污染,深海魚的供應已無法滿足日益增長的市場需求[4-6]。
自然界中ω-3 LC-PUFAs的合成起始于亞油酸(linoleic acid, LA, C18∶2)和γ-亞麻酸(γ-linoleic acid, ALA, C18∶3),經(jīng)過一系列脫飽和酶和延長酶的催化作用,最終轉化生成EPA和DHA。ω-3脂肪酸脫飽和酶(EC 1.14.19.25)位于內質網(wǎng)膜上,是ω-3 LC-PUFAs合成中的關鍵酶[7]。研究發(fā)現(xiàn),不同來源的ω-3脂肪酸脫飽和酶對不同碳鏈長度的脂肪酸具有不同的催化效率。早期研究發(fā)現(xiàn)來源于植物的ω-3脂肪酸脫飽和酶(FAD7, FAD8等)只能催化18C PUFAs[8]。而來源于秀麗隱桿線蟲(Caenorhabditiselegans)的ω-3脂肪酸脫飽和酶FAT 1則能同時催化18C和20C PUFAs,但對20C底物的催化活性較低[9]。2004年PEREIRA等人發(fā)現(xiàn)來源于異枝水霉(Saprolegniadiclina)的ω-3脂肪酸脫飽和酶SDD17對18C PUFAs沒有催化活性,卻能將二十碳四烯酸(Arachidonic acid, AA, C20∶4)催化為EPA,轉化率為25.9%[10]。同樣的,來源于致病疫霉(Phytophthorainfestans)的ω-3脂肪酸脫飽和酶OPIN 17也不能以18C PUFAs為底物,但是對AA的轉化率達到了30.94%[11]。2013年,XUE等人從瓜果腐霉(Pythiumaphanidermatum)、大豆疫霉菌(Phytophthorasojae)和櫟樹猝死病菌(Phytophthoraramorum)中分離出3種ω-3脂肪酸脫飽和酶——PaD 17、PsD 17和PrD 17,這3種酶都具有催化18C和20C PUFAs的能力,對20C 脂肪酸AA的轉化率分別為56%、47%和36%,并且這3種酶適應在28 ℃下進行催化反應[12]。這一類在28 ℃下偏好催化20C脂肪酸的ω-3脂肪酸脫飽和酶對ω-3 LC-PUFAs尤其是EPA的合成至關重要。本研究結合基因搜索和基因工程重組表達,篩選出了一種新的偏好催化20C脂肪酸的ω-3脂肪酸脫飽和酶。新ω-3脂肪酸脫飽和酶的發(fā)現(xiàn)為研究這類脫飽和酶的偏好性提供了新的資源,也為ω-3脂肪酸脫飽和酶的應用提供了更多選擇,同時為EPA的工業(yè)化生產(chǎn)奠定了理論基礎。
1 材料與方法
1.1材料
1.1.1菌株和載體
大腸桿菌(Escherichiacoli,E.coli)TOP 10由本實驗室保存。表達載體pYES2/NT C與尿嘧啶缺陷型釀酒酵母INVSc 1購于Invitrogen公司,現(xiàn)由本實驗室保存。
1.1.2試劑
KOD plus高保真DNA聚合酶購自Toyobo公司;限制性內切酶EcoR I和XhoI、T4DNA連接酶、Prime Script RT reagent kit試劑盒均購自TaKaRa生物技術公司(大連);凝膠回收試劑盒購自Fermentas公司;PCR產(chǎn)物純化試劑盒購自生物工程(上海)股份有限公司;TaqDNA聚合酶購自北京天根生化科技有限公司;BCA蛋白測定試劑盒購于上海碧云天公司;SYBR Green PCR Master Mix購自美國Bio-Rad公司;脂肪酸標準品LA、十八碳三烯酸(GLA)、二十碳三烯酸(DGLA)、AA購自美國Sigma公司。
1.1.3培養(yǎng)基及培養(yǎng)條件
LB培養(yǎng)基(g/L):胰蛋白胨 10,酵母提取物 5,氯化鈉 10,pH 7.0,固體培養(yǎng)基添加1.5%瓊脂粉,用于大腸桿菌的培養(yǎng),培養(yǎng)溫度37 ℃,搖床轉速200 r/min。
YPD培養(yǎng)基(g/L):酵母抽提物 10,蛋白胨 20,葡萄糖 20,pH 7.0,固體培養(yǎng)基添加1.5%瓊脂粉,用于釀酒酵母的培養(yǎng),培養(yǎng)溫度28 ℃,搖床轉速200 r/min。
SC-U(g/L):酵母氮源(無氨基酸有硫酸銨) 6.7,葡萄糖 20,腺嘌呤、 精氨酸、半胱氨酸、亮氨酸、賴氨酸、蘇氨酸、色氨酸、尿嘧啶各0.1,天冬氨酸、組氨酸、異亮氨酸、甲硫氨酸、苯丙氨酸、脯氨酸、絲氨酸、酪氨酸、纈氨酸各0.05,用于轉化子擴大培養(yǎng),培養(yǎng)溫度28 ℃,轉速200 r/min。誘導培養(yǎng)時更換碳源為10 g/L棉籽糖和20 g/L半乳糖,其余成分與SC-U培養(yǎng)基相同。
1.1.4實驗所用引物
本研究所用引物如表1所示,根據(jù)優(yōu)化得到的oAiFADS17基因序列設計引物擴增目的基因片段,同時引入EcoR I和XhoI酶切位點(表中下劃線所示)。根據(jù)oAiFADS17基因序列保守區(qū)設計RT-qPCR引物,使用釀酒酵母18S rRNA通用引物作為內參[13]。
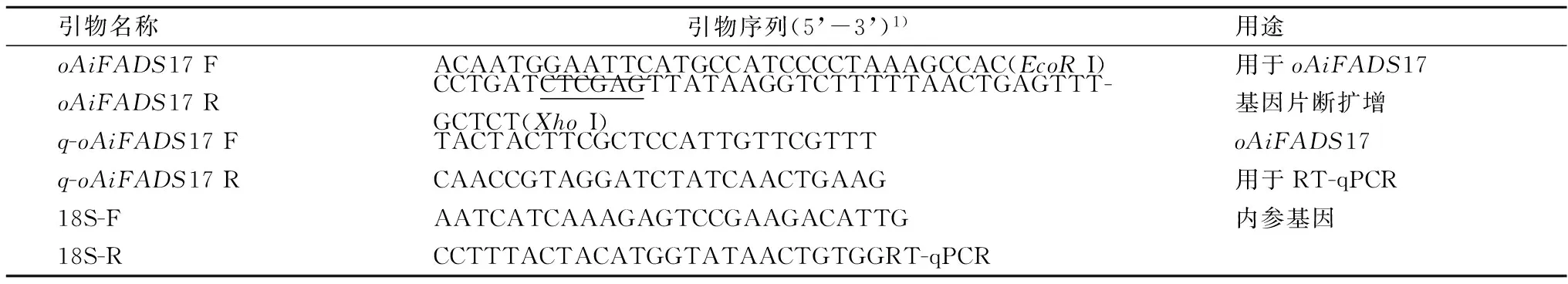
表1 本研究所用引物
注:1)表中下劃線表示引入的酶切位點,括號中的字母表示限制性酶切位點。
1.2實驗方法
1.2.1oAiFADS17基因的獲得
利用BLAST從Genbank數(shù)據(jù)庫篩選到一段來自真菌媒介絲囊菌(Aphanomycesinvadans)的基因序列(GenBank accession No. XM_008870610),與5種已知偏好催化20C PUFAs的ω-3脂肪酸脫飽和酶(SDD17, OPIN 17, PaD17, PsD17, PrD17)具有較高的同源性。將這段基因編碼的蛋白與上述5種ω-3脂肪酸脫飽和酶序列在Clustal W2中進行比對,同時利用TMHMM軟件預測此段序列編碼蛋白的跨膜域。進一步構建含此段序列的表達載體,首先利用Genscript Optimum GeneTMsystem軟件對其核酸序列進行密碼子優(yōu)化,人工合成優(yōu)化后的基因序列,命名為oAiFADS17(GenBank accession No. KT371999),再與pUC57-simple載體連接得pUC57-oAiFADS17,保存于大腸桿菌 TOP 10中(金斯瑞,南京,中國)。依據(jù)oAiFADS17基因序列設計引物oAiFADS17 F/oAiFADS17 R來擴增目的基因片段。
1.2.2表達載體構建及測序
用限制性內切酶EcoR I和XhoI雙酶切oAiFADS17基因片段和pYES2/NT C表達載體,酶切產(chǎn)物經(jīng)切膠回收純化后進行連接。取3 μL上述連接產(chǎn)物電轉化大腸桿菌TOP 10。挑取陽性轉化子,用目的基因引物進行PCR鑒定。PCR產(chǎn)物用1%瓊脂糖凝膠電泳驗證,條帶大小正確的轉化子進行測序驗證。
1.2.3釀酒酵母轉化及重組轉化子誘導蛋白表達
采用PEG/LiAc法[14]轉化釀酒酵母,從平板上挑取生長狀態(tài)良好的陽性克隆接種到5 mL YPD培養(yǎng)基中,30 ℃ 200 r/min培養(yǎng)48 h。提取質粒,用通用引物T7/T7 terminator(結合于多克隆位點上下游)進行PCR驗證并測序。挑取測序正確的釀酒酵母轉化子單菌落接種于SC-U培養(yǎng)基(含空質粒的釀酒酵母轉化子為陰性對照,每組3個平行),28 ℃培養(yǎng)48 h。測量OD600值,轉接入SC-U誘導培養(yǎng)基,調整OD600值達到0.4。同時根據(jù)需要,向培養(yǎng)基中添加不同碳鏈長度PUFAs(見2.3.3),28 ℃/12 ℃培養(yǎng)48 h后收集菌體。
1.2.4釀酒酵母轉化子轉錄水平測定
用Trizol提取釀酒酵母菌體總RNA。以1 μg總RNA為模板,根據(jù)Prime Script RT reagent kit(TaKaRa, Otsu, Shiga, Japan)試劑盒說明進行操作,獲得cDNA。使用Bio-Rad CFX ConnectTMsystem 按照 iTaq Universal SYBR Green Supermix說明進行RT-qPCR反應。反應體系為:8 μL無酶水,10 μL iTaq Universal SYBR Green Supermix,0.5 μL q-oAiFADS17 F,0.5 μL q-oAiFADS17 R,1 μL cDNA模板,總體積20 μL。以釀酒酵母的18S rRNA作為內參基因,根據(jù)2-△△Ct法計算基因的相對轉錄水平,所有實驗進行3次重復。(注明:①ΔCt=待測樣品目的基因Ct值-待測樣品內參Ct值;②ΔCt=實驗組ΔCt-對照組ΔCt。)
1.2.5蛋白表達水平測
收集誘導后的菌體2 mL,10 000 r/min離心棄去上清。加入適量體積的裂解液(50 mmol/L Na2HPO4-NaH2PO4, 1 mmol/L EDTA, 5% 1,2,3-丙三醇, 1 mmol/L PMSF, pH 7.4)使OD600值達到100,然后加入等體積的0.5 mm酸洗玻璃珠,置于漩渦振蕩器振蕩30 s后置于冰上30 s,如此重復4次后于13 000 r/min離心10 min,得到的上層懸液轉移至新的1.5 mL離心管中,用BCA試劑盒測定蛋白濃度后,以100 μg總蛋白的上樣量進行SDS-PAGE以及Western Blot[15]。
1.2.6底物特異性測定
收集誘導后的釀酒酵母菌體,真空冷凍干燥后置于研缽中充分研磨粉碎。稱取20 mg菌體粉末采用脂肪酸甲酯化法[16]進行提取,加入C15∶0(用于標定鏈長≤18C的脂肪酸)和C21∶0(用于標定鏈長>18C的脂肪酸)各100 μL作為內標。得到的脂肪酸甲酯使用GC-MS(Shimadzu Co., Japan)檢測[17]。色譜柱為Rtx-Wax(30 m×0.25 mm, 0.25 μm),汽化室和檢測器溫度分別為240 ℃和250 ℃,分流方式進樣1 μL,分流比10∶1,載氣為氦氣。程序升溫:初始溫度120 ℃保持3 min,以5 ℃/min升到190 ℃,再以4 ℃/min升到220 ℃,保持20 min。
2 結果與討論
2.1oAiFADS17序列的生物信息學分析
oAiFADS17基因來自真菌媒介私囊菌,全長1095 bp,編碼364個氨基酸,蛋白理論大小約41 kDa。為了考證oAiFADS17編碼的蛋白oAiFADS17是否與ω-3脂肪酸脫飽和酶同源,將其與5種已知的ω-3脂肪酸脫飽和酶氨基酸序列進行了比對(圖1)。
利用Clustal W2工具的比對結果表明,oAiFADS17與已知ω-3脂肪酸脫飽和酶的同源性高于59%,且具有與其他ω-3脂肪酸脫飽和酶一樣的3個高度保守的組氨酸保守區(qū)HX3HH,HX2HH,HX2HH。進一步通過脂肪酸拓撲結構模型分析發(fā)現(xiàn)(圖2),除位于親水區(qū)的3個組氨酸保守區(qū)外,oAiFADS17還具有位于疏水區(qū)的4個跨膜螺旋和2個膜內結構域,共同構成了ω-3脂肪酸脫飽和酶的催化活性中心,理論上符合ω-3脂肪酸脫飽和酶的基本特征。
2.2含oAiFADS17基因重組釀酒酵母菌的構建
利用oAiFADS17 F/oAiFADS17 R引物通過PCR擴增得到oAiFADS17基因序列,將其連接至pYES2/NT C載體,命名為pYES2-oAiFADS17。將重組質粒以及空質粒pYES2/NT C轉化入釀酒酵母INVSc 1,挑取陽性轉化子,利用通用引物T7/T7 terminator進行PCR驗證,驗證結果如圖3所示,2、3、4號轉化子擴增得到的片段在1 463 bp(1 095 bp+368 bp)左右,而轉入空質粒pYES2/NT C的1號轉化子在368 bp左右出現(xiàn)條帶,與理論大小一致。進一步通過測序驗證表明1號為轉入空質粒pYES2/NT C的轉化子,2、3、4號為轉入重組質粒pYES2-oAiFADS17的轉化子。將得到1-4號轉化子分別命名為INVSc 1(pYES2/NT C),INVSc 1 (pYES2-oAiFADS17)-1,INVSc 1 (pYES2-oAiFADS17)-2,INVSc 1 (pYES2-oAiFADS17)-3。

圖1 真菌媒介私囊菌的ω-3脂肪酸脫飽和酶(oAiFADS17)與偏好催化20C PUFAs的ω-3脂肪酸脫飽和酶氨基酸序列比對Fig.1 Multiple equence alignment of amino acids for ω-3 desaturases from Aphanomyces invadans (oAiFADS17) and which prefer to convert 20C PUFAs. PrD17:ω-3 desaturases from Phytophthora ramorum(GenBank accession No. FW362214.1); PsD17:ω-3 desaturases from Phytophthora sojae(GenBank accession No. FW362213.1); OPIN17:ω-3 desaturases from Phytophthora infestans(GenBank accession No. XM 002902553.1); PaD17:ω-3 desaturases from Pythium aphanidermatum(GenBank accession No. FW362186.1); SDD17:ω-3 desaturases from Saprolegnia diclina (GenBank accession No. AY373823.1). Black bars show the identical amino acid residues. Deletions are indicated by dashes. Boxes represent three typical histidine motifs

圖2 ω-3脂肪酸脫飽和酶oAiFADS17的拓撲結構模型Fig.2 Topology model of ω-3 desaturase oAiFADS17 Cylinders A-F indicate transmembrane helices, and the black solid circles indicate three conserved histidine-rich motifs. Prediction of transmembrane helices and topology was performed with TMHMM, HMMTOP and TOP-PRED . The predicted topology model was in qualitative agreement with other membrane-bound enzymes
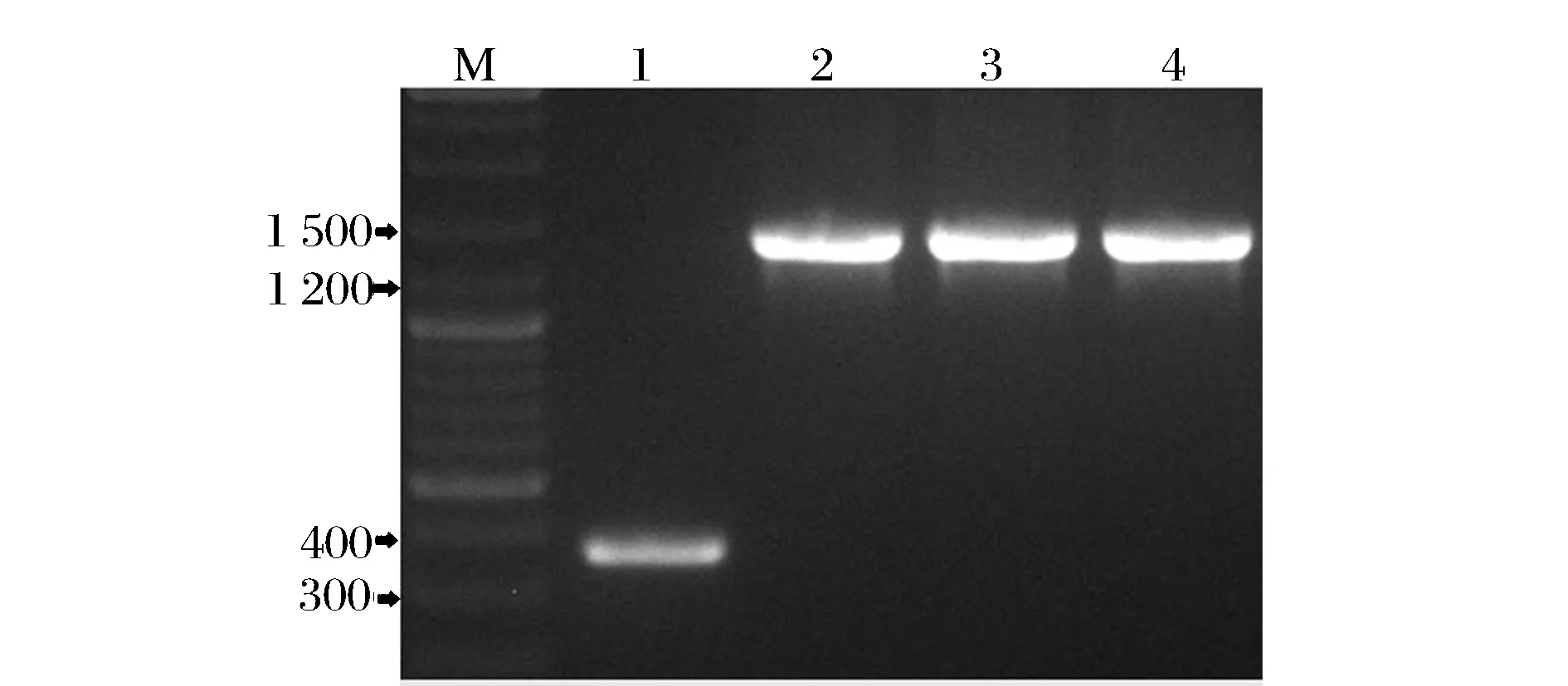
M-Marker; 1-對照, INVSc 1 (pYES2/NT C); 2-INVSc 1 (pYES2-oAiFADS17)-1; 3-INVSc 1 (pYES2-oAiFADS17)-2; 4-INVSc 1 (pYES2-oAiFADS17)-3圖3 釀酒酵母轉化子PCR驗證Fig.3 PCR verification of yeast transformation
2.3 oAiFADS17脫飽和酶重組釀酒酵母菌的活性驗證
2.3.1目的基因oAiFADS17轉錄水平測定
將篩選獲得的3株轉化子于SC培養(yǎng)基中28 ℃培養(yǎng)2 d后收集菌體,通過RT-qPCR方法測定目的基因在轉化子中的轉錄水平。轉錄水平分析結果表明在轉入空質粒的釀酒酵母轉化子INVSc 1(pYES2/NT C)中未檢測到目的基因轉錄,而在轉入重組質粒的3個釀酒酵母轉化子INVSc 1 (pYES2-oAiFADS17)中,目的基因均有顯著表達(圖4)。
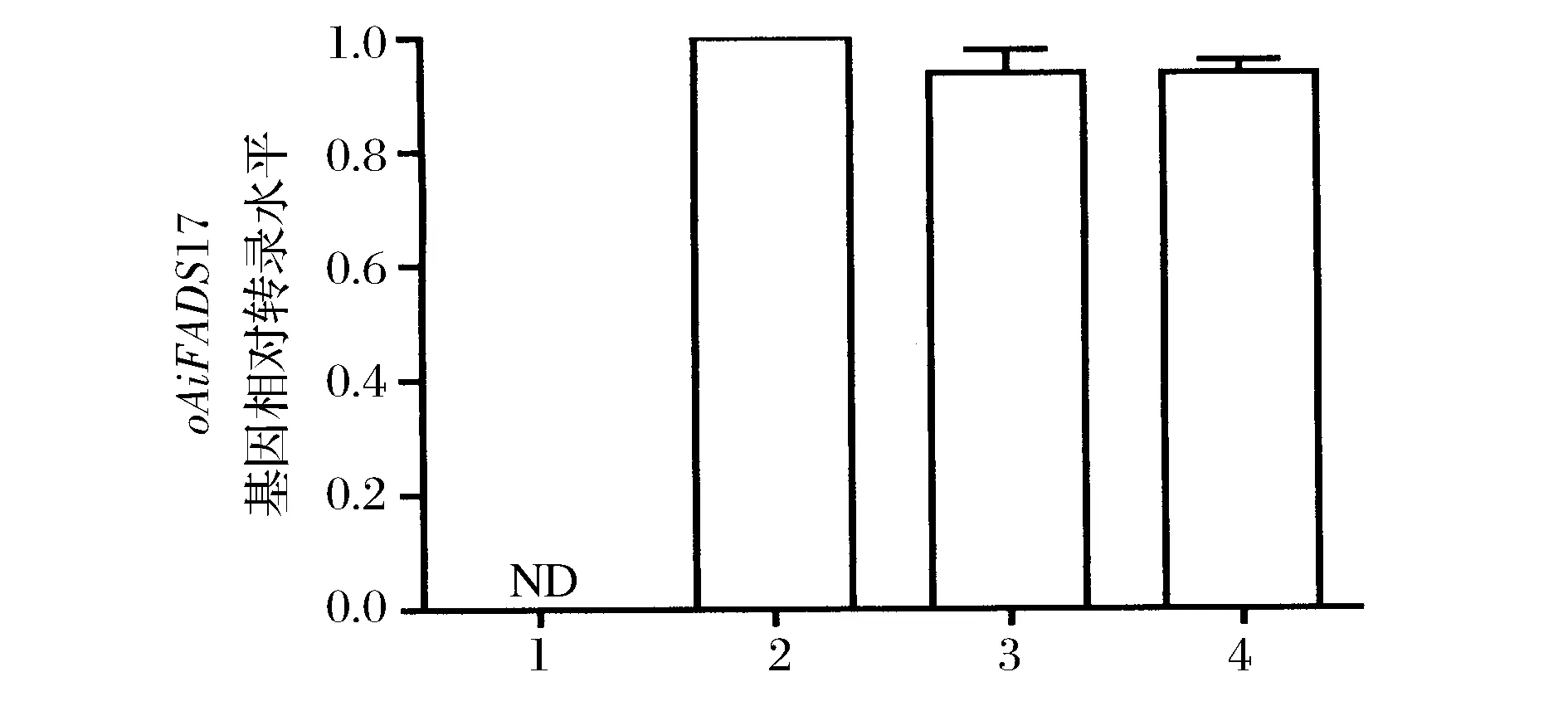
1-Control, INVSc 1 (pYES2/NT C); 2-INVSc 1 (pYES2-oAiFADS17)-1; 3-INVSc 1 (pYES2-oAiFADS17)-2; 4-INVSc 1 (pYES2-oAiFADS17)-3圖4 oAiFADS17基因相對表達量Fig.4 The relative expression level of oAiFADS17 ( **:P<0.001. All data are presented as mean ± SD.)
2.3.2目標蛋白oAiFADS17表達水平測定
利用Western blot對重組菌株中目的蛋白的表達水平進行分析,具體方法參見1.2.5。轉入重組質粒的3個釀酒酵母轉化子均在45 kDa附近(表達載體上帶有3.4 kDa His標簽,故檢測蛋白理論分子量大小約45 kDa)檢測到目標蛋白的雜交條帶(圖5),該蛋白的分子量大小與其理論值相符,說明在釀酒酵母3個轉化子INVSc 1 (pYES2-oAiFADS17)中目的蛋白正確表達。
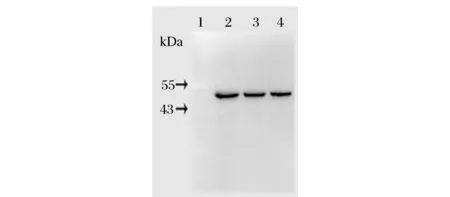
1-對照, INVSc 1 (pYES2/NT C); 2-INVSc 1 (pYES2-oAiFADS17)-1; 3-INVSc 1 (pYES2-oAiFADS17)-2; 4-INVSc 1 (pYES2-oAiFADS17)-3圖5 釀酒酵母轉化子的蛋白表達水平Fig.5 The protein expression level of yeast transformants
2.3.3釀酒酵母重組轉化子催化特性分析
為了檢測重組釀酒酵母菌株對AA的轉化率,將3個釀酒酵母轉化子在分別添加了0.05 ,0.1,0.2 mmol/L AA的誘導培養(yǎng)基中28 ℃培養(yǎng)2 d,收集菌體后檢測其脂肪酸組成。氣相色譜圖顯示,轉入目的基因的釀酒酵母轉化子INVSc 1 (pYES2-oAiFADS17)在37.5 min處出現(xiàn)了信號峰(圖6c),與EPA標準品出峰時間一致(圖6a),而轉入空質粒的對照組釀酒酵母INVSc 1 (pYES2/NT C)在相應的保留時間無信號(圖6b)。該信號峰與EPA標準品的出峰時間完全一致。進一步將該峰對應物質的質譜圖與EPA標準品質譜圖(圖7)比對,兩者一致,且均在m/z79、m/z91、m/z119處出現(xiàn)強的離子峰,確認此物質為EPA。以上GC-MS分析結果表明,釀酒酵母轉化子INVSc 1 (pYES2-oAiFADS17)能將ω-6系的AA轉化為ω-3系的EPA,oAiFADS17具有ω-3脂肪酸脫飽和酶活性。當外源添加不同濃度的AA時(表2),各轉化子對AA的轉化率非常接近。在AA濃度為0.05 mmol/L時,各轉化子的轉化率為36%~46%不等,而隨著AA濃度增高,各轉化子對AA的轉化率有所降低,原因可能是由于AA量達飽和導致。為了進一步探究oAiFADS17對不同鏈長脂肪酸的催化特性,選取釀酒酵母INVSc 1 (pYES2-oAiFADS17)-3以及對照組INVSc 1 (pYES2/NT C)轉化子在添加不同碳鏈長度脂肪酸底物的培養(yǎng)基中培養(yǎng),添加方式包括單一底物(0.2 mmol/L LA、GLA、DGLA、AA)及雙底物(0.1 mmol/L LA+0.1 mmol/L AA)形式,培養(yǎng)溫度分別為28 ℃和12 ℃。在兩種培養(yǎng)溫度下,釀酒酵母INVSc 1 (pYES2-oAiFADS17)-3對20CPUFAs的轉化率明顯高于18CPUFAs,且對AA轉化率最高,而在添加雙底物(LA+AA)條件下,轉化子對20C底物AA的偏好性更強,由此可知oAiFADS17偏好催化20CPUFAs,而對照組卻不能轉化任何一種底物。同時,釀酒酵母轉化子INVSc 1 (pYES2-oAiFADS17)在28 ℃下對各種脂肪酸的轉化率均高于12 ℃下的轉化率(表3),說明oAiFADS17在28 ℃下活性較高。綜上所述,oAiFADS17是一種在28 ℃對AA有較高催化活性的ω-3脂肪酸脫飽和酶。
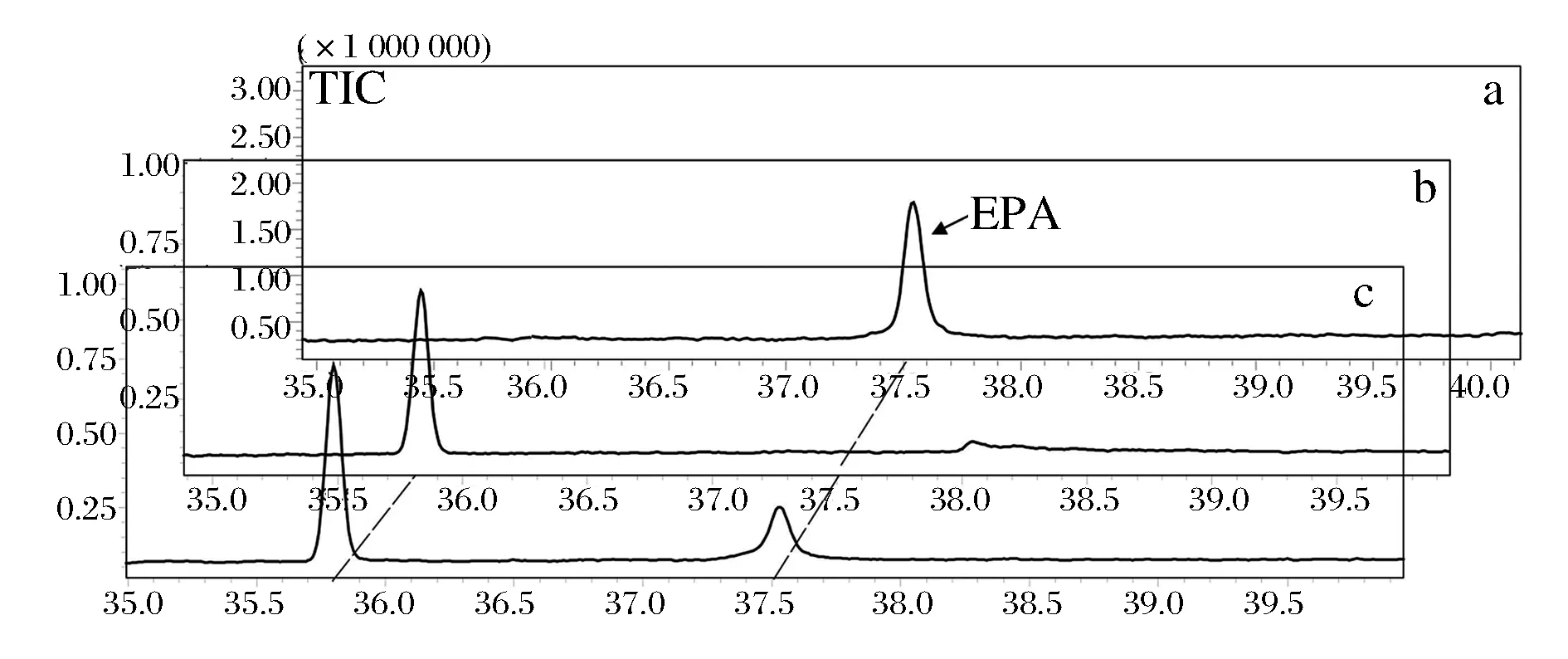
a-EPA standard; b-Control, INVSc 1 (pYES2/NT C); c-INVSc 1 (pYES2-oAiFADS17)圖6 釀酒酵母轉化子INVSc 1 (pYES2-oAiFADS17)脂肪酸色譜圖Fig.6 Gas chromatogram of fatty acid methyl esters from lipid fraction of INVSc 1 (pYES2-oAiFADS17)
3 結論
本研究從GenBank數(shù)據(jù)庫篩選到一個新ω-3脂肪酸脫飽和酶基因oAiFADS17,并將此基因成功地在釀酒酵母系統(tǒng)中進行了表達。通過外源添加底物,對該基因的轉錄水平、蛋白表達水平及底物特異性進行了分析。結果表明這種ω-3脂肪酸脫飽和酶(oAiFADS17)在28 ℃下可以催化18C和20C鏈長的ω-6 PUFAs,尤其偏好催化20C鏈長底物AA生成具更高附加值的EPA,對AA的轉化率達到了46.3%。本研究為ω-3脂肪酸脫飽和酶偏好性的研究提供了新的資源,也為EPA的工業(yè)化生產(chǎn)奠定了一定的理論基礎。
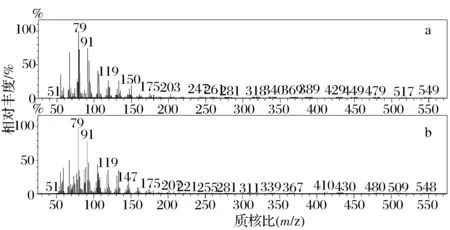
a-Mass spectrum of EPA in INVSc 1 (pYES2-oAiFADS17); b-Mass spectrum of EPA standard圖7 EPA質譜圖Fig.7 Mass spectrum of the EPA
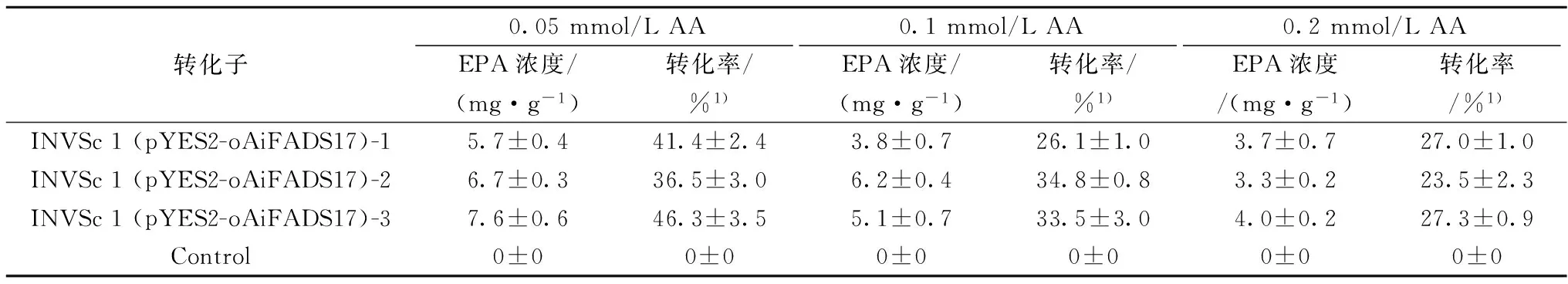
表2 釀酒酵母轉化子INVSc 1 (pYES2-oAiFADS17)對AA的轉化率
注:1)Conversion rate is calculated as 100% ×Product/(Product+Substrate).

表3 不同溫度下釀酒酵母轉化子對不同底物轉化率
注:1)Conversion rae is calculated as 100%×Product/(Product+Substrate).
[1]SAKURADANI E,SHIMIZU S.Single cell oil production byMortierellaalpina[J].Journal of Biotechnology, 2009,144(1):31-36.
[2]KHAWAJA O A,GAZIANO J M,DJOUSSE L.N-3 fatty acids for prevention of cardiovascular disease[J]. Current Atherosclerosis Reports,2014,16(11):450.
[3]LORENTE-CEBRIAN S,COSTA A G,NAVAS-CARRETERO S,et al.Role of omega-3 fatty acids in obesity, metabolic syndrome, and cardiovascular diseases:a review of the evidence [J].Journal of Physiology and Biochemistry,2013, 69(3):633-651.
[4]RACINE R A,DECKELBAUM R J.Sources of the very-long-chain unsaturated omega-3 fatty acids:eicosapentaenoic acid and docosahexaenoic acid [J].Current Opinion in Clinical Nutrition and Metabolic Care, 2007,10(2):123-128.
[5]杜冰,潘力,林煒鐵,等.多不飽和脂肪酸發(fā)酵的代謝調控育種[J].食品與發(fā)酵工業(yè),2000,26(5):52-55.
[6]徐天宇. 利用生物技術生產(chǎn)廿碳五烯酸和廿二碳六烯酸[J].食品與發(fā)酵工業(yè),1995,21(1):56-65.
[7]WANG M X,CHEN H Q,GU Z N,et al.ω 3 fatty acid desaturases from microorganisms:structure, function, evolution, and biotechnological use[J].Applied Microbiology and Biotechnology,2013,97(24):10 255-10 262.
[8]TOCHER D R,LEAVER M J,HODGSON P A.Recent advances in the biochemistry and molecular biology of fatty acyl desaturases[J].Progress in Lipid Research,1998,37(2/3):73-117.
[9]MEESAPYODSUK D,REED D W,SAVILE C K,et al.Characterization of the regiochemistry and cryptoregiochemistry of aCaenorhabditiselegansfatty acid desaturase (FAT-1) expressed inSaccharomycescerevisiae[J].Biochemistry-Us,2000,39(39):11 948-11 954.
[10]PEREIRA S L,HUANG Y S,BOBIK E G,et al.A novel omega 3-fatty acid desaturase involved in the biosynthesis of eicosapentaenoic acid[J].Biochemical Journal,2004,378:665-671.
[11]FU Y,FAN X Z,LI X Z,et al.The desaturase OPIN17 fromPhytophthorainfestansconverts arachidonic acid to eicosapentaenoic acid in CHO cells[J].Applied Biochemistry and Biotechnology,2013,171(4):975-988.
[12]XUE Z X,HE H X,Hollerbach D,et al.Identification and characterization of new delta-17 fatty acid desaturases [J].Applied Microbiology and Biotechnology,2013,97(5):1 973-1 985.
[13]CANKORUR-CETINKAYA A,DERELI E, Eraslan S,et al.A novel strategy for selection and validation of reference genes in dynamic multidimensional experimental design in yeast[J].Plos One,2012,7(6):e38 351.
[14]胡又佳,高楓,朱春寶,等.PEG/LiAc轉化酵母細胞方法的改進[J].生物技術,1998(5):22-26;49.
[15]CHEN H Q,GU Z N,ZHANG H,et al.Expression and purification of integral membrane fatty acid desaturases [J].Plos One, 2013,8(3). http://dx.doi.org/10.1371/journal pone.0058139.
[16]WANG L,CHEN W, FENG Y,et al.Genome characterization of the oleaginous fungusMortierellaalpina[J].Plos One, 2011,6(12).http://dx.doi.org/10.1371/journal.pone.0028319.
[17]施忠芬,王桂瑛,陳紅艷,等.GC-MS法測定宣威火腿中脂肪酸組成[J].食品與發(fā)酵工業(yè),2012,38(9):141-145.
Cloning, expression and characterization of a new ω-3 fatty acid desaturase
MEI Tian-tian, CHEN Hai-qin, HAO Guang-fei, GU Zhen-nan,CHEN Wei, CHEN Yong-quan
(School of Food Science and Technology, State Key Laboratory of Food Science and Technology, Jiangnan University,Wuxi 214122,China)
ω-3 fatty acid desaturase can convert ω-6 PUFAs to ω-3 PUFAs, which is significant for the synthesis of ω-3 LC-PUFAs. In order to realize the production of EPA (eicosapentaenoic acid; 20∶5) at 28 ℃, a new gene which shares high sequence similarity with reported ω-3 fatty acid desaturases was screened in GenBank. After bioinformatics analysis, the new sequence was expressedinSaccharomycescerevisiaein codon optimized form. Its activity and substrate specificity was investigated through exogenous feeding different fatty acid substrates at different temperature. As a result, the new sequence-encoded protein named as oAiFADS17 could desaturate not only 18C PUFAs but also 20C PUFAs, with a strong preference for AA (arachidonic acid; 20∶4). The new desaturase was able to convert the ω-6 AA to the ω-3 EPA with a substrate conversion rate of 46.3% at 28 ℃. The enzymatic conversion rate for substrates with different carbon length at 28 ℃ was higher than that at 12 ℃. In this research, a new ω-3 desatutase oAiFADS17 with specific catalyzing function for 20C PUFAs, especially AA at 28 ℃, was successfully identified. The new enzyme provide theoretical basis for constructing EPA cell factory and its industrial production.
ω-3 desaturases; conversion rate; eicosapentaenoic acid (EPA)
10.13995/j.cnki.11-1802/ts.201608006
碩士研究生(陳海琴教授為通訊作者,E-mail:haiqinchen@jiangnan.edu.cn)。
國家自然科學基金(21276108)
2015-1-18,改回日期:2016-2-19

