胡蘿卜肉桂醇脫氫酶基因的克隆及其對非生物脅迫的響應
劉堰珺,馬 靜,王廣龍,黃 蔚,王 楓,熊愛生
?
胡蘿卜肉桂醇脫氫酶基因的克隆及其對非生物脅迫的響應
劉堰珺,馬靜,王廣龍,黃蔚,王楓,熊愛生*
(南京農業大學作物遺傳與種質創新國家重點實驗室,園藝學院,農業部華東地區園藝作物生物學與種質創制重點實驗室,南京210095)
該實驗采用RT-PCR技術,從胡蘿卜‘黑田五寸’中克隆獲得了編碼肉桂醇脫氫酶(Cinnamyl alcohol dehydrogenase, CAD)的基因DcCAD。DcCAD序列長1 074 bp,編碼357個氨基酸。親水/疏水性分析表明,DcCAD屬于親水性蛋白。系統進化分析顯示,DcCAD與番茄CAD(XP_010314515.1)親緣關系最近。熒光定量PCR結果顯示,DcCAD基因在胡蘿卜根、葉片和葉柄中的表達量差異顯著,其相對表達量為葉片>根>葉柄。DcCAD基因對高溫(38 ℃)、低溫(4 ℃)、干旱(20% PEG)和鹽(0.2 mol·L-1NaCl)脅迫均有響應,尤其對高溫脅迫和低溫脅迫響應明顯,而且高溫處理后1 h和低溫處理后2 h表達量最高。研究推測,DcCAD基因對胡蘿卜抗逆性具有一定的作用。
肉桂醇脫氫酶;同源分析;非生物脅迫;表達分析;胡蘿卜
木質素(lignin)主要分布于木質化的次生細胞壁中,它為植物細胞壁提供了必要的強度、疏水性以及對外界惡劣環境的抗性[1-3]。木質素的生物合成是一個由多種酶參與的復雜調控網絡,肉桂醇脫氫酶(cinnamyl alcohol dehydrogenase, CAD)作為其中的關鍵酶,人們在很早之前就開始對其進行研究[4]。國內外已對植物中肉桂醇脫氫酶的結構性質、相應的功能以及轉基因方面進行了一些研究[5]。
由于肉桂醇脫氫酶對木質素單體的生物合成起著重要的作用,目前,已有多個編碼肉桂醇脫氫酶的基因從不同植物中被成功克隆,如火炬松(Pinustaeda)[6]、九眼獨活(Araliacordata)[7]、紫花苜蓿(Medicagosativa)[8-9]、多年生黑麥草(Loliumperenne)[10]和高粱(Sorghumbicolor)[11]等。研究也證實,調控木質素合成途徑中相關基因的表達,可以控制木質素的生物合成。通過分析楊樹(Populuslaurifolia)[12]、小麥(Triticumaestivum)[13]、二穗短柄草(Brachypodiumdistachyon)[14]中肉桂醇脫氫酶基因的表達模式發現,肉桂醇脫氫酶基因在植物中以多基因家族的形式存在。植物在受到各種生物及非生物脅迫時,其木質素代謝相關基因的表達會發生變化,其木質素含量也會相應改變。
胡蘿卜(DaucuscarotaL.)是傘形科胡蘿卜屬的一種重要蔬菜,在世界各地廣泛栽培。胡蘿卜以肉質根為食用部位,含有豐富的營養物質和水溶性纖維[15]。作為根用型蔬菜,胡蘿卜肉質根中木質素含量的增加會影響胡蘿卜的品質。目前,有關傘形科蔬菜作物中木質素代謝相關研究報道很少。
本研究以胡蘿卜‘黑田五寸’為實驗材料,基于轉錄組和基因組數據,通過RT- PCR方法克隆出木質素合成關鍵酶—肉桂醇脫氫酶的基因(DcCAD),并對其進行了較為詳盡的分析。通過實時定量PCR技術,研究DcCAD基因在胡蘿卜不同組織及不同逆境處理下的表達情況,為進一步深入研究胡蘿卜中肉桂醇脫氫酶的功能及應用奠定基礎。
1 材料和方法
1.1材料
胡蘿卜‘黑田五寸’于2015年10月份種植于南京農業大學作物遺傳與種質創新國家重點實驗室人工氣候室。胡蘿卜植株種植在蛭石和有機質1∶1的混合基質中,光照和溫度設定為:14 h 25 ℃光照以及10 h 18 ℃黑暗,相對濕度60%~70%以及240 μmol·m-2·s-1光照強度。
大腸桿菌菌株DH5α由本實驗室保存;質粒載體pMD19-T vector simple、ExTaq酶、反轉錄酶MluI、DL marker 2000和dNTP熒光定量染料SYBR Green I等均購自大連TaKaRa公司。
1.2方法
1.2.1取樣對‘黑田五寸’2月齡植株分別進行高溫(38 ℃)、低溫(4 ℃)、干旱(20% PEG)和鹽(0.2 mol·L-1NaCl)處理,分別取處理 0、1、2、4、8和24 h的成熟健康葉片,同時取未處理植株葉片為對照。取2月齡‘黑田五寸’植株,剪取其生長健康的葉片用于葉片總RNA提取,反轉錄成cDNA,用于克隆DcCAD基因。取2月齡‘黑田五寸’植株的葉片、葉柄、根為材料進行組織表達分析。每次取樣3個重復,取樣后立即用液氮速凍,并保存于-80 ℃冰箱。
1.2.2RNA提取及cDNA合成用RNA simple Total Kit(Tiangen公司,中國北京)提取總RNA,RNA濃度和質量利用Nanodrop ND-100分光光度計(NanoDrop公司,美國)檢測。利用Prime Script RT reagent Ki(TaKaRa公司,中國大連)將提取的總RNA反轉錄成cDNA。所有cDNA樣品稀釋10倍用于PCR。
1.2.3胡蘿卜DcCAD基因序列的獲得與克隆基于本實驗室測定的胡蘿卜轉錄組數據和基因組數據[16],拼接得到DcCAD基因的序列,并設計正向引物DcCAD-F(5′-ATGGCTCAAACAACTCCCAATCACA-3′)和反向引物DcCAD-R(5′-CTATGGTTTTCCAGCAATATCAAT-3′)。以2月齡‘黑田五寸’新鮮主根cDNA第一鏈為模板進行擴增,反應條件為94 ℃預變性4 min;94 ℃變性30 s;54 ℃退火30 s;72 ℃延伸1 min;35個循環;72 ℃延伸10 min。利用1.2%瓊脂糖凝膠電泳分離擴增產物,回收后連接到pMD19-T simple載體上,并轉化到大腸桿菌DH5α。隨后提取質粒進行PCR鑒定,委托南京金斯瑞生物科技有限公司進行DNA測序。
1.2.4序列分析不同植物相關基因和氨基酸序列均來自NCBI數據庫;利用BioXM 2.6軟件對DcCAD基因的可閱讀框長度進行預測,并翻譯成對應的氨基酸序列。通過DNAMAN進行氨基酸序列比對及親水性/疏水性分析,然后用Mega 5構建進化樹和編輯并生成報告圖形[17]。蛋白質基本性質分析使用ExPASy相關軟件完成[18]。通過SWISS-MODEL(http://swiss-model.expase.org)預測DcCAD蛋白的三維結構。
1.2.5實時定量PCR反應實時定量PCR采用SYBR PremixExTaq試劑盒(大連TaKaRa公司),在ABI 7300熒光定量PCR儀上完成,采用iQTM5 Real-time PCR System完成,按照操作說明進行。相對定量方法參照2-ΔΔCt法。以胡蘿卜Tubulin為參照基因[19],引物為DcA15-F(5′-GAGTGGAGTTACCTGCTGCCTTC-3′)和DcA15-R(5′-ATGTAGACGAGGGAACGGAATCCAG-3′)。DcCAD基因表達分析引物為DcCAD-F(5′-CGGATAAGCCTCTGGACCTACCT-3′)和DcCAD-R(5′-TTGACAACTTCGATACCACACCTGAT-3′),目標基因與Tubulin一起擴增。實驗進行3次生物學重復,對數值進行了差異顯著水平分析。
1.2.6數據分析采用 Excel 2003和SPSS 16.0進行數據整理和基因表達分析,采用Duncan測驗分析數據差異顯著性。
2 結果與分析
2.1胡蘿卜DcCAD基因克隆
PCR結果(圖1)顯示,以胡蘿卜‘黑田五寸’的cDNA為模版擴增得到1 000 bp左右產物,其長度與
預期相一致。測序結果顯示,DcCAD基因長1 074 bp, 編碼357個氨基酸(圖2),預測其蛋白質相對分子質量為39.09 kD,等電點6.7。
2.2氨基酸序列比對和理化性質分析
對DcCAD的氨基酸序列進行BLASTp同源性檢索,結果(圖)表明:DcCAD的氨基酸序列與擬南芥(Arabidopsisthaliana,NP_195643.1)、黃蒿(Artemisiaannua,ACB54931.1)、茶樹(Camelliasinensis,ACV74414.1)、辣椒(Capsicumannuum,ACF17645.1)、黃瓜(Cucumissativus,XP_004136373.1)、黑麥草(Loliumperenne,AAL99536.1)、蘋果(Malusdomestica,XP_008359757.1)、煙草(Nicotianaattenuata,AFP43763.1)、馬鈴薯(Solanumtuberosum,XP_006356896.1)、小麥(Triticumaestivum,CDM85988.1)、玉米(Zeamays,NP_001136687.1)、水稻(Oryzasativa,NP_001052290.1)、高粱(Sorghumbicolor,XP_002446076.1)、菠菜(Spinaciaoleracea,KNA09004.1)和蓖麻(Ricinuscommunis,XP_011083448.1)等植物的氨基酸序列具有較高的相似度,相似性均在60%以上(圖3)。
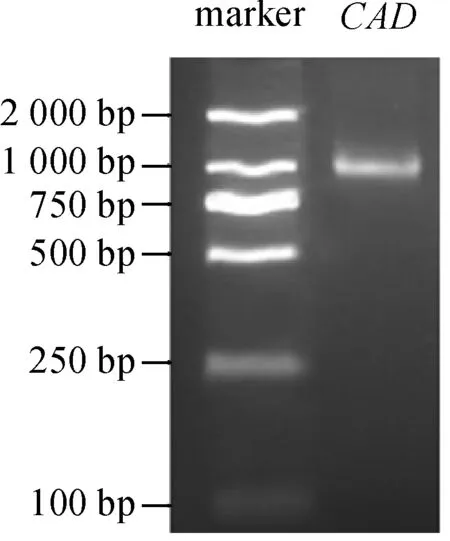
圖1 胡蘿卜DcCAD基因克隆圖譜Fig. 1 PCR amplification of DcCAD gene from carrot

圖2 胡蘿卜DcCAD基因及其編碼氨基酸序列Fig. 2 The nucleotide acid and deduced amino acid sequences of DcCAD gene from carrot
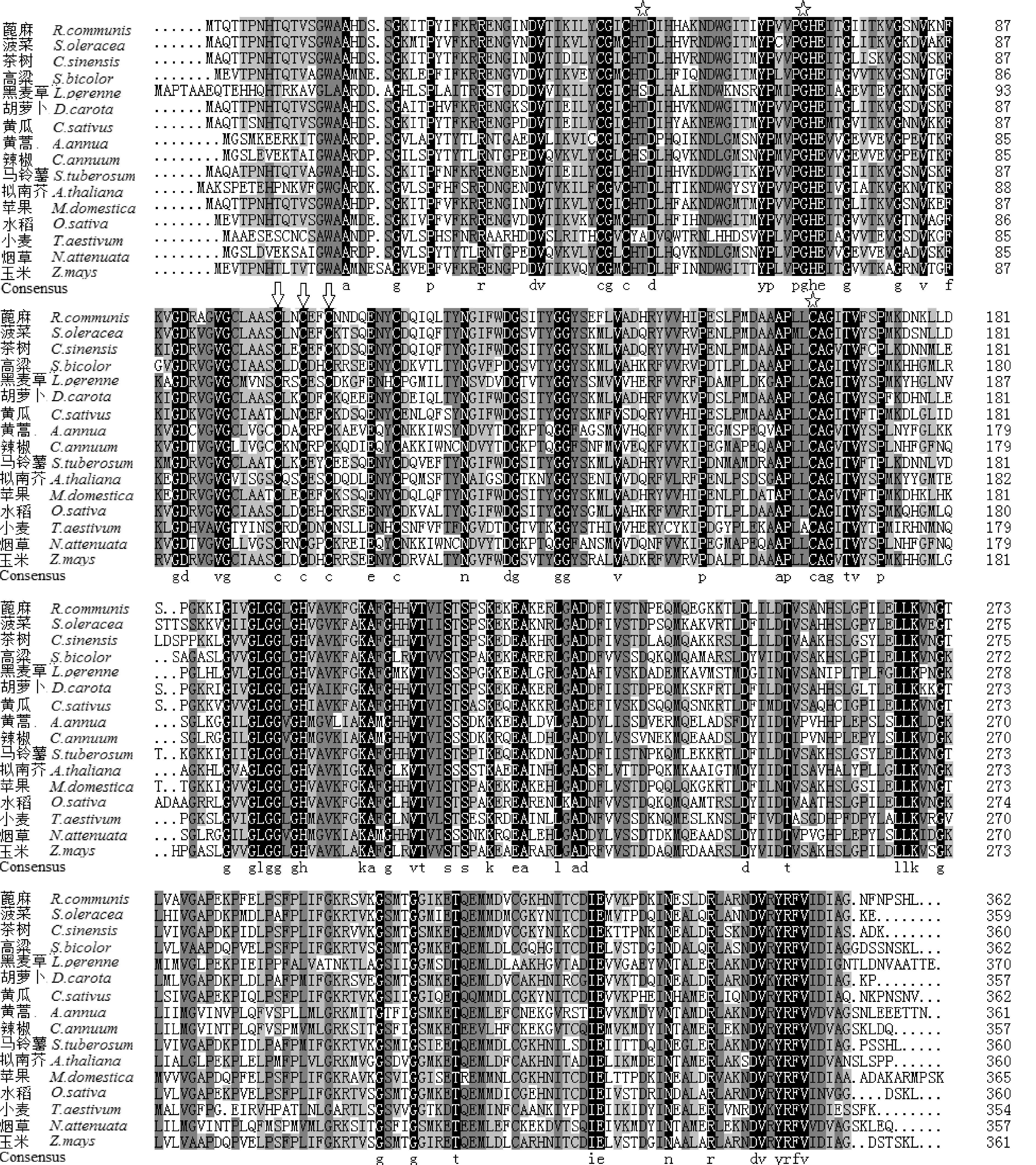
星號表示3個催化鋅結合位點;箭頭表示3個鋅結合位點; CAD蛋白登錄號依次為:蓖麻(XP_011083448.1)、菠菜(KNA09004.1)、茶樹(ACV74414.1)、高粱(XP_002446076.1)、黑麥草(AAL99536.1)、胡蘿卜(ALN38479.1)、黃瓜(XP_004136373.1)、黃蒿(ACB54931.1)、辣椒(ACF17645.1)、馬鈴薯(XP_006356896.1)、擬南芥(NP_195643.1)、蘋果(XP_008359757.1)、水稻(NP_001052290.1)、小麥(CDM85988.1)、煙草(AFP43763.1)和玉米(NP_001136687.1)圖3 胡蘿卜DcCAD的氨基酸序列與其他物種類似蛋白質氨基酸序列的多重比對The asterisk marks the 3 catalytic Zn binding sites; The arrow marks the 3 structural Zn binding sites; The accession numbers of CAD from different plants were: Ricinus communis (XP_011083448.1), Spinacia oleracea (KNA09004.1), Camellia sinensis(ACV74414.1), Sorghum bicolor (XP_002446076.1), Lolium perenne (AAL99536.1), Daucus carota(ALN38479.1), Cucumis sativus (XP_004136373.1), Artemisia annua (ACB54931.1), Capsicum annuum (ACF17645.1), Solanum tuberosum (XP_006356896.1), Arabidopsis thaliana (NP_195643.1), Malus domestica (XP_008359757.1), Oryza sativa (NP_001052290.1), Triticum aestivum (CDM85988.1), Nicotiana attenuata (AFP43763.1), and Zea mays (NP_001136687.1)Fig. 3 Alignment of amino acid sequences of carrot DcCAD and other different plants
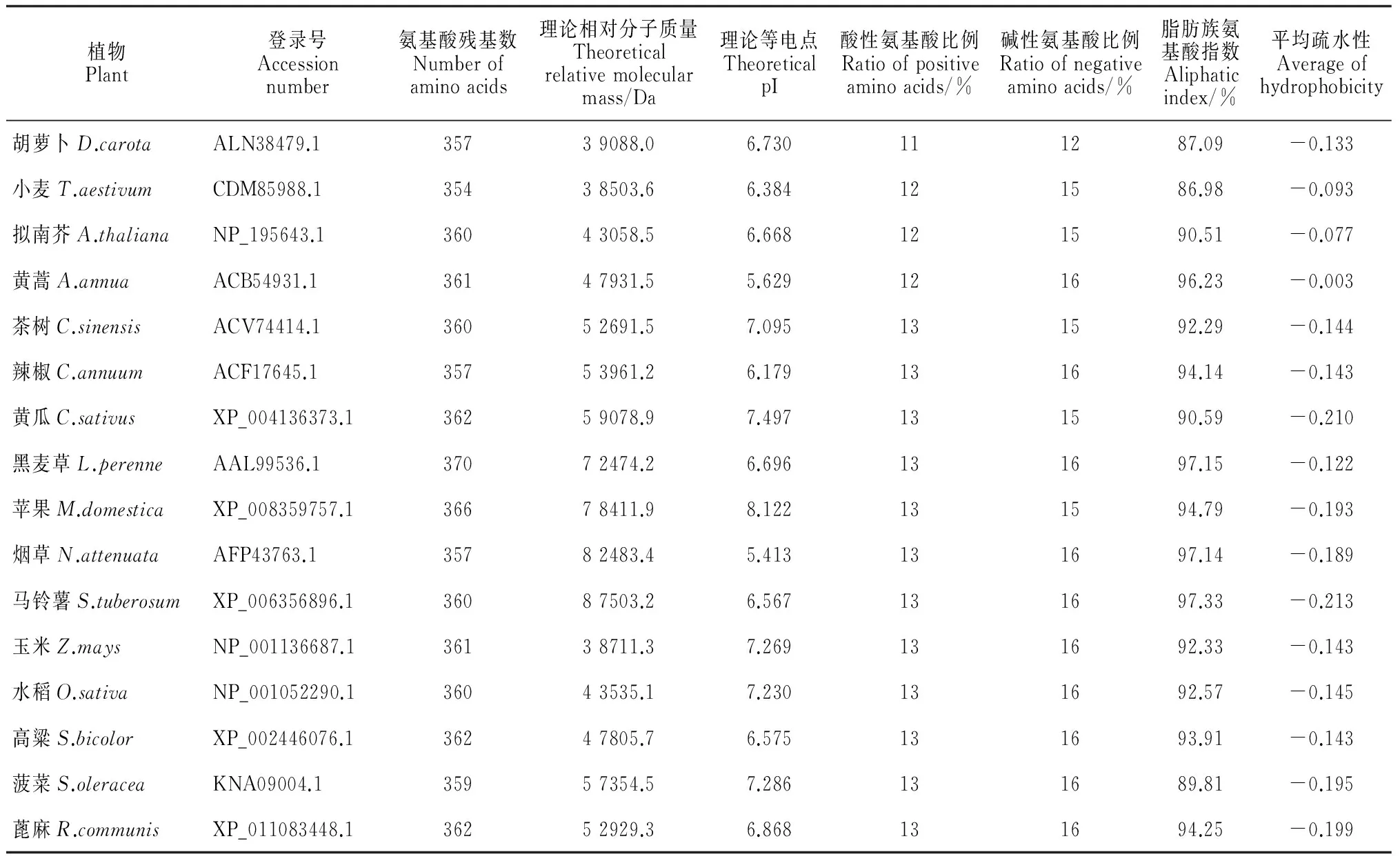
植物Plant登錄號Accessionnumber氨基酸殘基數Numberofaminoacids理論相對分子質量Theoreticalrelativemolecularmass/Da理論等電點TheoreticalpI酸性氨基酸比例Ratioofpositiveaminoacids/%堿性氨基酸比例Ratioofnegativeaminoacids/%脂肪族氨基酸指數Aliphaticindex/%平均疏水性Averageofhydrophobicity胡蘿卜D.carotaALN38479.135739088.06.730111287.09-0.133小麥T.aestivumCDM85988.135438503.66.384121586.98-0.093擬南芥A.thalianaNP_195643.136043058.56.668121590.51-0.077黃蒿A.annuaACB54931.136147931.55.629121696.23-0.003茶樹C.sinensisACV74414.136052691.57.095131592.29-0.144辣椒C.annuumACF17645.135753961.26.179131694.14-0.143黃瓜C.sativusXP_004136373.136259078.97.497131590.59-0.210黑麥草L.perenneAAL99536.137072474.26.696131697.15-0.122蘋果M.domesticaXP_008359757.136678411.98.122131594.79-0.193煙草N.attenuataAFP43763.135782483.45.413131697.14-0.189馬鈴薯S.tuberosumXP_006356896.136087503.26.567131697.33-0.213玉米Z.maysNP_001136687.136138711.37.269131692.33-0.143水稻O.sativaNP_001052290.136043535.17.230131692.57-0.145高粱S.bicolorXP_002446076.136247805.76.575131693.91-0.143菠菜S.oleraceaKNA09004.135957354.57.286131689.81-0.195蓖麻R.communisXP_011083448.136252929.36.868131694.25-0.199
對上述植物進行氨基酸組成成分及理化性質分析,結果(表1)發現,各植物氨基酸殘基數在354~370之間不等;理論相對分子質量的差異較大,理論等電點在5.413~8.122之間;堿性氨基酸所占比例略高于酸性氨基酸,脂肪族氨基酸的比例在86.98%~97.33%之間;蛋白質可溶性預測中平均疏水性為-0.213~-0.003,其中黃蒿的疏水性最強,馬鈴薯的疏水性最弱。
2.3氨基酸親水性/疏水性分析及結構域分析
對DcCAD氨基酸序列進行疏水性/親水性分析,結果(圖4)表明,DcCAD疏水區域中,第187~202位疏水性較強,第165位的丙氨酸(Ala)和第189位、276位的纈氨酸(Val)疏水性最強。親水區域中182~187位親水性較強,第33位的天冬酰胺(Asn)、112位的谷氨酸(Glu)和223位的賴氨酸(Lys)疏水性最強。結果表明,DcCAD屬于親水性蛋白。對胡蘿卜DcCAD編碼的氨基酸序列進行Blastp同源性檢索,結果顯示胡蘿卜DcCAD屬于MDR超家族,歸為CAD1家族,具有多個Zn結合位點,屬于多結構域蛋白序列。

圖4 胡蘿卜DcCAD氨基酸序列的親水性和疏水性Fig. 4 Predicted hydrophilicity and hydrophobicty of deduced amino acid sequence of DcCAD from carrot
2.4蛋白三級結構預測和進化分析
胡蘿卜DcCAD進行蛋白質三級結構同源建模分析的結果如圖5所示。胡蘿卜DcCAD蛋白有9個α螺旋和16個β折疊,與擬南芥的CAD蛋白有較高的相似性。通過Blastp檢索,將DcCAD的氨基酸序列與GenBank已報道的其他植物的CAD蛋白序列進行比對,用Mega 5構建系統進化樹(圖6)。

圖6 胡蘿卜DcCAD與其他植物CAD的系統進化樹Fig. 6 Phylogenetic tree of DcCAD from carrot and CAD from other species
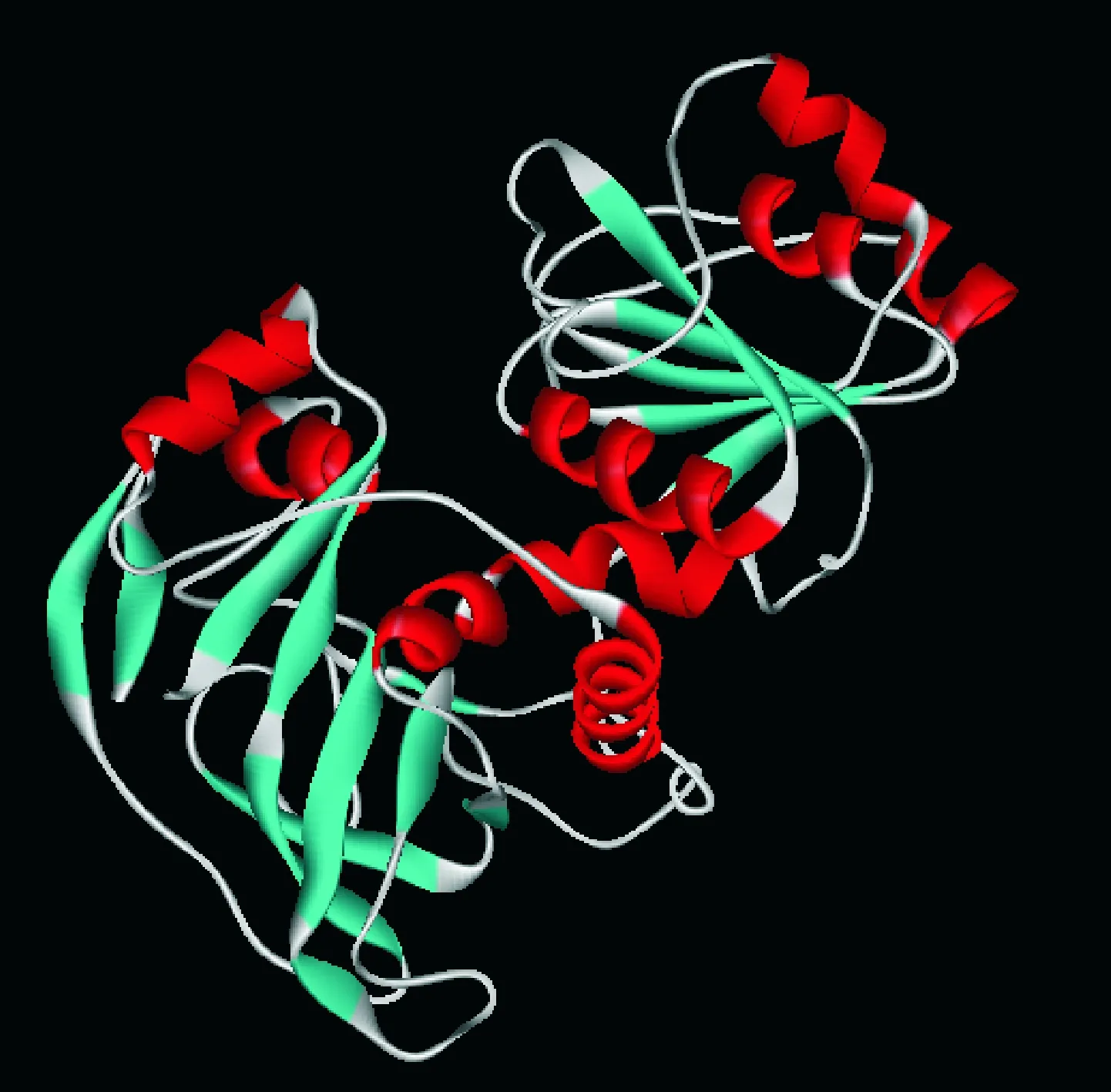
圖5 胡蘿卜DcCAD三級結構Fig. 5 The three-dimension structural model of DcCAD from carrot
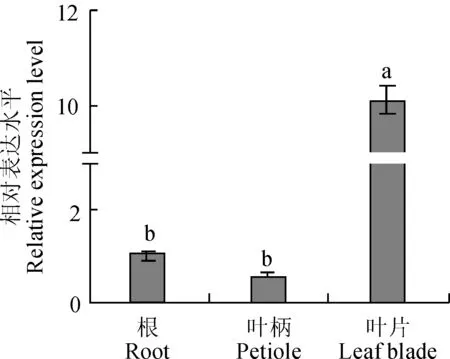
圖中小寫字母表示0.05水平差異顯著性圖7 胡蘿卜DcCAD基因在不同組織中的表達情況The different letters mean significant difference at 0.05 levelFig. 7 Expression analysis of the DcCAD gene in different tissues of carrot
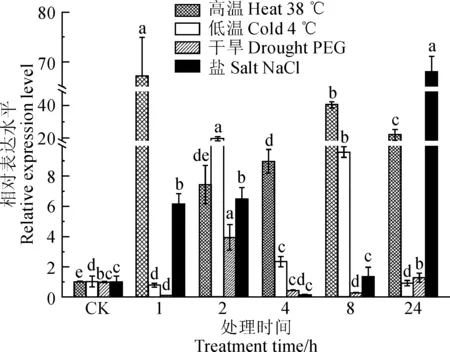
豎條上方不同字母代表同一處理在0.05水平上的差異顯著性圖8 胡蘿卜DcCAD基因在不同逆境以及不同時間段處理的表達Different letters on the vertical bars indicate significant difference in the same treatment at 0.05 levelFig. 8 Expression profiles of the DcCAD gene under different abiotic stress treatments in carrot
結果表明:胡蘿卜與茄科的番茄親緣關系最近,兩者氨基酸相似性達78.89%。與菠菜、茶樹、葡萄CAD進化關系較近,與薔薇科的蘋果、胡麻科的芝麻、山茶科的蓖麻以及楊柳科的楊樹和毛果楊CAD進化關系較遠,與葫蘆科的黃瓜、甜瓜以及桉樹CAD進化關系最遠。
2.5胡蘿卜DcCAD基因在不同組織及不同非生物脅迫條件下的表達
以胡蘿卜‘黑田五寸’的根、葉柄和葉片為材料,通過熒光定量PCR檢測DcCAD基因在胡蘿卜不同組織中的表達情況。結果(圖7)顯示,DcCAD基因在胡蘿卜不同組織中的表達量不同,且差異較大。葉片中表達量高于葉柄和根,葉片中表達量分別是根的10倍、葉柄的17.7倍。
用熒光定量PCR方法測定胡蘿卜‘黑田五寸’在高溫(38 ℃)、低溫(4 ℃)、干旱(20% PEG)和鹽(0.2 mol·L-1NaCl)4種脅迫處理后的表達情況。結果(圖8)顯示,38 ℃處理下,1、2、4、8和24 h表達量均較高,分別是未處理對照的65.8、7.28、8.79、39.66和21.47倍。4 ℃處理下隨著處理時間的延長,DcCAD基因的表達量呈現低-高-低的趨勢,且處理2 h后表達量最高,是對照的18.75倍。20% PEG處理下,除2 h和24 h外,其余處理時間的表達低于對照,2 h表達量達到峰值,是對照的3.95倍。鹽處理下,DcCAD基因總體表現為先上升,后下降,再上升,并于24 h達到峰值。
3 討 論
肉桂醇脫氫酶在植物木質素生物合成中發揮著重要的作用,其活性的高低影響著植物木質素的含量和木質素單體的組成[20-21]。目前已經從多種植物中分離編碼肉桂醇脫氫酶基因,并通過轉基因方法對其功能進行了驗證。本研究以‘黑田五寸’胡蘿卜為研究對象,克隆獲得編碼肉桂醇脫氫酶基因DcCAD。氨基酸理化性質分析結果顯示,DcCAD屬親水性蛋白,但不同物種間的氨基酸理化性質存在一定差異,這可能與其參與不同的逆境調控有關[22]。通過對胡蘿卜DcCAD基因及編碼蛋白的同源性分析發現,與DcCAD相似性最高的是番茄CAD。DcCAD與葡萄科、山茶科、藜科的CAD相似性較高,與單子葉禾本科、葫蘆科、楊柳科的CAD親緣關系較遠。植株中的CAD在不同科之間保守性并不高,DcCAD與其他物種同源基因的相似性只有60 %左右。本研究通過對不同物種CAD進化關系進行分析,為探討植物中CAD起源和進化提供一定的借鑒。
植物對外界環境變化的調控網絡十分復雜,當處于逆境條件下時,各組織器官會行使其特定的生理生化功能,產生相應的應對機制[23-25]。木質化過程也是植物響應逆境脅迫的一種方式,它可能被臭氧、重金屬、干旱、紫外線、機械損傷、病蟲害等逆境所誘導[26-29]。肉桂醇脫氫酶通過將醛類轉化為相應的醇類來參與木質素的生物合成,同時它還與植物的逆境應答有關[26,30-31]。當植株受到外界環境的傷害時,會合成木質素來響應逆境脅迫。本研究中對胡蘿卜植株進行高溫、低溫、干旱、鹽脅迫處理,發現DcCAD基因對非生物脅迫響應明顯。此外DcCAD基因在胡蘿卜不同組織中的表達量不同,在葉片中的表達量最高,葉柄中的表達量最低。這些結果說明胡蘿卜DcCAD基因對逆境脅迫有響應,由此可推測它對胡蘿卜的抗逆性起著一定的作用。
[1]PETER G, NEALE D. Molecular basis for the evolution of xylem lignification[J].CurrentOpinioninPlantBiology, 2004, 7(6): 737-742.
[2]GUO D M, RAN J H, WANG X Q. Evolution of the cinnamyl/sinapyl alcohol dehydrogenase (CAD/SAD) gene family: The emergence of real lignin is associated with the origin of bona fide CAD[J].JournalofMolecularEvolution, 2010, 71(3): 202-218.
[3]WENG J K, CHAPPLE C. The origin and evolution of lignin biosynthesis[J].NewPhytologist, 2010, 187(2):273-285.
[4]張魯斌, 谷會, 弓德強, 等. 植物肉桂醇脫氫酶及其基因研究進展[J].西北植物學報, 2011, 31(1):204-211.
ZHANG L B, GU H, GONG D Q,etal. Research progress of cinnamyl alcohol dehydrogenase and its gene[J].ActaBotanicaBoreali-OccidentaliaSinica, 2011, 31(1):204-211.
[5]曹佳強, 李波, 楊洋, 等. 木質素生物合成中肉桂醇脫氫酶基因(CAD)的研究進展[J].分子植物育種, 2014,10(5):1 034-1 043.
CAO J Q, LI B, YANG Y,etal. Advanced progress in the cinnamyl alcohol dehydrogenase gene involved in lignin biosynthesis[J].MolecularPlantBreeding, 2014, 10(5):1 034-1 043.
[6]O’MALLEY D M, PORTER S, SEDEROFF R R. Purification, characterization, and cloning of cinnamyl alcohol dehydrogenase in loblolly pine (PinustaedaL.)[J].PlantPhysiology, 1992, 98(4), 1 364-1 371.
[7]HIBINO T, SHIBATE D, CHEN J Q,etal. Cinnamyl alcohol dehydrogenase fromAraliacordata: cloning of the cDNA and expression of the gene in lignified tissues[J].PlantCellPhysiology, 1993, 34(5): 659-665.
[8]BRILL E M, ABRAHAMS S, HAYES C M,etal. Molecular characterization and expression of a wound-inducible cDNA encoding a novel cinnamyl-alcohol dehydrogenase enzyme in lucerne (MedicagosativaL.)[J].PlantMolecularBiology, 1999, 41(2): 279-291.
[9]VAN DOORSSELAERE J, BAUCHER M, FEUILLET C,etal. Isolation of cinnamyl alcohol dehydrogenase cDNAs from two important economic species: alfalfa and poplar. Demonstration of a high homology of the gene within angiosperms[J].PlantPhysiologyandBiochemistry, 1995, 33(1): 105-109.
[10]LYNCH D, LIDGETT A, MCINNES R,etal. Isolation and characterisation of three cinnamyl alcohol dehydrogenase homologue cDNAs from perennial ryegrass (LoliumperenneL.)[J].JournalofPlantPhysiology, 2002, 159(6):653-660.
[11]TSURUTA S, EBINA M, NAKAGAWA H,etal. Isolation and characterization of cDNA encoding cinnamyl alcohol dehydrogenase (CAD) in sorghum (Sorghumbicolor(L.) Moench)[J].JapaneseSocietyofGrasslandScience, 2007, 53(2): 103-109.
[12]EUDES A, GEORGE A, MUKERJEE P,etal. Biosynthesis and incorporation of side-chain-truncated lignin monomers to reduce lignin polymerization and enhance saccharification[J].PlantBiotechnologyJournal, 2010, 10(5): 609-620.
[13]MA Q H. Functional analysis of a cinnamyl alcohol dehydrogenase involved in lignin biosynthesis in wheat[J].JournalofExperimentalBotany, 2012, 61(10): 2 735-2 744.
[14]BUKH C, NORD-LARSEN P H, RASMUSSEN S K. Phylogeny and structure of the cinnamyl alcohol dehydrogenase gene family inBrachypodiumdistachyon[J].JournalofExperimentalBotany, 2012, 63(17): 6 223-6 236.
[15]莊飛云, 歐承剛, 趙志偉, 等. 胡蘿卜育種回顧及展望[J].中國蔬菜, 2008, 15(3): 41-44.
ZHUANG F Y, OU C G, ZHAO Z W,etal. Review and prospect of carrot breeding[J].ChinaVegetables, 2008, 15(3):41-44.
[16]XU Z S, TAN H W, WANG F,etal. CarrotDB: a genomic and transcriptomic database for carrot[J].DatabasetheJournalofBiologicalDatabases&Curation, 2014, 40 (5):1-8.
[17]TAMURA K, PETERSON D, PETERSON N,etal. MEGA5: molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods[J].MolecularBiologyandEvolution, 2011, 28(10): 2 731-2 739.
[18]GASTEIGER E, GATTIKER A, HOOGLAND C,etal. ExPASy: the proteomics server for in-depth protein knowledge and analysis[J].NucleicAcidsResearch, 2003, 31(13): 3 784-3 788.
[19]TIAN C, JIANG Q, WANG F,etal. Selection of suitable reference genes for qPCR normalization under abiotic stresses and hormone stimuli in carrot leaves[J].PLOSOne, 2015, 10(2):128-136.
[20]PILATE G, GUINEY E, HOLT K. Field and pulping performances of transgenic trees with altered lignification[J].NatureBiotechnology,2002, 20(4):607-612.
[21]CHABANNES M, BARAKATE A, LAPIERRE C,etal. Strong decrease in lignin content without significant alteration of plant development is induced by simulataneous downregulation of cinnamoyl CoA reductase (CCR) and cinnamyl alcohol dehydrogenase (CAD) in tobacco plants[J].PlantJournal, 2001, 28(3):257-270.
[22]YAMAGUCHI-SHINOZAKI K, SHINOZAKI K. Transcriptional regulatory networks in cellular responses and tolerance to dehydration and cold stresses[J].AnnualReviewofPlantBiology, 2006, 57(6): 781-803.
[23]HU Y, LI W C, XU Y Q,etal. Differential expression of candidate genes for lignin biosynthesis under drought stress in maize leaves[J].JournalofAppliedGenetics, 2009, 50(3): 213-223.
[24]ZELLER G, HENZ S R, WIDMER C K,etal. Stress-induced changes in theArabidopsisthalianatranscriptome analyzed using whole-genome tiling arrays[J].PlantJournal, 2009, 58(6): 1 068-1 082.
[25]WALLEY J W, DEHESH K. Molecular mechanisms regulating rapid stress signaling networks inArabidopsis[J].JournalofIntegrativePlantBiology, 2010, 52(4): 354-359.
[26]RICHET N, TOZO K, ATIF D,etal. The response to daylight or continuous ozone of phenylpropanoid and lignin biosynthesis pathways in poplar differs between leaves and wood[J].Planta, 2012, 236(2): 727-737.
[27]BETZ G, KNAPPE C, LAPIERRE C,etal. Ozone affects shikimate pathway transcripts and monomeric lignin composition in European beech (FagussylvaticaL.)[J].EuropeanJournalofForestResearch, 2009, 128(2):109-116.
[28]TRONCHET M, BALAGUé C, KROJ T,etal. Cinnamyl alcohol dehydrogenases-C and D, key enzymes in lignin biosynthesis, play an essential role in disease resistance inArabidopsis[J].MolecularPlantPathology, 2010, 11(1):83-92.
[29]ZHAO Q, DIXON R A. Transcriptional networks for lignin biosynthesis: more complex than we thought[J].TrendsinPlantScience, 2011, 16(4): 227-233.
[30]BI C, CHEN F, JACKSON L,etal. Expression of lignin biosynthetic genes in wheat during development and upon infection by fungal pathogens[J].PlantMolecularBiologyReporter, 2011, 29(1): 149-161.
[31]KAUR H, SHAKER K, HEINZEL N,etal. Environmental stresses of field growth allow cinnamyl alcohol dehydrogenase-deficientNicotianaattenuataplants to compensate for their structural deficiencies[J].PlantPhysiology, 2012, 159(4): 1 545-1 570.
(編輯:宋亞珍)
Cloning and Expression Profile Analysis of the Gene Encoding Cinnamyl Alcohol Dehydrogenase under Abiotic Stress in Carrot
LIU Yanjun, MA Jing, WANG Guanglong, HUANG Wei, WANG Feng, XIONG Aisheng*
(State Key Laboratory of Crop Genetics and Germplasm Enhancement, Ministry of Agriculture Key Laboratory of Biology and Germplasm Enhancement of Horticultural Crops in East China, College of Horticulture, Nanjing Agricultural University, Nanjing 210095, China)
Here, the gene encoding DcCAD were cloned from carrot cultivar ‘Kurodagosun’ with RT-PCR method. The length ofDcCADwas 1 074 bp, and encoding 357 amino acids. The DcCAD may belong to hydrophilic protein. Phylogenetic analysis showed that DcCAD closed to CAD from tomato(XP_010314515.1). Quantitative real-time PCR analysis showed that the expression profiles of theDcCADgene are significantly different in different tissues (root, leaf blade, and petiole) of carrot. Relative expression level of theDcCADgene is leaf blade>root>petiole. TheDcCADgene is responsive to various abiotic stresses, such as heat (38 ℃), cold (4 ℃), drought (20% PEG) and salt (0.2 mol·L-1NaCl). The expression level was significantly increased after high temperature or low temperature treatments, respectively. The expression levels peaked at 1 h heat treatment and 2 h cold treatment, respectively. These results suggested thatDcCADgene participate in the response process of carrot under the abiotic stress.
cinnamyl alcohol dehydrogenase; homologous analysis; abiotic stress; expression analysis;Daucuscarota
1000-4025(2016)07-1294-08
10.7606/j.issn.1000-4025.2016.07.1294
2016-04-20;修改稿收到日期:2016-06-13
江蘇省自然科學基金杰出青年基金(BK20130027);江蘇高校優勢學科建設項目(PAPD)
劉堰珺(1993-),女,碩士研究生,主要從事蔬菜分子生物學研究。E-mail: 2015104076@njau.edu.cn
熊愛生,教授、博士生導師,主要從事蔬菜遺傳育種與分子生物學研究。E-mail: xiongaisheng@njau.edu.cn
Q785; Q786; S631.2
A

