藥用動物烏梢蛇線粒體基因組全序列分析*
劉 杰,田曉軒,崔 英,朱 彥
(天津中醫藥大學,天津市現代中藥重點實驗室-省部共建國家重點實驗室(培育),天津 300193)
?
·中藥研究·
藥用動物烏梢蛇線粒體基因組全序列分析*
劉杰,田曉軒,崔英,朱彥
(天津中醫藥大學,天津市現代中藥重點實驗室-省部共建國家重點實驗室(培育),天津300193)
摘要:[目的]獲取藥用動物烏梢蛇線粒體基因組全序列。[方法]采用Ion Torrent PGM技術,對烏梢蛇全基因組DNA進行提取測序,采用MIRA方法從頭組裝方法和近緣種mapping方法進行組裝拼接,應用MITOS等方法進行注釋。[結果]獲得17 162 bp烏梢蛇線粒體基因組全序列。其共編碼22個tRNA,13個蛋白編碼基因,2個rRNA (16S rRNA、12S rRNA)并有1個輕鏈(L鏈)復制起始區。[結論]獲取烏梢蛇線粒體基因組全序列,并可用于該藥用動物的資源調查與保護研究。
關鍵詞:烏梢蛇;線粒體基因組;線粒體DNA;序列分析
烏梢蛇Ptyas dhumnades(Cantor,1842)在分類上屬于蛇亞目(Serpentes)游蛇科(Colubridae)游蛇亞科(Colubrinae)鼠蛇屬(Ptyas)。歷史上,其異名包括Zaocys dhumnad(Cope,1860),Ablabes vittatus(Duméril,Bibron&Duméril,1854)及Coluberdhumnades(Cantor,1842)等,后被歸入Ptyas屬[1-2]。本物種俗名中國鼠蛇(Chineseratsnake)、大眼鼠蛇(Big-eyedratsnake),或俗稱過山刀。該物種主要分布于中國,生存于海拔50~1 570 m的區域,在河北、河南、山西、陜西、甘肅、四川、湖北、湖南、江西、江蘇、福建、云南、貴州、廣東、廣西、臺灣等省份均有發現[3]。烏梢蛇屬無毒蛇類,其分類除去內臟的干燥體,有祛風、通絡、止痙的功效,常用于治療風濕頑痹、抽搐痙攣、瘰癘惡瘡等癥[4]。其分類特征包括頭部鱗呈黑褐色,脊背聳立成屋脊狀,背鱗有端窩兩個,行數為偶數等[5-6]。近年來,基于CYTB及CO1基因序列以及高特異性聚合酶鏈式反應(PCR)的烏梢蛇分子鑒定方法也得到建立與應用[7-9]。
動物線粒體基因組包含了從分子序列到基因結構的全面信息,其基因組成排列相對保守,具母系遺傳特征,且易于擴增測序[10],被廣泛用于動物系統發育,以及種群保護等領域研究。蛇類的線粒體基因組編碼2個rRNA、22個左右的tRNA和13個蛋白,1~2個非編碼的控制區,并常包括1個輕鏈復制起始區。本文采用Ion torrent PGM二代測序技術[11],獲得了烏梢蛇線粒體DNA全序列,此為國內外有關鼠蛇屬動物線粒體基因組的首次公開報道。
1 材料與方法
1.1動物烏梢蛇購于福建福州,并經形態學及CO1基因分子鑒定。取新鮮肌肉組織立即使用。Voucher ID為wss-1。
1.2總DNA提取參照經典的酚氯仿抽提方法,獲得總基因組DNA,并經A260/A280粗略估計濃度及純度。
1.3Ion Torrent PGM高通量測序
1.3.1鳥槍法建庫鳥槍法是指將目的DNA隨機打斷成大小不同的片段,再將這些片段的序列連接起來的測序方法。
1.3.2OneTouch(油包水PCR) 每個微小磁珠通過接頭連接相應的DNA片段,然后把含有DNA片段的磁珠和擴增試劑的溶液注入礦物油中,形成了油包水的小體系,使得每個小水滴只含一個磁珠,每個小水滴就是一個微反應體系。通過擴增,DNA片段可以增加幾百萬個拷貝,并且都連接在磁珠上。
1.3.3ES富集油包水PCR之后,加入Dynabeads? MyOneTM Streptavidn C1 beads,它通過親和素-生物素與帶有雙鏈模板的磁珠結合,最后加入氫氧化鈉(NaOH)使得雙鏈解鏈,制備單鏈模板。
1.3.4PGM上機測序將帶有單鏈模板的磁珠、測序引物、DNA聚合酶混勻,離心使其進入318芯片的微孔中,然后上機測序。
1.4序列拼接從測序服務器上下載鳥槍法文庫測序的原始數據至本地,預處理過濾掉低質量序列、過短序列以及錯誤序列,根據目的基因組大小(約16K bp),分割文件,調整合適的覆蓋倍數(基因組大小的20X為佳),使用MIRA 4.0進行de novo拼接,結合近緣物種mapping組裝,經反復迭代補全控制區(重復區)后,進行最高質量堿基合意,得到合意后的完整線粒體全基因組序列。
1.5注釋分析和提交使用MITOS server線粒體在線注釋系統[12],對全序列進行注釋,結合tRNAscan-SE 1.21軟件,進行tRNA定位。以游蛇亞科Elaphe anomala,Elaphe bimaculata,Oligodon ningshaanensis 和Euprepiophis perlacea的線粒體基因組全序列為參照,對蛋白質編碼基因、tRNA、rRNA及控制區等進行注釋,L鏈復制起始區二級結構由RNA structure折疊生成。使用軟件MEGA 4.0及Bioedit等統計序列總長、堿基組成和GC含量等。完整的烏梢蛇線粒體基因組全序列經Sequin注釋后,提交GenBank,登錄號為KR812112。
2 結果
2.1序列拼接結果獲取烏梢蛇線粒體基因組后,經bowtie2比對[13],確認共189 977條序列(reads)可拼接至該基因組,序列平均長度214.4 bp,測序深度達1 958倍。
2.2整體結構烏梢蛇線粒體基因組結構見圖1、表1。與大多數脊椎動物類似,烏梢蛇共編碼37個基因,包括了22個tRNA,13個蛋白編碼基因,2個rRNA(16S rRNA、12S rRNA)并有1個輕鏈(L鏈)復制起始區。其中除ND6和8個tRNA(Ala、Asn、Cys、Gln、Glu、Pro、Ser和Tyr)由L鏈編碼,其余28個基因均由重鏈(H鏈)編碼。其基因的排布順序與大部分游蛇類動物相同。

圖1 烏梢蛇線粒體基因組結構圖
2.3堿基組成烏梢蛇線粒體基因組全長17162bp,其中L鏈的堿基含量為:A=5 932 bp(34.6%),C=4 843 bp(28.2%),G=2 139 bp(12.5%),T=4 248 bp (24.8%),GC=6982bp(40.7%),A>C>T>G,AT%>GC%。且呈AT偏斜(0.165)和CG偏斜(0.387),這也與蛇類普遍情況相同。
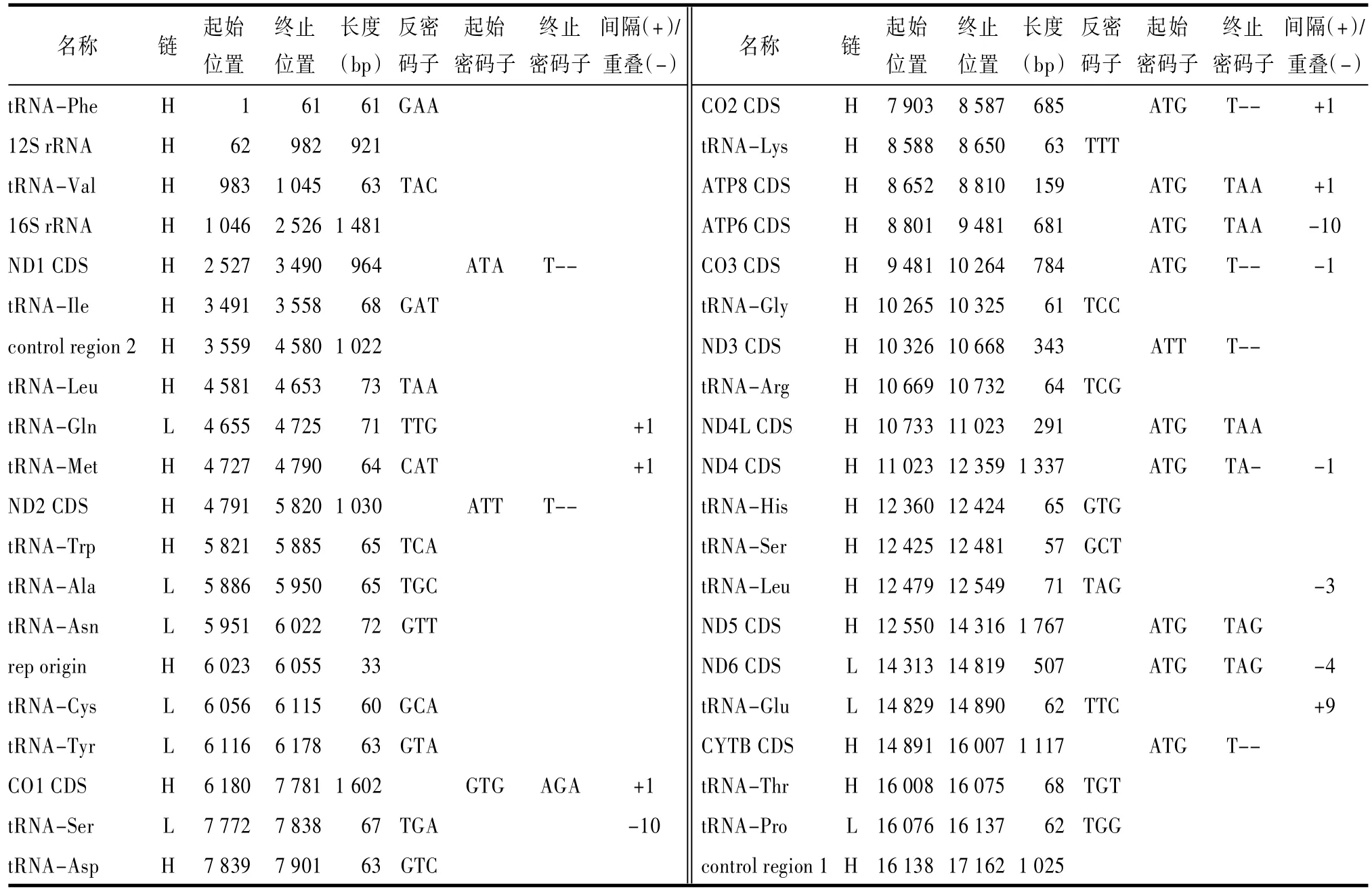
表1 烏梢蛇線粒體基因組結構
2.4蛋白編碼基因烏梢蛇線粒體基因組中蛋白編碼基因全長11 267 bp,約占全基因組的65.65%。A=3 967 bp(35.2%),C=3 298 bp(29.3%),G=1 267 bp (11.2%),T=2 735 bp(24.3%),GC=4 565 bp(40.5%),A>C>T>G,AT%>GC%。所有蛋白編碼基因無內含子。除ND1以ATA為起始密碼子,ND2和ND3以ATT起始,CO1以GTG起始外,其余均使用ATG為起始密碼子。除CO1以AGA為終止密碼子,ND5和ND6以TAG終止,其余蛋白均使用T或TA堿基終止,并通過轉錄過程中3’端的poly A補全,形成完整終止密碼子。氨基酸使用頻率的分析見表2。在一共3 747個氨基酸中,出現頻率較低的為Cys (0.77%)、Asp(1.52%)和Arg(1.65%),而頻率較高的為Leu(14.87%)和Thr(12.09%)。
2.5rRNA和tRNA基因烏梢蛇的12S rRNA和16S rRNA長度分別為921 bp和1 481 bp,GC含量分別為44.7%和40.2%。22個tRNA基因中,最短的為tRNA-Ser(anticodon=GCT,57 bp),最長的為tRNA-Leu(anticodon=TAA,73 bp)。大部分tRNA可折疊成三葉草二級結構,但tRNA-Ser(anticodon= GCT)折疊后缺失D臂,tRNA-Cys缺失TΨC環,見圖2。
2.6非編碼區烏梢蛇的 1號控制區(control region 1)位于tRNA-Pro與tRNA-Phe之間,2號控制區(control region 2)位于tRNA-Ile與tRNA-Leu (anticodon=TAA)之間,長度分別為1 025 bp和1 022 bp,比對后發現兩者一致堿基為1 018 bp,一致率為99.5%。除控制區外,烏梢蛇線粒體基因組中有14 bp的6個非編碼間隔區域,其中最長的間隔區為9 bp,位于ND6和tRNA-Glu之間,顯示該物種線粒體基因組結構較為緊湊。
2.7L鏈復制起始區烏梢蛇基因組的L鏈復制起始區,長度為33 bp,可折疊成穩定的莖環結構,見于圖2C。其中莖由9對堿基組成,環為11個堿基。
3 討論
線粒體全基因組測序方法,在以往多基于PCR技術[14],該方法需根據目標物種的近緣物種,設計合適引物,進行普通PCR或Long-PCR(>4 kb),并對擴增產物進行測序或步移測序。而近年來,隨著高通量測序技術的成熟,測序成本的大幅下降,更多的目光集中在利用總基因組DNA測序結果,從中篩選拼接線粒體基因組序列[15-17]。本實驗即采取后者策略,建立了基于高通量測序結果的線粒體基因組拼接工作流程。此方法可快速、低成本的得到目標物種的高深度線粒體基因組,并避免PCR過程中產生的堿基錯配可能。
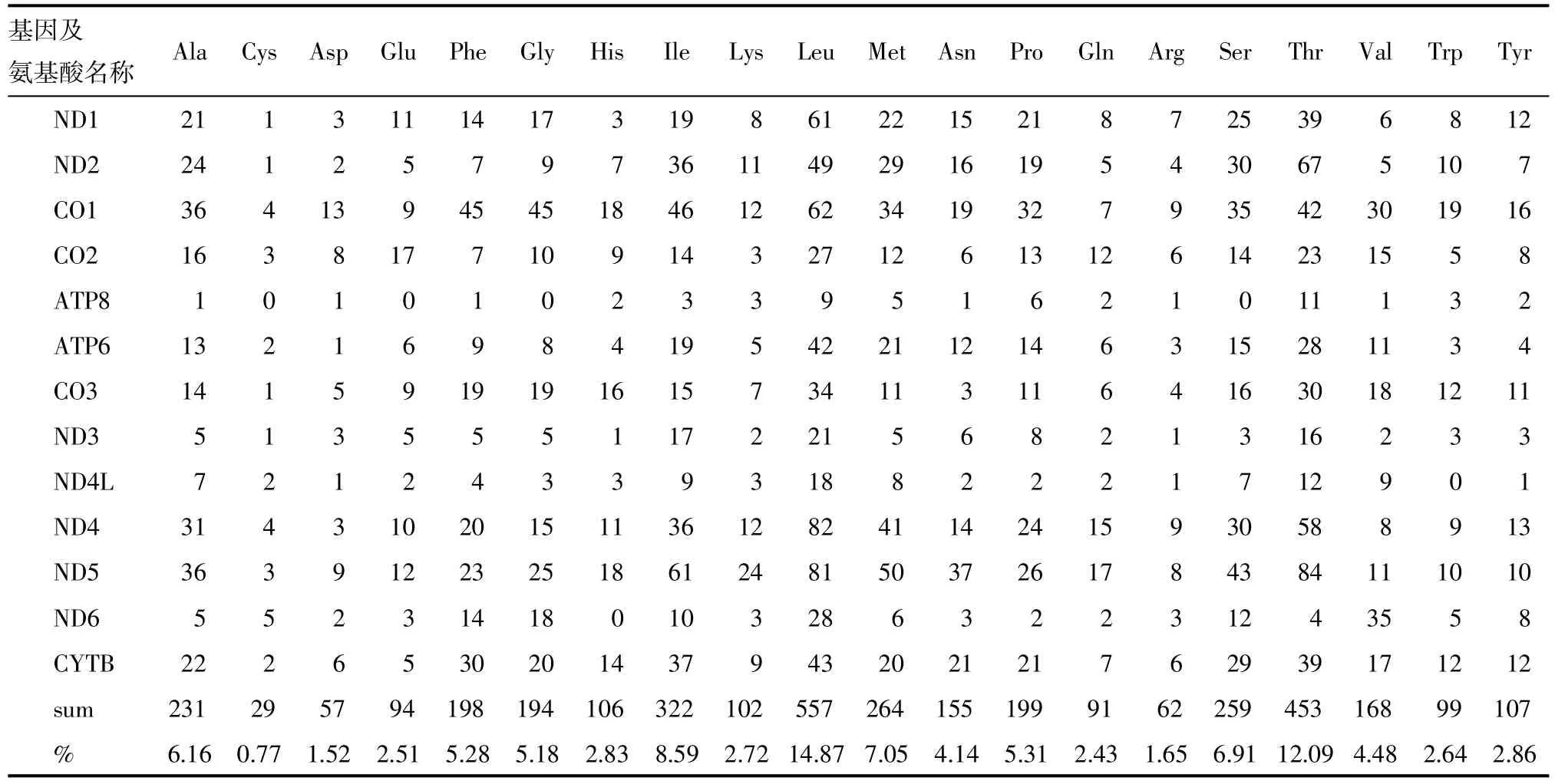
表2 烏梢蛇線粒體基因組蛋白編碼基因氨基酸組成分析

圖2 tRNA-Ser(anticodon=GCT),tRNA-Cys及L鏈復制起始區二級結構圖
在系統發育學研究中,線粒體中蛋白編碼區以及tRNA區的結構變化應用較多。而另一方面,控制區(重復區)序列由于受進化壓力較小,其堿基替換速率較快,這對藥用動物的種下研究尤有意義。與其他真蛇類動物類似,本研究同樣在結構緊湊的線粒體基因組中,發現了兩個控制區序列,其形成原因尚無定論。但已有研究發現,蛇類中這兩個控制區在同一物種內相似度很高,而不同的物種間又有較大的分歧。這提示了其可能是協同進化的結果[18]。
中藥資源的研究是目前中藥研究的熱點問題,較多的工作集中在藥用植物上[19-20],而動物上開展較少。通過線粒體基因組數據的累積,研究以上雙控制區序列在種內(種群間)的變化,也可作為研究藥用蛇類種群分化的分子標記,為藥用蛇類的資源普查及保護提供參考。
參考文獻:
[1]David P,Vogel G.The Snakes of Sumatra:an annotated checklist and key with natural history notes[M].Germany: Frankfurt am Main:Chimaira,1996.
[2]David P,Das I.On the grammar of the gender of Ptyas Fitzinger,1843(Serpentes:Colubridae)[J].Hamaddryad,2004,28(1-2):113-116.
[3]李軍德,黃璐琦,曲曉波.中國藥用動物志[M].福州:福建科學技術出版社,2013.
[4]國家藥典委員會.中華人民共和國藥典(一部)[S].北京:中國醫藥科技出版社,2010.
[5]宋秀菊.動物烏梢蛇的鑒別[J].時珍國醫國藥,2012,23(7): 1824.
[6]林喆,胡麗娜,李娜.動物藥整理研究——烏梢蛇[J].吉林中醫藥,2009,29(11):982-984.
[7]廖婧,梁鎮標,張亮,等.常見藥用蛇類的DNA條形碼研究[J].中國藥學雜志,2013,48(15):1255-1260.
[8]唐曉晶,馮成強,黃璐琦,等.高特異性PCR方法鑒別烏梢蛇及其混淆品[J].中國藥學雜志,2007,42(5):333-336.
[9]王義權,周開亞,徐珞珊,等.中藥材烏梢蛇及其混淆品的DNA序列分析鑒別[J].藥學學報,1999,34(1):68-72.
[10]Wolstenholme DR.Animal mitochondrial DNA:structure and evolution[J].Int Rev Cytol,1992,141(6):173-216.
[11]Morinha F,Clemente C,Cabral JA,et al.Next-generation sequencing and comparative analysis of Pyrrhocorax pyrrhocorax and Pyrrhocorax graculus(Passeriformes:Corvidae)mitochondrial genomes[J].Mitochondrial DNA,2014(28): 1-4.
[12]Bernt M,Donath A,Juhling F,et al.MITOS:improved denovo metazoan mitochondrial genome annotation[J].Mol Phylogenet Evol,2013,69(2):313-319.
[13]Langmead B,Salzberg SL.Fast gapped-read alignment with Bowtie 2[J].Nat Methods,2012,9(4):357-359.
[14]Zardoya R,Suárez M.Sequencing and phylogenomic analysis of whole mitochondrial genomes of animals[J]. Methods in Molecular Biology,2007,422:185-200.
[15]Tang M,Tan M,Meng G,et al.Multiplex sequencing of pooled mitochondrial genomes-a crucial step toward biodiversity analysis using mito-metagenomics[J].Nucleic Acids Res,2014,42(22):e166.
[16]Gillett CP,Crampton-Platt A,Timmermans MJ,et al.Bulk de novo mitogenome assembly from pooled total DNA elucidatesthephylogenyofweevils(Coleoptera:Curculionoidea)[J]. Mol Biol Evol,2014,31(8):2223-2237.
[17]Hahn C,Bachmann L,Chevreux B.Reconstructing mitochondrial genomes directly from genomic next-generation sequencing reads--a baiting and iterative mapping approach[J].Nucleic Acids Res,2013,41(13):e129.
[18]Jiang ZJ,Castoe TA,Austin CC,et al.Comparative mitochondrial genomics of snakes:extraordinary substitution rate dynamics and functionality of the duplicate control region[J].BMC Evol Biol,2007(7):123.
[19]李天祥,李國輝,張春艷,等.天津黃精屬植物的資源考察[J].天津中醫藥,2013,30(12):749-753.
[20]田春雨,薄海美,許靜,等.唐山山區野生藥用植物資源及其分布組成特點的初步研究[J].天津中醫藥,2014,31(1):56-58.
中圖分類號:R284
文獻標志碼:A
文章編號:1673-9043(2016)03-0187-05
DOI:10.11656/j.issn.1673-9043.2016.03.10
收稿日期:(2016-02-24)
*基金項目:國家自然科學基金項目(81202874);高等教育博士學科點專項科研基金(20121210120006);國家科技支撐計劃項目(2012BAI29B01)。
作者簡介:劉杰(1990-),女,碩士研究生,研究方向為中藥學。
通訊作者:朱彥,E-mail:yanzhu.harvard@gmail.com;田曉軒,E-mail:tian_xiaoxuan@tjutcm.edu.cn。
Analysis of mitochondrial genome of Ptyas dhumnades
LIU Jie,TIAN Xiao-xuan,CUI Ying,ZHU Yan
(Tianjin University of Traditional Chinese Medicine,Tianjin State Key Laboratory of Modern Chinese Medicine,Tianjin 300193,China)
Abstract:[Objective]To investigate the complete mitochondrial genome of medical animal Ptyas dhumnades (Cantor,1842).[Methods]The Ion Torrent PGM technique was chosed to sequence genomic DNA of Ptyas dhumnades,and then the strategy of de novo assembly and mapping versu mtgenome of closely related species by MIRA 4.0 was adopted to get the complete mitochondrial sequence.This genome was annotated by Online server MITOS etc.[Results]The 17 162 bp complete mitochondrial genome of Ptyas dhumnade with high coverage contains 22 transfer RNA,13 protein coding genes,2 ribosomal RNA genes and 1 putative L-strand replication origin. [Conclusion]The mtgenome of Ptyas dhumnades was obtained,and could be furher applied in the resources survey and protection of this medical animal.
Key words:Ptyas dhumnades;mitochondrial genome;mitochondrial DNA;sequence analysis

