兒童感染沙門菌耐藥特點及鑒定方法的研究
胡立新 宋文琪 蘇建榮 董 方 王 艷
(首都醫科大學附屬北京兒童醫院檢驗中心,北京 100045)
?
兒童感染沙門菌耐藥特點及鑒定方法的研究
胡立新宋文琪*蘇建榮董方王艷
(首都醫科大學附屬北京兒童醫院檢驗中心,北京 100045)
【摘要】目的 研究引起兒童感染的沙門菌耐藥特點及快速鑒定方法。方法選擇沙門菌75株,使用標準生物化學和血清學試驗對所有菌株進行鑒定;使用16S rRNA基因測序法鑒定;使用基質輔助激光解析電離飛行時間質譜(matrix assisted laser desorption ionization time of flight mass spectrometry, MALDI-TOF-MS)法鑒定,以上兩種方法結果與傳統培養方法結果進行比對分析。使用Kerby-Bauer紙片擴散法對常用抗生素進行敏感性試驗,使用聚合酶鏈反應 (polymerase chain reaction, PCR)測定對廣譜β-內酰胺類藥物耐藥的基因。結果75株沙門菌中,鼠傷寒沙門菌和腸炎沙門菌檢出率最高,分別為32株和26株。16S rRNA基因測序法與傳統培養法的符合率為100%。MALDI-TOF-MS法的屬符合率為100%,其中鑒定率在99.9%以上的為17株,90%以上的為32株,80%以上的為26株,結果均可信,但其對具體血清型的符合率不高。75株沙門菌中,耐藥率最高的是氨芐西林(69.3%,52/75),其次是氨芐西林/舒巴坦(44.0%,33/75),第三是萘啶酸(42.7%,32/75)。75株沙門菌中共檢出19株對廣譜β-內酰胺類藥物耐藥,占25.3%。19株中有15株攜帶blaCTX-M基因(8株為blaCTX-M-14,2株為blaCTX-M-9,1株為blaCTX-M-34,2株為blaCTX-M-3,2株為blaCTX-M-15),8株攜帶blaTEM-1基因,4株攜帶blaOXA-1基因。結論16S rRNA基因測序法作為細菌鑒定的金標準,以其快速準確著稱,可作為傳統鑒定法的補充方法。MALDI-TOF-MS法可以快速鑒定沙門菌,但僅在科研模式下指向性地提示血清型,因此需采用其他輔助技術進行鑒定。引起兒童腹瀉的沙門菌對多種抗生素耐藥,對3代頭孢菌素類的耐藥主要由blaCTX-M型引起。
【關鍵詞】沙門菌;16S rRNA基因測序;基質輔助激光解析電離飛行時間質譜;耐藥;blaCTX-M
沙門菌病是全球性(尤其是發展中國家)嚴重的公共衛生問題。沙門菌廣泛存在,主要污染動物性食品,尤其是禽肉和蛋類,因此極易引起人類感染。沙門菌中毒的癥狀主要以急性胃腸炎為主,一般預后良好,但是老人、兒童和體弱者如不及時處理感染者可死亡,是危害全球兒童健康的重要因素之一[1-3]。在臨床工作中,對標本中分離得到的沙門菌進行鑒定并分型是診斷過程中的關鍵問題。而當前沙門菌傳統培養和分型一般存在敏感性低、耗時長、操作復雜等缺點。因此建立一種快速、簡便、準確的沙門菌鑒定和分型對輔助臨床診斷和治療是十分必要的。
1材料與方法
1.1菌株與試劑
收集2012年3月至2014年7月首都醫科大學附屬北京兒童醫院分離的沙門菌,剔除以同一患者分離的重復菌株,共計75株。質控菌株:大腸埃希菌ATCC25922、大腸埃希菌ATCC35218、大腸埃希菌ATCC8739。MALDI-TOF-MS儀:法國生物梅里埃公司。沙門氏菌診斷血清(60瓶):泰國S&A公司。細菌基因組DNA提取試劑盒:北京天根公司。PCR擴增試劑盒:上海生工公司。引物:北京天一輝遠公司。
1.2細菌傳統法鑒定
用傳統培養法和生物化學反應來鑒定沙門菌,采用血清凝集法對血清型做進一步鑒定。
1.316S rRNA基因序列分析法鑒定沙門菌
采用天根公司的細菌基因組DNA提取試劑盒,引物序列參照文獻[1],采用細菌通用引物,目的片段長度約為1 500 bp,由北京天一輝遠公司進行合成,其序列如下:27F:(5′-AGAGTTTGATCCTGGCTCAG-3′);1492R:(5′-GGTTACCTTGTTACGACTT-3′);后進行PCR擴增,擴增體系為50 μL,具體成分見表1。
在5 μL PCR終產物中加入1 μL的6×Loading Buffer,混勻后加入到1.2%瓊脂糖凝膠的上樣孔中,同時在第一個上樣孔中加入6 μL的相對分子質量為 2 000 DNA Marker。在0.5×TBE電泳緩沖液中,以120 V電壓,75 mA電流電泳40 min。在紫外光凝膠成像分析系統中觀察并記錄結果。結果顯示在1 500 bp處出現單一條帶為陽性產物。PCR終產物測序工作由北京天一輝遠測序公司完成。測序結果提交到http://blast.ncbi.nlm.nih.gov中進行Blast序列比對進行分析,找出最相似的已知序列。

表1 PCR反應體系
The reaction conditions are as follows:94 ℃ predegeneration 5 min; 94 ℃ degeneration 1 min, annealed at 55 ℃ 1 min,72 ℃ extension 1.5 min, 40 cycles; 72 ℃ and then extended 5 min.
1.4MALDI-TOF-MS質譜法鑒定沙門菌
從培養基上選取純培養18~24 h的單個沙門菌菌落。依次進行涂菌,加基質,進行質譜分析。通過SARAMIS Premium軟件與儀器自帶數據庫進行分析比較。用相匹配的百分率來量化結果,并給出最終的鑒定結果。
1.5沙門菌的藥敏試驗及耐藥基因的檢測
選用11種抗生素紙片對沙門菌進行K-B法藥敏試驗,具體的名稱及劑量見表2;按照美國臨床和實驗室標準協會(Clinical and Laboratory Standards Institute, CLSI)[2]2014手冊的說明,對廣譜β-內酰胺類耐藥的菌株進行表型初篩和確證試驗;進行β-內酰胺類耐藥基因的檢測,引物序列參照文獻[4],包括blaTEM,blaSHV,blaOXA,blaCTX-M(表3)。所有引物由北京天一輝遠公司進行合成。
2結果
2.1傳統培養方法
對75株沙門菌鑒定表明:分屬于9種血清型,其中鼠傷寒沙門菌和腸炎沙門菌檢出率最高,分別檢出32株和26株,其他血清型的沙門菌檢出率處于較低的水平。各血清型檢出率及構成比見表4。好發季節以5~9月為最多,其余各月發病數量比較零散。
2.216S rRNA基因序列分析法
用上述方法提取細菌DNA進行PCR擴增后,均將在1 500 bp處獲得陽性條帶,與預期片段相吻合,如圖1所示。

表2 抗生素紙片的名稱及劑量

表3 PCR引物序列
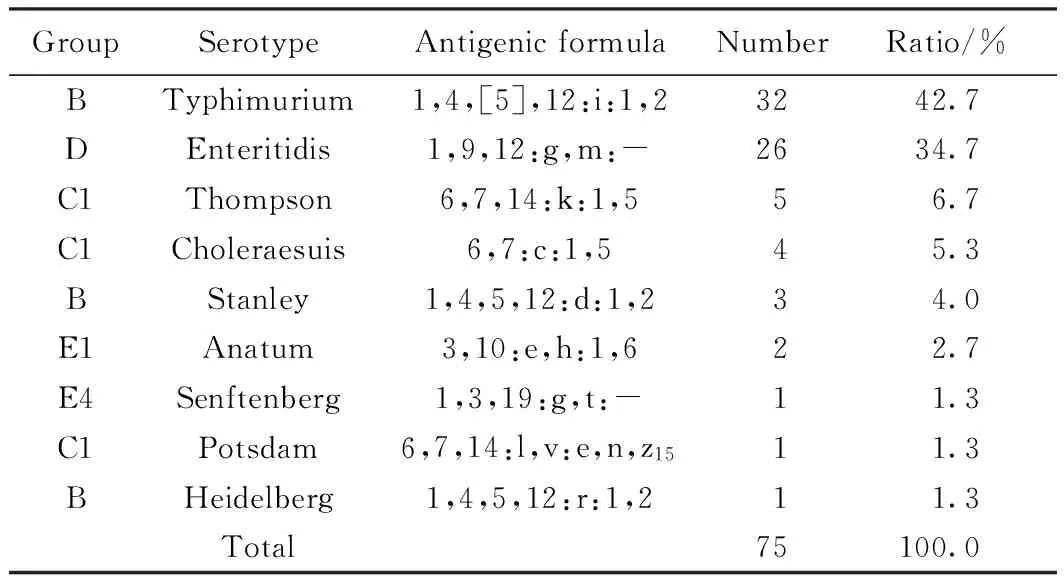
表4 兒童大便分離出的沙門菌血清型檢出數及構成比
16S rRNA基因測序提交到http://blast.ncbi.nlm.nih.gov中進行Blast序列比對進行分析,比對結果顯示與實驗室常規鑒定方法的結果完全一致,可鑒定到具體的血清型,符合率為100%。16S rRNA基因測序鑒定細菌準確,可用于細菌的分類鑒定。
2.3MALDI-TOF-MS法分析
臨床模式下鑒定結果均為沙門菌屬,菌屬鑒定正確率為100%。其中6株鑒定為沙門菌屬,其余69株鑒定為腸道沙門氏菌亞種。MALDI-TOF-MS相匹配的百分率,匹配度在99.9%以上的為17株,顯示為墨綠色;90%以上的為32株,顯示為綠色;80%以上的為26株,顯示為黃色,結果均可信,在科研模式下與儀器自帶數據庫進行比對,質譜法能指向性地提示血清型,有利于臨床上的快速鑒定。
2.4沙門菌的耐藥性分析
所有的菌株對亞胺培南和美羅培南均敏感。75株沙門菌株中,21株對所有的抗生素均敏感,15株對1種抗生素耐藥,5株對2種抗生素耐藥,34株對3種及以上的抗生素耐藥。其中耐藥率最高的是氨芐西林(69.3%,52/75),其次是氨芐西林/舒巴坦(44.0%,33/75),萘啶酸(42.7%,32/75),詳見表5。75株沙門菌中共檢出19株對廣譜β-內酰胺類耐藥,占25.3%。
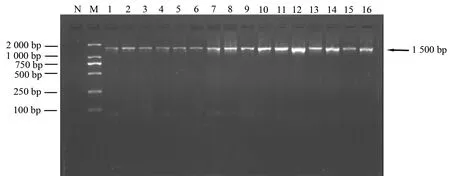
圖1 PCR擴增電泳圖
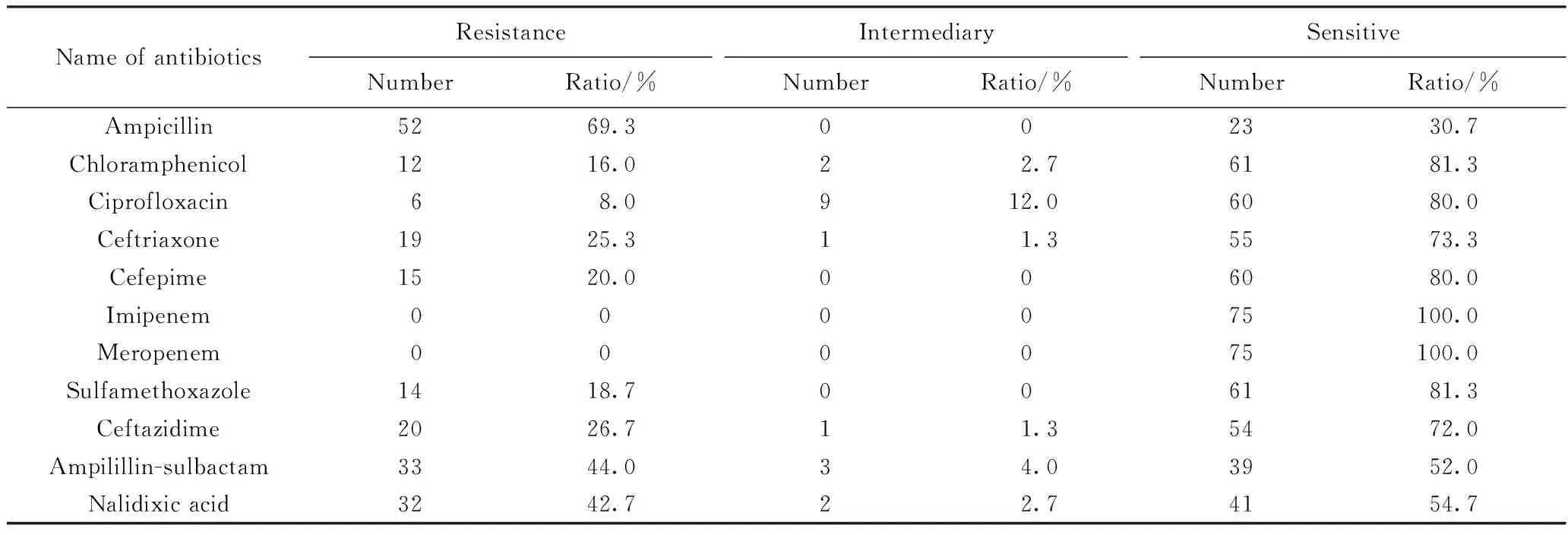
NameofantibioticsResistanceNumberRatio/%IntermediaryNumberRatio/%SensitiveNumberRatio/%Ampicillin5269.3002330.7Chloramphenicol1216.022.76181.3Ciprofloxacin68.0912.06080.0Ceftriaxone1925.311.35573.3Cefepime1520.0006080.0Imipenem000075100.0Meropenem000075100.0Sulfamethoxazole1418.7006181.3Ceftazidime2026.711.35472.0Ampilillin-sulbactam3344.034.03952.0Nalidixicacid3242.722.74154.7
75株沙門菌中共檢出對廣譜β-內酰胺類耐藥的菌株19株,占25.3%。19株沙門菌中有2株未擴增出β-內酰胺類耐藥基因,其中攜帶blaTEM基因的有8株,經DNA測序表明均為blaTEM-1型;攜帶blaOXA基因的有4株,經DNA測序表明均為blaOXA-1型;攜帶blaCTX-M基因的有15株,包括blaCTX-M-1型5株,blaCTX-M-9型10株。經DNA測序表明,5株blaCTX-M-1型中,1株為blaCTX-M-1亞型,2株為blaCTX-M-3亞型,2株為blaCTX-M-15亞型。10株blaCTX-M-9型中,2株為blaCTX-M-9亞型,8株為blaCTX-M-14亞型。未檢出blaSHV基因、blaCTX-M-2基因和blaCTX-M-8基因。具體的基因型及分布見表6。
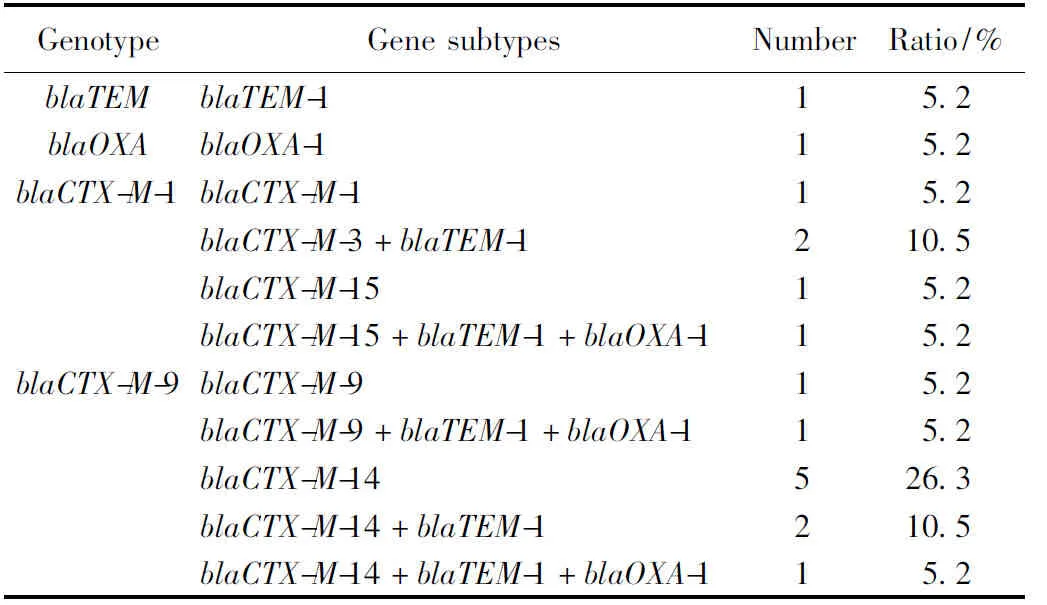
表6 19株對廣譜β-內酰胺類耐藥沙門菌的β-內酰胺類耐藥基因型別及分布
3討論
從75名患病兒童統計結果來看,2歲以下患兒為主要發病人群,其中男孩居多。從沙門菌血清學分型結果來看,以B群和D群沙門菌為優勢菌。鼠傷寒沙門菌與腸炎沙門菌為主要的致病菌。這與國內外報道[4]的非傷寒沙門菌血清型以腸炎型及鼠傷寒為主是基本一致的。但是其他血清型,如湯卜遜、豬霍亂、斯坦利、鴨沙門菌等也有所分離,說明沙門菌的血清型趨于多樣化。這可能與兒童腸道免疫功能尚未發育成熟、抵抗力低有關系。
傳統沙門菌鑒定到血清型需要大量的血清凝集工作。但有時血清效價低,血清凝集反應弱甚至為陰性,這些都加大了沙門菌鑒定的難度,因而需要其他方法對其進行準確快速鑒定。16S rRNA基因是編碼細菌染色體上rRNA相對應的DNA序列,它存在于所有的原核生物中,片段大小約為1 500 bp。存在著保守區和可變區的基因,兩者之間的序列編碼成為互相交替排列。因此,16S rRNA可以作為細菌群落結構分析最常用的系統進化標記分子[5]。本實驗中其對沙門菌的鑒定率要優于MALDI-TOF-MS法,因其準確性高,成為傳統培養鑒定方法中不可或缺的輔助手段。
MALDI-TOF-MS是近幾年發展起來的一種全新的微生物鑒定和分型的技術,己廣泛地用于微生物的檢測與鑒定。它對樣本的要求比較低,且能在短時間內對菌株進行高通量檢測,因而越來越受到人們的關注[6]。但是在國內外用該方法對沙門菌的研究還不多。其不足是與傳統培養血清型的符合率不高,原因可能是MALDI-TOF-MS分析的是細胞膜上易于通過激光解吸的蛋白質如細胞受體、黏附素等。而血清學分型中檢測的細胞膜上的蛋白質未必是在MALDI-TOF-MS分析中被解吸的蛋白質,這兩種檢測分型方法在原理上不是直接對應的關系[7]。因此需采用其他的輔助技術進行鑒定,在本研究中采用血清學進一步進行驗證。在實際檢測中,如果儀器自帶數據庫信息不足,就需要研究人員豐富數據庫,提高鑒定準確率。胡驍等[8]用Bruker質譜法建立沙門菌標準菌庫。沙門菌的MALDI-TOF-MS法還處于初期階段,受細菌庫的限制,鑒定也僅限于方法學之間的比較。
由于氟喹諾酮類和第三代頭孢菌素具有廣譜、高效的特點,是治療沙門菌感染的主要藥物。但隨著耐藥菌株的播散和抗生素的濫用,使沙門菌的治療面臨越來越大的困難。本研究結果表明,75株沙門菌中共檢出對廣譜β-內酰胺類耐藥的菌株19株,占25.3%,與陳強等[9]關于兒童感染沙門菌報道符合。本研究中頭孢類耐藥率較高,分析其原因可能為:(1)喹諾酮類藥物和氨基糖苷類藥物在兒童應用中存在爭議,導致兒科臨床應用的抗生素以β-內酰胺類為主,其中三代頭孢菌素更是廣泛使用。(2)北京兒童醫院為三級甲等兒童專科醫院,與患者病情較重,三代頭孢菌素使用率高,住院時間長,因而導致產酶株的院內播散概率大。因此,關注沙門菌的流行和耐藥情況,研究其相關耐藥基因,對了解菌株的分布,指導臨床合理應用抗生素十分必要。
對廣譜β-內酰胺類耐藥的基因可分為以下幾類:TEM型、SHV型、OXA型、CTX-M型和其他型(如CMY型、VIM型、PER型等)。本研究中19株對廣譜β-內酰胺類耐藥沙門菌中共檢出15株攜帶blaCTX-M基因,其中8株為CTX-M-14亞型,這一基因在我國對廣譜β-內酰胺類耐藥的腸桿菌科細菌中最常見[10-14]。同時檢出了較少見的CTX-M-3,CTX-M-15型,顯示了CTX-M型在本地區的主導地位。TEM基因型是對廣譜β-內酰胺類耐藥基因中常見的基因型,可水解青霉素類、頭孢菌素類抗生素,至今已有178種,并呈全球性分布。本試驗中有8株攜帶TEM型基因,并且伴有CTX-M型和OXA型基因,說明細菌耐藥基因的多樣性和復雜性。
4參考文獻
[1]Frank J A, Reich C I, Sharma S, et al. Critical evaluation of two primers commonly used for amplification of bacterial 16S rRNA genes[J]. Appl Environ Microbiol, 2008,74(8):2461-2470.
[2]Clinical and Laboratory Standards Institute. Performance standards for antimicrobial susceptibilitv testing:Twenty-Fourth informational supplement[C]. Wayne, PA:CLSI, 2014.
[3]紀文靜, 徐樨巍,董方.兒科產超廣譜β-內酰胺酶志賀菌的基因型和耐藥性研究[J].中華微生物學和免疫學雜志,2010,30(5):472-476.
[4]Ran L, Wu S, Gao Y, et al. Laboratory-based surveillance of nontyphoidal salmonella infections in China[J]. Foodborne Pathog Dis,2011,8(8):921-927.
[5]Rajendran J, Gunasekaran P. Microbial phylogeny and diversity: small subunit ribosomal RNA sequence analysis and beyond[J]. Microbiol Res,2011,166(2):99-110.
[6]Tan K E, Ellis B C, Lee R, et al. Prospective evaluation of a matrixassisted laser desorption ionization-time of flight mass spectrometry system in a hospital clinical microbiology laboratory for identification of bacteria and yeasts: a bench-by-bench study for assessing the impact on time to identification and cost-effectiveness. [J]. Clin Microbiol, 2012,50(10):3301-3308.
[7]王曄茹,崔生輝,李鳳琴.基質輔助激光解吸/電離飛行時間質譜在沙門菌檢測和鑒定分型中的應用研究[J].衛生研究,2008,37(6):685-689.
[8]胡驍,林肖惠,佟巍,等.沙門菌MALDI-TOF-MS標準菌庫的建立及應用研究[J].衛生研究,2014,43(1):40-46.
[9]陳強,余曉君,李俏俏,等.引起兒童腹瀉的沙門菌屬臨床分離株的耐藥特點及分子流行病學研究[J].中華檢驗醫學雜志,2011,34(3):249-253.
[10]Cui S, Li J, Sun Z, et al. Characterization of Salmonella enterica isolates from infants and toddlers in Wuhan, China[J]. J Antimicrob Chemother, 2009,63(1):87-94.
[11]陳娜,蘇建榮,丁軍穎,等. 混合菌感染中氨芐西林耐藥的大腸埃希菌β-內酰胺酶基因的研究[J]. 解放軍醫藥雜志,2015,27(10):76-79.
[12]韋志友,丁軍穎,劉清泉. β-內酰胺酶耐藥機制及其中醫藥相關研究進展[J]. 解放軍醫藥雜志,2015,27(10):82-87.
[13] 桂紅,丁軍穎,高翔,等. 醫院感染產β-內酰胺酶金黃色葡萄球菌的耐藥分析[J]. 解放軍醫藥雜志,2015,27(10):79-81.
[14] 張海譜,李仕英,王縛鯤,等. 112例慢性腹瀉飛行員腸道菌群中產超廣譜β-內酰胺酶菌的檢測與分析[J]. 解放軍醫藥雜志, 2014,26(9):89-92.
編輯慕萌
Research on children infected withSalmonellaidentification methodology and antimicrobial resistance characteristics
Hu Lixin, Song Wenqi*, Su Jianrong, Dong Fang, Wang Yan
(ClinicalLaboratory,BeijingChildren’sHospital,CapitalMedicalUniversity,Beijing100045,China)
【Abstract】ObjectiveTo investigate the rapid identification and the antimicrobial resistance characteristics of Salmonella recovered from children. MethodsSelected 75 strains Salmonella which were isolated from children. Via standard biochemical and serological tests for all strains were identified. 16S rRNA sequencing method was used to identify. Matrix assisted laser desorption ionization time of flight mass spectrometry (MALDI-TOF-MS) method was used to identify. The results of the above two methods with traditional culture methods were compared and analyzed. Kerby-Bauer disk diffusion method for commonly used antibiotic sensitivity test was used. Polymerase chain reaction (PCR) method was used to analyze extended-sptectrum beta-lactam resistant gene. ResultsSeventy-five Salmonella, Salmonella Typhimurium and Salmonella enteritidis were the predominant serotypes, respectively 32 and 26. The results of 16S rRNA gene sequencing analysis compared with the traditional culture method revealed the coincidence rate was 100%. Via the MALDI-TOF-MS method, the genus coincidence rate was 100%. The identification rate above 99.9% was 17, above 90% was 32, above 80% was 26. All the results were credible. But its coincidence rate of serotypes is not high. 75 Salmonella, the highest rate of drug resistance was Ampicillin (69.3%, 52/75), followed by ampicillin/sulbactam (44%, 33/75) and nalidixic acid (42.7%, 32/75). 75 Salmonella were detected 19 strains of extended-sptectrum beta-lactam antimicrobial resistance, accounting for 25.3%. Among 19 strains had 15 carrying blaCTX-M gene (8 were blaCTX-M-14, 2 were blaCTX-M-9, 1 was blaCTX-M-34, 2 were blaCTX-M-3, 2 were blaCTX-M-15), 8 strains carried blaTEM-1 gene, 4 strains carried blaOXA-1 gene.Conclusion16S rRNA gene sequencing method is as the gold standard for identification of bacteria, known for its fast and accurate, a supplement traditional methods of identification method. MALDI-TOF-MS method can quickly identify Salmonella, but only directional prompts serotypes in the research mode, and therefore it needs to be identified by other assistive technologies. Salmonella causes diarrhea in children resistant to multiple antibiotics, third generation cephalosporins resistance is mainly caused by CTX-M type.
【Key words】Salmonella; 16S rRNA gene sequencing analysis; matrix assisted laser desorption ionization time of flight mass spectrometry; antimicrobial resistant; blaCTX-M
Corresponding author, E-mail:songwenqi1218@163.com
[doi:10.3969/j.issn.1006-7795.2016.03.023]
【中圖分類號】R 446.1
(收稿日期:2015-12-08)
網絡出版時間:2016-06-0718∶39網絡出版地址:http://www.cnki.net/kcms/detail/11.3662.r.20160607.1839.020.html
· 基礎研究 ·

