關于基因重組中OLC算法的改進研究
買阿麗, 楊雯雯
(運城學院 應用數學系 山西 運城 044000)
關于基因重組中OLC算法的改進研究
買阿麗, 楊雯雯
(運城學院 應用數學系 山西 運城 044000)
針對基因組組裝問題,從數據預處理,利用KMP算法在O(m+n)的時間上快速確定某兩個堿基片段的最大重復度,將讀長序列依據Overlap圖連成Contigs鏈以及ContigsN50的確定4個環節,改進現有的OLC拼接技術,并給出優化后的模型和算法,較好地解決了基因組組裝問題.
基因重組; KMP算法; Overlap圖; Contigs鏈; ContigsN50
0 引言
確定基因組堿基對序列的過程,稱為測序.目前,測序技術正向著高通量、低成本的方向發展[1-3].但能直接讀取的堿基對序列長度遠小于基因組序列長度,因此需要利用一定的方法將測序得到的短片段序列組裝成更長的序列.
常用的組裝算法主要基于OLC(Overlap/Layout/Consensus)方法、貪婪圖方法及De Bruijn圖方法等[1-2,4].一個好的算法應具備組裝效果好、時間短和內存小等特點[5-7].新一代測序技術的性能還有較大的改善空間,曾培龍分析基因組序列拼接所面臨的主要挑戰,比如大量重復片段的存在,reads數據海量、長度短及含有測序錯誤等,探討了當前基因組序列拼接所采用的貪心策略、交疊-排序-生成共有序列(OLC)策略和De Bruijn圖策略等,總結了不同算法的優勢及不足,提出了序列拼接算法的改進方向,率先在拼接算法中提出了基于信息累計和數據特征相結合的評分方法[4].鄭緯名等提出了歐拉超路拼接算法,由于該算法要求構造一個復雜的De Bruijn圖,因此用歐拉超路算法拼接大規模全基因組存在存儲瓶頸問題[8].王旭提出了一種新的重疊群生成算法SRGA,該算法基于De Bruijn圖,將從頭測序問題轉化成一個四叉樹的搜索問題,并采用啟發式搜索策略,能夠快速地處理海量測序數據,能得到質量較高的重疊群[9].
基因組組裝就是將得到的有ATCG4個字符組成的字符串,拼接成更長的字符串,本質上就是字符串匹配問題,利用KMP算法可以較快速地實現基因重組.KMP算法如圖1所示[10].本文以2014年深圳杯全國大學生數學建模B題(以下簡稱2014B題)附件為例在現有的OLC拼接技術上提出改進.2014B題中數據為一個全長約為120 000個堿基對的細菌人工染色體(BAC),采用Hiseq2000測序儀進行測序,基因組中每個位置平均被測到約70次[11].
1 數據預處理
2014B題得到的長數據存儲在fastq文件中,首先將數據讀取到Matlab中,并依據以下原則對數據進行篩選,去除不準確的數據.
1) 整條read堿基中無N堿基.N代表模糊堿基,可能是由于測序熒光強度不夠造成,將其刪除[12].
2) 整條read中質量值低于20的堿基不超過20%.根據文獻[12]中給出的公式:字符的ASCII碼-64=質量值.也即將read_zhiliang矩陣中ASCII碼值中小于84的數據占該行比例小于20%的行刪除,對應將reads1、reads2中的該堿基片段刪除.
3) 整條read中質量值低于13的堿基不超過10 %.也即將read_zhiliang矩陣中ASCII碼值中小于77的數據占該行比例小于10 %的行刪除,對應將reads1、reads2中的該堿基片段刪除[12].
4) 整條read的堿基平均值不小于20.將不符合的片段刪除[12].
依據刪除數據的4個標準,reads1共刪掉2 805行,reads2共刪掉4 487行.得到43 982×89的reads1_used矩陣和42 360×89的reads2_used矩陣,其中第89列記錄該行在reads1、reads2中的位置.
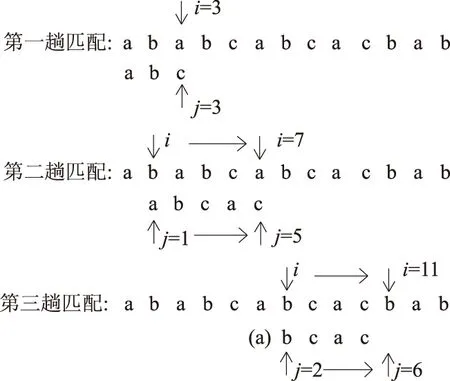
圖1 KMP算法Fig.1 KMP algorithm
2 建立Overlap有向圖
若有兩個短片段序列分別為AGTACCTTGCTAGCGT 和GCTAGCGTAGGTCTGA.則有可能基因組序列中包含有AGTACCTTGCTAGCGTAGGTCTGA這一段.事實上,一個片段可能與多個片段都有重復部分,但是重復的堿基數量不同,記此數量為重復度.認為重復越大,這兩個片段越有可能連在一起.因此,找到reads1中每個片段與之重復度最大的片段,記下此時的重復度,命為最大重復度.整理這些最大重復度,建立一個Overlap有向圖.
2.1 計算兩個片段最大重復度
若逐一比較整段基因片段,則需將兩個基因片段從不同的位置開始依次比較,這個過程比較復雜,效率很低.因此,在比較兩段基因之前先進行查找,快速鎖定可能的重復位置,根據可能的重復位置再從最大的可能重復的位置開始比較.以片段a:TCGATACTAG 和片段b:ACTAGGCTAG為例說明.
1) 在b片段中利用KMP算法查找a片段最后3個字符串TAG,稱為子串,并記下子串在b中出現的每個起始地址.

3 7
2) 先從位置7開始,倒序向前逐一比較每個字符.
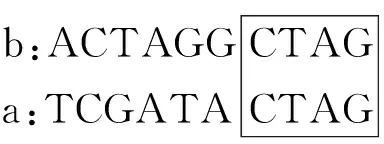
倒序第5位對應不相等,于是再從位置3開始比較.

此時重疊部分相同,重疊部分有5個堿基,認為a、b的最大重復度為5. 稱此時子串的起始地址為重復地址,即a與b重復地址為3.
2.2 在多個片段尋找某個片段的后繼片段
以a:TCGATACTAG; b:ACTAGGCTAG; c:TAGCCAATTA; d:GATACTAGAC4個片段為例,尋找a的后繼片段:

3 7
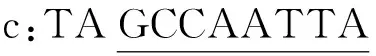
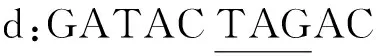
6
1) 用上述1中的方法得出b與a的重復地址為3,最大重復度為5.
2) 在c片段大于等于3的位置上利用KMP算法尋找“TAG”,不是從c的第一個位置開始找,這樣就減少了查找次數,提高了執行效率.此例中,發現沒有找到.此時仍然認為b最有可能是a的后繼片段.
3) 在d片段大于等于3的位置上利用KMP算法尋找“TAG”,發現位置6出現“TAG”,再用2.1中的方法比較d與a,得出重復度為8,大于b與a的重復度,于是認為在b、c、d中d最可能為a的后繼片段.
2.3 建立Overlap圖算法
Overlap圖模型算法1:等長reads重復度計算.
算法描述:對每個ri片段在剩余的片段中尋找后繼片段,并計算最大重復度.
算法輸入:(1) reads片段集合,即ri集合; (2) 最小重復度d.
算法輸出:每個ri片段最大重復度,及其后繼片段在ri集合中的序號.
P1初始化,i=1.
P2 若i>n,則結束,否則轉P3.
P3 1) 取出ri的最后一個長度為d的子串,記為rd;
2) 令max_p=1及j=1;
3) 若j>n,則轉P4;否則轉4);
4) 若j≠i,轉5,否則轉8);
5) 令p=max_p;
6) 在ri中從p開始調用KMP算法找到所有子串rd出現的位置,記為B={b-1,b-2,…b-k}其中bi+1>bi,k表示在rd中rj出現的次數;
7) 令l=k,若l<1,則轉8),否則執行以下操作:
① 依次比較rj中第bi之前位置是否與ri中對應字符相等,若直到rj中第一個字符均相等,則令max_p=bi+1,j=j+1,轉3).否則轉②
②l=l-1,轉①;
8)j=j+1,轉3).
P4 記錄ri的最大重復度為max_p+d-1及對應后繼片段rj;i=i+1,轉P2.
當rd越大,兩個reads片段重復的概率越小,算法執行越快,但會使許多reads無法連接到一起.另一方面,若rd越小,兩個reads片段連接的概率越大,算法執行越慢,同時錯連概率增大.因此,先取rd=30建立Overlap圖,之后對無后繼節點取rd=10,完善Overlap圖. Matlab編程得到最后的44 042×2的矩陣readsl_used_youxiangtu,第ri行第1列表示第ri個片段的后繼片段是rj,第2列表示ri與rj的最大重復度.
3 Contigs的建立
基于 reads 之間的 Overlap 有向圖,拼接獲得的序列稱為 Contigs(重疊群),Contigs的建立是連接成更長堿基鏈的過渡環節.
3.1 模型建立
先列舉10個片段進行說明,假設這10個片段建立了如圖2的Overlap圖,第二列表示兩個片段的最大重復度.
Overlap圖說明:1的后繼片段是7,7的后繼片段是10,10沒有后繼片段.所以得出Contigs單鏈;2的后繼片段是3,4的后繼片段也是3,3的后繼片段是5,5沒有后繼片段,所以得出Contigs分叉鏈;6的后繼片段是8,8的后繼片段是9,9的后繼片段是8,所以得出Contigs環鏈,如圖3所示.
產生分叉和環的原因是在整條基因中存在很多堿基序列相同的區域,如圖4所示,陰影部分為堿基序列相同的兩個區域,虛線是切割位置,與4片段重復度最大的是3片段,于是錯把3片段當作4片段的后繼片段,因此產生了分叉.環的產生也類似.
實際上,基因中存在許多堿基序列一樣的區域,在這種情況下出現分叉和環的概率很大,而且還會出現分叉和環相組合的情況.

圖2 10個片段的Overlap圖Fig.2 Overlap figure of ten Congtigs
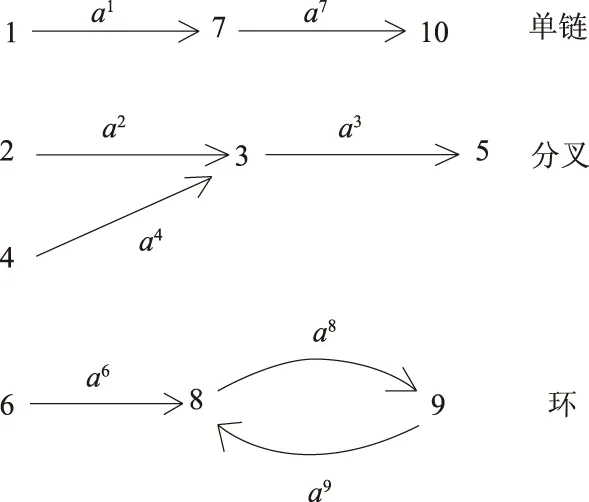
圖3 Contigs鏈Fig.3 Chain of Contigs

圖4 有重復片段基因圖Fig.4 Figure of duplication Contigs
3.2 數據結構
基于以上分析,為了從Overlap有向圖找到Contigs,構造如下的數據結構Contigs.
1) 第i行表示第i條Contigs的信息;
2) 每一行有5個元素,第1列為該條Contigs的起點,第2列為終點,第3列為該條Contigs的類型,0表示單鏈,1表示分叉,2表示環;
3) 如果為分叉,第4列表示分叉接入的Contigs編號,第5列表示接入的位置;
4) 如果為環,第2列表示接入位置的前驅位置,第4列為0,第5列表示環接入的位置.
3.3 模型算法
Contigs連接算法描述:對建立的Overlap有向圖,尋找每一個有向路徑,即連接reads片段形成單支、環及分叉.
算法輸入:Overlap有向圖.
算法輸出:Contigs矩陣.
1) 將44 042×2的矩陣reads1_used_youxiangtu對應建立一個44 042×2的矩陣biao_hao,將有后繼的節點標記為0,無后繼的節點標記為-1,即若biao_hao(i,1)=0表示第i個片段有后繼,biao_hao(i,1)=-1表示第i個片段無后繼.
2) 找到第1個標號為0的行,記為zerosl,并令Contigs_num=1.
3) 從zerosl之后開始,在矩陣biao_hao中找到第1個標號為0的行,若找到,則該行為第i行,做為第Contigs_num條Contigs的開頭片段;令zerosl=i,轉4);否則結束.
4) 在對應到Overlap圖中沿后繼往后找,同時將經過的每個節點對應的biao_hao改為Contigs_num.
如果節點biao_hao(i,1)=-1,Contigs_num=Contigs_num+1,則轉3);如果節點標號biao_hao(i,1)=contigs_num,則轉5);否則轉6).
5) (形成環)記錄Contigs中Contigs_num行為環及相應元素值;
6) (形成分叉)記錄Contigs中Contigs_num行為分叉及相應元素值;
將形成的矩陣reads1_used_contigs按其類型分為矩陣danzhi_xulie和矩陣fencha_xulie,對形成的環Contigs從接入位置展開,合并到danzhi_xulie矩陣中. Matlab以此算法編程得到矩陣reads1_used_danzhi_xulie.
從矩陣reads1_used_danzhi_xulie中發現有的堿基片段無后繼片段,形成單個孤立的片段.單個孤立片段之所以屬于矩陣danzhi_xulie,是因為基因被切割后很多堿基序列是完全一樣的,這些片斷有后繼而且彼此之間的最大重復度是88,從接入位置展開后依然是片段本身.對于這些長度仍為88 bp的單鏈,Contigs將其從矩陣中刪除.Matlab編程不斷調試后發現rd=10時,即在ri片段中選取倒數10個堿基作為子串,建立Overlap圖和Contigs鏈效果最佳,reads1形成了3 574條Contigs單鏈,reads2形成3 415條Contigs單鏈.
Matlab依據Contigs算法編程得到矩陣reads1_used_fencha_xulie,如圖5所示.
圖5 Contigs矩陣Fig.5 Contigs matrix
對于形成的矩陣fencha_xulie,會形成比較復雜的情況,但都嫁接在單鏈上, 考慮選取一簇中最長的一條,Matlab編程實現找到每簇分叉中最長的單鏈,最后將reads1形成的3 574條和reads2形成的3 415條單鏈分別存入矩陣reads1_contigs_max_dazhi和reads2_contigs_max_dazhi中,再將其轉為字符串堿基存入Matlab軟件cell中,其中一條長度為282 bp的reads1_contigs堿基,如圖6所示:
圖6 單鏈Contigs Fig.6 Single stranded Contigs
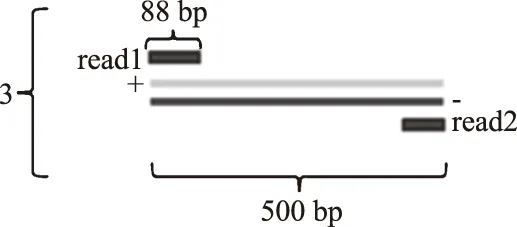
圖7 堿基片段Fig.7 Basic group
如圖7, reads1、reads2是同一基因堿基片段互補鏈上的堿基,但reads2與reads1的測序方向相反,故將reads2形成的單鏈Contigs中的堿基先倒序然后再依據堿基互補原則(與堿基A結合的堿基必為T,與堿基C結合的堿基必為G)翻譯,并與reads1形成的單鏈Contigs合并,形成一個總共含有6 989個Contigs鏈的Contigs庫,對所有Contigs鏈進行一次Overlap圖和Contigs鏈建立,最終在10重復度下形成1 500條Contigs鏈.
4 ContigsN50的確定
4.1 結果分析
reads 拼接后會獲得一些不同長度的 Contigs.將所有的 Contigs長度相加,能獲得一個 Contigs 總長度.將所有的 Contigs 按照從長到短進行排序,再按照這個順序依次相加,當相加的長度達到 Contigs 總長度的一半時,最后一個加上的 Contigs 長度即為 ContigsN50.舉例:Contigs1+Contigs2+ Contigs3+Contigs4=Contigs 總長度的1/2 時,Contigs4 的長度即為 ContigsN50[3].ContigsN50 可以作為基因組拼接的結果好壞的一個判斷標準.
4.2 ContigsN50的確定
統計得到的1 500條Contigs鏈的長度,所有長度相加得到Contigs的總長為371 311 bp,將所有的 Contigs 按照從長到短進行排序,將這Contigs的長度從大到小依次累加,最終加到第462條Contigs時,達到總長的一半,第462條Contigs長度為284 bp,所以ContigsN50=284 bp,最長Contigs為1 226 bp .ContigsN50和最長Contigs的堿基序列如圖8、圖9所示.
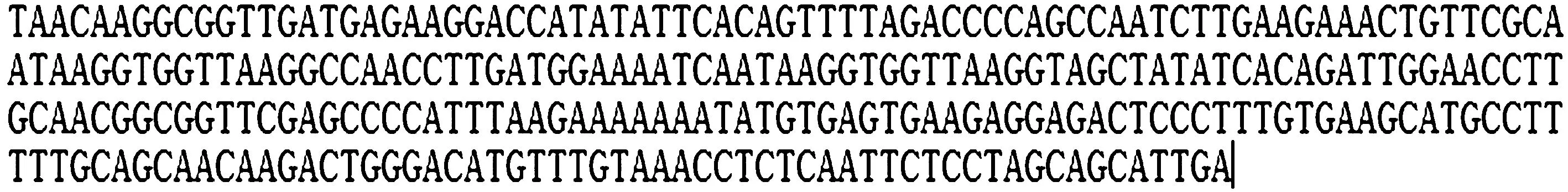
圖8 ContigsN50的堿基序列Fig.8 Base sequence of ContigsN50

圖9 最長Contigs的堿基序列Fig.9 the longest base sequence of Contigs
5 模型計算時間
Overlap圖的運行時間:在子串最大重復度為30的情況下,對于reads1和reads2建立Overlap圖的運行時間分別在9 638.8 s、9 416 s;最大重復度為10的情況下, reads1和reads2分別在1 283.4 s、1 336 s建立了Overlap圖.
r1_r2_Contig的運行時間:重復度30運行時間為498.71 s;重復度10運行時間為485.701 s.
6 結語
對于海量的堿基片段,對數據依據相關文獻進行預處理,可以有效避免錯誤數據對后續拼接的干擾.處理中人工干預較少.在傳統的OLC算法上,運用KMP算法將原本的O(m×n)時間縮短為O(m+n),在保證準確性和連續性不會降低的情況下提高了堿基段的拼接速度.進一步拼接形成3種Contigs鏈,對于單鏈和環給出了解決方法,但是出于拼接完整性的考慮,對分叉型Contigs處理還需進一步研究,另外重復度的選取也需進一步探討.
[1] 駱志剛,方小永,丁凡.DNA 序列拼接的研究進展及挑戰[J].計算機工程與科學,2007,29(8):127-127.
[2] 孫海汐,王秀杰.DNA測序技術發展及其展望[J].科研信息化技術與應用,2009(3):18-18.
[3] 徐魁,陳科,徐君,等. CGDNA:基于簇圖的基因組序列集成拼接算法[J].計算機科學, 2015, 42(9):235-239.
[4] 曾培龍.基于 reads 引導的基因組序列拼接算法[J]. 智能計算機與應用,2015, 5(3):23-25.
[5] 汪勇,張新,徐瓊,等.基因重組算法設計及多目標旅行商問題求解[J].系統工程, 2015(2):68-73.
[6] 耿麗,張仁杰.對基因組組裝算法的分析和研究[J].世界最新醫學信息文摘,2015(88):169-170.
[7] 毛華, 趙小娜, 史田敏,等. 多部圖的最大匹配算法[J]. 鄭州大學學報(理學版), 2013, 45(1):27-29.[8] 鄭緯民,林皎,羅水華.DNA 序列拼接中歐拉超路算法的新并行策略[J].計算機學報,2006,29(1): 139-139.
[9] 王旭.基于De Bruijn圖的DNA contig生成算法[D].哈爾濱:哈爾濱工業大學,2011.
[10]嚴蔚敏.數據結構(C語言版)[M].北京:清華大學出版社,2007.
[11]數學建模全國組委會.全國大學生數學建模競賽[EB/OL].[2014-04-19]. http://www.Mcm.edu.cn.
[12]美吉生物網.Illumina測序reads過濾[EB/OL]. [2014-05-19]. http://www.majorbio.com.
(責任編輯:方惠敏)
The Improvement and Research about the OLC Algorithm in the Genetic Recombination
MAI Ali, YANG Wenwen
(DepartmentofAppliedMathematics,YunchengUniversity,Yuncheng044000,China)
It was great significance to obtain the genetic information of the organism quickly and accurately, then to obtain the sequence information of target creature genome for life science research. For genome assembly problem, four steps were proposed .They were the data preprocessing, the use of KMP algorithm in the time ofO(m+n) to quickly determine the maximum of certain segments of two bases the duplication, Contigs chain according to the Overlap figure, and the determination of ContigsN50. They could considerably improve existing OLC splicing technology. The optimized model and algorithm was presented, and the problem of the genome assembly could be solved in an easy way.
genetic recombination; KMP algorithm; Overlap figure; the chain of Contigs; ContigsN50
2015-11-17
國家自然科學基金資助項目(11526183);山西省基礎研究項目(2015021015);運城學院數學學科研究項目(XK-2014035, XK-2014030);運城學院博士啟動項目(YQ-2014011).
買阿麗(1981—),女,山西運城人,副教授,博士,主要從事微分方程理論及應用研究,E-mail: maialiy@126.com.
買阿麗,楊雯雯.關于基因重組中OLC算法的改進研究[J].鄭州大學學報(理學版),2016,48(2):34-39.
Q784
A
1671-6841(2016)02-0034-06
10.13705/j.issn.1671-6841.2015266

