藏豬、長白豬腸道微生物菌群的定量分析及比較研究
李江凌 陳曉暉 劉 銳 王秋實 龔建軍 曾 凱 廖黨金 高 榮 呂學斌
(1四川畜牧科學研究院,四川成都610066;2四川大學,四川成都610063)
?
藏豬、長白豬腸道微生物菌群的定量分析及比較研究
李江凌1陳曉暉1劉銳1王秋實1龔建軍1曾凱1廖黨金1高榮2呂學斌1
(1四川畜牧科學研究院,四川成都610066;2四川大學,四川成都610063)
摘要:為研究藏豬腸道微生物菌群特性及耐粗飼特性,本試驗選擇生長發育和營養狀況正常的藏豬和長白豬各18頭,隨機分成兩組即常規組和粗糧組,每組9頭。分別于0、30、60日齡采集腸道內容物,應用熒光定量PCR的方法對腸道中四種細菌(大腸桿菌、乳酸桿菌、芽孢桿菌、梭狀桿菌)開展定量分析及對比研究,結果顯示:在出生時,藏豬、長白豬腸道內四種細菌的數量差異不顯著;隨著生長發育,斷奶后藏豬腸道內大腸桿菌、乳酸桿菌數量顯著多于長白豬(<0.05),梭狀桿菌數量極顯著多于長白豬(<0.01),芽孢桿菌屬兩個豬種之間無明顯差異。飼喂粗飼料后,藏豬腸道內梭狀桿菌、芽孢桿菌數量極顯著增加(<0.01),而長白豬乳酸桿菌數量顯著增加(<0.05)。這些結果為進一步研究藏豬、長白豬耐粗飼性狀差異的遺傳機理提供了科學依據。
關鍵詞:藏豬;長白豬;耐粗飼性能;腸道微生物;熒光定量PCR
耐粗飼性狀主要指生豬對飼料營養的要求低,可以適應低能量的飼料來源,比如青綠飼料、纖維素含量高的飼料等,耐粗飼的豬品種對纖維類物質的利用率較高,可以充分利用各種來源的飼料資源,緩解農業資源的供需矛盾。生豬耐粗飼的能力與其腸道特定的微生態區系密切相關。動物腸道相對穩定的微生態系統和腸道微生物間的相互穩定作用促進了特定動物復雜共生體的共同進化,并發揮了促進動物正常生長發育和健康的重要作用。因此,開展對藏豬、長白豬腸道微生物區系的研究,對于生豬耐粗性狀的遺傳機理研究尤為關鍵。
過去研究動物體內的腸道微生物通常應用傳統離體培養方法,這種方法的主要缺點是對于嚴格厭氧菌和一些尚未得到鑒定的細菌不能被正常分離純化,在離體培養狀態下的微生物在形態和特征與在腸道自然狀態下均存在顯著差異。隨著科技的發展,目前普遍應用不依賴微生物培養的分子生物技術研究動物胃腸道微生物區系,主要方法有應用16SrDNA、DGGE、定量PCR等,可研究土壤、動物腸道、食品微生物等的群落變化。
本試驗應用熒光定量PCR技術研究藏豬、長白豬腸道中四種細菌(大腸桿菌、乳酸桿菌、芽孢桿菌、梭狀桿菌)的多樣性,分析常規飼養及粗糧飼養條件下藏豬、長白豬四種細菌數量的變化情況,為揭示藏豬的耐粗性狀提供依據。
1 材料與方法
1.1試驗動物
選擇生長發育和營養狀況正常的0月齡藏豬、長白豬各18頭,試驗前驅蟲、去勢,并免疫相關疫苗。
1.2試驗設計
藏豬和長白豬隨機分成兩組即常規組和粗糧組,每組9頭。參照地方豬、外種豬的飼養標準,根據本場豬群情況,以玉米和大豆為主要原料配制育肥豬基礎日糧,基礎日糧均為粉料。對照組飼喂基礎日糧,試驗組飼喂由90%的基礎飼糧和10%的菊苣混合而成(菊苣切碎后與飼料混勻)的粗糧。
1.3樣品采集及DNA提取
分別于0、30、60日齡屠宰豬只后采集結腸部位內容物,-20℃保存,取回實驗室后用天根公司TIANamp Stool DNA Kit糞便基因組DNA提取總DNA,-20℃保存待用。
1.4腸道內常見菌群的定量檢測
1.4.1熒光定量PCR標準曲線的建立
對腸道內常見的大腸桿菌、乳酸桿菌、芽孢桿菌、梭狀桿菌等4種菌群做定量分析。設計引物熒光定量PCR反應的條件如表1~3。

表1 熒光定量PCR所用引物及退火溫度
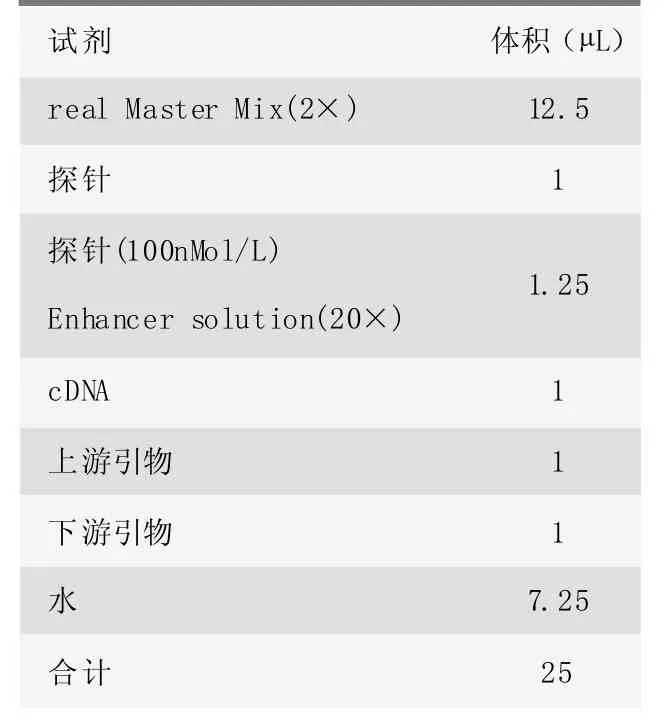
表2 實時定量PCR反應體系設置

表3 PGR擴增反應程序
采用常規方法分別完成了熒光定量標準質粒構建、建立標準曲線及線性范圍的確定。
(1)使用E.Z.N.A.TM plasmid Mini Kit I提取經測序鑒定的陽性克隆重組質粒,然后用紫外可見分光光度計測定重組質粒DNA的濃度,并按如下公式換算成拷貝數。
DNA(copy)=50(μg/mL)× 10-6×OD×NA(阿佛加德羅常數:6.02×1023分子/摩爾)/660(堿基對平均分子量)×擴增堿基數
(2)標準曲線的制作是在Real-time PCR的檢測范圍內,以各濃度梯度起始拷貝數的對數值為X軸,相應的Ct值為Y軸作回歸曲線,得出Real-time PCR標準曲線的表達式。
1.4.2樣品的Real-time PCR測定
對上述重組質粒DNA構建的標準曲線樣品和各DNA樣品進行熒光定量PCR檢測。每個樣品作3次重復,采用TaqMan Real-Time PCR Premix Kit試劑盒于BiONEER公司ExicyclerTM96實時熒光定量PCR進行絕對定量PCR分析。熒光定量PCR反應體系和反應條件同標準曲線的建立。根據相應的標準曲線和相應的Ct值,計算出各個樣品中4種菌群的拷貝數和每克糞便中的數量。
1.5數據整理分析
Real-time PCR結果采用SPSS 21.0進行顯著性分析。
2 結果分析
2.1標準曲線及方程
四個菌屬實時熒光定量PCR標準曲線,重復管Ct值之差在0.5以內,以1×109~1×104拷貝數/μL標準品陽性模板進行熒光定量,連續10倍梯度濃度之間Ct值之差在3.33左右;以初始標準品模板量的對數為橫坐標,Ct值為縱坐標,分別繪制出4種菌屬的熒光定量PCR標準曲線方程,結果見表4。
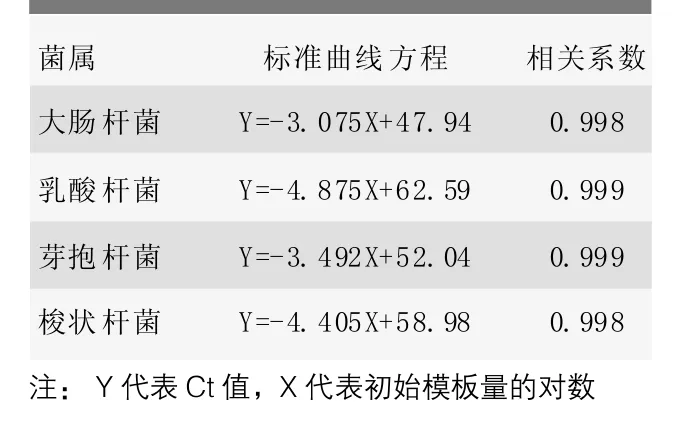
表4 4個菌屬的標準曲線方程
2.2實時熒光定量檢測藏豬和長白豬糞便中4種菌群
使用已經建立起來的標準曲線和方法對試驗藏豬、長白豬糞便DNA中4種菌屬數量進行檢測。由于兩樣本的總體方差d2均未知,且為小樣本,用t檢驗,結果如表5。
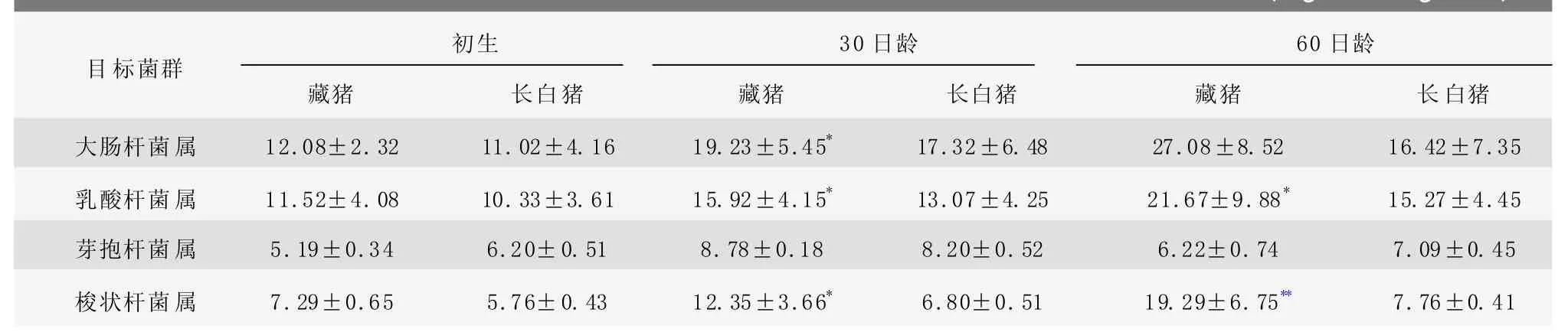
表5 常規組腸道內容物樣品中幾種菌群數量 (log拷貝數//gg樣品)
表5結果顯示,在出生時,藏豬、長白豬腸道內四種細菌的數量差異不顯著。隨著生長發育,斷奶后藏豬腸道內大腸桿菌、乳酸桿菌顯著多于長白豬(<0.05);藏豬腸道內梭狀桿菌明顯多于長白豬,30日齡時差異顯著(<0.05),60日齡時差異極顯著(<0.01);芽孢桿菌屬兩個豬種之間無明顯差異。
2.3添加粗飼料后腸道微生物菌群的變化
表6結果顯示,(1)添加粗飼料后,藏豬腸道內梭狀桿菌、芽孢桿菌數量明顯增加,與長白豬腸道內梭狀桿菌、芽孢桿菌相比,差異極顯著(<0.01);(2)添加粗飼料后,長白豬乳酸桿菌數量增加顯著,而藏豬腸道內乳酸桿菌數量下降,差異顯著(<0.05);(3)添加粗飼料后,兩個豬種之間的大腸桿菌數量無明顯差別。
3 小結與討論
在哺乳動物中,腸道微生物在營養物質的攝取、上皮細胞的生長發育、免疫方面起著重要作用。豬的腸道中寄存著大量的微生物,復雜的腸道微生物區系對于腸道健康有著重要貢獻[1]。近年來,隨著分子生物學技術的發展,人們越發了解動物的腸道微生物群落,腸道微生物不僅是動物的一個重要代謝“器官”,而且也是動物自身重要的免疫“器官”,研究表明腸道有益微生物區系的建立對于避免仔豬腹瀉和刺激腸道的生長、發育具有重要作用[2]。仔豬從剛出生到斷奶后,直至體成熟,其腸道微生物的組成受到日糧、環境及宿主等因素共同影響,形成了一個相對穩定的多樣化的微生物生態環境[3]。腸道微生物與宿主在漫長的協同進化過程中,相互選擇并形成了一個相互依賴和相互制約的共生生態系統,腸道微生物是腸道生態系統的組成成員,并與宿主構成了一個統一的整體[4]。然而,腸道微生物區系的建立是一個非常復雜的過程,受品種、環境、發育階段、消化道各部位組織結構和生理特性不同的影響,導致體內微生物菌落的數量、組成有所差異[5]。因此研究腸道微生物菌群的發育變化過程對于研究豬種的消化能力形成、以及對于粗飼料分解、營養成分吸收等具有重要意義。
我國的地方豬種普遍有耐粗飼的特性,肖文萍等[6]采用PCR/DGGE技術和16SrDNA序列分析方法,研究了普通豬、藏豬的腸道細菌優勢菌群結構,發現豬腸道微生物區系的品種間差異大于品種內差異[6]。本研究結果顯示:在出生時,藏豬、長白豬腸道內四種細菌的數量差異不顯著;隨著生長發育,斷奶后藏豬大腸桿菌、乳酸桿菌數量顯著多于長白豬(<0.05),藏豬梭狀桿菌極顯著地多于長白豬(<0.01),芽孢桿菌屬兩個豬種之間無明顯差異。在添加粗飼料后,藏豬腸道內梭狀桿菌、芽孢桿菌數量明顯增加,與長白豬腸道內梭狀桿菌、芽孢桿菌相比,差異極顯著(<0.01);(2)添加粗飼料后,長白豬乳酸桿菌數量增加顯著,藏豬腸道內乳酸桿菌數量降低,兩者之間差異顯著(<0.05);(3)兩個豬種之間的大腸桿菌無明顯差異。這些結果說明:不同品種的仔豬之間,腸道優勢菌群呈現較大差異,腸道微生物群落差異與品種有直接相關。隨著生豬日齡的增長,豬斷奶及其對飼料(如增加粗飼料)與環境的適應,各品種豬糞樣中微生物菌群發生了很大的變化,這些結果與楊柳等對地方品種榮昌豬[7]的研究結果類似。
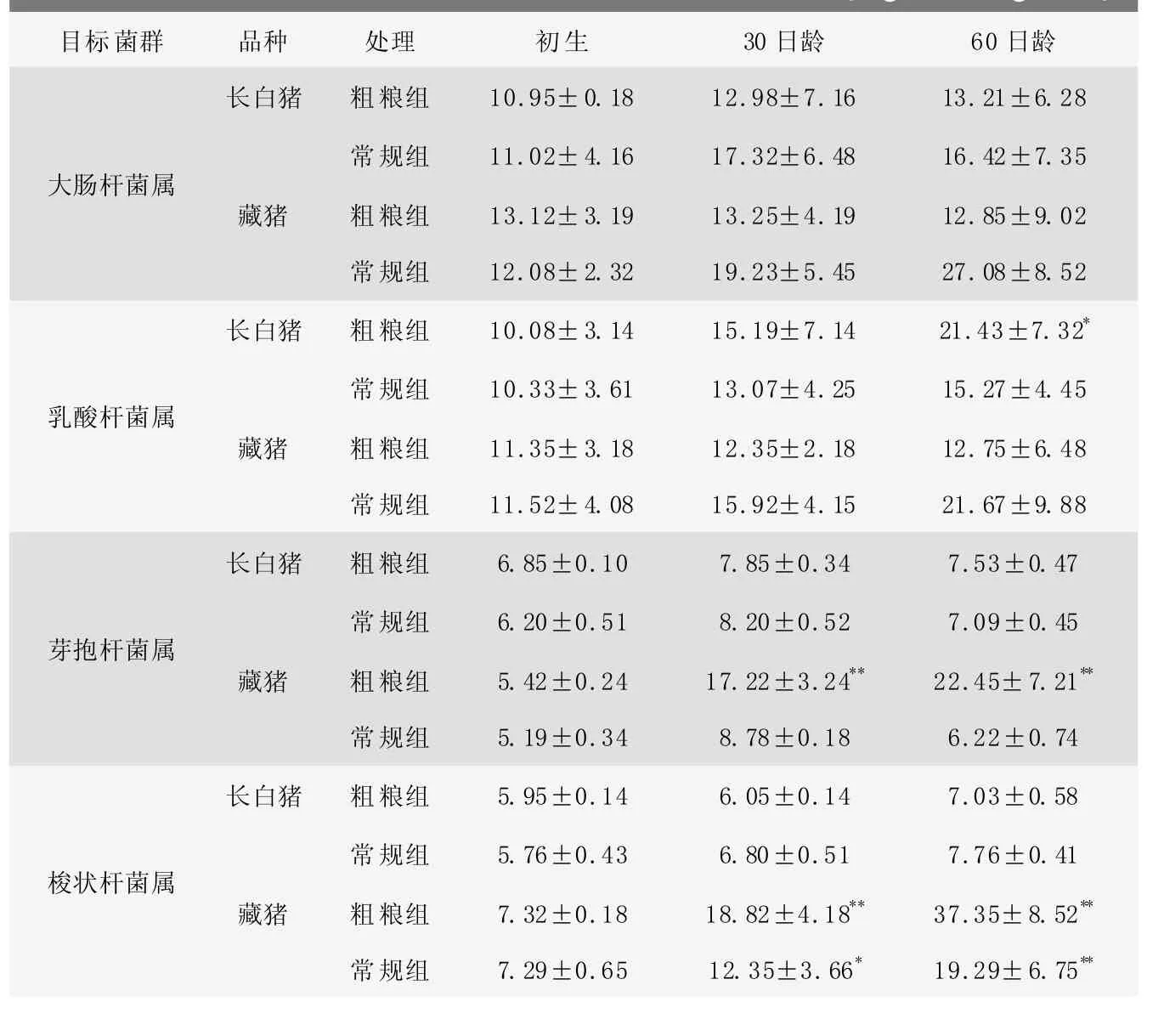
表6 常規組、粗糧組腸道內容物樣品中幾種菌群數量 (log拷貝數//gg樣品)
4 結論
本研究結果表明,在出生時,藏豬、長白豬腸道內四種細菌的數量差異不顯著;隨著生長發育,斷奶后藏豬腸道內大腸桿菌、乳酸桿菌數量顯著多于長白豬(<0.05);梭狀桿菌數量極顯著多于長白豬(<0.01);芽孢桿菌屬兩個豬種之間無明顯差異。添加粗飼料后,藏豬腸道內梭狀桿菌、芽孢桿菌數量極顯著增加(<0.01);長白豬腸道內乳酸桿菌數量顯著增加(<0.05),兩個豬種之間大腸桿菌無明顯差異。可見,藏豬的耐粗性狀與腸道內梭狀桿菌、芽孢桿菌增加相關。
參考文獻
[1]Mai V,Draganov PV.Recent advances and remaining gaps in our knowledge of associations between gut microbiota and human health[J].World J Gastroenterol,2009,15(1):81-85.
[2]Danielsen M,Hornshoj H,Siggers R H,et al.Effeets of baeterial colonization on the Porcine intestinal proteome[J].J Proteome Res,2007,6(7):2596-2604.
[3]Leser TD,Lindeerona RH,Jensen TK,et al.Changes in baeterial community structure in the colon of pigs fed different experimental diets and after infection with Brachyspira hyodysenteriae[J].APP Environ Microbiol,2000,66(8):3290-3296.
[4]Konstantinov SR,Awati AA,Williams BA,et al.Post -natal development of the porcine microbiota composition and activities[J].Environ Microbiol,2006,8(7):1191-1199.
[5]Ryo Inoue TT.Development of the intestinal microbita in the Piglets[J].J Gen APP Microbiol,2005,(51):257-265.
[6]肖文萍,劉海艷,趙海波,等.藏豬食草機理的研究——藏豬腸道微生物的多樣性分析[J].中國獸醫學報,2013,33(3):472-476.
[7]楊柳,張邑帆,鄭華,等.榮昌、長白、杜洛克豬腸道微生物ERIC -PCRDGGE指紋圖譜比較分析[J].家畜生態學報,2011,32(5):21-25.
作者簡介:李江凌(1972-),女,研究員,從事動物遺傳學研究,E-mail:yujiang1465@126.com
基金項目:四川省應用基礎項目(2013JY0112);四川省財政基礎科研項目;四川省生豬創新團隊項目
收稿日期:2016-01-20
中圖分類號:S828;S813.9
文獻標識碼:B
文章編號:1673-4645(2016)03-0061-04

