釀酒酵母轉氨酶Ⅰ基因的克隆及序列分析
劉曉蓉,郭麗瓊,林俊芳,*,康林芝,徐曉煒,文源華,陳 賓,黎穎茵
(1.華南農業大學食品生物技術研究所,華南農業大學食品學院,廣東廣州510640;2.廣東輕工職業技術學院食品系,廣東廣州510300;3.廣州市澳鍵豐澤生物科技有限公司,廣東廣州510760)
釀酒酵母轉氨酶Ⅰ基因的克隆及序列分析
劉曉蓉1,2,郭麗瓊1,林俊芳1,*,康林芝3,徐曉煒1,文源華1,陳賓1,黎穎茵1
(1.華南農業大學食品生物技術研究所,華南農業大學食品學院,廣東廣州510640;2.廣東輕工職業技術學院食品系,廣東廣州510300;3.廣州市澳鍵豐澤生物科技有限公司,廣東廣州510760)
利用釀酒酵母YT0801的總DNA為模板,采用PCR技術克隆出轉氨酶I基因的DNA序列,利用生物信息學工具對其核酸序列和蛋白序列進行分析。測序結果表明DNA序列含有一個1503bp的開放閱讀框,編碼500個氨基酸,推測等電點為5.81,相對分子質量為56.2ku。同源性比對結果顯示,該基因及推測的氨基酸序列與已報道的ARO8基因(GenBank Accession No:NM 001181067.1)的同源性均為100%。此外,對其理化性質、信號肽、疏水性、親水性、二級結構和三級結構進行了分析。該基因的成功克隆,為其功能研究和生物合成2-苯乙醇提供了分子基礎。
釀酒酵母,轉氨酶I,基因克隆,序列分析
2-苯乙醇(2-phenylethanol,簡稱2-PE)是一種具有玫瑰香氣的芳香醇[1],在化妝品和食品等領域應用廣泛。目前2-PE大多通過化學合成[2],但原料毒性大,副產物難以除去[3],人們傾向于生物合成。很多酵母能合成2-PE[4-10],但產量低;以L-苯丙氨酸為唯一氮源,酵母可通過艾利希途徑提高2-PE的產量,但高濃度的2-PE會抑制酵母生長。針對產物抑制問題進行了大量研究[11-13],但酵母轉化2-PE尚未能實現工業化生產。因而,人們迫切希望通過基因工程技術來生產2-PE或其前體物質。
酵母通過艾利希途徑合成2-PE,是分別在轉氨酶、苯丙酮酸脫羧酶和醇脫氫酶3種酶的作用下完成的。轉氨酶能催化L-苯丙氨酸形成苯丙酮酸。目前已知轉氨酶有芳香族氨基轉氨酶Ⅰ和芳香族氨基轉氨酶Ⅱ兩種,分別由基因aro8和aro9編碼[14],芳香族氨基轉氨酶Ⅰ是細胞內普遍存在的、主要的轉氨酶[15]。而芳香族氨基轉氨酶Ⅱ在正常生長過程中不存在,只有當aro8基因缺失時,才通過苯丙氨酸等芳香族氨基酸誘導產生。近年來,國內外已開展艾利希途徑相關酶基因克隆及表達的研究[16-17]。本研究從釀酒酵母YT0801中克隆出轉氨酶Ⅰ基因YT0801-aro8,并對該基因的結構與推導的蛋白質序列進行分析,旨為構建高產2-PE的轉基因菌株奠定基礎。
1 材料與方法
1.1材料與儀器
釀酒酵母(Saccharomyces cerevisiae)YT0801菌株、大腸桿菌(Escherichia coli)菌株DH5α華南農業大學食品生物技術研究所保存;TaKaRa Ex Taq酶、T4 DNA連接酶、EcoRⅠ酶等購自大連寶生物工程有限公司;pGEM-T Easy克隆載體購自Promega公司;質粒小量提取試劑盒、瓊脂糖凝膠DNA回收試劑盒等購自天根生化科技有限公司;常規試劑均為國產分析純;PCR引物由上海捷瑞生物工程公司合成;測序由華大基因完成。
5417C微量離心機德國Eppendorf公司;CHB-100恒溫金屬浴杭州博日科技有限公司;PCR擴增儀德國Biometra公司;DYY-6C電泳儀北京市六一儀器廠;Bio-Rad凝膠成像系統美國Bio-Rad公司。
1.2培養基
LB培養基胰蛋白胨10g/L,酵母提取物5g/L,NaCl 10g/L,瓊脂粉15g/L(固體),121℃高壓滅菌20min。抗性篩選時加入氨芐青霉素,終濃度為80mg/L。
YPD培養基酵母提取物10g/L,蛋白胨20g/L,葡萄糖20g/L,pH自然,瓊脂粉20g/L(固體),121℃高壓滅菌20min。
1.3實驗方法
1.3.1釀酒酵母YT0801總DNA的提取收集過夜培養20h的釀酒酵母YT0801菌體,參照文獻[18],采用珠磨法提取總DNA。
1.3.2轉氨酶Ⅰ基因YT0801-aro8的克隆
1.3.2.1引物的設計根據已報道的釀酒酵母S288c轉氨酶Ⅰ基因序列(GenBankAccessionNo:NM_001181067.1),設計PCR擴增引物,上游引物aro8F:5’-ATGACTTTACCTGAATCAAAA-GACT-3’;下游引物aro8R:5’-CTATTTGGAAATACCAAATT CTTCG-3’。
許沁沒有貿然去找葛局長,那樣必定遭到拒絕。許沁找了玉敏,玉敏大包大攬地應下了。她之前答應過許沁,不能言而無信。玉敏表現出了一副成竹在胸的樣子。
1.3.2.2轉氨酶Ⅰ基因YT0801-aro8的PCR擴增以提取的總DNA為模板進行PCR擴增。反應體系為:10×Ex Taq Buffer(Mg2+Plus)5.0μL,dNTP Mixture(2.5mmol/L each)4μL,DNA模板3μL,引物aro8F(10μmol/L)和aro8R(10μmol/L)各2μL,TaKaRa Ex Taq(5U/μL)0.5μL,ddH2O 33.5μL。PCR反應程序為:94℃預變性5min后,94℃變性45s,48℃退火45s,72℃延伸90s,33個循環,72℃延伸10min,結束反應。PCR產物經1%瓊脂糖凝膠電泳檢測。
1.3.2.3PCR產物的回收、轉化及重組質粒的鑒定采用凝膠回收試劑盒回收PCR擴增中的目標條帶,與pGEM-T Easy載體連接,將連接產物轉化E.coli DH5α感受態細胞,挑取藍白斑篩選中獲得的白色單菌落,提取質粒,并進行EcoRⅠ單酶切鑒定。將陽性重組質粒提交華大基因公司進行序列測定。
1.3.3轉氨酶Ⅰ基因YT0801-aro8的序列分析登陸http://www.ncbi.nlm.nih.gov/網站,利用Blast對獲得的核苷酸序列及推測的氨基酸序列進行比對。登陸http://blast.ncbi.nlm.nih.gov進行核酸序列比對,登陸http://web.expasy.org/protparam/將氨基酸序列進行分子量、理論等電點、氨基酸組成、原子組成和半衰期等的預測;登陸http://web.expasy.org/protscale/進行蛋白質親水性和疏水性預測;登陸http://www.cbs.dtu. dk/services/SignalP/進行信號肽預測;登陸http:// services.cbib.u-bordeaux2.fr/pise/toppred.html進行跨膜分析;登陸https://npsa-prabi.ibcp.fr/cgi-bin/ npsa_automat.pl?page=/NPSA/npsa_sopma.html進行蛋白質二級結構預測;登陸http://www.cbs.dtu.dk/services/蛋白質三級級結構預測。
2 結果與分析
2.1轉氨酶Ⅰ基因YT0801-aro8的擴增
提取釀酒酵母YT0801的總DNA,以aro8F和aro8R為引物進行PCR擴增。通過凝膠電泳,得到的PCR產物為一條約1500bp的片段,結果如圖1所示。其片段大小與預期相符,且擴增特異性好,純度高。
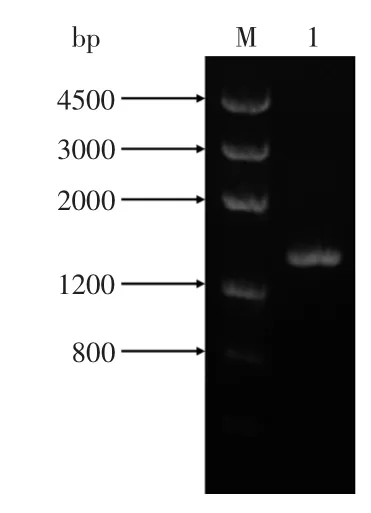
圖1 YT0801-aro8的PCR擴增電泳圖Fig.1 Electrophoresis pattern PCR amplification of YT0801-aro8
2.2陽性克隆質粒的鑒定
2.3轉氨酶Ⅰ基因YT0801-aro8基因序列分析
2.3.1理化性質預測與分析測序結果(GenBank Accession No:KC422723.1)見圖3,克隆基因片段全長1503bp。經BLAST比對,該基因及推測的氨基酸序列與已報道的釀酒酵母S288c ARO8(GenBank Accession No:NM_001181067.1)的相似性均為100%。ORF查找結果表明,擴增基因片段的閱讀框架長度為1503bp,其中A、G、T和C分別為438個(占29.14%)、298個(占19.83%)、436個(占29.01%)和331個(占22.02%)。共編碼500個氨基酸殘基。利用在線工具ProtScale Server計算此蛋白質,相對分子質量大小為56.2ku,理論等電點為5.81,其半衰期在體外哺乳動物網織紅細胞為4.4h,在酵母體內半衰期大于20h,在大腸桿菌體內大于10 h。不穩定系數(instability index)為36.51,屬于穩定蛋白。脂溶系數(Aliphatic index)為82.72,總平均疏水指數(Grand average of hydropathicity)為-0.371,表明其為親水性蛋白。該氨基酸序列功能位點的預測由在線分析工具Predict Protein完成,預測結果顯示其包含多個作用位點,如N-糖基化作用位點、蛋白激酶C磷酸化作用位點等。
2.3.2疏水性和親水性預測與分析登陸http://web. expasy.org/protscale/,分析此蛋白質的疏水性和親水性,結果見圖4,橫坐標表示氨基酸殘基的序號,縱坐標表示氨基酸殘基的疏水/親水特性。由圖4可知,第274位的賴氨酸殘基和275位的谷氨酰胺殘基的親水性最強,分值為-2.700;第41位的甘氨酸殘基的疏水性最強,分值為1.844;該蛋白的親水性氨基酸略多于疏水性氨基酸,可推測此酶是親水性蛋白質。

圖2 YT0801-aro8重組質粒的酶切鑒定Fig.2 Enzyme digestion identification of recombinant plasmid YT0801-aro8

圖3 YT0801-aro8基因序列及推導的氨基酸序列Fig.3 Nucleotide sequences and its deduced amino acid sequence of YT0801-aro8 from Saccharomyces cerevisiae
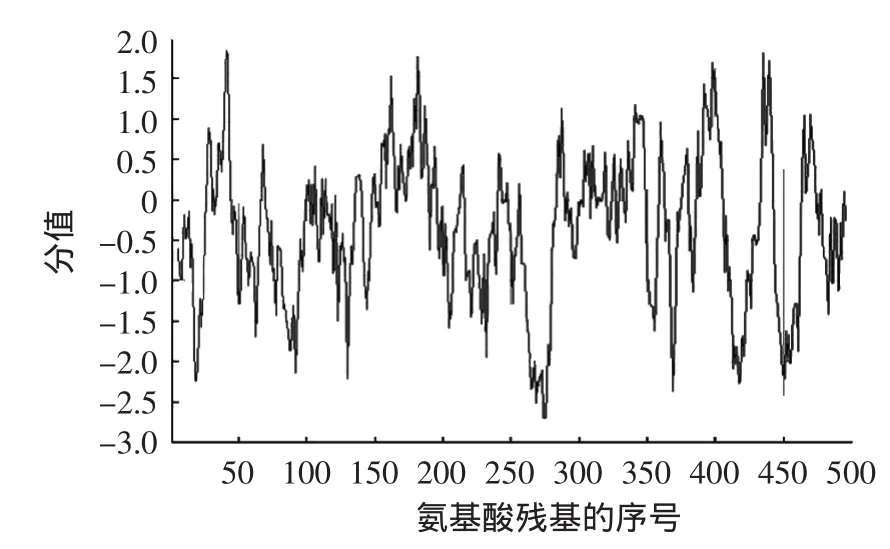
圖4 氨基酸序列親水性和疏水性分析Fig.4 The hydrophilicity and hydrophobicity analysis of deduced amino acid sequence
2.3.3信號肽預測和分析利用在線網站iPSORT prediction預測信號肽定位。結果顯示,該氨基酸序列不含信號肽或線粒體轉運肽,為非分泌性蛋白,沒有跨膜結構,這與2.3.2在線分析氨基酸序列親疏水性相符。
2.3.4二級結構預測與分析此蛋白質的二級結構預測如圖5所示,α-螺旋結構占37.4%,β-折疊占4.8%,延伸鏈和無規則卷曲分別占16.2%和41.6%。由此可知,此蛋白含有較多的α-螺旋和無規則卷曲,而延伸鏈和β-折疊則隨機散布于整個蛋白中。

圖5 蛋白質二級結構預測圖譜Fig.5 The predicted map of target protein secondary structure
2.3.5三級結構預測與分析用SAM-T08[19]預測該蛋白質的三級結構,結果見圖6。由圖6可知,此蛋白具有明顯的空間結構,主要由α螺旋和無規則卷曲構成,與NPS@的分析預測結果一致。
2.3.6多重序列比對采用DNAMAN version 5.2.2,將YT0801-aro8推測的氨基酸序列與GenBank中有關的轉氨酶基因進行氨基酸多重序列比對。結果表明,它與其他微生物來源的轉氨酶一級結構相似性差異較大;與Lachancea thermotolerans CBS 6340(GenBank Accession No:XP_002555128)相似性最大,為64.75%;與Colletotrichum fioriniae PJ7(XP_007602696)的相似性最小,為24.80%;與Talaromyces marneffei ATCC 18224(XP_002145224)及Togninia minima UCRPA7(XP_007913717)的相似性都是35.77%;與Candida albicansSC5314(XP_719544)、Aspergillusflavus NRRL3357(XP_002379696)及Eutypa lata UCREL1(XP_007799376)的相似性分別為51.69%、36.69%和32.93%。
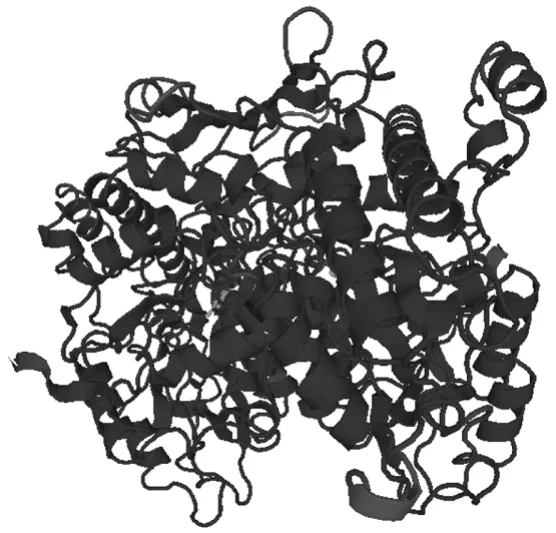
圖6 蛋白質三級結構預測圖譜Fig.6 The predicted map of target protein three-dimensional structure
3 結論
以釀酒酵母YT0801基因組DNA為模板,采用PCR技術克隆出轉氨酶I的DNA序列。該DNA的開放閱讀框長為1503bp,編碼500個氨基酸,與GenBank中釀酒酵母S288c(NM 001181067.1)ARO8基因的相似性為100%,但與其他7種微生物來源的轉氨酶一級結構相似性差異較大。二級結構預測該酶由較多α-螺旋、β-折疊和少量延伸鏈、無規則卷曲構成,而三級結構主要由α螺旋和無規則卷曲構成,表明芳香族氨基轉氨酶Ⅰ具有復雜的空間結構,對催化與苯丙氨酸相關的轉氨反應具有重要作用。還通過生物信息學軟件和在線分析網站,預測了該基因編碼的氨基酸序列的功能位點及其他蛋白質屬性。為下階段利用所克隆的YT0801-aro8基因進行表達生產2-苯乙醇和中間產物奠定了分子基礎。
[1]Jollivet N,Bezenger M C,Vayssier Y,et al.Production of volatile compounds in liquid cultures by six strains of coryneform bacteria[J].Appl Microbiol Biotechnol,1992,36:790-794.
[2]劉麗琴,郭廷翹,左宏,等.玫瑰型香料β-亞甲基苯乙醇的合成[J].精細化工,2000,17(5):257-259.
[3]Clark GS.Phenethyl alcohol[J].Perfum Flavor,1990,15:37-44.
[4]Etschmann M M W,Sell D,Schrader J.Screening of yeasts for the product ion of the aroma compound 2-phenylethanol in a molasses-based medium[J].Biotechnology Letters,2003,25:531-536.
[5]Stark D,Munch T,Sonnleitner B,et al.Extractive Bioconversion of 2-phenylethanol from L-Phenylalanine by Saccharcmyces cererisiae[J].Journal of Biotechnology Progress,2002,18:514-523.
[6]Etschmann M M W,Schrader J.An aqueous-organic twophase bioprocess for efficient production of the natural aroma chemicals 2-phenylethanol and 2-phenylethylacetate with yeast[J].Applied Microbiology and Biotechnology,2006,71:440-443.
[7]Stark D,Zala D,M nch T,et al.Inhibition aspects of the bioconversion of L-phenylalanineto2-phenylethanolby Saccharomyces cerevisiae[J].Enzyme and Microbial Technology,2003,32:212-223.
[8]Fabre C E,Duviau V J,Blanc P J,et al.Identification of volatile flavour compounds obtained in culture of Kluyveromyces marxianus[J].Biotechnol Lett,1995,17:1207-1212.
[9]Huang C J,Lee S L,Chou C C.Production of 2-phenylethanol,a flavor ingredient,by Pichia fermentans L-5 under various culture conditions[J].Food Res Int,2001,34:277-282.
[10]唐育岐,劉天明,趙修報,等.高產2-苯乙醇酵母菌株的篩選及培養基優化[J].中國食品添加劑,2012,3:137-142.
[11]王航,董清風,孟春.高產2-苯乙醇釀酒酵母的選育[J].福州大學學報:自然科學版,2010,38(1):153-156.
[12]梅建鳳,閔航,呂鎮梅.利用酵母細胞生物催化合成2-苯乙醇[J].催化學報,2007,28(11):993-998.
[13]劉國,陸軍,張偉國.兩相體系中菌體轉化法制備2-苯乙醇的研究[J].生物技術,2009,19(3):67-70.
[14]楊霄,崔志峰.酵母生物轉化生產2-苯乙醇的研究進展[J].應用與環境生物學報,2006,12(1):140-144.
[15]Urrestarazu A,Vissers S,Iraqui I,et al.Phenylalanine-and tyrosine-auxotrophicmutantsofSaccharomycescerevisiae impaired in transamination[J].Mol Gen Genet,1998,257:230-237.
[16]William E Karsten,Zoraya L Reyes,Kostyantyn D Bobyk,et al.Mechanism of the aromatic aminotransferase encoded by the Aro8genefromSaccharomycescerevisiea[J].Archivesof Biochemistry and Biophysics,2011,516:67-74.
[17]劉麗,尹勝,梁婧如,等.Saccharomyces cerevisiae氨基轉移酶基因ARO8克隆及對3-甲硫基丙醇合成的影響[J].食品工業科技,2014,35(9):132-135,139.
[18]趙宏宇,李琣,趙珗,等.4種酵母基因組提取方法的比較[J].食品科學,2011,32(9):170-173.
[19]Sol Katzman,Christian Barrett,Grant Thiltgen,et al.Predict-2nd:a tool for generalized protein local structure prediction[J]. Bioinformatics,2008,24(21):2453-2459.
Cloning and sequence analysis of aromatic aminotransferaseⅠgene from Saccharomyces cerevisiae
LIU Xiao-rong1,2,GUO Li-qiong1,LIN Jun-fang1,*,KANG Lin-zhi3,XU Xiao-wei1,WEN Yuan-hua1,CHEN Bin1,LI Ying-yin1
(1.Institute of Food and Biotechnology,College of Food Science,South China Agriculture University,Guangzhou 510640,China;2.Department of Food,Guangdong Industry Technical College,Guangzhou 510300,China;3.Alchemy Biotechnology Co.Ltd.of Guangzhou City,Guangzhou 510760,China)
Total DNA from Saccharomyces cerevisiae YT0801 was used as the template to clone aromatic aminotransferaseⅠgene YT0801-aro8 by PCR amplification.Bioinformatics softwares were used to analyse the DNA and protein sequence.The result of DNA sequencing showed that the amplified YT0801-aro8 had an open reading frame of 1503bp,which coded 500 amino acid residues.The molecular mass was 56.2ku and the theoretical isoelectric point was 5.81.Blastn and Blastx alignment results demonstrated that both the nucleotide sequence and the deduced amino acid sequence of YT0801-aro8 exhibited 100%of similarities to ARO8(GenBank Accession number:NM 001181067.1).Besides,the bioinformatics,including physical and chemical properties,the signal peptide,hydrophobicity,hydrophilicity,secondary structure and tertiary structure,were analyzed.The cloning of YT0801-aro8 could provide the molecular base for study of its functions and biosynthesizing 2-phenylethanol.
Saccharomyces cerevisiae;aromatic aminotransferaseⅠ;gene cloning;sequence analysis
TS201.1
A
1002-0306(2015)14-0180-04
10.13386/j.issn1002-0306.2015.14.029
2014-09-26
劉曉蓉(1973-),女,博士研究生,研究方向:食品生物技術。
林俊芳(1962-),男,博士,研究員,研究方向:生物活性天然產物。
國家自然科學基金項目(31272217);農業部能源植物資源與利用重點實驗室開放課題。

