巴西橡膠樹乳管細胞中5個橡膠合成關鍵酶基因節律性表達研究
楊鵬等
摘 要 以巴西橡膠樹無性系PR107、RRIM600、CATAS7-33-97和CATAS8-79的未開割樹為材料,采用熒光定量PCR技術,分析乳管細胞中HMGR1、FDPS、SRPP、REF和HRT2等5個橡膠生物合成關鍵酶基因的晝夜表達模式。結果表明:5個基因的表達均具有節律性,而且不同基因在同一無性系中以及同一基因在不同無性系間的節律性表達模式都有一定程度的差異。結果為分析不同環境因素對橡膠生物合成相關酶基因節律性表達的影響具有一定參考價值。
關鍵詞 巴西橡膠樹;膠乳;橡膠生物合成相關酶;節律性表達
中圖分類號 S794.1 文獻標識碼 A
Rhythmic Expression of Genes Encoding Enzymes for
Rubber Biosynthesis in Rubber Trees
YANG Peng1,2, ZHANG Shixin2, CHEN Huiping1, TIAN Weimin1,2 *
1 College of Horticulture and Landscape Architecture, Hainan University, Haikou, Hainan 570228, China
2 Rubber Research Institute, CATAS/Key Laboratory of Biology and Genetic Resources of Rubber Tree, Ministry of Agriculture/State Key Laboratory Incubation Base for Cultivation and Physiology of Tropical Crops, Danzhou, Hainan 571737, China
Abstract The day and night expression pattern of FDPS, SRPP, HMGR1, REF and HRT2 encoding the enzymes for rubber biosynthesis in rubber tree clones(PR107, RRIM600, CATAS7-33-97and CATAS8-79)were analyzed by quantitative PCR technique. The expression of all the five genes appeared to be day and night rhythm. The rhythmic expression pattern was different to some extent either among different genes in the same clone or among different clones for the same gene. This is valuable for investigating the effects of environmental factors on the rhythmic expression of genes encoding the key enzymes for rubber biosynthesis in rubber tree.
Key words Hevea brasiliensis Muell. Arg.; Latex; Key enzymes for rubber biosynthesis; Rhythmic expression
doi 10.3969/j.issn.1000-2561.2015.09.007
巴西橡膠樹(Hevea brasiliensis Müll. Arg.)是天然橡膠的主要來源,而天然橡膠生物合成是典型的植物類異戊二烯代謝途徑,有多種酶類參與,如HMG-CoA還原酶1(3-Hydroxy-3-Methylglutaryl Coenzyme A Reductase 1, HMGR1)、法尼基焦磷酸合成酶(Farnesyl Diphosphate Synthase, FDPS)、小橡膠粒子蛋白(Small Rubber Particle Protein, SRPP)、橡膠延伸因子(Rubber Elongation Factor, REF)和橡膠轉移酶(Rubber Transferase, HRT)等。目前對這些酶的研究較多:HMGR催化由HMG-CoA形成甲羥戊酸(Mavalonic acid, MVA),繼而合成異戊烯焦磷酸(IPP)[1],在煙草中表達HMGR1、HMGR2基因,導致甾醇的含量增加[2-3],在小粒種咖啡(Coffea arabica)中,HMGR1表達存在時間差異性[4];FDPS催化一分子二甲基丙烯焦磷酸和兩分子異戊烯基焦磷酸以1、4頭尾連續縮合反應,而形成法尼基焦磷酸(FPP)[5],橡膠生物合成的效率與細胞內的FPP濃度呈正相關[6];FDPS在植物中起到甾醇合成和三萜類生物合成的調節作用[7];SRPP和REF是橡膠粒子膜蛋白,兩者與脂質膜的結合方式不同,REF嵌入到半個單位膜中,SRPP僅結合在膜的表面[8-9],REF的表達與橡膠合成量成正相關[10],在蒲公英中,SRPP對橡膠合成效率和橡膠合成穩定性具有非常關鍵的作用[11-12];HRT1和HRT2在橡膠樹的膠乳中特異表達[13],在俄羅斯蒲公英中,HRT參與橡膠合成[14]。
生物節律在植物生命過程中扮演著重要的角色,是植物對環境的一種適應性機制[15-16],這種生物節律在分子水平上表現為基因的節律性表達[17]。模式植物擬蘭芥大約有1/3基因屬于時鐘調節基因[18]。由于橡膠生物合成關鍵酶基因不是典型的時鐘基因,所以幾乎所有關于環境因素對橡膠生物合成關鍵酶基因表達的研究都沒有考慮基因的節律性表達。因此,本文以橡膠樹無性系品系PR107、RRIM600、CATAS7-33-97和CATAS8-79的4 a未開割樹為材料,采用熒光定量PCR技術,分析5個橡膠生物合成關鍵酶基因的晝夜表達模式,以期為基因表達分析研究提供一定參考。
1 材料與方法
1.1 植物材料
巴西橡膠樹無性系PR107、RRIM600、CATAS7-33-97和CATAS8-79的4 a未開割樹,種植于中國農業科學院橡膠研究所試驗基地。
1.2 方法
1.2.1 樣品采集 選取大小和長勢一致的3株PR107植株,采用針刺法從主干不同部位收集膠乳。第一個部位距芽接樁30 cm,從該部位往上,以正面-背面交錯方式再選取8個部位,相鄰位置間相隔15 cm,同時在9個位置用針刺法采集膠乳樣品。每株樹每個部位的膠乳分別提取總RNA,通過熒光定量PCR分析基因在樹干不同部位的表達。
選取6株大小和長勢較一致的PR107植株,其中,3株作針刺處理,3株為對照。針刺處理的部位距離芽接樁30 cm。針刺處理4 h 后,在處理位置上方15 cm處的樹干背面采集膠乳。對照組在對應高度采集膠乳。每株樹的膠乳分別提取總RNA,通過熒光定量PCR分析針刺處理對臨近部位基因表達的影響。
分別從4個無性系中選取3株大小和長勢一致的植株,分別在6 : 00、10 : 00、14 : 00、18 : 00、22 : 00、次日2 : 00、次日4 : 00、次日6 : 00、次日10 : 00等9個時間點,用針刺法由下往上從樹干的9個部位采集膠乳。每株樹的每個部位的膠乳分別提取總RNA,通過熒光定量PCR分析基因的晝夜表達模式。
1.2.2 膠乳中總RNA提取與反轉錄 使用天根生化科技(北京)有限公司多糖多酚植物總RNA提取試劑盒(DP441)提取膠乳總RNA,方法參照試劑盒說明書。提取的膠乳總RNA通過瓊脂糖凝膠電泳檢測完整性并使用美國Thermo Electron Corporation的微量紫外分光光度計(Gene company Limited NO2000)測定濃度,各取800 ng RNA進行等量反轉錄,方法參照試劑盒(#K1621)說明書。
1.2.3 熒光定量PCR 將反轉錄產物稀釋10倍作為模版,使用美國Bio-Rad Laboratories, Inc.的CFX96/384 Touch Real-Time PCR Detection System進行熒光定量PCR分析。使用寶生物工程(大連)有限公司的SYBRR Premix Ex TaqTM II(RR820A)配制反應體系,以HbACTIN為內參基因。熒光定量PCR反應體系見表1。引物序列及擴增效率見表2。
熒光定量PCR反應程序:95 ℃ 30 s;95 ℃ 5 s,62 ℃/63.3 ℃ 30 s,72 ℃ 10 s,40個循環;95 ℃ 10 s;60~95 ℃做熔解曲線分析,每個溫度以每步0.2 ℃上升,每個溫度停留5 s。
1.2.4 數據分析 使用SAS8.1 軟件進行數據統計分析。采用One-way ANOVA 中的Duncan多重對比分析不同數據組間的差異。
2 結果與分析
2.1 橡膠樹樹干不同高度的膠乳中基因表達分析
熒光定量PCR分析結果表明,HMGR1、FDPS、SRPP、REF和HRT2等5個基因在樹干不同部位的表達存在一定程度的差異(圖1),這可能與砧木的不同導致株間差異有關。但通過SAS軟件進行單樣本T檢測,這些差異并未達到顯著水平。
2.2 針刺處理對鄰近部位橡膠合成相關基因表達的影響
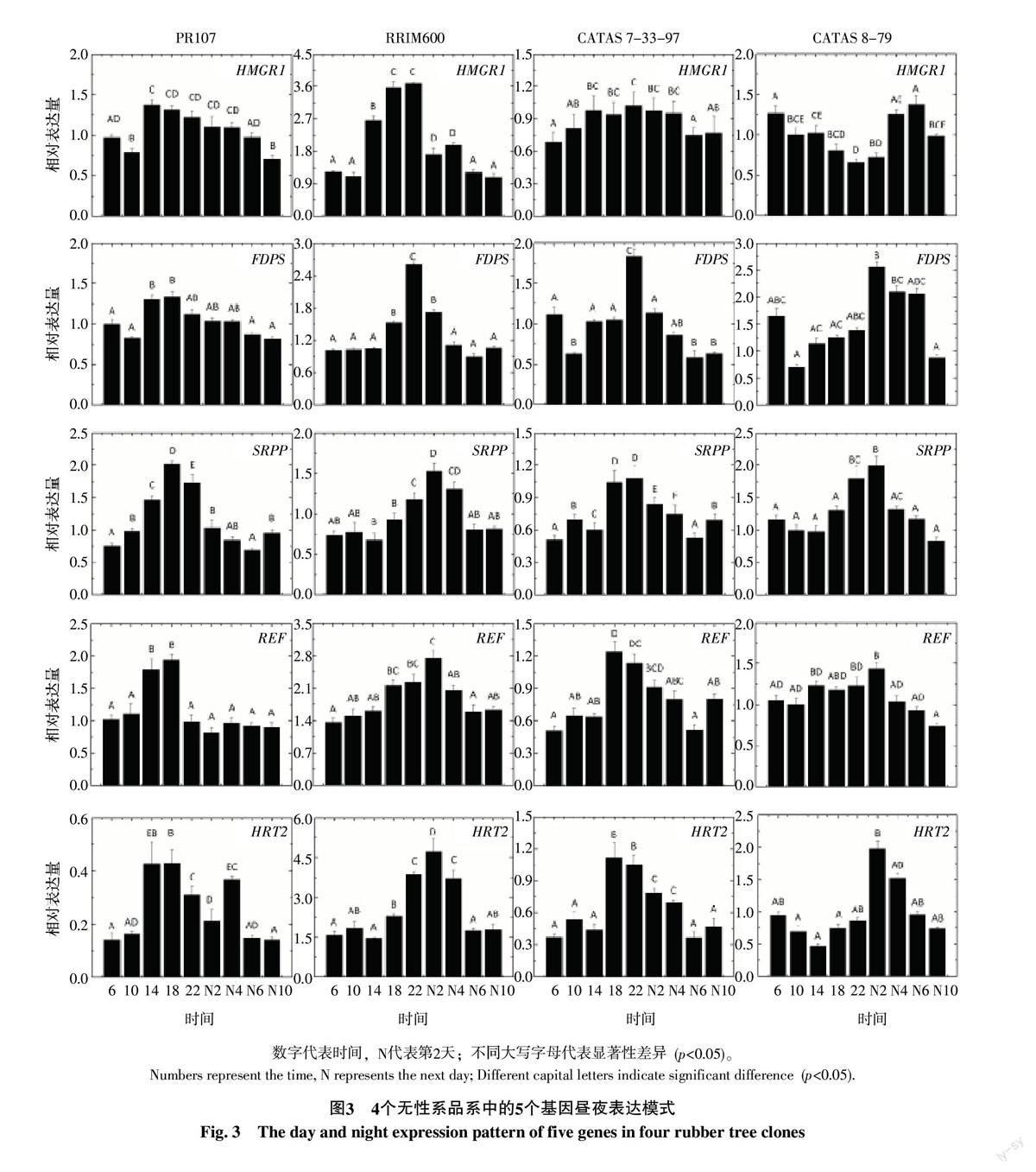
離芽接樁30 cm處的樹干部位作針刺處理,4 h后在距離處理位置15 cm處的樹干背面采集膠乳。對照不作任何處理,在相應高度處采集膠乳。結果表明,HMGR1、FDPS、SRPP、REF和HRT2等5個基因表達量在處理和對照間存在一定程度的差異,但差異不顯著(p>0.05)(圖2)。
2.3 不同基因在同一無性系中的表達分析
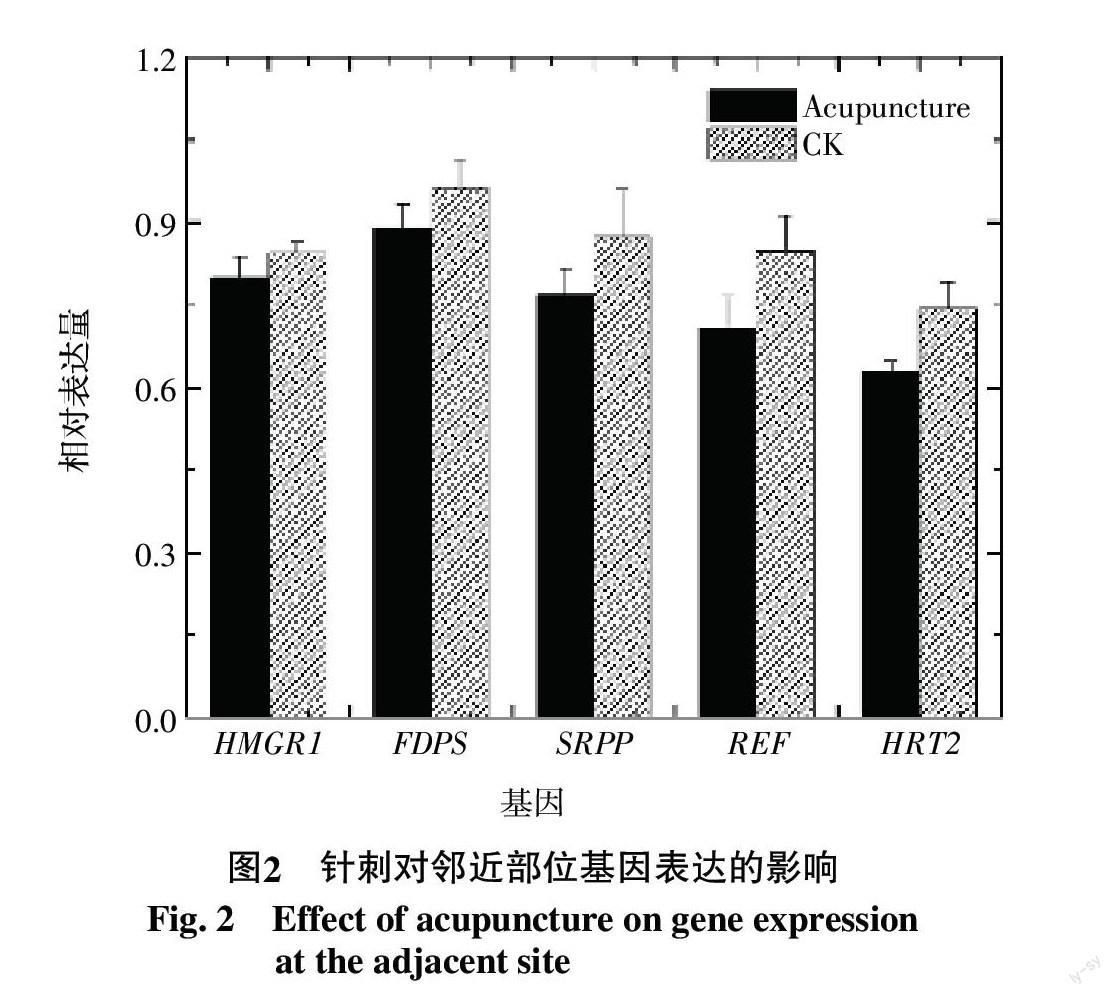
在無性系PR107中,膠乳中的5個基因在白天的表達呈上升趨勢,到18 : 00達到最高峰,隨后在22 : 00開始下調。但是表達量處于低谷的時間點存在一定差異,其中,HMGR1和FDPS的低谷出現在第2天10 : 00,而SRPP、REF和HRT2的表達最低點在第2天06 : 00。在RRIM600中,5個基因在白天都持續上調表達,但是,表達高峰出現的時間點存在差異。HMGR1和FDPS的表達高峰出現在22 : 00,而SRPP、REF和HRT2的表達高峰在第2天02 : 00。在CATAS7-33-97中,5個基因在白天持續上調表達,最高峰出現在18 : 00到22 : 00之間,但表達低谷都在06 : 00。在CATAS8-79中,5個基因表達在白天(06 : 00至14 : 00)都呈下調表達,但是表達低谷出現的時間點存在差異,其中HMGR1的表達低谷出現在22 : 00,FDPS、SRPP和REF的表達低谷出現在10 : 00,HRT2的表達低谷出現在14 : 00(圖3)。
2.4 同一基因在不同無性系間的表達分析
HMGR1在PR107、RRIM600、CATAS7-33-97和CATAS8-79中的表達高峰分別出現在14 : 00、18 : 00、14 : 00和06 : 00,其表達低谷分別出現在10 : 00、10 : 00、6 : 00和22 : 00。在CATAS8-79中,該基因在白天的表達呈下調趨勢,而在其他3個無性系中,該基因在白天的表達呈上調趨勢。FDPS表達的上下調趨勢在PR107和CATAS8-79中相似,但是表達高峰出現的時間點不同。在PR107中,表達高峰出現在14 : 00,而在CATAS8-79中,表達高峰出現在02 : 00。FDPS在RRIM600和CATAS7-33-97中都是白天緩慢上調,表達高峰出現在22 : 00。SRPP、REF和HRT2基因在PR107和CATAS7-33-97中的表達高峰出現在18 : 00,而在RRIM600和CATAS8-79中的表達高峰出現在02 : 00(圖3)。
3 討論與結論
本研究結果表明,相同基因在樹干不同部位的表達量沒有顯著性差異,局部針刺對鄰近部位的基因表達也沒有明顯影響,因此,不同時間點的膠乳樣品可以采自同一植株樹干的不同部位,可避免株間差異的干擾。
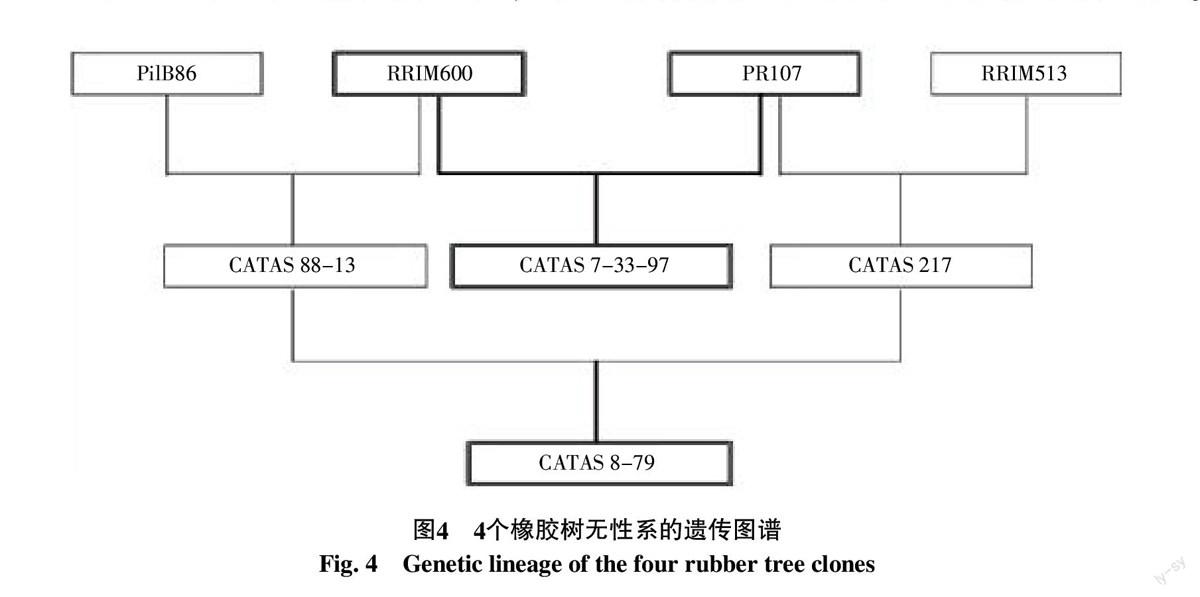
本文所選的5個橡膠生物合成關鍵酶基因表現出晝夜節律性表達,但是橡膠樹無性系PR107和RRIM600之間的基因表達模式存在較大差異,每個基因表達高峰出現的時間點在這2個品系間都不相同。SRPP、REF和HRT2基因在無性系CATAS7-33-97中的表達模式與PR107相似,而REF、HRT2和SPRR基因在CATAS8-79中的表達模式相對傾向于RRIM600。由于這些材料都種植在同一環境中,所以基因表達的差異性和相似性主要取決于遺傳因素。根據4個品系的遺傳圖譜(圖4),無性系CATAS7-33-97的親本是RRIM600和PR107[19],而CATAS8-79分別與RRIM600和PR107有1/4的血緣[20]。
有研究結果表明 :在擬蘭芥中時間調控的核心時鐘基因受到EARLY FLOWERING 3(AtELF3)的調控[21];在一些組織中有數千個基因在時鐘基因的調控下做規律性震蕩[22]。因此,時鐘基因的應答網絡分為三個層次:核心時鐘基因、上游生物鐘調節器和下游時鐘應答基因[23-24]。通過下游節律性表達基因找到上游核心時鐘調控基因,繼而發現時鐘調節器,有助于研究生物的生理表現,改良生物的適應能力等[25]。雖然本研究所選擇的橡膠生物合成關鍵酶基因不屬于核心時鐘基因,但是這些基因表達具有明顯的節律性,而且這種節律性表達在不同無性系中都存在,表明其具有一定的代表性。從研究結果可推測這些基因可能處于鐘基因應答網絡的下游,而對于環境因素對基因表達的影響還有待進一步研究。
參考文獻
[1] Chye M L, Kush A, Tan C T, et al. Characterization of cDNA and genomic clones encoding 3-hydroxy-3-methylglutaryl-coenzyme A reductase from Hevea brasilienis[J]. Plant Molecular Biology, 1991, 16: 567-577.
[2] van Deenen N, Bachmann A L, Schmidt T, et al. Molecular cloning of mevalonate pathway genes from Taraxacum brevicorniculatum and functional characterisation of the key enzyme 3-hydroxy-3-methylglutaryl-coenzyme A reductase[J]. Molecular Biology Reports, 2012, 4: 4 337-4 349.
[3] Suwanmanee P, Sirinupong N, Suvachittanont W. Regulation of 3-hydroxy-3-methylglutaryl-CoA synthase and 3-hydroxy-3-methylglutaryl-CoA reductase and rubber biosynthesis of Hevea brasiliensis(BHK)Mull[A]//Bach T J, Rohmer M(eds)Isoprenoid Synthesis in Plants and Microorganisms: New Concepts and Experimental Approaches. Springer, New York, 2013: 315-327.
[4] Tiski I, Marraccini P, Pot D, et al. Characterization and expression of two cDNA encoding 3-hydroxy-3-methylglutaryl coenzyme A reductase isoforms in coffee(Coffea arabica L.)[J]. Journal of Integrative Biology, 2011, 15: 719-727.
[5] Cornish K. The separate roles of plant cis and trans prenyl transferases in cis-1, 4-polyisoprene biosynthesis[J]. European Journal of Biochemistry, 1993, 218: 267-271.
[6] Cornish K, Backhaus R A. Rubber transferase activity in rubber particles of guayule[J]. Phytochemistry, 1990, 29: 3 809-3 813.
[7] Ok T K, Kim S H, Ohyama K, et al. Upregulation of phytosterol and triterpene biosynthesis in Centella asiatica hairy roots overexpressed ginseng farnesyl diphosphate synthase[J]. Plant Cell Reports, 2010, 4: 403-411.
[8] Oh S K, Kang H, Shin D H, et al. Isolation, characterization and functional analysis of a novel cDNA clone encoding a small rubber particle protein from Hevea brasiliensis[J]. Journal of Biological Chemistry, 1999, 274: 17 132-17 138.
[9] Berthelot K, Lecomte S, Estevez Y, et al. Rubber elongation factor(REF), a major allergen component in Hevea brasiliensis latex has amyloid properties[J]. Plos One, 2012, 7: e48065.
[10] Priya P, Venkatachalam P, Thulaseedharan A. Differential expression pattern of ruber elongation factor(Ref)mRNA transcripts from high andlow yielding clones of rubber tree(Hevea Brasiliensis Muell. Arg.)[J]. Plan Cell Reports, 2007, 10: 1 833-1 838.
[11] Collins-Silvaa J, Nurala A T, Skaggsa A, et al. Altered levels of the Taraxacum Kok-Saghyz(Russian dandelion)small rubber particle protein, TkSRPP 3, result in qualitative and quantitative changes in rubber metabolism[J]. Phytochemistry, 2012, 79: 46-56.
[12] Hillebrand A, Post J J, Wurbs D, et al. Down-regulation of small rubber particle protein expression affects integrity of rubber particles and rubber content in Taraxacum brevicorniculatum[J]. Plos One, 2012, 7: e41874.
[13] Asawatreratanakul K, Zhang Y W, Wititsuwannakul D, et al. Molecular cloning, expression and characterization of cDNA encoding cis-prenyltransferases from Hevea brasiliensis - a key factor participating in natural rubber biosynthesis[J]. European Journal of Biochemistry, 2003, 270: 4 671-4 680.
[14] Schmidt T, Lenders M, Hillebr R, et al. Characterization of rubber particles and rubber chain elongation in Taraxacum koksaghyz[J]. BMC Biochemistry, 2010, 11: 1 471-2 091.
[15] 門中華, 李生秀. 植物生物節律性研究進展[J]. 生物學雜志, 2009, 26: 53-55.
[16] Mas P, Yanovsky M J. Time for circadian rhythms: plants get synchronized[J]. Plant biology, 2009, 12: 574-579.
[17] Harmer S L, Hogenesch J B, Straume M, et al. Orchestrated transcription of key pathways in Arabidopsis by the circadian clock[J]. Science, 2000, 290: 2 110-2 113.
[18] Covington M F, Maloof J N, Straume M, et al. Global transcriptome analysis reveals circadian regulation of key pathways in plant growth and development[J]. Genome Biology, 2008, 9: R130.
[19] 黃華孫, 梁茂寰, 吳運通, 等. 中規模推廣級橡膠樹優良品系CATAS7-33-97的選育[J]. 熱帶作物學報, 1994, 15(2): 1-6.
[20] 方家林, 張曉飛, 李偉國, 等. 橡膠樹野生種質59號雜交子代早期鑒定初報[J]. 熱帶農業科學, 2013, 33(8): 20-23.
[21] Dixon L E, Knox K, Kozma-Bognar L, et al. Temporal repression of core circadian genes is mediated through EARLY FLOWERING 3 in Arabidopsis[J]. Current Biology, 2011, 21: 120-125.
[22] Yan J, Wang H, Liu Y, et al. Analysis of gene regulatory networks in the mammalian circadian rhythm[J]. PLOS Computational Biology, 2008, 4: e1000193.
[23] McCarthy M J, Nievergelt C M, Kelsoe J R, et al. A survey of genomic studies supports association of circadian clock genes with bipolar disorder spectrum illnesses and lithium response[J]. PLOS One, 2012, 7: e32091.
[24] Polhilko A, Fermandez A P, Edwards K D, et al. The clock gene circuit in Arabidopsis includes a repressilator with additional feedback loops[J]. Molecular Systems Biology, 2012, 8: 1-13.
[25] Helfer A, Dmitri A, Nusinow, et al. Lux arrhythom encodes a nighttime repressor of circadian gene expression in the Arabidopsis core clock[J]. Current Biology, 2011, 21: 126-133.

