阪崎克羅諾桿菌的16S rDNA 鑒定分型
婁彬彬 ,滿朝新,費 鵬,牛婕婷,李 然,柴云雷,李 理,姜毓君,
(1東北農業大學食品學院/乳品科學教育部重點實驗室,哈爾濱 150030;2東北農業大學/國家乳業工程技術研究中心,哈爾濱 150028)
阪崎克羅諾桿菌是一種含周身鞭毛、能運動并兼性厭氧的革蘭氏陰性無芽孢的粗短桿菌。目前克羅諾桿菌屬有7 個種[1,2],包括阪崎克羅諾桿菌(C.sakazakii)、丙二酸鹽陽性克羅諾桿菌(C.malonaticus)、蘇黎世克羅諾桿菌 (C.turicensis)、穆汀斯克羅諾桿菌(C.muytjensii)、都柏林克羅諾桿菌(C.dublinensis)、傳奇克羅諾桿菌 (C.universalis)、調味品克羅諾桿菌(C.condimenti)。阪崎克羅諾桿菌是重要的食源性條件致病菌,能夠引起嚴重的新生兒腦膜炎[3]、壞死性小腸結腸炎[4]和菌血癥[5]。該菌能分離自廣泛的食品來源(包括肉、奶酪、蔬菜、藥草、調料和嬰幼兒配方粉等)[6,7]、環境、以及昆蟲[8,9]。
傳統的生化鑒定方法(如API20E),表現出了很大的局限性,如重復性差、分辨力低、易出現假陽性結果等。因此,依據細菌遺傳性特征的分子分型(亦稱基因分型)技術逐步應用于致病菌的溯源分析及數據庫的構建中。本研究利用核糖體分型中以16sRNA 基因序列為研究對象,其在生物體中種類少,含量較多,存在與所有細胞中,功能同源且最為古老,序列變化與進化距離相適應[10]。可通過對保守序列設計通用引物可實現對細菌的鑒定,對可變序列設計特異性引物,用于檢測菌種或菌屬間的差異并對細菌進行分類。本研究利用16S rDNA 基因測序的方法對37 株阪崎克羅諾桿菌進行鑒定和分型,鑒定結果表明細菌在分子水平上的鑒定較生化鑒定具有更好的準確性和重復性。通過構建系統發育樹分析并根據序列同源性比對,可將所有菌株進行分型,這表明16S rDNA 基因測序可以對阪崎克羅諾桿菌進行分型,并進一步揭示其系統發育關系。說明16S rDNA基因測序能夠對阪崎克羅諾桿菌進行鑒定和分型。
1 材料與方法
1.1 試驗菌株
37 株阪崎克羅諾桿菌,分離自2009—2012年期間不同地區生產的乳粉和嬰幼兒配方粉樣品及某一濕法加工嬰幼兒配方粉生產企業的加工環境和成品的分離菌株,以及來自實驗室間菌株交換和相關單位的贈送的標準菌株。
1.2 試劑與儀器
細菌基因組DNA 提取試劑盒,北京天根生物技術有限公司;SeaKem Gold 瓊脂糖,美國Amresco 公司;引物,北京invitrogen 公司合成;胰蛋白胨大豆瓊脂(TSA),青島海博生物有限公司;Bcn1360 型生物超凈工作臺:上海佳勝儀器制造有限公司;9700 PCR 擴增儀:美國Applied Biosystems 公司;DYY-10C 型電泳儀:北京市六一儀器廠;UVP 凝膠成像系統:美國UVP 公司;TGC-16G 型離心機:上海安亭科學儀器廠;高壓蒸汽滅菌鍋:上海三申醫療器械有限公司;DHP-9272 型電熱恒溫培養箱:上海一恒科技有限公司。
1.3 試驗方法
1.3.1 菌株的培養、活化與純化
取出-80 ℃甘油保存的含有阪崎克羅諾桿菌的凍存管,將菌株恢復至室溫后,按2%的接種量接種于LB液體培養基,37 ℃培養8~12 h 進行活化。用接種環蘸取活化后的菌液于TSA 平板上劃線,37 ℃過夜培養。使平板上長出阪崎克羅諾桿菌單菌落,已達到純化的目的。
1.3.2 基因組總DNA 的提取
挑取TSA 平板中的單菌落,在LB 培養液中過夜培養。取2mL 培養液,進行阪崎克羅諾桿菌全基因組提取,采用細菌基因組DNA 提取試劑盒,具體操作步驟參照試劑盒說明。
1.3.3 16S rDNA 序列的擴增
阪崎克羅諾桿菌16S rDNA 序列擴增:采用16S rDNA 通用引物27f:‘5-AGTCTCTGATCATGC-CTCAG-3’和1492r:‘5-AAGGAGGTGCTCCAGCC-3’。擴增體系為50μL:10 × Buffer (含MgCl2),5.0 μL;dNTPs (10 mmol/L),2.0μL;引物27f (10 μmol/L),1.0 μL;引物1 492r (10 μmol/L),1.0μL;Pyrobest DNA Polymerase (2.5 U/μL),1.0μL;DNA 模板 (20~50 ng),2.0μL,補加ddH2O 至50μL。
PCR 擴增條件:95 ℃預變性5 min;95 ℃變性30 s,58 ℃退火30 s,72 ℃延伸1 min;循環數為30 個;72℃終延伸5 min;將PCR 擴增產物在1 %的瓊脂糖凝膠(含EB)上進行電泳檢測,100 V 電壓和100 A 電流電泳30 min,在凝膠成像系統下觀察,檢測是否有擴增條帶和條帶大小。
1.3.4 16S rDNA 序列的測序
將所有菌株的16S rDNA PCR 擴增產物和引物,送到上海立菲生物技術有限公司進行雙向測序。測序結果用Chromas 1.45 軟件進行查閱,用DNAMAN 5.29 軟件將雙向測序結果進行剪切與拼接,并將拼接后的序列在NCBI 上進行Blast 序列比對,實現對分離菌株的鑒定。從GenBank 核酸數據庫中提取IVERSEN 等遞交的克羅諾菌屬的相關菌種和亞種的模式菌株及腸桿菌屬的相關菌株的16S rDNA 序列作為參比,利用MEGA 4 軟件構建系統發育樹。
2 結果與分析
2.1 基因組總DNA 的提取與鑒定
將試驗菌株參照細菌基因組DNA 提取試劑盒,提取基因組總DNA。將提取后的產物于1%的瓊脂糖凝膠(含EB)上進行電泳檢測,電泳結果如圖1。

圖1 試驗菌株基因組總DNA 提取電泳圖
由圖1 可知,基因組DNA 條帶清晰單一、無雜帶,可知DNA 提取效果良好,可作為模板進行后續16S rDNA 序列擴增。
2.2 16S rDNA 序列的擴增
采用通用引物27f 和1492r 對菌株16S rDNA 序列進行擴增,擴增產物經1 %瓊脂糖凝膠電泳進行檢測,檢測結果圖2 所示。

圖2 試驗菌株16S rDNA PCR 產物電泳圖
由圖2 可知,擴增產物條帶單一明亮,且大小在1 500 bp 左右,與目的條帶大小一致。說明產物為16S rDNA 序列擴增產物,可送去測序。
2.3 16S rDNA 序列的測定
將37 株分離菌株的16S rDNA PCR 擴增產物和引物,送到上海立菲生物技術有限公司進行雙向測序。測序結果用軟件剪切和拼接后,將序列在NCBI 上進行Blast 序列比對。比對結果顯示,所有菌株(包括ES59)除了ES4 外,其余菌株與Cronobacter sakazakii 的同源性最高,相似性達99%,而ES4 的比對結果發現它與Citrobacter freundii 的同源性最高,達99%,因此ES4 被鑒定為弗氏檸檬酸桿菌,其余36 株菌株均鑒定為阪崎克羅諾桿菌。這一說明表明除菌株ES4 外,其余66 株分離菌株均鑒定為阪崎克羅諾桿菌。以分離菌株ES1 為例,經Blast 比對后得到的分值較高的菌株為Cronobacter sakazakii,且相似性達99% (附表),其他菌株的同源性分析結果與ES1 相似。
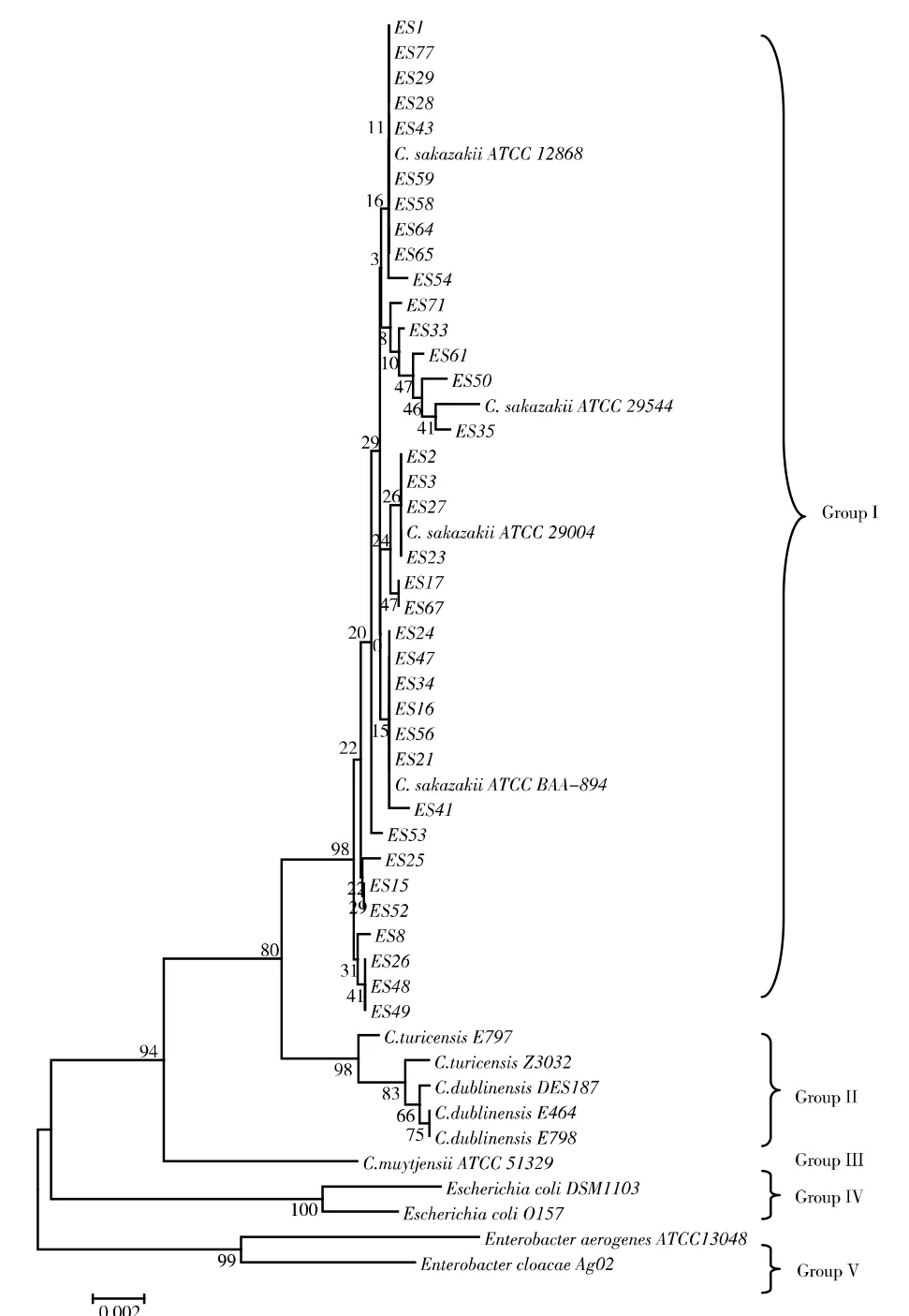
圖3 基于16S rDNA 序列構建的系統發育樹

附表 分離菌株ES1 在GenBank 中同部分16S rDNA 序列同源性比對結果
2.4 16S rDNA 序列構建系統發育樹及分析
因菌株ES4 被鑒定為弗氏檸檬酸桿菌,所以不參與后續試驗分析。利用MEGA 4.1 軟件對36 株分離菌株、阪崎克羅諾桿菌標準菌株,克羅諾菌屬的相關菌種和亞種的模式菌株(包括蘇黎世克羅諾桿菌和都柏林克羅諾桿菌)及腸桿菌屬的相關菌株(包括大腸埃希氏菌、產氣腸桿菌和陰溝腸桿菌),共計50 株菌株構建系統發育樹(圖3),參數設置為鄰接相鄰算法,Kimura 2-parameter 模式,重復次數為1 000 次。
對系統發育樹分析,結果顯示所有菌株被分成五個類群(Group I~Group V),其中分離菌株與阪崎克羅諾桿菌ATCC 標準菌株(除ATCC51329)在同一系統發育分支Group I,說明親緣關系最近,這與鑒定結果相符。克羅諾桿菌屬的其它菌種組成Group II 類群,ATCC51329 呈單一分支,為Group III。此外,大腸埃希氏菌和大腸埃希氏O157 屬于Group IV,產氣腸桿菌和陰溝腸桿菌屬于Group V。對Group I 中菌株進行多重序列比對分析,發現菌株間的相似度為97.35%,有研究學者認為,當16S rDNA 序列的相似度小于98.3 %時,可以對該組群進一步分型[11]。
在阪崎克羅諾桿菌的分支下可分成7 個子群,子群1 包含10 株分離菌株和1 株ATCC12868,它們之間的相似性為98.51%;子群2 含有5 株分離菌株和1 株ATCC29544,群內相似性為98.30%;子群3 包含6 株分離菌株和1 株ATCC29004,群內相似性為98.37%;子群4 包含8 株分離菌株和1 株ATCCBAA-894,群內相似性為98.82%;子群5 只含有一株菌株ES53;子群6 和7 分別含有3 株分離菌株和4 株分離菌株,它們的群內相似性分別為98.34%和98.46%。
3 結論
本試驗結果顯示,利用API20E 鑒定為阪崎克羅諾桿菌的ES4 被進一步鑒定為弗氏檸檬酸桿菌(Citrobacter freundii),其余分離菌株的16S rDNA 序列與Cronobacter sakazakii 的同源性最高,相似性達99%,鑒定為阪崎克羅諾桿菌。這表明基因水平的鑒定具有一定的準確性和可靠性,與傳統鑒定方法相比,具有更好的靈敏性,較優于傳統生化反應的鑒定。此外利用MEGA 4.1 軟件構建的系統發育樹顯示,所有分離菌株位于同一類群中(Group I),依據群內菌株間基因序列的同源性比對,又將Group I 中的菌株分成7 個子群,這說明16S rDNA 測序具有一定的分型能力,可以幫助我們更好的了解不同克羅諾桿菌之間的系統發育關系。
[1]董曉暉,李程思,吳清平,等.食品污染克羅諾桿菌(阪崎腸桿菌)的分離和鑒定[J].微生物學報,2013,53(5):429-436.
[2]JOSEPH S,CETINKAYA E,DRAHOVSKA H,et al.Cronobacter condimenti sp.nov.,isolated from spiced meat and Cronobacter universalis sp.nov.,a novel species designation for Cronobacter sp.genomospecies 1,recovered from a leg infection,waterand food ingredients [J].Int J Syst Evol Microb,2012,62:1277-1283.
[3]Biering G,Karlsson S,Clark N C,et al.Three cases of neonatal meningitis caused by Enterobacter sakazakii in powdered milk [J].Journal of Clinical Microbiology,1989,27(9):2054-2056.
[4]Van Acker J,de Smet F,Muyldermans G,et al.Outbreak of necrotizing enterocolitis associated with Enterobacter sakazakii in powdered milk formula [J].Journal of Clinical Microbiology,2001,39(1):293-297.
[5]Muytjens H L,Zanen H C,Sonderkamp H J,et al.Analysis of eight cases of neonatal meningitis and sepsis due to Enterobacter sakazakii [J].Journal of Clinical Microbiology,1983,18(1):115-150.
[6]TURCOVSK I,KUNIKOV K,DRAHOVSK H,et al.Biochemical and molecular characterization of Cronobacter spp.(formerly Enterobacter sakazakii)isolated from foods [J].Anton Leeuw,2011,99(2):257-269.
[7]王艷,焦彥朝.辣椒制品中阪崎腸桿菌的檢測與鑒定[J].貴州農業科學,2012,40(6):198-200.
[8]Hamilton,J.V.,Lehane,M.J.,Braig,H.R.,Isolation of Enterobacter sakazakii from midgut of Stomoxys calcitrans[J].Emerging Infectious Diseases,2003,9:1355-1356.
[9]Kandhai,M.C.,Reij,M.W.,van Puyvelde,K.,Guillaume-Gentil,O.,Beumer,R.R.,van Schothorst,M.,Occurrence of Enterobacter sakazakii in food production environments and households [J].Lancet,2004,363:39-40.
[10]雷正瑜.16S rDNA 序列分析技術在微生物分類鑒定中的應用[J].湖北生態工程職業技術學院學報,2006,4(1):4-7.
[11]Rabeb Miled-Bennour,Timothy C.Ells.Genotypic and phenotypic characterization of a collection of Cronobacter (Enterobacter sakazakii)isolates [J].International Journal of Food Microbiology,2010,139:116-125.

