PowerPlex21系統在親子鑒定中的應用評估
陳 玲,邱平明,陸慧潔,黃恩平,余嘉欣
(南方醫科大學 法醫學系;南方醫科大學司法鑒定中心,廣東 廣州 510515)
PowerPlex21是美國Promega公司2012年推出的常染色體檢測試劑盒。該試劑盒除包括CODIS推薦的13個核心基因座外,還增加了D1S1656、D6S1043、Penta E、D2S1338、Penta D、D12S391 和 D19S433 這7個基因座。本研究對該系統在1704例親子鑒定中的應用及相關問題進行了分析,以評估該系統在中國人群親子鑒定中的效力。
1 材料和方法
1.1 樣本
南方醫科大學司法鑒定中心親子鑒定檢案1704例(其中1937個無關個體),檢材類型包括人體血樣、唾液斑、帶毛囊毛發、羊水等。
1.2 儀器和試劑
9700擴增儀(美國AB公司);3130xL遺傳分析儀(美國AB公司);PowerPlex21試劑盒(美國Promega公司)。
1.3 方法
1.3.1 DNA提取、PCR擴增、STR分型檢測
采用chelex100法提取DNA,擴增體系和熱循環反應按PowerPlex21試劑盒說明書進行,擴增產物在3130xL遺傳分析儀電泳,Genemapper ID v3.2軟件分析結果。
1.3.2 統計分析
應用PowerStats V12.xls軟件,計算STR基因座的雜合度(H)、個體識別力(DP)、非父排除率(PE)、多態信息含量(PIC)等法醫學應用參數。累積非父排除率(CPE)和累積個體識別力(CDP)參照文獻[1]進行計算。
2 結果
2.1 PowerPlex21系統的法醫學應用參數
用PowerPlex21系統對1937個無關個體進行DNA分型,對分型結果進行法醫學應用參數統計,結果見表1。除TH01和TPOX基因座外,其余基因座均H>0.7;除 TPOX 基因座外,其余基因座均 DP>0.8;除D3S1358、CSF1PO、TH01和TPOX基因座外,其余基因座均 PE>0.5、PIC>0.7。
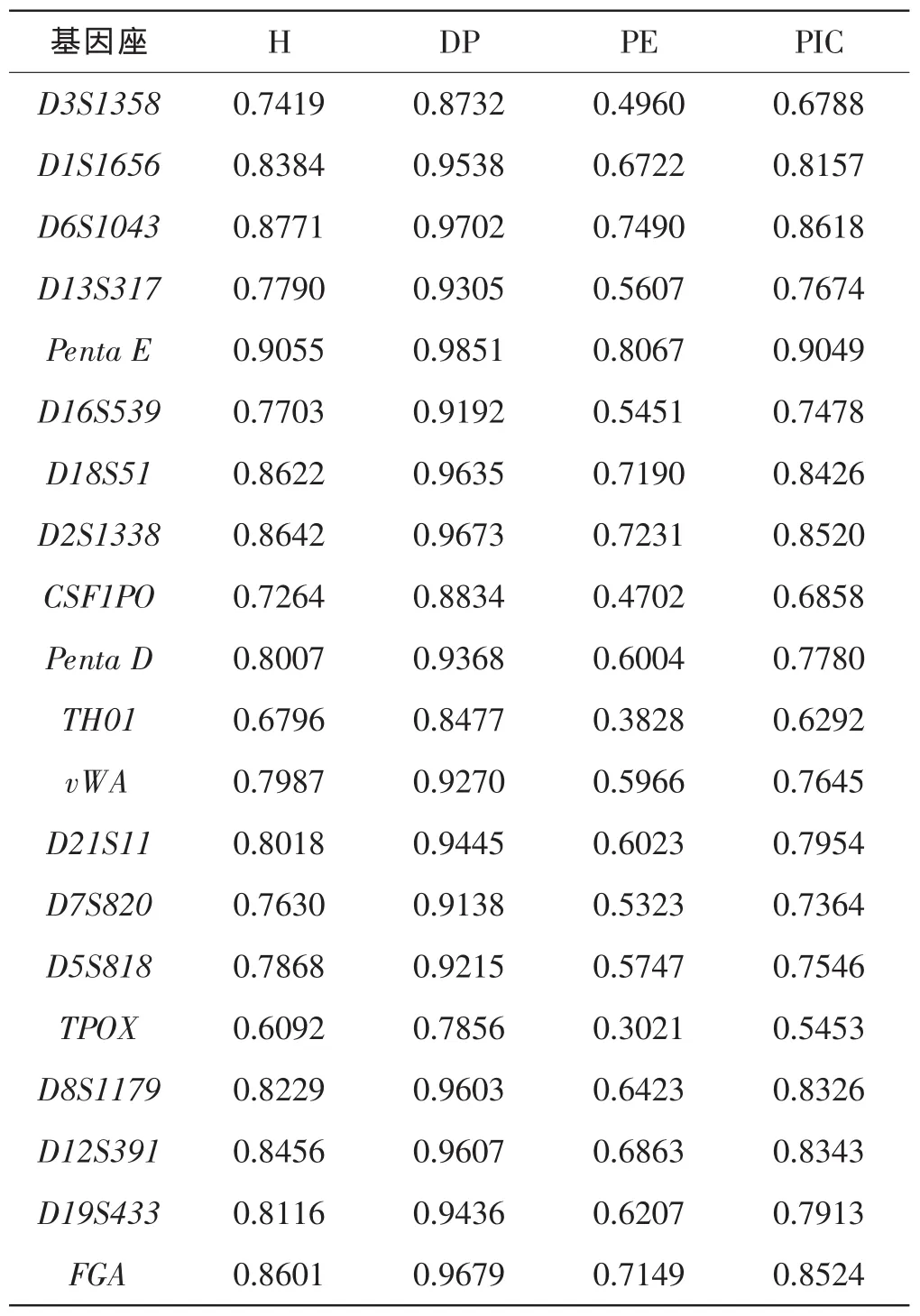
表1 中國漢族人群20個STR基因座的法醫學常用參數
2.2 PowerPlex21在親子鑒定中的應用
用PowerPlex21系統檢測1704例親子鑒定案件,認定親子關系的三聯體案件為82例、二聯體案件為1357例,排除親子關系265例,其中Penta E基因座的排除概率最高,為71.54%;TPOX的排除概率最低,為 20.97%(見表 2)。
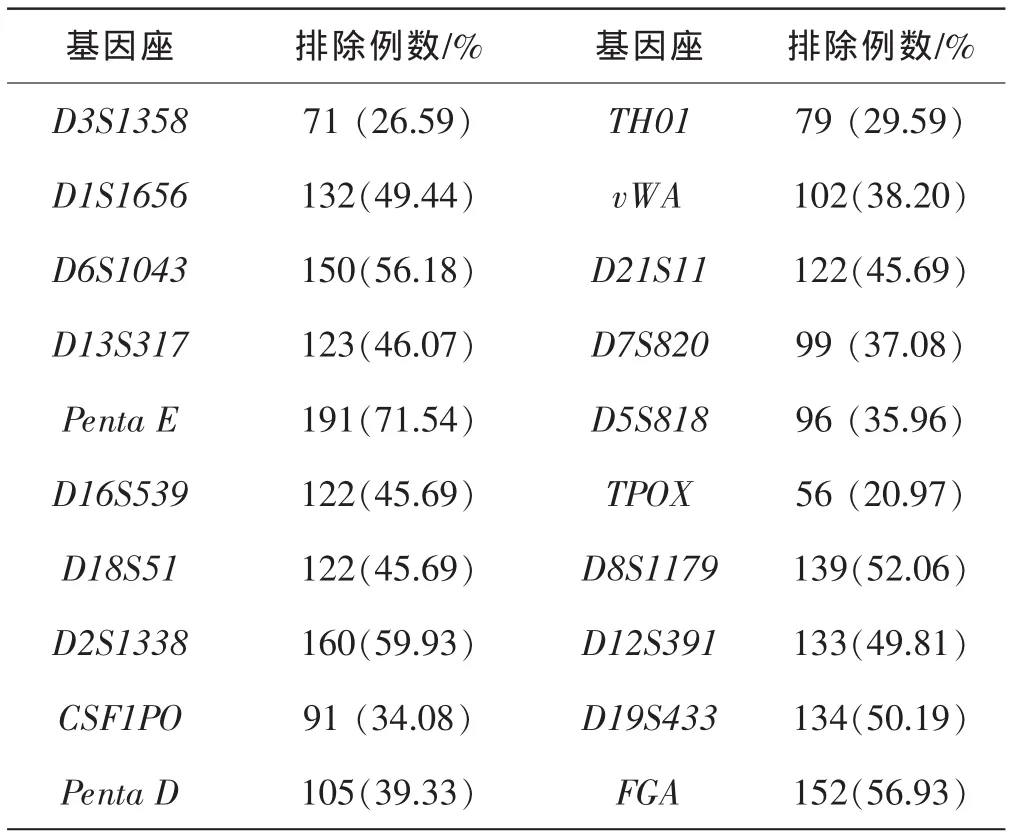
表2 PowerPlex21系統20個基因座作為排除基因座的概率
2.3 PowerPlex21系統20個STR基因座的突變情況
在認定的1439例親子鑒定中,其中44例表現為1個基因座違反遺傳規律。發現的突變基因座共15個,按突變率從高到低排列如下:D12S391、Penta E、D18S51、vWA、FGA、D5S818、D13S317、CSF1PO、D8S1179、D19S433、D3S1358、D1S1656、D2S1338、TH01和 D21S11,見表 3。
2.4 PowerPlex21系統的等位基因丟失
用PowerPlex21系統檢測的1704例親子鑒定案件中,2例親子鑒定經PowerPlex21系統檢測,父和子在D5S818基因座均表現為純合子、且峰高約為附近純合子基因座峰高的一半,經Sinofiler檢測證實PowerPlex21系統在D5S818基因座出現等位基因丟失,見圖1。

圖1 PowerPlex21和Sinofiler系統檢測兩例案件在D5S818基因座的分型圖譜
3 討論
Shriver等[2]認為當 DP>0.80、PE>0.50 時,該基因座屬于高度多態性。Gill等[3]認為,DP≥0.9、H≥0.7 的基因座具有高鑒別力。與目前常用的試劑盒相比,PowerPlex21試劑盒增加了D1S1656基因座,該基因座的H、DP、PE、PIC值均顯示滿足高度多態性基因座的標準,可見D1S1656基因座在親子鑒定和個體識別方面有較大的應用價值。
對于認定親子關系的案件,PowerPlex21檢測三聯體的CPI值均大于10000,檢測二聯體CPI小于10000的案例百分比低于其他檢測系統。這表明PowerPlex21可更好地滿足親子鑒定的要求。對于排除親子關系的案件,PowerPlex21系統在Penta E基因座的排除概率最高,在TPOX基因座的排除概率最低,這與統計得到的PE值一致。PowerPlex21系統認定的1439例親子鑒定中,有15個STR基因座檢出突變,突變率較高的基因座是 D12S391、Penta E、D18S51、vWA、FGA。
PowerPlex21檢測時出現了2例親子鑒定在D5S818基因座發生等位基因丟失的現象。這兩例案件在分析DNA圖譜時,根據該基因座的峰高特征(峰高約為附近雜合子基因座峰高的一半),且僅該基因座違反遺傳規律,初步預測可能是等位基因丟失,因此加做其他公司的擴增試劑盒(Sinofiler系統)檢測,確認用PowerPlex21檢測時在D5S818基因座出現等位基因丟失。這2宗案例易誤認為是發生一步突變,因此對于親代和子代為純合子的基因座判斷突變時要特別慎重。
[1] 侯一平.法醫物證學[M].北京:人民衛生出版社,2009:21-23.
[2] Shriver MD,Jin L,Boerwinkle E,A novel measure of genetic distance for highly polymorphic tandem repeat loci.Mol Biol Evol[J].1995, 12(5):914-920.
[3] Gill P.A new method of STR interpretation using in feren tial logic developm ent of a crim in al in telligence database.In t J Leg Med, 1996, 109(1):14-22.

