三角帆蚌四核苷酸重復微衛星的開發及特征分析
韓學凱李家樂,2王照旗白志毅
(1.上海海洋大學 農業部淡水水產種質資源重點實驗室,上海 201306;2.上海高校水產養殖學 E-研究院,上海 201306)
三角帆蚌四核苷酸重復微衛星的開發及特征分析
韓學凱1李家樂1,2王照旗1白志毅1
(1.上海海洋大學 農業部淡水水產種質資源重點實驗室,上海 201306;2.上海高校水產養殖學 E-研究院,上海 201306)
用磁珠富集法和生物素標記(ATAC)5為探針構建了三角帆蚌基因微衛星富集文庫。利用PrimerSelect軟件對得到的微衛星序列進行引物設計并合成得到43對引物,初篩后發現有20對引物可以擴增出穩定、清晰的目的產物帶。熒光標記這20對引物,然后用36只洞庭湖三角帆蚌擴增,發現17對引物呈現多態性。17個微衛星位點等位基因數的范圍為6-28。期望雜合度(He)、觀測雜合度(Ho)的范圍分別為0.677 6-0.946 4、0.638 9-1.000 0。多態信息含量(PIC)在0.630-0.929之間。統計分析結果顯示,其中有12個微衛星位點滿足Hardy-Weinberg平衡條件。與本實驗室之前開發的二核苷酸重復微衛星相比,該17對四核甘酸重復微衛星具有更高多態性,且更易于采用聚丙烯酰胺凝膠電泳等傳統方法分析基因型,有利于節約成本,從而為三角帆蚌群體遺傳分析、親子鑒定等技術提供了準確、價格低廉的微衛星標記。
三角帆蚌 微衛星 四核苷酸重復 多態性
三角帆蚌(Hyriopsis cumingii)是我國特有的淡水珍珠蚌,其產珠質量佳,所產珍珠占全世界淡水珍珠的90%以上[1-3]。近些年來,養殖三角帆蚌中出現的產珠質量差、個體變小等一系列問題,對珍珠的產量和效益都造成較大的影響[4]。因而篩選培育抗逆性好、生長性狀佳、產珠質量優的品種,促進三角帆蚌養殖業持續健康發展,是目前三角帆蚌遺傳育種研究的主要方向。
由于微衛星的突變率高,可產生大量多態性豐富的位點,如今已作為一種良好的分子標記廣泛應用于個體識別、種質資源鑒定、生物遺傳作圖、遺傳多樣性分析等方面的研究[5,6]。關于貝類微衛星開發與應用研究,國外已見較多報道[7-10],國內對淡水貝類如三角帆蚌[11-14]、背瘤麗蚌(Lamprotula leai)[15]、褶紋冠蚌(Cristaria plicata)[16]和背角無齒蚌(Anodonta woodiana)[17]的微衛星研究也逐漸開展起來。但目前國內淡水貝類開發的微衛星標記主要是雙核苷酸重復微衛星,用傳統銀染法檢測 PCR 產物時,擴增產物常常會出現影子帶,對于PCR產物長度的正確分析干擾較大。研究人類基因組發現,四堿基重復微衛星序列具有豐富的多態性,易于 PCR 擴增,具有明顯的等位基因差異,極少出現影子帶[18],故分型效果較二核苷酸微衛星要好。魯翠云等[19]研究鯉魚(Cyprinus carpio)后發現,在黑龍江鯉野生群體中,四核苷酸重復的微衛星標記表現出的多態性水平高于雙核苷酸重復。同樣,譚照君等[20]在研究鰱魚(Hypophthalmichtysmolitrix)中不同核苷酸重復微衛星特征時,發現四核甘酸重復微衛星不但多態性更高,而且遺傳穩定性更好,更加適合作為種群分析的工具標記。在我國特有淡水貝類三角帆蚌中,四核苷酸重復微衛星是否同樣具有高度多態性及其特征如何,還未見到有關研究報道。本研究通過生物素標記的微衛星探針獲得并篩選一些三角帆蚌四核苷酸重復微衛星位點,通過對其多態性初步分析,以期為三角帆蚌群體遺傳分析、親子鑒定技術、種質資源的保護等研究提供更多高質量的微衛星分子標記。
1 材料與方法
1.1 材料
本研究使用在洞庭湖采集到的三角帆蚌野生群體作為試驗樣本。
1.2 方法
1.2.1 三角帆蚌基因組DNA的提取及富集文庫的構建 取三角帆蚌外套膜組織作為提取材料,用苯酚-氯仿提取法提取其基因組DNA。基因組DNA的酶切和富集文庫的構建主要參照羅明等[14]和Li等[21]的方法。用RsaⅠ內切酶進行三角帆蚌基因組DNA的酶切,用試劑盒回收200-1 000 bp大小的片段用于文庫構建。微衛星序列的富集采用生物素標記的(ATAC)5探針,具體操作步驟與羅明等[14]所述相同。1.2.2 微衛星引物設計 先用Sequencher軟件去除載體及接頭后,再使用SSRhunter軟件來查找其中微衛星位點,之后用GeneQuest軟件查找并去除冗余序列。最后用PrimerSelect軟件在微衛星序列上進行引物設計。
1.2.3 多態性微衛星引物的初篩及熒光標記引物分析 引物設計好后,首先送上海邁浦生物科技有限公司合成普通引物,然后用3個三角帆蚌的基因組DNA作為模板來初篩這些引物。經PCR擴增反應后,用2%瓊脂糖凝膠電泳檢測分析擴增產物,從中挑選能夠穩定擴增出條帶且與設計引物片段大小相符的引物,經初步篩選的引物用來合成5'上游熒光引物(FAM、HEX)。從洞庭湖中采集36只野生三角帆蚌,取每只蚌的外套膜組織進行基因組DNA提取。用熒光引物對36個模板DNA進行PCR擴增,擴增后的熒光PCR產物送上海邁浦生物科技有限公司進行STR測序分析。
1.2.4 數據分析 用GeneMapper v4.0軟件對公司送來的基因組掃描結果進行分析并估算等位基因大小,利用PopGen32軟件計算以下參數:等位基因數(N)、有效等位基因數(A)、觀測雜合度(Ho)、期望雜合度(He),并進行Hardy-Weinberg平衡檢測(HWE)。用Cervus軟件來分析多態信息含量(PIC)。
2 結果
2.1 多態性引物的設計與篩選結果
用Sequencher軟件去除接頭及載體序列后,利用PrimerSelect設計并合成引物43對。用3個三角帆蚌的基因組DNA為模板對這43對引物進行初步篩選,PCR擴增產物經過2%瓊脂糖凝膠電泳檢測得到20對穩定擴增、條帶清晰并且大小相符的引物。對這20對引物的5'上游熒光引物分別用 FAM和 HEX 兩種熒光基團來修飾,用36只洞庭湖野生三角帆蚌個體對合成的20對熒光引物進行PCR擴增。用GeneMapper v4.0軟件對公司送來的基因組掃描結果進行分析后顯示,合成的20對引物中17對均具有良好的多態性(引物信息見表1),并對這17
對引物多態性進行深入評價。
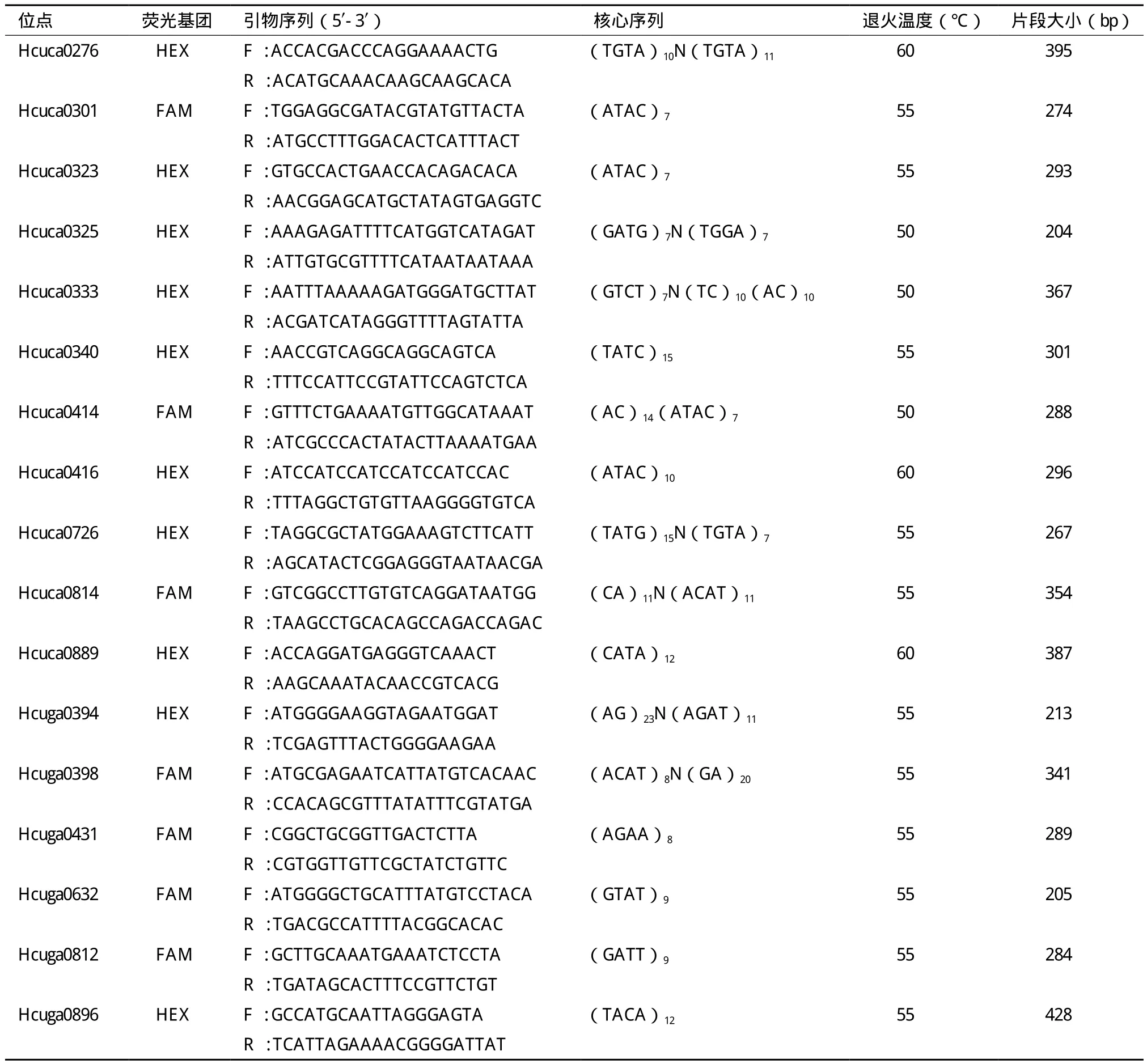
表 1 三角帆蚌 17個微衛星位點的核心序列及其引物特征
2.2 微衛星多態性引物對群體遺傳評估分析
用PopGen32軟件對這17對引物擴增的產物進行多態性評價分析,結果(圖1)顯示擴增產物大小在163-438 bp之間,17個微衛星位點等位基因數變動范圍為6-28,平均每對引物獲得等位基因數為17.52,其中位點Hcuca301獲得等位基因數最少,僅為 6個,位點Hcuga726獲得等位基因數最多,為28個,有效等位基因數在3.014 0-14.982 7之間,平均值為9.365 8,Ho在0.638 9-1.000,平均值為0.833 3;而He在0.677 6-0.946 4之間,平均值為0.877 9。用Cervus軟件來分析多態信息含量,多態信息含量 PIC在0.630-0.929之間,平均值為0.853 1。經Hardy-Weinberg平衡檢測顯示,17個微衛星位點中有12個滿足Hardy-Weinberg平衡條件(P>0.05)。17個微衛星位點多態性統計數據見表2。
3 討論
雖然近些年微衛星標記在水產動物中得到大
量開發和應用,但是目前多數水產動物開發的微衛星標記多為雙核苷酸重復。在有關人類二核苷酸重復微衛星研究中,發現PCR產物常常會出現比主帶小2 bp的影子帶[22,23]。本實驗室利用二核苷酸重復微衛星開展三角帆蚌種質資源評價及其育種工作中也發現同樣問題。研究發現,多核苷酸重復微衛星標記可以減少擴增產物中“影子帶”的出現,比二核苷酸重復序列遺傳穩定性更高且擴增更忠實[24],故基因分型效果較二核苷酸微衛星要好,且更易于采用聚丙烯酰胺等傳統方法分析基因型。目前國內四核苷酸重復微衛星標記在水產動物中開發應用較少,僅見鯉魚(Cyprinus carpio)[19]、鰱魚(Hypophthalmichtysmolitrix)[20]、方正銀鯽(Carassius auratus gibelioBloch)[25]、草魚(Ctenopharyngodon idellu)[26]中有相關研究,而淡水貝類中關于四核苷酸的開發與研究卻鮮有報道。而隨著磁珠富集法的發展和完善,四核苷酸重復的微衛星篩選比例得到提高,并逐漸被開發應用。本研究通過構建三角帆蚌ATAC四核苷酸重復的富集文庫,從中篩選和開發出17對四核甘酸重復微衛星標記用于研究。

表 2 三角帆蚌17個微衛星位點的遺傳參數

圖1 引物Hcuca0340在部分個體的擴增結果
多態信息含量(PIC)可以較好地反映片段的多態性和遺傳信息含量。在一個群體中,多態信息含量高低與其提供的遺傳信息多少成正比[27]。參照Botstein等[28]提出的衡量多態信息含量大小的指標,本研究中的17個微衛星位點的PIC在0.630-0.929之間,平均值為0.828 2,PIC值均大于0.5,為高度多態性位點,表明篩選微衛星位點多態性信息含量豐富。本實驗室羅明等[14]曾用20對微衛星引物對洞庭湖野生三角帆蚌進行了遺傳評估,本研究與其所用的洞庭湖樣本為同一群體,并采用相同的分析方法,不同的是本研究使用的是四核甘酸重復微衛星,因此可以通過對兩者結果進行對比,反映出雙核苷酸重復與四核苷酸重復微衛星標記在多態性和遺傳多樣性評價方面差別(表3)。比較發現,本研究的四核苷酸重復微衛星多態信息含量(PIC=0.853 1)高于羅明用雙核苷酸重復在同一群體中得到結果(PIC=0.823 9)。同樣,與許巧晴等[13]用13個雙核苷酸重復標記對洞庭湖野生三角帆蚌的
研究結果(PIC=0.519 8)相比,本研究四核苷酸重復微衛星評價的群體多態性也明顯高。有效等位基因數反映了群體中等位基因的分布均勻度,而雜合度可以反映群體中遺傳多樣性高低。本研究17個微衛星位點在洞庭湖三角帆蚌野生群體擴增到的有效等位基因數(A=9.365 8)和期望雜合度(He=0.877 9)兩項群體遺傳多樣性評估指標均高于羅明等用雙核苷酸重復在同一群體得到的結果(A=6.582 2,He=0.823 9),表明四核甘酸重復評價的群體遺傳多樣性更高,遺傳變異更大。與羅明和許巧晴研究結果相比,雖然本研究所用樣本量稍多,但總體可以判斷在三角帆蚌中四核苷酸重復微衛星多態性水平高于二核苷酸重復微衛星。此結果與譚照君等[20]通過研究鰱魚和魯翠云等[19]研究鯉魚后得出結論相似。四核苷酸重復的微衛星多態性更高可能是真核生物中四核苷酸重復多存在于非編碼區,在生物進化過程中承受的選擇壓力較小,因而可以較為“自由”突變的緣故[29]。
對17個微衛星位點進行Hardy-Weinberg平衡檢測,發現其中有5個位點偏離了平衡,這可能是雜合子缺失或研究采用樣本較少等緣故,這種情況在研究二核苷酸重復微衛星時也經常出現,屬合理現象。選取本研究中開發的幾對四核甘酸重復微衛星進行PCR擴增,通過傳統銀染法來檢測PCR產物,發現擴增帶譜清晰,“影子帶”出現較少。這與Edwards等[18]研究人類中四核甘酸重復微衛星特征所得結果相一致。因此本研究開發的四核甘酸微衛星可作為理想的分子標記。

表3 不同類型核心序列的微衛星對洞庭湖三角帆蚌的評估效果比較
4 結論
本研究開發的17對四核苷酸重復的微衛星標記,不但具有高度的多態性,而且是較二核苷酸重復微衛星更為高效的分子標記,可以在不采用SSR熒光標記分析技術,節約成本情況下,用傳統非熒光電泳檢測方法同樣取得較好效果,能夠為三角帆蚌群體遺傳分析、親子鑒定技術等研究提供廉價、準確的分子標記,是很好的微衛星資源。
[1] Hua D, Gu R. Freshwater pearl culture and production in China[J]. Aquaculture Asia, 2002, 7(1):6-8.
[2] Li JL, Li YS. Aquaculture in China—Freshwater pearl culture[J]. World Aquaculture, 2009, 40(1):60-62.
[3] 劉月英, 張文珍, 王躍先, 等.中國經濟動物志——淡水軟體動物[M]. 北京:科學出版社, 1979:114-115.
[4] 張元培.淡水珍珠養殖技術[M].長沙:湖南科學技術出版社, 1981.
[5] Lagercrantz U, Ellegren H, Andersson L. The abundance of various polymorphic microsatellite motifs differs between plants and vertebrates[J]. Nucleic Acids Research, 1993, 21(5):1111-1115.
[6] 盛巖, 鄭蔚紅, 裴克全, 等.微衛星標記在種群生物學研究中的應用[J]. 植物生態學報, 2002, 26(s):119-126.
[7] Sato M, Kawamata K, Zaslavskaya N, et al. Development of microsatellite markers for Japanese scallop(Mizuhopecten yessoensis)and their application to a population genetic study[J]. Mar Biotechn, 2005, 7(6):713-728.
[8] Geist J, Geismar J, Kuehn R. Isolation and characterization of the first microsatellite markers for the endangered swan musselAnodonta cygneaL.(Bivalvia:Unionoidea)[J]. Conserv Genet, 2009, 11(3):1103-1106.
[9] Jones JW, Culver M, David V, et al. Development and characterization of microsatellite loci in the endangered oyster musselEpioblasma capsaeformis(Bivalvia:Unionidae)[J]. Mol Ecol Not, 2004, 4(4):649-652.
[10] Shaw KM, King TL, Lellis WA, et al. Isolation and characterization of microsatellite loci inAlasmidonta heterodon(Bivalvia:Unionidae)[J]. Molec Ecol Not, 2006, 6(2):365-367.
[11] Bai ZY, Yin YX, Hu SN. Identification of genes involved in immune response, microsatellite, and SNP markers from expressed sequence tags generated from hemocytes of freshwater pearl mussel (Hyriopsis
cumingii)[J]. Mar Biotechnol, 2009, 11(4):520-530.
[12] Li JL, Wang GL, Bai ZY. Genetic diversity of freshwater pearlmussel(Hyriopsis cumingii)in populations from the five largest lakes in China revealed by inter-simple sequence repeat(ISSR)[J]. Aquacult Int, 2009, 17(4):323-330.
[13] 許巧情, 謝駿, 張書環, 等.三角帆蚌微衛星富集文庫的構建、鑒定及多態性分析[J].中國水產科學, 2010, 17(6):1200-1207.
[14] 羅明, 白志毅, 李應森, 等.三角帆蚌微衛星位點篩選及多態性分析[J].淡水漁業, 2012, 42(1):81-83.
[15] Xu B, Li JL, Wang GL. Development and characteriza tion of microsatellite loci inLamprotula leai, with cross-amplication inHyriopsis cumingii[J]. Conserv Genet Resour, 2011, 3(3):545-547.
[16] Jia MJ, Li JL, Wang SL. Isolation and characterization of 18 polymorphic microsatellite loci from freshwater pearl mussel(Cristaria plicata)[J]. Conserv Genet, 2010, 11(3):1131-1133.
[17] 汪桂玲, 蘇翔, 李家樂, 等.背角無齒蚌基因組(GT)n微衛星DNA特征[J].生態學雜志, 2011, 30(1):1-6.
[18] Edwards A, Civitello A, Hammond HA, et al. DNA typing and genetic mapping with trimeric and tetrameric tandem repeats[J]. American Journal of Human Genetics, 1991, 49(4):746-756.
[19] 魯翠云, 毛瑞鑫, 李歐, 等.鯉魚三、四核苷酸重復微衛星座位的篩選及特征分析[J].農業生物技術學報, 2009, 17(6):979-987.
[20] 譚照君, 張天奇, 魯翠云, 等.鰱三、四核苷酸重復微衛星標記的篩選及其特征分析[J].上海海洋大學學報, 2011, 20(3):329-335.
[21] Li JL, Wang GL, Bai ZY, et al. Ten polymorphic microsatellites from freshwater pearl musselHyriopsis cumingii[J]. Mol Ecol Notes, 2007, 7(6):1357-1359.
[22] Hauge XY, Litt M. A study of the origin of‘shadow bands’seen when typing dinucleotide repeat polymorphisms by the PCR[J]. Human Molecular Genetics, 1993, 2(4):411-415.
[23] Murray V, Monchawin C, England PR. The determination of the sequences present in the shadow bands of a dinucleotide repeat PCR[J]. Nucleic Acids Research, 1993, 21(10):2395-2398.
[24] Gastier JM, Pulido JC, Sunden S, et al. Survey of trinucleotide repeats in the human genome:assessment of their utility as genetic markers[J]. Human Molecular Genetics, 1995, 4(10):1829-1836.
[25] 曹柱, 魯翠云, 鄧先虎, 等.磁珠富集法篩選方正銀鯽的三、四核苷酸重復微衛星 DNA[J].淡水漁業, 2011, 41(1):23-28.
[26] 李文升, 劉翠, 魯翠云, 等.草魚三、四核苷酸重復微衛星標記的分離與特征分析[J].中國水產科學, 2011, 18(4):742-750.
[27] 田艷苓, 畢偉偉, 楊海玲, 等.濟寧百日雞群體遺傳多樣性的微衛星標記分析[J].經濟動物學報, 2010, 14(1):41-45.
[28] Botstein D, White RL, Skolnick M, et al. Construction of a genetic linkage map in man using restriction fragment length polymorphisms[J]. American Journal of Human Genetics, 1980, 32(3):314-331.
[29] 高煥, 孔杰.串聯重復序列的物種差異及其生物功能[J].動物學研究, 2005, 26(5):555-564.
(責任編輯 馬鑫)
Development and Characteristics of Tetranucleotide Repeat Microsatellite Loci in Hyriopsis cumingii
Han Xuekai1Li Jiale1,2Wang Zhaoqi1Bai Zhiyi1
(1. Key Laboratory of Freshwater Fishery Germplasm Resources, Ministry of Agriculture, Shanghai Ocean University, Shanghai 201306;2. Aquaculture Division,E-Institute of Shanghai Universities,Shanghai 201306)
A microsatellite-enriched library of the Hyriopsis cumingii was constructed using repeat-enrichment method with biotinlabeled oligo(ATAC)5and streptavidin coated magnetic beads. Forty-three pairs of primers designed by software PrimerSelect according to the sequences were synthesized for the pre-screening. Twenty pairs of primers which could amplify constant and legible expected DNA products were labeled by fluorescence. Seventeen microsatellite loci showed high levels in genetic polymorphism testing on 36 individuals sampled from Dongting Lake. The number of alleles at each locus ranged from 6 to 28. The expected and observed heterozygosities varied from 0.677 6 to 0.946 4 and from 0.638 9 to 1.000, respectively. The PIC value ranged from 0.630 to 0.929. Twelve microsatellite loci fitted to Hardy-Weinberg equilibrium. Compared with the previous dinucleotide repeat microsatellite developed in our laboratory, these 17 tetranucleotide repeat microsatellite loci showed higher polymorphism. Also, these 17 microsatellite loci can make genotyping easier by conventional methods such as polyacrylamide gel electrophoresis and save cost. Therefore, it offered accurate and inexpensive microsatellite markers for population genetic analysis and paternity test in Hyriopsis cumingii.
Hyriopsis cumingii Microsatellite Tetranucleotide repeat Polymophism
2013-12-16
上海市高校知識服務平臺項目(ZF1206),國家科技支撐計劃項目(2012BAD26B04),國家自然科學基金項目(31272657),上海市青年科技啟明星(A類)(12QA1401400),上海工程技術中心能力提升項目(13DZ2280500)
韓學凱,男,碩士,研究方向:種質資源與遺傳育種;E-mail:15618062362@163.com
白志毅,男,博士,副教授,研究方向:種質資源與遺傳育種;E-mail:zybai@shou.edu.cn

