EPSP合酶的研究進展
徐杰 蔣世云 傅鳳鳴 耿鵬飛 黃凱
(廣西科技大學生物與化學工程學院,柳州 545006)
EPSP合酶的研究進展
徐杰 蔣世云 傅鳳鳴 耿鵬飛 黃凱
(廣西科技大學生物與化學工程學院,柳州 545006)
5-烯醇式丙酮酰莽草酸-3-磷酸合酶(5-Enolpyruvylshikimate-3-phosphate synthase,EPSP合酶)是莽草酸途徑中的第六位酶,參與合成芳香族氨基酸以及部分次生代謝的產物,同時EPSP合酶不僅是除草劑草甘膦、抗菌素、抗寄生蟲藥物的作用靶酶,而且也是促進生物體內莽草酸積累的重要調控位點。近年來,隨著分子生物學技術的快速發展和對EPSP合酶的深入研究,EPSP合酶基因在耐草甘膦轉基因作物、醫藥衛生等方面被廣泛應用。對EPSP合酶的研究進展進行綜述及展望。
莽草酸途徑 EPSP合酶 莽草酸積累 耐草甘膦
1969年研究人員在研究真菌體中芳香族氨基酸的生物合成過程中發現EPSP合酶的存在,并將該酶命名為3-phosphoshikimate-1-carboxyvinyl transferase[1],1974年將其名改為5-Enolpyruvylshikimate-3-phosphate synthase,即5-烯醇式丙酮酰莽草酸-3-磷酸合酶,簡稱EPSP合酶。EPSP合酶主要存在真菌、細菌、藻類、高等植物及寄生于脊椎動物的Apicomplexan(頂復蟲)體內,該酶催化莽草酸途徑中的倒數第二步反應,是合成芳香族氨基酸以及部分次生代謝物過程的關鍵酶。迄今為止,EPSP合酶已經從原核和真核生物中純化出來,且在很多生物中分離克隆出能編碼EPSP合酶的基因(aroA)[2]。
由于EPSP合酶是農藥草甘膦作用的靶標而受到廣泛研究。草甘膦是目前使用最廣泛的一種廣譜性除草劑,但是作為一種非選擇性除草劑,能夠專一的抑制EPSP合酶的活性,從而阻斷植物內輔酶Q、葉酸、芳香族氨基酸等化合物的生物合成,使植物細胞分裂、葉綠素合成、蒸騰、呼吸及蛋白質等代謝受到影響而死亡[3,4]。隨著分子生物學技術的快速發展和對EPSP合酶的深入研究,1996年,美國Monsanto公司利用CP4農桿菌的耐草甘膦EPSP合酶的編碼基因研發出第一種耐草甘膦作物-耐草甘膦大豆(Roundup ready)。雖然研究人員已經發現上千種抗草甘膦的EPSP合酶,但是大多數不符合轉基因的要求[5]。目前,研究人員已經研發出許多耐草甘膦的轉基因作物,如棉花、玉米、油菜、水稻、番茄和楊樹等,但現在只有大豆、棉花、玉米、油菜和轉基因作物用于商業化大規模種植[6]。
EPSP合酶不僅是除草劑草甘膦作用的靶標,也可以作為抗菌素和抗寄生蟲藥物的靶標。另一方面,EPSP合酶是莽草酸代謝下游的一個重要酶,研究表明,減弱或切斷該酶基因的表達或抑制其酶活性也有利于莽草酸的合成積累,因此EPSP合酶也是促進生物體內莽草酸積累的重要調控位點[7-12]。
1 莽草酸途徑簡介
莽草酸途徑是存在于植物、細菌和真菌中的一條重要代謝途徑,而動物體內的代謝中是不存在此途徑的,此途徑使糖代謝和次生代謝緊密地聯系在一起。該途徑有7個酶參與催化反應,并且這7個酶都可以通過真核和原核細胞來源的基因表達獲得[13]。第一步反應是磷酸烯醇式丙酮酸(PEP)和赤蘚糖-4磷酸(E4P)的反應。PEP和E4P分別是由糖酵解途徑和戊糖磷酸途徑產生的物質。最后一步反應是分支酸的生成。分支酸不僅是莽草酸途徑的終產物,也是3種芳香族氨基酸和一些次生代謝產物的主要前體[14]。分支酸的去向如圖1所示。植物的次生代謝產物合成途徑包括苯丙烷代謝途徑、異戊二烯代謝途徑和生物堿合成途徑等。研究表明,許多次生代謝產物的芳香族氨基酸前體都是來自莽草酸途徑產生的芳香族氨基酸及其中間產物,因此莽草酸途徑在合成有商業價值的天然產物過程中起著非常重要的作用。莽草酸途徑在植物中的重要性也得到證實,由莽草酸途徑產生的物質大約占植物干重的35%以上[15]。
由于莽草酸途徑在哺乳動物、鳥類、魚類、爬行動物和昆蟲中是不存在的,近年來它逐漸成為抗菌素、除草劑和活體疫苗的靶標[16]。例如,由惡性瘧原蟲引起的瘧疾,每年大約200多萬人死于這種疾病,科學家利用莽草酸途經尋找新型的抗瘧疾藥,目前草甘膦作為瘧疾的療效藥已經在小鼠身上成功試驗[17]。
2 EPSP合酶的分類、細胞中定位及不同組織中的分布
目前EPSP合酶被分為兩種類型,類型I包括來源于大腸桿菌(Escherichia coli)和鼠傷寒沙門氏菌(Salmonella typhimurium)的EPSP合酶;類型II包括來源于覆盆子土壤桿菌(Agrobacterium tumefacienssp.CP4)、無色桿菌(Achromobactersp.LBAA)、假單胞菌(Pseudomonassp.PG2982)等[18]。類型II的EPSP合成酶多克隆抗體與類型I的EPSP合酶不會發生抗原抗體交叉反應,而且兩種類型序列一致性30%左右[19]。I型EPSP合酶對除草劑草甘膦敏感。II型EPSP合酶在草甘膦存在時,相對于I型EPSP合酶表現出更高的催化效率。研究人員將II型EPSP合酶轉入作物中可以對除草劑產生抗性,顯著有效的控制了田間雜草[20]。目前已知的II型EPSP合酶都是在微生物中存在的[13,21]。在今天市場上銷售的大多數耐草甘膦產品都含有農桿菌CP4-EPSPS[22]。
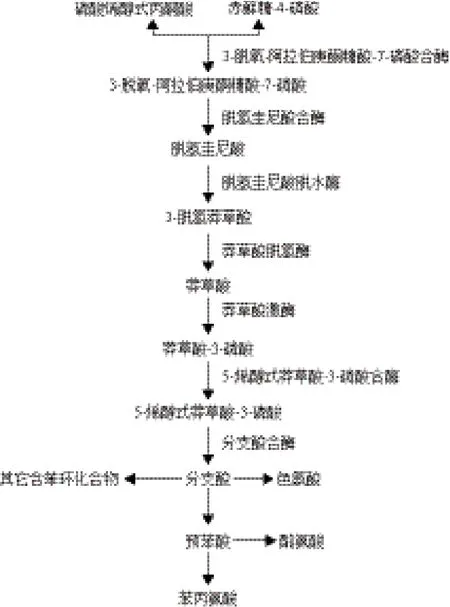
圖1 莽草酸途經
Della-Cioppa等[23]發現在植物細胞中,前體EPSP合酶存在于細胞核中,但是當該酶變為成熟的EPSP合酶(Mature enzyme)時,是存在于葉綠體內。EPSP合酶是通過前體EPSP合酶合成的。絕大多數植物的EPSP合酶的活性中心存在于葉綠體中。前體EPSP合成酶也具有催化活性,并且它的催化活性也受到草甘膦的抑制[24]。前體EPSP合酶的N端為信號肽所在的位置。信號肽的主要作用是引導EPSP合酶進入葉綠體基質,之后信號肽被水解,前
體蛋白就變成成熟的EPSP合酶。例如,童旭宏等[25]發現陸地棉EPSP合酶基因編碼521個氨基酸殘基,前74個氨基酸殘基為運輸肽,當前體EPSP合酶進入葉綠體后經加工剪切信號肽,變成成熟EPSP合酶[26]。成熟EPSP合酶包含447個氨基酸,比前體EPSP合酶氨基酸少了74個氨基酸,分子量約為48 kD[25]。信號肽對于EPSP合酶定位于葉綠體起著決定性的作用。微生物的EPSP合酶存在于細胞質中,并且沒有一段氨基酸序列在N端,所以微生物EPSP合酶無信號肽。我們通過轉基因技術把微生物的EPSP合酶基因導入植物中雖然能夠成功表達EPSP合酶,但是不能進入葉綠體,如果加上信號肽序列,就能把表達的EPSP合酶定位到葉綠體上。
植物中的EPSP合酶主要存在葉片、根、頂端分生組織、嫩葉和胚芽鞘中,葉片中合成大量的芳香族氨基酸,而在根、頂端分生組織、嫩葉和胚芽鞘中既能夠自身合成,也能吸收葉片中的芳香族氨基酸[27]。
3 EPSP合酶的三維結構與活性區
Krekel 等[28]成功純化出大腸桿菌的EPSP合酶。通過對此酶核磁共振及晶體衍射研究分析發現,其合酶由兩個結構域組成,每個結構域包含3個相同的βαβαββ折疊單位,每個折疊單位由兩個平行的α螺旋和4個β折疊組成(圖2)[29]。研究人員發現大腸桿菌EPSP合酶共有427個氨基酸殘基,PEP結合位點與Lys-22、Arg-124、Asp-313、Arg-344、Arg-386和Lys-411這6個氨基酸殘基有關,Arg-27與莽草酸-3-磷酸(S3P)結合位點有關[15]。EPSP合酶蛋白的N端和C端都在同一個結構域中,PEP主要與EPSP合酶的C端結構域結合,而S3P與EPSP合酶主要結合在N端結構域[29-31]。兩個平行的α螺旋在兩個結構域間形成了可以吸引負電荷基團的正電荷區。EPSP合酶存在狀態分為兩種:一種是開放(Open)狀態(圖3-a),當沒有底物與合酶結合,且兩個結構域相距較遠時存在的狀態[32];當酶與底物S3P結合時,兩個結構域相互靠近,并且在兩個結構域之間裂縫中出現酶的活性位點[33],這時合成酶存在的狀態為關閉(Closed)狀態(圖3-b)。
在關閉狀態下,采用限制性胰消化酶也很難使其消化,在開放口的90-102位氨基酸及附近的1區(123-134位氨基酸)、2區(140-152位氨基酸)、3區(355-366位氨基酸)與底物結合,從而避免了這些區域被消化,通過這個試驗確定了EPSP合酶的活性區域。如果將EPSP合酶與底物結合區域的某些氨基酸殘基替代,如將Gly96改為Ala、Pro101改為Ser,則草甘膦就很難與合酶結合,這時該酶表現出耐草甘膦的特性[34]。
易弋等[2]對鹽藻的EPSP合酶三級結構進行預測并對氨基酸序列進行多重比對發現,鹽藻EPSP合酶的氨基酸序列與高等植物、細菌一樣,都有開放和關閉兩種狀態,并且都含有3處氨基酸序列非常保守的區域,這些非常保守的區域就是EPSP合酶與酶底物PEP、S3P或草甘膦結合的活性位點,如果改變保守區域中的一個位點,那么酶的活性將會改變,草甘膦的抑制作用也會減弱。
研究人員發現細菌和植物的EPSP合酶是一個單體酶,分子量大約為44-48 kD[35]。而真菌的EPSP合酶屬于AROM蛋白結構域中的其中一個,AROM蛋白通常是以二聚體形式存在的,單體形式下沒有催化活性,分子量比較大,最多含有1 618個氨基酸,最少含有1 563個氨基酸,而且AROM蛋白是一個同時具有脫氫奎尼酸合酶、脫氫奎尼酸脫水酶、莽草酸脫氫酶、莽草酸激酶和EPSP合酶活性的5功能多肽,催化真菌中芳香族氨基酸合成途徑的第3步到第6步的反應[36,37]。
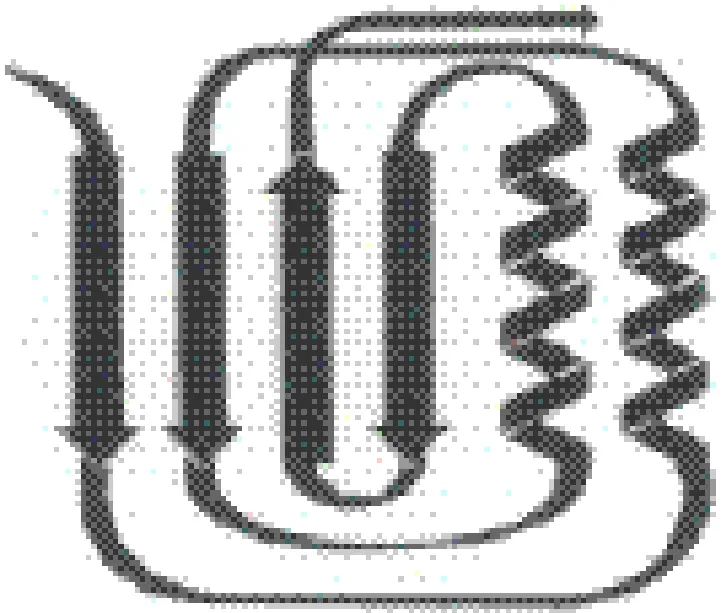
圖2 EPSP合酶二級結構[13]
4 EPSP合酶的催化機制
EPSP合酶作為莽草酸途徑的第6位酶,參與合
成芳香族氨基酸(苯丙氨酸、酪氨酸及色氨酸)和許多次生代謝產物,如泛醌(Ubinquinone),維生素K(Vitamink),維生素K2類(Menaquinone)等。EPSP合酶催化烯醇式丙酮酸基團從PEP轉化到S3P上形成產物EPSP,并釋放出一分子無機磷,這個化學反應是PEP的C-O鍵裂解而不是經過P-O鍵的裂解,并且在PEP上發生乙烯基質子的交換[38]。之前有研究者認為合酶與底物的結合是隨機的,但是通過用動力學優化的底物誘導試驗證實了酶與底物結合是有先后順序的[39]。1988年,Wibbenmeyer等[40]將大腸桿菌EPSP合酶純化,當純度達到97%以上時,通過核磁共振(NMR)和快速淬火動力學(Raid quench kinetics)方法得到了EPSP合酶催化S3P和PEP的反應是一個縮合反應,EPSP合酶和S3P及PEP形成了一個四面體過渡態Ⅰ,并伴隨著生成一個EPSP縮酮副產物Ⅱ,過渡態Ⅰ隨后生成EPSP并釋放一分子無機磷(圖4-a)。1997年,Studelska等[41]利用固體核磁共振(Solid-state NMR)方法對EPSP合酶的催化機理重新進行了研究,發現PEP首先和EPSP合酶形成過渡態A、B,之后與S3P結合形成合酶縮酮過渡態C,而后生成EPSP(圖4-b),這種催化機理也得到了研究者的證實[42,43]。
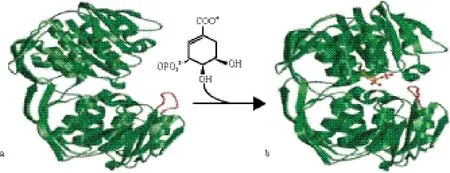
圖3 EPSP合酶開放和關閉結構示意圖[13]
5 EPSP合酶基因及表達
目前已經從細菌,真菌和植物中分離克隆出許多EPSP合酶基因[44-46]。在原核生物中,許多EPSP合酶基因被克隆與分析。1984年,Duncan等[44,47]首次克隆出長度為1 284 bp,編碼427個氨基酸殘基的E.coliEPSP合酶基因,并且該基因內沒有內含子。Comai等[48]對鼠傷寒沙門氏菌(Salmonellavtyphimurium)EPSP合酶基因進行克隆發現,基因編碼427個氨基酸,合酶蛋白的分子量約為46 kD。有研究者對大腸桿菌與鼠傷寒沙門氏菌的EPSP合酶基因進行了對比發現,二者的核苷酸序列差異為21%,同義密碼子有78%在第1位或第二位堿基上發生變化,65%在第3位堿基上變化,氨基酸序列的同源性為93%,并且兩種多肽的15個N端氨基酸殘基也是相同的,在86-131位氨基酸之間的區域為高度保守區域,而在302-371和381-422位氨基酸之間的區域為C端高度保守區域。脯氨酸分別位于這兩種合酶序列中的不同位置,位于鼠傷寒沙門氏菌EPSP合酶序列的85位上,而在大腸桿菌中位于合酶序列的81位上,并且在81-85位的氨基酸殘基在鼠傷寒沙門氏菌EPSP合酶序列中是不存在的,兩者其它部位的脯氨酸是完全保守的[49]。
在真菌中,許多EPSP合酶基因也被克隆與分析。Charles等[45]報道了真菌構巢曲霉(Aspergillus nidulans)的EPSP合酶基因,其合酶屬于AROM蛋白結構域中的其中一個,基因中含有一個單一的閱讀框架,長度為5 311 bp。于海濤等[36]克隆分離得到了假絲酵母TY-JM和黃曲霉TZ1985的EPSP合酶蛋白編碼序列。假絲酵母TY-JM 的EPSP合酶編碼序列長度為1 311 bp,編碼436個氨基酸,理論分子量為46 kD,黃曲霉TZ1985的EPSP合酶編碼序列長度為1 353 bp,編碼450個氨基酸,理論分子量為48 kD。通過與其它物種EPSP合酶氨基酸序列對比分析,TY-JM EPSP合酶基因與杜氏假絲酵母、熱帶假絲酵母和白假絲酵母等物種的同源性為80%,而與棒曲霉、核盤菌的同源性為66%,TZ1985 EPSP合酶基因與土曲霉、煙曲霉、白曲霉和黑曲霉的同
源性為90%,而與白假絲酵母、杜氏假絲酵母和熱帶假絲酵母的同源性為68%。在進化關系上,TYJM EPSP合酶和TZ1985 EPSP合酶與真菌的EPSP合酶有很近的親緣關系,而與植物和細菌的EPSP合酶親緣關系比較遠。
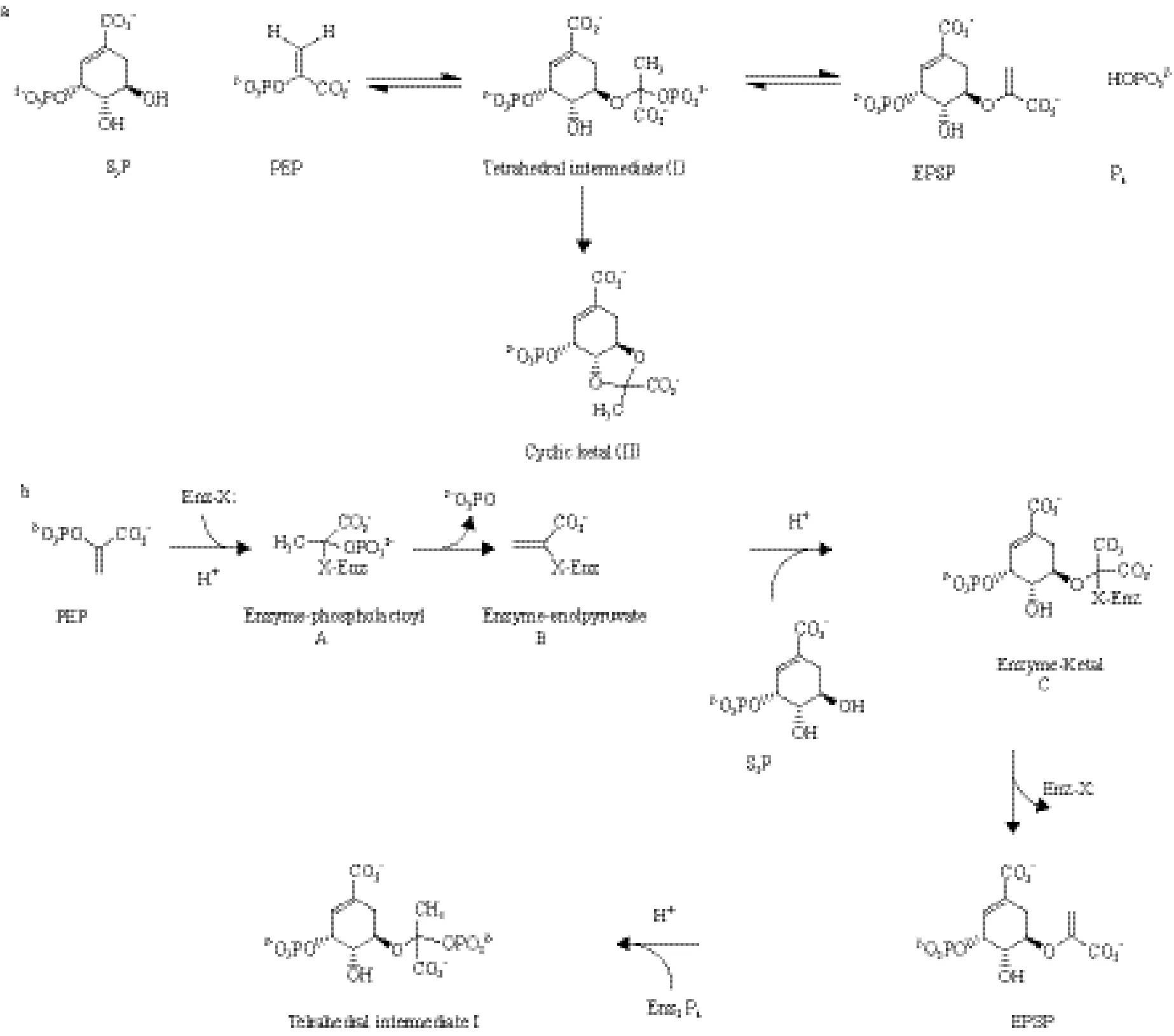
圖4 EPSP合酶的催化反應[38]
在植物中,一些EPSP合酶基因也被克隆與分析。Wang等[31]發現煙草中有兩種EPSP合酶基因的cDNA,兩種cDNA的長度分別為2 000 bp和1 300 bp,二者編碼的氨基酸序列和核苷酸序列的同源性分別為95.9%和89%,但是兩種EPSP合酶的酶活力相同。劉曉軍等[24]從諸葛菜中也克隆出兩段EPSP合酶基因片段,其中一段長為797 bp,另一段長為1 157 bp,兩個片段的外顯子序列是完全相同的,內含子相差很大,但是這兩段基因編碼出相同的EPSP合酶,這兩個片段所編碼氨基酸序列與歐洲油菜、玉米、擬南芥、黑麥草、水稻及矮牽牛的一致性分別為85%、80%、83%、80%、79%和79%。
Gasser等[50]通過對比細菌、植物和真菌的EPSP合酶編碼區,獲得了一個比較一致的模式(圖5),三者EPSP合酶中約有38%的氨基酸殘基相同,細菌和植物之間EPSP合酶的同源性為54%,真菌和植物之間EPSP合酶的同源性為38%,這些數據表明真菌和植物中的EPSP合酶的分枝比細菌和植物中此酶的分枝出現的早。
研究者發現EPSP合酶基因在生物不同的組織
中表達也是有差異的,在矮牽牛中其基因表達量最高的組織是花瓣[50],在銀杏中表達量最高的是在果實和葉子中;其次為莖,最低的是在根中[51]。木本植物喜樹EPSP合酶基因表達量最高的組織是在葉子,根中表達量最低[52]。薤白EPSP合酶基因在幼葉中表達量最高,而在壯葉和莖中表達量最低[53]。出現這種表達差異的原因可能是由于EPSP合酶基因在不同的組織中發生轉錄的起始位點不相同,也有研究者認為黃酮含量也與不同組織中EPSP合酶基因的表達量有關[54]。
另一方面,有研究者報道,改變高度保守區域的氨基酸殘基,可使EPSP合酶與草甘膦的結合能力發生改變,從而使生物表現出抗草甘膦特性[55]。
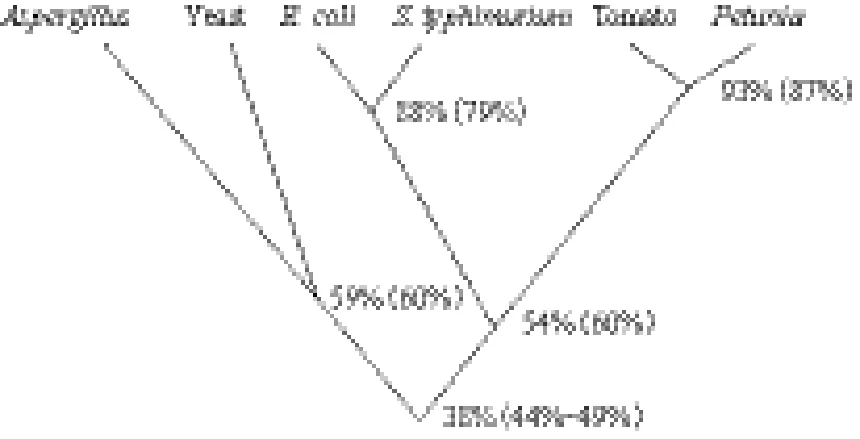
圖5 EPSP合成酶的系統進化樹[50]
6 EPSP合酶基因的應用
EPSP合酶不僅是除草劑草甘膦作用的靶標,也可以作為抗菌素和抗寄生蟲藥物的靶標;另一方面,EPSP合酶活性大小對生物體內莽草酸的積累有重要影響[7-12]。
6.1 草甘膦及抗草甘膦EPSP合酶基因
6.1.1 草甘膦及其與EPSP合酶作用機制 草甘膦作為一種非選擇性除草劑,在殺死雜草的同時,也會殺死農作物,從而限制了草甘膦的使用范圍和時間,自1974年在美國注冊登記以來,已成為當今世界上銷售量最大且使用最廣的農藥品種,問世30多年來經久不衰[18]。草甘膦與EPSP合酶的作用機制是:草甘膦與PEP在結構上非常相似,在莽草酸途徑中草甘膦占據了EPSP合酶和PEP的連接位點,形成EPSP合酶·S3P ·glyphosate的復合物,使EPSP合酶催化烯醇式丙酮酸基團從PEP轉化到S3P上形成EPSP的過程停止,競爭性抑制EPSP合酶的活性導致芳香族氨基酸和一些芳香化合物合成受阻,最終導致一些關鍵性代謝物和激素如酚類化合物、木質素、代謝失調,使生物體不能進行正常的氮代謝而死亡,而且EPSP合酶進入葉綠體的過程也受草甘膦的抑制[56-58]。
6.1.2 抗草甘膦EPSP合酶基因來源 1983年,Comai等[59]從鼠傷寒沙門菌中分離克隆出了抗草甘膦的突變基因——aroA基因,此后許多研究者對抗草甘膦基因進行了深入廣泛的研究。抗草甘膦的EPSP合酶基因主要來源于微生物和植物,而且研究者發現植物中的EPSP合酶比細菌中EPSP合酶對草甘膦的抗性大約低了兩個數量級。表1為目前已獲得的主要抗草甘膦EPSP合酶基因及其來源。目前獲得抗草甘膦EPSP合酶編碼基因的途徑主要有兩種:第1種是從天然抗草甘膦的物種或突變體中克隆分離出抗草甘膦基因或突變基因。在天然抗草甘膦基因中,土壤農桿菌CP4-EPSP合酶基因是應用最廣泛的抗草甘膦EPSP合酶基因,并形成商業化生產。除此之外,研究者從一些物種的突變體中克隆分離出抗草甘膦突變基因,如研究者克隆大豆、油菜、馬鈴薯及擬南芥抗草甘膦的EPSP合酶突變基因發現,大豆EPSP合酶基因編碼的104Gly突變為Ala,油菜EPSP合酶基因編碼的Gly96突變為Ala,馬鈴薯和擬南芥EPSP合酶第101位上Gly變為Ala[60]。Shah等[61]從篩選出的抗草甘膦矮牽牛MP4——G細胞系中克隆出EPSP合酶的cDNA,將其cDNA導入矮牽牛葉片后,對草甘膦的抗性明顯提高。Scott等[62]發現牛筋草的EPSP合酶基因發生突變,第101位脯氨突變為色氨酸后,對草甘膦具有很低的敏感度,表現出抗草甘膦。第2種是利用遺傳學方法改造EPSP合酶編碼基因,提高對草甘膦的抗性:定點突變可以減小酶與草甘膦的親和力或提高酶與底物的親和力,從而提高對草甘膦的抗性。Tian等[63]利用定點突變使蘋果的EPSP合酶基因編碼的Thr101變為Ala,Ala187變為Thr,通過動力學分析證實兩個氨基酸突變后提高了對草甘膦的抗性;Ming等[64]使用易錯傾向PCR隨機突變技術使來源于大腸桿菌和沙門氏菌的aroA基因發生突變和重組,得到aroM1、aroM2、aroM3和aroM44種突變體,通過EPSP合酶活性的動力學分析表明,
4種突變體的4個突變基因編碼的EPSP合酶的活性比突變前提高了2-10倍,與烯醇式丙酮酸的親和性提高了2.5-19倍,與草甘膦的Ki值提高了0.4-8倍;Lebrun等[65]使用定點突變,從玉米中克隆獲得了抗草甘膦的CP4基因,而且先正達公司通過轉基因技術開發出了耐草甘膦玉米品種GA21。研究者發現,通過遺傳學方法改造EPSP合酶編碼基因,雖然提高了生物對草甘膦的抗性,但是也降低了EPSP合酶的催化活性[21],因此,目前改造成功并用于商業化生產的抗草甘膦轉基因作物的基因幾乎全部是CP4-EPSP合酶基因。
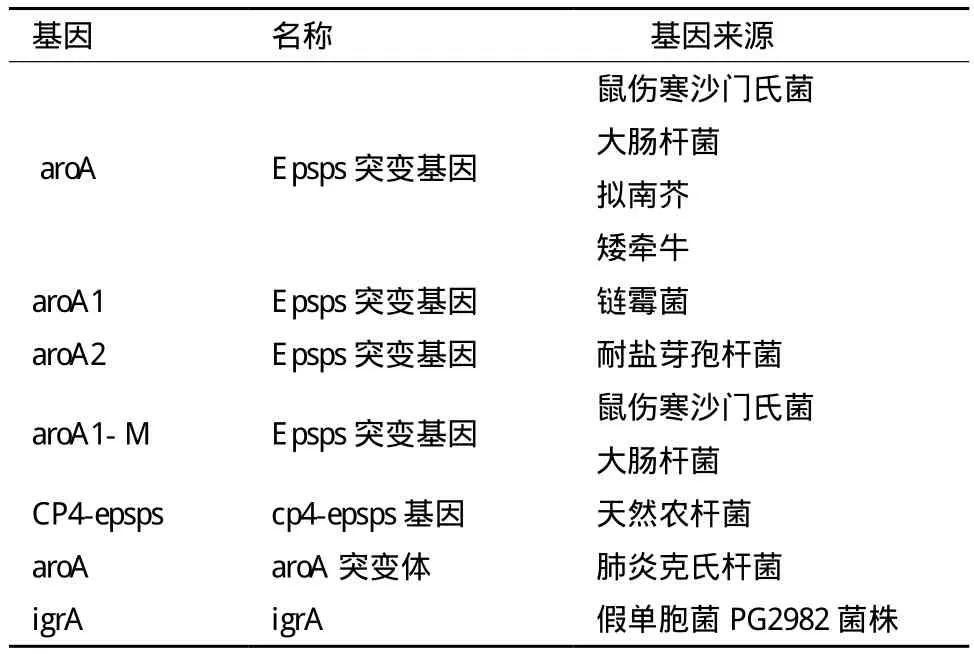
表 1 主要抗草甘膦的EPSP合酶基因及其來源[66,67]
6.2 在抗菌素和抗寄生蟲藥物上的應用
EPSP合酶可以作為抗菌素的靶標,如研究者通過試驗刪除肺炎鏈球菌和支氣管炎博德特菌的EPSP合酶的基因后,發現二者的毒性減弱[68,69]。在抗寄生蟲藥物方面,研究者已經證實草甘膦能夠抑制Toxoplasm gondii、Cryptosporidium以及Plasmodium falciparum(malaria)等頂復門寄生蟲的生長[17,70]。
6.3 EPSP合酶新型抑制劑
草甘膦作為EPSP合酶的抑制劑是眾所周知的,但隨著EPSP合酶晶體與催化底物機理的深入研究,也推動了EPSP合酶新型抑制劑的研發。如研究者根據S3P·glyphosate、EPSP合酶的分子模型,設計并合成了與除草劑草甘膦具有類似抑制作用的化合物4[71,72],用等溫低定量熱法測定EPSP合酶與化合物4的連接常數值Kd為0.53±0.04 μmol/L,而EPSP合酶·S3P + glyphosate的Kd值為0.15±0.03 μmol/L,前者Kd值僅僅是后者的3.5倍,而且通過動力學、光譜學和微量熱法證實了化合物4與EPSP合酶的結合位點和S3P與酶的結合位點是相同的,從而抑制EPSP合酶的催化活性。研究者報道了莽草酸衍生物、羥基丙二酸衍生物等都可以與EPSP合酶形成四面體過渡態,從而使EPSP合酶活性受到抑制[73]。目前,草甘膦依然是作用于該靶標的有效抑制劑,還沒有新的EPSP合酶抑制劑商業化應用,但隨著除草劑草甘膦的使用越來越廣,抗草甘膦雜草也越來越多,所以EPSP合酶新的抑制劑研究必將會成為熱點[74]。
6.4 EPSP合酶對莽草酸代謝的影響
莽草酸是生物體內許多物質合成代謝的中間體,也是人工化學合成許多生物堿、芳香氨基酸與吲哚衍生物、手性藥物(如抗病毒藥)的原料。近年來,禽流感爆發此起彼伏,危害人類健康安全。莽草酸是合成目前在臨床上唯一一種有效抗禽流感藥物——磷酸奧斯米韋(商品名為達菲,為瑞士Roche制藥公司生產)的關鍵原料[75-77],因此生物合成莽草酸的研究是目前的一個研究熱點。EPSP合酶是莽草酸代謝下游的一個重要酶,研究表明,減弱或切斷該酶基因的表達或抑制其酶活性也有利于莽草酸的合成積累[7-12]。Kramer等[7]報道,敲除EPSP合酶基因,使莽草酸和莽草酸-3-磷酸大量累積,后者去磷酸化后就獲得目標產物。2011年公布的美國專利[9]“草甘膦在微生物發酵法生產莽草酸中的使用”就是利用草甘膦對EPSP合酶的抑制作用來提高微生物發酵生產莽草酸產量。Bresnahan等[10]研究報道,在給小麥施加非選擇性除草劑草甘膦后,不僅有助于農作物的豐收,而且草甘膦還可以被吸收并轉移到植物的活性生長區內,通過抑制EPSP合酶的合成,能夠干擾莽草酸的正常代謝途徑。在試驗中觀察到,在小麥乳熟期使用草甘膦會使其內部的莽草酸最多增加24倍,若在蠟熟期使用,其內部的莽草酸能增加3倍。
7 結語
自EPSP合酶發現至今已有40多年,其理論與應用研究都取得了顯著的成果。目前已發現并克隆出許多種EPSP合酶基因,特別在農業上的應用,
隨著許多耐草甘膦的EPSP合酶基因被克隆出來,有力的推動了耐草甘膦作物的發展。雖然通過遺傳學方法改造EPSP合酶編碼基因,提高了生物對草甘膦的抗性,但是也降低了EPSP合酶的催化活性,所以目前改造成功并用于商業化生產的抗草甘膦轉基因作物的基因幾乎全部是CP4-EPSP合酶基因,因此該領域有待進一步深入研究。另一方面,EPSP合酶也可以作為抗菌素和抗寄生蟲藥物的靶標。因此對EPSP合酶與草甘膦以及其它抑制劑的結合模式的研究,能夠為設計出新的除草劑、抗寄生蟲藥物以及抗菌素提供有力的理論依據。此外,EPSP合酶也是促進生物體內莽草酸積累的重要調控位點,因此隨著人們對EPSP合酶的全面深入研究,相信EPSP合酶基因會在醫藥衛生、農業生產等方面發揮更重要的作用。
雖然現在一些EPSP合酶的三維晶體結構已通過物理化學方法和X-射線衍射得到,但是許多研究只報道了未與配體結合的EPSP合酶三級結構,所以不能完全確定酶的活性位點,但隨著計算機技術的發展,可利用計算機模擬出EPSP合酶與底物的結合位點;另一方面通過計算機模擬,可為篩選出較高活性的EPSP合酶及抗草甘膦較強的EPSP合酶基因節省大量人力物力。本課題組首先利用分子對接軟件進行反對接計算配體小分子化合物與不同生物的EPSP合酶結合自由能,同時根據計算結合自由能最低的結合模式分析底物、抑制劑(草甘膦)與EPSP合酶作用機理及其構效關系,提出EPSP合酶基因的改造方案,應用基因工程手段對目標生物EPSP合酶基因進行遺傳改造,改造選育出有利于莽草酸積累的生物突變體或耐草甘膦作物;另一方面根據底物、抑制劑(草甘膦)與EPSP合酶作用機理及其構效關系,也可提出抑制劑改造方案,合成新的抑制劑備選化合物,通過試驗驗證最終篩選獲得更有效的除草劑、抗菌素或抗寄生蟲藥物。
[1] Ahmed SI, Giles NH. Organization of enzymes in the common aromatic synthetic pathway:evidence for aggregation in fungi[J]. Bacteriology, 1969, 99(1):231-237.
[2] 易弋. 鹽生杜氏藻EPSP合成酶基因的克隆、功能鑒定及其結構的光譜學性質分析[D]. 成都:四川大學, 2007.
[3] 游大慧, 騫宇, 王健美, 等. 蕓苔EPSPS基因cDNA的克隆和表達載體的構建[J]. 四川大學學報, 2004, 41(3):661-664.
[4] Priestman MA, Healy ML, Funke T, et al. Molecular basis for the glyphosate-insensitivity of the reaction of 5-enoipyruvylshikimate 3-phosphate synthase with shikimate[J]. FEBS Letters, 2005, 579(25):5773-5780.
[5] 劉樹鵬, 李剛強, 王楠, 等.CP4-EPSPS蛋白在大腸桿菌中的表達與制備[J]. 中國農業科技導報, 2012, 14(1):91-97.
[6] 童漢華, 曹一平, 章善慶. 耐草甘膦作物的選育研究概況[J].中國稻米, 2006(6):12-14.
[7] Kramer M, Bongaerts J, Bovenberg R, et al. Metabolic engineering for microbial production of shikimic acid[J]. Metabolic Engineering, 2003, 5(4):277-283.
[8] Iomantas YAV, Abalakina EG, Polanuer BM, et al. Method for producing shikimic acid:US, B1, 6436664[P]. 2002-8-20.
[9] Bogosian G, Frantz JP. Use of glyphosate to produce shikimic acid in microorganisms:US, A1, 20110020885[P]. 2011-1-27.
[10] Bresnahan GA, Manthey FA, Howatt KA, et al. Glyphosate applied preharvest induces shikimic acid accumulation in hard red spring wheat(Triticum aestivum)[J]. Journal of Agricultural and Food Chemistry, 2003, 51(14):4004-4007.
[11] Marchiosi R, Ferrarese MLL, Bonini EA, et al. Glyphosate-induced metabolic changes in susceptible and glyphosate-resistant soybean(Glycine maxL. )roots[J]. Pesticide Biochemistry and Physiology, 2009, 93(1):28-33.
[12] Tong XH, Daud MK, Sun YQ, et al. Physiological and molecular mechanisms of glyphosate tolerance in anin vitroselected cotton mutant[J]. Pesticide Biochemistry and Physiology, 2009, 94(2-3):100-106.
[13] 李亮. Ⅱ型EPSP合酶的功能域鑒定及大腸桿菌在草甘膦沖擊下的基因表達譜分析[D]. 北京:中國農業科學院, 2010.
[14] Knaggs AR. The biosynthesis of shikimate metabolites[J]. Natural Product Report, 2003, 20(1):119-136.
[15] 聶燕芳. T42M突變型EPSP合成酶基因的克隆和研究[D].廣州:中山大學, 2004.
[16] Pereira JH, Canduri F, Basso LA, et al. Structural bioinformatics study of EPSP synthase fromMycobacterium tuberculosis[J]. Biochemical and Biophysical Research Communications, 2003, 312(3):608-614.
[17] Roberts F, Roberts CW, Johnson JJ, et al. Evidence for the shikimate pathway inapicomplexan parasites[J]. Nature, 1998, 393(6699):801-805.
[18] 王宏偉, 梁業紅, 史振聲, 等. 作物抗草甘膦轉基因研究概況[J]. 作物雜志, 2007(4):9-12.
[19] Tian YS, Xu J, Han J, et al. Complementary screening, identification and application of a novel class II 5-enopyruvylshikimate-3-phosphate synthase fromBacillus cereus[J]. World J Microbiol Biotechnol, 2013, 29(3):549-557.
[20] Barry GF, Kishore GM, Padgette SR, et al. Glyphosate-tolerant 5-enolpyruvylshikimate-3-phosphate synthases:US, B1, 6248876[P]. 2001-6-19.
[21] Funke T, Han HJ, Healy-Fried ML, et al. Molecular basis for the herbicide resistance of Roundup Ready crops[J]. Proceedings of the National Academy of Sciences of the United States of America, 2006, 103(35):13010-13015.
[22] 柏亞羅. 耐草甘膦作物的發展歷程和展望[J]. 現代農藥, 2005, 4(5):26-30.
[23] Della-Cioppa G, Bauer SC, Klein BK, et al. Translocation of the precursor of 5-enolpyruvylshikimate-3-phosphate synthase into chloroplasts of higher plantsin vitro[J]. Proceedings of the National Academy of Sciences of the United states of America, 1986, 83(18):6873-6877.
[24] 劉曉軍. 諸葛菜(Olychophragmus violaceus)EPSP合成酶基因的克隆與分析[D]. 成都:四川大學, 2002.
[25] 童旭宏, 吳玉香, 祝水金. 陸地棉EPSPS基因的克隆及其組織特異性表達分析[J]. 棉花學報, 2009, 21(4):259-264.
[26] Zhao J, Hemnann KM. Cloning and sequeneing of a second cDNA encoding 3-deoxy-D-arabino-Heptulosonate7-phosphate synthase fromSolanum tuberosuL.[J]. Plant Physiol, 1992, 100(2):1075-1076.
[27] Lynda GB, Celine BS. Subtle effects of herbicide use in the context of genetically modified crops:a case study with glyphosate(Roundup)[J]. Ecotoxicology, 2003, 12(1-4):271-285.
[28] Krekel F, Oecking C, Amrhein N, et al. Substrate and inhibitorinduced conformational changes in the structurally related enzymes UDP-N-acetylglucosamine enolpyruvyl transferase(MurA)and 5-enolpyruvylshikimate3-phosphate synthase(EPSPS)[J]. Biochemistry, 1999, 38(28):8864-8878.
[29] William WC, Abdel-Meguid SS, Lim LW, et al. Structure and topological symmetry of the glyphosate target 5-enolpyruvylshikimate-3-phosphate synthase:A distinctive protein fold[J]. Proceedings of the National Academy of Sciences of the USA, 1991, 88(11):5046-5050.
[30] 劉光全, 劉柱, 陸偉, 等. 草甘膦抗性新基因的克隆、序列分析及其蛋白高級結構預測[J]. 中國生物化學與分子生物學報, 2006, 22(3):197-203.
[31] Wang YX, Jones JD, Weller SC, et al. Expression and stability of amplified genes encoding 5-enolpyruvylshikimate-3-phosphate synthase in glyphosate-tolerant tobacco cells[J]. Plant Molecular Biology, 1991, 17(6):1127-1138.
[32] Schonbrunn E, Eschenburg S, Shuttleworth WA. Interaction of the herbicide glyphosate with its target enzyme 5-enolpyruvylshikimate-3-phosphate synthase in atomic detail[J]. Proceedings of the National Academy of Sciences of the USA, 2001, 98(6):2944-2946.
[33] Li L, Lu W, Han YL, et al. A novel RPMXR motif among class II 5-enolpyruvylshikimate-5-enolpyruvyl synthases is required for enzymatic activity and glyphosate resistance[J]. Journal of Biotechnology, 2009, 144(4):330-336.
[34] 劉錫娟. 轉EPSPS基因抗草甘膦煙草棉花的研究[D]. 北京:中國農業科學院, 2007.
[35] 徐軍望, 馮德江, 李旭剛, 等. 水稻EPSP合酶基因的克隆、結構分析和定位[J]. 中國科學, 2002, 32(2):97-104.
[36] 于海濤. 抗草甘膦真菌的分離及EPSPS基因克隆與表達研究[D]. 哈爾濱:東北農業大學, 2012.
[37] 于寒穎, 楊謙. 核盤菌arom基因的克隆及其在釀酒酵母中的表達[J]. 微生物學報, 2006, 46(1):43-47.
[38] Lewis J, Johnson KA, Anderson KS, et al. The catalytic mechanism of EPSP synthase revisited[J]. Biochemistry, 1999, 38(22):7372-7379.
[39] Ream JE, Yuen HK, Frazier RB, et al. EPSP synthase:binding studies using isothermal titration microcalorimetry and equilibrium dialysis and their implications for ligand recognition and kinetic mechanism[J]. Biochemistry, 1992, 31(24):5528-5534.
[40] Wibbenmeyer J, Brundage L, Stephen R, et al. Mechanism of the EPSP synthase catalyzed reaction:Evidence for the lack of a covalent carboxyvinyl intermediate in catalysis[J]. Biochemical and Biophysical Communications, 1988, 153(2):760-766.
[41] Studelska DR, McDowell LM, Espe MP, et al. Slowed enzymatic
turnover allows characterization of intermediates by solid-state NMR[J]. Biochemistry, 1997, 36(50):15555-15560.
[42] Jakeman DL, Mitchell DJ, Shuttleworth WA, et al. On the mechanism of 5-enolpyruvylshikimate-3-phosphate synthase[J]. Biochemistry, 1998, 37(35):12012-12019.
[43] 向文勝, 張文吉, 王相晶, 等. EPSP合成酶的特性及新抑制劑的研究進展[J]. 農藥學學報, 2000, 2(2):1-8.
[44] Duncan K, Lewendon A, Coggins JR. The complete amino acid sequenee ofEseheriehia coli5-enolpyruvylshikimate-3-phosphate synthase[J]. FEBS Lett, 1984, 170(1):59-63.
[45] Charles G, Keyte JW, Brammar WJ, et al. Structure and nucleotide sequence of the complex AROM locus ofAspergillus nidulans[J]. Nucleic Acids Res, 1986, 14(5):2201-2213.
[46] Klee HJ, Muskopf YM, Gasser CS. Cloning of anArabidopsis thalianagene encoding 5-enolpyruvylshikimate-3-phosphate synthase:sequence analysis and manipulation to obtain glyphosate-tolerant plants[J]. Mol Genet, 1987, 210(3):437-442.
[47] Duncan K, Coggins JR, TheserC-aroAoperon ofEscherichia coli. A mixed function operon encoding enzymes from two different amino acid biosynthetic pathways[J]. Biochem J, 1986, 234(1):49-57.
[48] Comai L, Facciotti D, Hiatt WR, et al. Expression in plants of a mutantaroAgene fromSalmonella typhimuriumconfers tolerance to glyhosate[J]. Nature, 1985, 317:741-744.
[49] Maskell DJ, Morrissey P, Dougan G. Cloning and nueleotide sequence of thearoAgene ofBordetella pertussis[J]. J Baeteriol, 1988, 170(6):2467-2471.
[50] Gasser CS, Winter JA, Hironaka CM, et al. Structure, expression, and evolution of the 5-enolpyruvylshikimate-3-phosphate synthase genes of petunia and tomato[J]. The Journal of Biological Chemistry, 1988, 263:4280-4287.
[51] 程華, 李琳玲, 王燕, 等. 銀杏EPSPS基因克隆及表達分析[J].西北植物學報, 2010, 30(12):2365-2372.
[52] Gong Y, Liao Z, Chen M, et al. Characterization of 5-enolpyruvylshikimate 3-phosphate synthase gene fromCamptotheca acuminata[J]. Biologia Plantarum, 2006, 50(4):542-550.
[53] Jiang X, Dai XZ, Li YQ, et al. Semiquantitative analysis ofEPSPSgene expression in tissues ofAllium macrostemon Bunge[J]. Journal of Hunan Agricultural University, 2007, 33(5):542-545.
[54] Benfey PN, Takatsuji H, Ren L, et al. Sequence requirements of the 5-enolpyruvylshikimate-3-phosphate synthase 5[prime]--upstream region for tissue-specific expression in flowers and seedlings[J]. Plant Cell, 1990, 2(9):849-856.
[55] 向文勝. 抗除草劑草甘麟轉基因作物[J]. 東北農業大學學報, 1998, 29(1):92-98.
[56] Vanghn KC, Duke SO. Biochemical basis of herbicide resistance[J]. Chem Plant Protect, 1991, 7:141-169.
[57] Dill GM. Glyphosate-resistant crops:history, status and future[J]. Pest Manage Sci, 2005, 61(3):219-224.
[58] Tan S, Evans R, Singh B. Herbicidal inhibitors of amino acid biosynthesis and herbicide-tolerant crops[J]. Amino Acids, 2006, 30(2):195-204.
[59] Comai L, Sen LC, Stalker DM. An alteredaroAgene product confers resistance to the herbicide glyphosate[J]. Science, 1983, 221(4608):370-371.
[60] 周曉卉, 黃麗華, 蔣向, 等. 薤白EPSP合成酶基因cDNA的克隆與原核表達分析[J]. 中國農業科學, 2009, 42(7):2297-2304.
[61] Shah DM, Horsch RB, Klee HJ, et al. Engineering herbicide tolerance in transgenic plants[J]. Science, 1986, 233(4762):478-481.
[62] Scott R, Damian J, Rodriguez MT, et al. Glyphosate-resistant goosegrass identification of a mutation in the target enzyme 5-enolpyruvylshikimate -3-phosphate synthase[J]. Plant Physiology, 2002, 129(3):1265-1275.
[63] Tian YS, Xu J, Peng RH, et al. Mutation by DNA shuffling of 5-enolpyruvylshikimate-3-phosphate synthase from Malus domestica for improved glyphosate resistance[J]. Plant Biotechnology, 2013, 11(7):829-838.
[64] Ming H, Zhong YY, Yan FN, et al. A new type of class I bacterial 5-enopyruvyl -shikimate-3-phosphate synthase mutants with enhanced tolerance to glyphosate[J]. Biochimica et Biophysica Acta(BBA)General Subjects, 2001, 1568(1):1-6.
[65] Lebrun M, Sailland A, Freyssinet G, et al. Mutated 5-enolpyruvylshikimate-3-phosphate synthase, gene coding for said protein and transformed plants containing said gene:US, B1, 6566587[P]. 2003-5-20.
[66] 阮燕曄, 薛寥莎. 植物抗除草劑基因研究進展[J]. 南方農業學報, 2012, 43(4):462-466.
[67] 周心童. 抗除草劑基因EPSPS在棉花雜種優勢中的利用極其
快速轉育[D]. 保定:河北農業大學, 2012.
[68] McDevitt D, Payne DJ, Holmes DJ, et al. Novel targets for the future development of antibacterial agents[J]. Applied Microbiology, 2002, 92(S1):28-34.
[69] McArthur JD, West NP, Cole JN, et al. An aromatic amino acid auxotrophic mutant ofBordetella bronchisepticais attenuated and immunogenic in a mouse model of infection[J]. FEMS Microbiology Letters, 2003, 221(1):7-16.
[70] Mckinney JD, JacobsJr WR, Bloom BR. 3 Persisting problems in tuberculosis[R]. New York:Emerging Infections, Academic Press, 1998:51-146.
[71] Marzabadi MR, Gruys KJ, Walker MC, et al. An EPSP synthase inhibitor joining shikimate 3-phosphate with glyphosate: synthesis and ligand binding studies[J]. Biochemistry, 1996, 35(13):4199-4210.
[72] Wang JH, Lv ZJ. Design and synthesis of a novel inhibitor of 5-enolpyruvylshikimate-3-phosphate synthase[R]. Germany:Magjarevic R, 2009:100-101.
[73] Miller MJ, Cleary DG, Ream JE, et al. New EPSP synthase inhibitors:Synthesis and evaluation of an aromatic tetrahedral intermediate mimic containing a 3-malonate ether as a 3-phosphate surrogate[J]. Bioorganic & Medicinal Chemistry, 1995, 3(12):1685-1692.
[74] 劉柱. 可變鹽單胞菌中草甘膦抗性EPSP合酶新基因克隆、大腸桿菌表達及其抗性機制的研究[D]. 成都:四川大學, 2004.
[75] Ghosh S, Chisti Y, Banerjee UC. Production of shikimic acid[J]. Biotechnology Advances, 2012, 30(6):1425-1431.
[76] Nie LD, Shi XX. A novel asymmetric synthesis of oseltamivir phosphate(Tamiflu)from(-)-shikimic acid[J]. Tetrahedron:Asymmetry, 2009, 20(1):124-129.
[77] Satoh N, Akiba T, Yokoshima S, et al. A practical synthesis of(-)-oseltamivir[J]. Tetrahedron, 2009, 65(16):3239-3245.
(責任編輯 狄艷紅)
The Research Progress of EPSP Synthase
Xu Jie Jiang Shiyun Fu Fengming Geng Pengfei Huang Kai
(College of Biological and Chemical Engineering,Guangxi University of Science and Technology,Liuzhou 545006)
5-Enolpyruvylshikimate-3-phosphate synthase(EPSP synthase for short), is the sixth enzyme of shikimic acid pathway and participates in the synthesis of aromatic amino acids and some of secondary metabolites. Meanwhile, EPSP synthase are not only targets of the herbicide(glyphosate), antibiotics, anti-parasitic drugs, but also is important regulatory site of promoting the accumulation of shikimic acid in the organism. In recent years, with the rapid development of molecular biology technology and the in-depth study of EPSP synthase, EPSP synthase genes have been widely used in resistance to glyphosate genetically modified crops, medicine and health, etc. The research progress of EPSP synthase were reviewed and prospects in this paper.
Shikimate pathway EPSP synthase Shikimic acid accumulation Resistance to glyphosate
2013-10-24
廣西自然科學基金項目(2013GXNSFAA019168)
徐杰,男,碩士研究生,研究方向:生物制藥;E-mail:xj6176512@126.com
蔣世云,男,博士,教授,研究方向:生物制藥;E-mail:jiangshiyun@126.com

