風向因素對轉基因抗蟲棉花基因漂移效率的影響
朱家林,賀 娟,牛建群,張青文,劉小俠
(中國農業大學昆蟲學系, 北京 100193)
風向因素對轉基因抗蟲棉花基因漂移效率的影響
朱家林,賀 娟,牛建群,張青文,劉小俠*
(中國農業大學昆蟲學系, 北京 100193)
在轉基因作物獲準進行環境釋放并實行大面積商品化推廣的同時,基因漂移所引起的生態環境安全問題不容忽視。以含有雙價抗蟲基因(Bt/CpTI)的轉基因棉花SGK321為花粉供體材料,以常規非轉基因棉花品種石遠321、中棉35、吉扎1號為花粉受體材料,在溫室中人工創造定向風和非定向風條件,應用PCR與蛋白檢測相結合的方法,檢測外源基因發生基因漂移的效率。結果表明:隨著與轉基因棉花SGK321距離的增加,外源基因轉移至非轉基因棉花的基因漂移頻率呈現波動性變化。在定向風處理中,基因漂移頻率在距離轉基因棉花6.4m處達到峰值33.33%,在測定范圍內基因漂移最遠距離為25.6m;而在非定向風處理中,基因漂移頻率在距離轉基因棉花12.8m處達到峰值36.67%,在測定范圍內基因漂移最遠距離為36m。非定向風可顯著提高轉移至海島棉吉扎1號的基因漂移頻率。外源基因從SGK321轉移至其非轉基因親本石遠321的基因漂移頻率顯著高于轉移至陸地棉中棉35和海島棉吉扎1號的漂移頻率。為轉基因棉花的生態安全性分析提供一定的理論參考價值。
轉基因棉花;Bt基因;基因逃逸;定性PCR;生物安全性
轉基因生物技術的迅速發展和轉基因作物的不斷培育推廣,為全球特別是第三世界國家的糧食保障和經濟發展帶來了新的機遇[1]。但是,轉基因作物的環境釋放和商品化生產同時引起了全世界對其潛在的環境生物安全問題的極大關注和爭議[2- 3],如外源基因向非轉基因植物逃逸的生態風險[4- 7]、轉基因作物長期種植導致靶標生物的抗性進化[8- 11]、以及轉基因作物對非靶標生物[12- 13]、土壤生物群落[13- 15]、農業生態系統及系統外生物多樣性[16- 17]、轉基因產品食品安全性[18]的潛在影響等。
在轉基因作物商品化推廣的過程中,基因漂移是引起生態環境安全問題的最主要風險。基因漂移,又稱為基因流(gene flow),是指遺傳物質(一個或多個基因)從某一個生物群體(或居群)轉移到另一個生物群體(或居群)的過程[19- 21],是生物進化的一個重要過程和非常普遍的自然現象。在植物中,基因漂移的途徑大致分為自然雜交和基因水平轉移兩種[21- 22]。花粉介導,作為自然雜交的主要途徑,會引起不同的植物個體之間發生雜交和滲入,從而導致一系列的生態學和進化生物學反應。由于通過花粉介導的基因漂移有其規律性可循,其研究成果既具有重要的理論意義,又具有指導生產實踐的應用價值,因而是植物基因漂移研究中最受關注的焦點[23- 26]。
棉花是常異花授粉,花朵較大,易造成外媒傳粉的發生。雖然棉花花粉自身的傳播距離十分有限[27],但是如果借助昆蟲和風力的傳播就可使漂移距離明顯提高。目前關于轉基因棉花基因漂移的研究主要集中在棉花品種間的異花傳粉的雜交親和性[28- 31]、自然條件下基因漂移的可能性和空間范圍[29,32]、昆蟲傳粉對基因漂移的影響[31- 32]等方面。在我國,除新疆地區外,棉花種植區多屬于季風氣候區[33]。轉基因棉花體內的外源基因在風力條件下向周圍漂移的距離和頻率以及是否受到風向的影響引起我們的關注。明確這些問題,充分利用氣候條件,從而有效地設置隔離帶,減少甚至杜絕基因漂移的發生。目前有關這方面的報道并不多見。因此,為初步確定風向和基因漂移的關系,本研究通過設置定向風和非定向風不同處理,對溫室內轉基因棉花基因漂移效率進行分子生物學檢測,從而有效評估轉基因抗蟲棉花環境釋放的生態風險性。
1 材料與方法
1.1 供試材料
花粉供體材料為轉基因抗蟲棉花SGK321,SGK321具有人工合成的GFMCryⅠA(C)和經過修飾的CpTI的高效雙價殺蟲基因。花粉受體材料為SGK321的非轉基因親本陸地棉石遠321和陸地栽培種中棉35。鑒于海島棉與陸地棉可以雜交,故本試驗常規棉品種選用了世界栽培種海島棉吉扎1號。以上材料均由中國農業科學院棉花研究所提供。
1.2 試驗設計
試驗于2010年在北京市中國農業大學上莊試驗基地(40°08′22.92″N, 116°12′16.98″E)的溫室(60m×8m)內進行。試驗分別在兩個溫室內進行,見圖1。溫室1設置A、C處理區,溫室2設置B、C處理區,A為定向風處理區,B為非定向風處理區,C為對照處理區,均自北向南依次種植非轉基因品種中棉35、石遠321、吉扎1號。不同常規棉品種種植區域之間用透明薄膜隔開,以防止非轉基因棉花之間發生傳粉。轉基因棉花SGK321分別種植于兩個溫室的中部,種植面積為32m2。株距30cm,行距80cm。栽培方法和田間管理均按常規棉花生產方式進行。
在SGK321種植區各設置了3臺落地電風扇(FS40-8A2型,廣東美的環境電器制造公司),扇葉5片,其直徑約為50cm,高度約為2m,面向A、B處理區用于鼓風,以使轉基因棉花花粉充分擴散。A、B處理區在每個品種分布區的上方分別設置了8臺風程約為5m的電風扇(FB40- 1205型,上海華生電器有限公司),扇葉3片,其直徑約為50cm。電風扇設置于距地面垂直高度2m的欄桿處,水平傾角約為25°,每隔4.5m設置1臺,以使處理區內均勻持久受風。由于棉花僅白天開花,電風扇工作時間設置為每天6:00—21:00。A處理為定向風處理,風向垂直于棉花栽種行的方向,自西向東,保持恒定不變;B處理為非定向風處理,風向以棉花栽種行的垂直方向為中心,左右呈90°勻速變化;C處理為對照處理,僅作種植,不設人工風力條件。
風速測定使用熱球微風儀(ZRQF F303型,北京檢測儀器有限公司),在A、B、C三個處理區內,每隔4.5m為一個測量點,水平測定處理區的風速數值。為了避免誤差,于當天的上午、下午各測量兩次,記錄數值并分析匯總測定結果。

圖1 溫室各處理的種植布局Fig.1 Field design
1.3 研究方法
1.3.1 非轉基因棉花F1代種子采集處理
棉花吐絮后,對非轉基因棉花F1代種子進行采集。各處理以與轉基因棉田交界處記為0m,A、B處理在距離轉基因棉田0.8、1.6、3.2、6.4、12.8、19.2、25.6、36m處進行取樣;C處理在距離轉基因棉田0.8、1.6、3.2、6.4、12.8、19.2m處進行取樣。有研究表明轉基因棉花的異交率與所采種子在其植株上的位置基本不存在相關性[29],因此,每個取樣點分別自常規棉品種的上、中、下部各摘取棉桃,作為3次重復,分別置于低溫干燥環境下保存。待軋花脫絨后,采用土培法(蛭石∶草炭=3∶1)室內種植,每個樣點每個品種隨機選擇F1代棉苗30株,用于棉葉總DNA提取,取樣共計2520株棉苗。
1.3.2 PCR分析
取1cm2幼嫩棉花葉片,采用改良的CTAB抽提法提取樣品的總DNA[34],利用NanoDrop2000測定DNA的濃度和質量,將DNA稀釋到40ng/μL,置于-20℃冰箱中保存,待PCR時使用。
選用特異性引物對樣品內的CryⅠA(c)基因進行PCR定性檢測。上下游引物分別為5′ GAAGGATTGAGCAATCTCTAC 3′ 和5′ CAATCAGCCTAGTAAGGTCGT 3′。 PCR反應體系:2×Taq PCR Master Mix(PC0902,北京艾德萊生物科技有限公司)10μL,上下游引物(10u/mol)各0.5μL(上海生工生物技術有限公司合成),DNA模板2μL,雙蒸水7μL。
反應程序采用:預變性95℃4min,變性94℃1min、退火56℃1min、延伸72℃1.5min,30個循環,循環結束后總延伸72℃5min,4℃保存。擴增基因CryⅠA(c)的目的片段長度大約為340bp,使用2%的瓊脂糖凝膠電泳檢測[35]。出現相應條帶的樣本記為陽性結果,其余樣本記為陰性結果。
1.3.3 蛋白檢測
對PCR檢測出含有CryⅠA(c)基因的F1代樣品進行Bt蛋白表達的定性檢測[36]。取所測樣品幼嫩葉片1cm2,按1g葉片組織對10ml水的比例加入蒸餾水,充分研磨,3000r/min離心5min,取上清,插入蛋白試紙條(金標BT-Cry1Ab/Ac免疫檢測試劑盒,北京銀土地生物技術有限公司),5min后觀察結果。檢測線和質控線均出現紫紅色條帶的樣本記為陽性結果,說明Bt蛋白在F1代植株體內正常表達;檢測線未出現條帶,質控線出現紫紅色條帶的樣本記為陰性結果,說明Bt蛋白在轉入Bt基因的F1代植株體內未表達;檢測線和質控線均未出現條帶,說明紙條存在質量問題或試驗中操作不當,需重新測量。
1.4 數據處理
將PCR檢測結果和蛋白試紙檢測結果均呈陽性的樣本記為陽性結果,將PCR檢測結果呈陽性、蛋白試紙檢測結果呈陰性的樣本記為假陽性結果,將PCR檢測結果和蛋白試紙檢測結果均呈陰性的樣本均計為陰性結果。記錄陽性結果和假陽性結果的取樣位點和數目。
基因漂移頻率=(含外源Bt基因并表達的F1代個體數,即陽性結果個數/總檢測個數)×100%
假陽性頻率=(假陽性結果個數/PCR檢測陽性結果個數)×100%
所得數據采用Duncan法多重比較常規棉品種之間和與轉基因棉不同距離之間的差異水平,采用多因素方差分析方法比較風向和與轉基因棉花的距離二者對基因漂移影響的大小,采用獨立樣本t檢驗比較定向風和非定向風之間的差異水平。Plt; 0.05視為有顯著差異。
2 結果與分析
2.1 處理區風速測定結果
通過風速測定可知(圖2), 定向風區(A區)最高風速為0.78m/s,最低風速為0.39m/s;非定向風區(B區)最高風速為0.74m/s,最低風速為0.39m/s,對照區(C區)最高風速為0.04 m/s,最低風速為0.01 m/s。結果表明,在不同的測量位點,A區和B區全程風速均顯著高于C區(Plt;0.05),而二者之間無顯著性差異(Pgt;0.05),二者的區別在于前者風向恒定,后者風向不恒定,因而達到了實驗設計的風速相近而風向不同的預期效果。

圖2 A、B、C三個處理區的風速測定結果Fig.2 The measurement of wind velocities in A, B, C treatments小寫字母表示特定樣點距離A、B、C區之間風速差異的比較結果
2.2 轉基因棉花的基因漂移
2.2.1 定向風對轉基因棉花基因漂移的影響
定向風區(A區)存在陽性結果共計32個,對照區(C區)全部取樣位點基因漂移頻率皆為0,A定向風處理區發生的基因漂移頻率顯著地高于C對照處理區(Plt;0.05)。
對轉移至不同常規棉品種的基因漂移頻率進行比較分析(表1),結果表明在距離轉基因棉區1.6、6.4、19.2m處,SGK321的非轉基因親本石遠321相對于中棉35和吉扎1號存在顯著差異,轉移至前者的基因漂移頻率顯著地高于轉移至后兩個常規棉品種。轉移至亞洲棉中棉35和海島棉吉扎1號的基因漂移頻率在所有的取樣位置并未出現顯著差異。
對轉移至不同距離位點常規棉的基因漂移頻率進行比較分析(表1),結果表明在距轉基因棉區的不同距離發生的基因漂移頻率呈較大波動性變化。石遠321在距轉基因棉區1.6、6.4、19.2、25.6m處檢測到基因漂移的發生,在6.4m處達到最高值33.33%,最遠漂移距離為25.6m,漂移頻率在不同距離間呈現顯著差異(F7,16=5.481,Plt;0.05)。中棉35在距轉基因棉區6.4、19.2m處檢測到基因漂移的發生,頻率均為3.33%,最遠漂移距離為19.2m,漂移頻率在不同距離間差異不顯著(F7,16=0.857,Pgt;0.05)。吉扎1號在距轉基因棉區0.8、3.2、6.4、12.8m處檢測到基因漂移的發生,漂移頻率均為3.33%,最遠漂移距離為12.8m,漂移頻率在不同距離間差異不顯著(F7,16=0.571,Pgt;0.05)。

表1 定向風區(A區)SGK321發生的基因漂移頻率
平均值(±標準誤)為3次重復的均值,不同的小寫字母表示不同品種在相同距離的取樣位點差異顯著(Plt;0.05)
2.2.2 非定向風對轉基因棉花基因漂移的影響
非定向風區(B區)存在陽性結果共計40個,對照區(C區)全部取樣位點基因漂移頻率皆為0,B非定向風處理區發生的基因漂移頻率顯著地高于C對照處理區(Plt;0.05)。
對轉移至不同常規棉品種的基因漂移頻率進行比較分析(表2),結果表明在距離轉基因棉區3.2、12.8、36m處,SGK321的非轉基因親本石遠321相對于中棉35和吉扎1號存在顯著差異,轉移至前者的基因漂移頻率顯著地高于轉移至后兩個常規棉品種;在距離轉基因棉區0.8、6.4m處,海島棉吉扎1號相對于石遠321和中棉35存在顯著差異,轉移至前者的基因漂移頻率顯著地高于轉移至后兩個常規棉品種;在距離轉基因棉區25.6m處,轉移至石遠321和吉扎1號的基因漂移頻率差異不顯著,并在此處顯著地高于轉移至中棉35的漂移頻率。中棉35在B區并未檢測出基因漂移的樣本。
對轉移至不同距離位點常規棉的基因漂移頻率進行比較分析(表2),結果表明在距轉基因棉區的不同距離發生的基因漂移頻率呈較大波動性變化。石遠321在距轉基因棉區3.2、12.8、25.6、36m處檢測到基因漂移的發生,在12.8m處達到最高值36.67%,最遠漂移距離為36m,漂移頻率在不同距離間呈現顯著差異(F7,16=15.121,Plt;0.05)。中棉35在所有設定的取樣位置均未檢測出基因漂移的樣本。吉扎1號在距轉基因棉區0.8、6.4、12.8、25.6m處檢測到基因漂移的發生,在6.4m處達到最高值23.33%,最遠漂移距離為25.6m,漂移頻率在不同距離間呈現顯著差異(F7,16=9.143,Plt;0.05)。
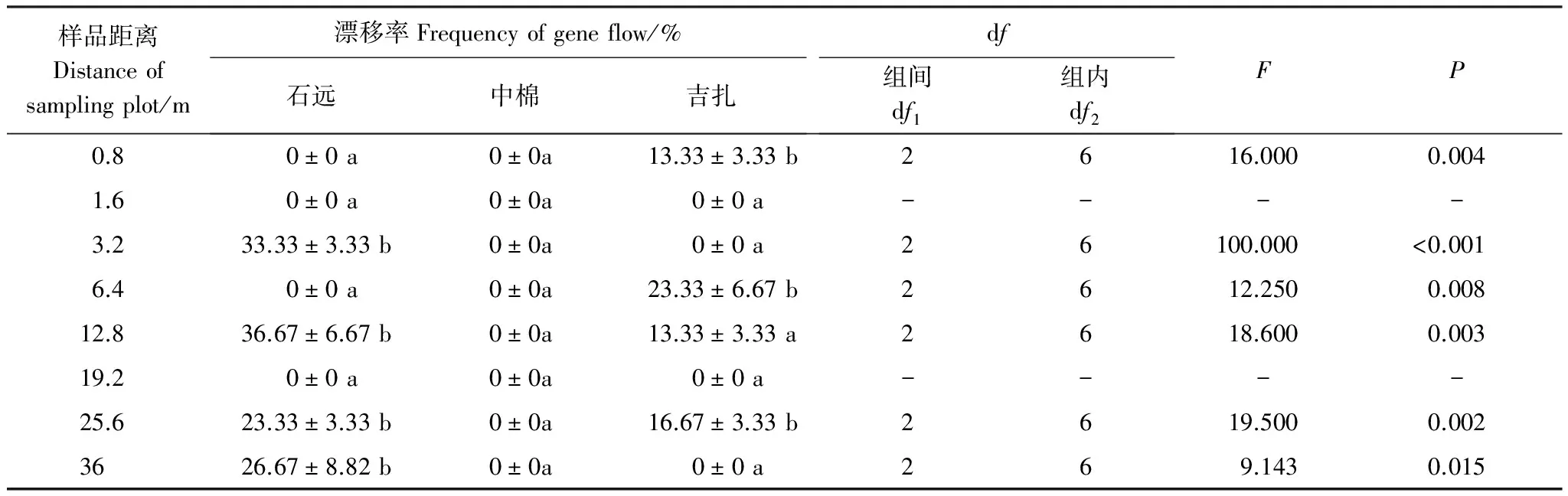
表2 非定向風區(B區)SGK321發生的基因漂移頻率
平均值(±標準誤)為3次重復的均值,不同的小寫字母表示不同品種在相同距離的取樣位點差異顯著(Plt;0.05)
2.2.3 定向風和非定向風處理間轉基因棉花基因漂移頻率的比較
對在定向風區和非定向風區所有取樣位點的基因漂移頻率進行比較分析,結果表明,對于常規棉石遠321和中棉35,定向風區和非定向風區檢測到的基因漂移頻率之間沒有顯著差異;而對于常規海島棉吉扎1號,定向風區和非定向風區檢測到的基因漂移頻率之間存在顯著差異(Plt;0.05),非定向風區的漂移頻率顯著地高于定向風區(表3)。由此推測,風向是否恒定對Bt基因漂移至海島棉吉扎1號的影響較顯著,且相對于定向風,非定向風可以顯著提高轉移至海島棉吉扎1號的基因漂移頻率。
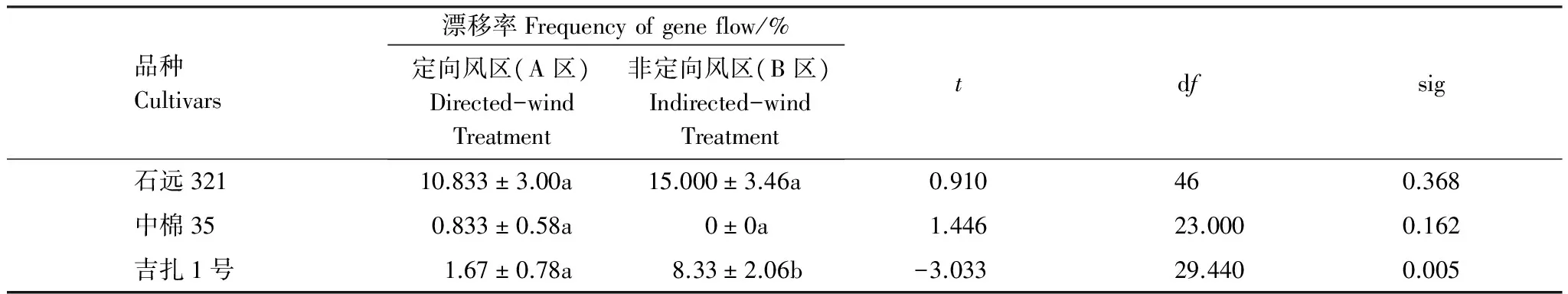
表3 不同風向因素下SGK321發生的基因漂移頻率
平均值(±標準誤)為同一常規棉品種在該處理區內所有取樣點的均值,不同的小寫字母表示同一常規棉品種在定向風區和非定向風區差異顯著(Plt;0.05)
2.2.4 風向和距離因素間的交互作用對轉基因棉花基因漂移的影響
對于常規棉品種石遠321,風向是否恒定對基因漂移頻率影響不顯著(F石遠風向=2.857,Pgt;0.05),而與轉基因棉田的距離對漂移頻率存在顯著影響(F石遠距離=3.706,Plt;0.05),風向和距離因素間的交互作用對漂移頻率影響顯著(F石遠交互=14.416,Plt;0.05)。對于常規棉品種中棉35,風向是否恒定和與轉基因棉田的距離對漂移頻率均不存在顯著影響(F中棉風向=0.857,Pgt;0.05;F中棉距離=0.857,Pgt;0.05)。對于常規棉品種吉扎1號,風向是否恒定和與轉基因棉田的距離對漂移頻率均存在顯著影響(F吉扎風向=23.273,Plt;0.05;F吉扎距離=7.065,Plt;0.05),且相對于與轉基因棉田的距離,風向是否恒定對漂移頻率的影響更大(F吉扎風向=23.273gt;F吉扎距離=7.065),二者之間的交互作用顯著影響漂移頻率(F吉扎交互=4.987,Plt;0.05)。
由此可知,對于常規陸地棉(石遠321)和海島棉(吉扎1號),與轉基因棉田的距離對轉移至二者的基因漂移頻率影響顯著;且相對于與轉基因棉田的距離,風向是否恒定對轉移至常規海島棉的基因漂移影響更大。
2.2.5 基因轉入和蛋白表達的一致性分析
試驗檢測常規棉F1代樣本共2520株,測得陽性結果72株,假陽性結果3株,陰性結果2445株,假陽性頻率為4.17%。假陽性樣本全部為石遠321的F1代,中棉35和吉扎1號未測得假陽性結果。定向風處理檢測F1代樣本共計720株,陽性結果32株,假陽性結果3株,陰性結果685株,假陽性頻率為9.38%;非定向風處理未測得假陽性結果。對定向風處理和非定向風處理的假陽性頻率使用獨立樣本t檢驗,結果表明,不同風向對假陽性頻率影響不顯著(Pgt;0.05)。
3 結論與討論
本研究發現,轉基因抗蟲棉花基因漂移的頻率最高為36.67%(非定向風區、石遠321、距轉基因棉花12.8m處),其他研究者得到的最高漂移頻率均低于本研究結果[29,35,37- 39]。在本試驗測定范圍內,轉基因抗蟲棉花基因漂移最遠檢測距離為36m(非定向風區、石遠321、此處漂移頻率為26.67%)。不同研究者所得到的轉基因棉花基因漂移最遠距離存在明顯差異,大都介于20m至100m之間[29,35,37- 39],但也有研究發現最遠可達1625m[32]。本試驗采用相對封閉的溫室環境,設置充足集中的風力條件,可能導致溫室內空氣中花粉密度的相對提高。同時相對于大田環境,溫室內部的溫度略高,加劇了花粉微粒的布朗運動,增加了花粉的擴散速率,導致基因漂移頻率在一定程度上增加。另外,不同轉基因花粉供體所攜帶不同目的基因的第二效應(如在自然狀態下的發芽率和生存能力)所選用的不同轉基因花粉受體的品種特性(如自身花粉量、自交進程速度及接受外源花粉能力)均可以在一定程度上影響基因漂移結果[40]。其他因素,諸如實驗進行的自然條件(風力、降雨、溫濕度)、蟲媒的種類和數量、轉基因作物釋放的面積[41]等都會影響基因漂移的最遠距離。
轉基因棉花基因漂移頻率變化的趨勢是備受關注的焦點。相關研究證實,隨著轉基因作物種植區與非轉基因作物之間距離的增加,基因漂移頻率不斷降低[42- 43]。這是由于隨著與轉基因花粉源距離的增加,空氣中的花粉密度逐漸降低[44]。同時,在非轉基因花粉受體周圍存在其他非轉基因花粉時,不同來源的花粉相互競爭導致了基因漂移頻率的迅速下降[45]。而本研究發現基因漂移頻率并未出現隨著與轉基因棉花距離的增加而下降的趨勢,而是呈現較大的波動性。不同取樣位點微環境風速的差異可能影響基因漂移頻率。為使轉基因棉區花粉充分擴散,本試驗在轉基因棉區設置了3臺電風扇,這造成了A、B處理區初始取樣位點附近風速的相對提高。因此,3個常規棉品種初始取樣位點(0.8、1.6m)檢測到的漂移頻率較小,可能與該處風速較大有關。同時,本試驗在定向風區和非定向風區每間隔4.5m設置一臺電風扇以造成持續的風力條件,因此每隔4.5m都存在風速不恒定的梯度風場,造成基因漂移頻率的波動性較大。這種波動性還可能是由于每個樣點采集的種子范圍和檢測數量有限造成的。另外,有研究表明基因漂移PCR檢測和蛋白檢測結果不能完全吻合,存在基因轉入但未表達的假陽性檢測結果[46]。本研究發現假陽性樣本3株,假陽性頻率為4.17%,基因轉入和蛋白表達檢測結果呈現較高的一致性。因此,檢測方法的靈敏度也影響著基因漂移的結果。Rieger[47]等研究結果表明,在授粉媒介和花粉源的雙重效應下,隨著與花粉源距離的增加,花粉活力下降的趨勢較不明顯,基因漂移發生的分布性較為隨機。
本試驗設置了兩種理想狀況,即風向恒定不變和勻速變化,發現非定向風可以顯著提高轉移至海島棉吉扎1號的基因漂移頻率,且相對于與轉基因棉田的距離,風向是否恒定對其基因漂移的影響更大。原因可能在于海島棉植株茂密,株型大,對攜帶有外源基因的花粉和人工設置的風力在空間的傳播形成了較大的阻力;而植株內部風速小,增加了授粉的可能性。另外,由于風向并不恒定,水平方向上缺少恒定氣流,因而延長了花粉定向水平傳送的時間,影響花粉顆粒的沉降速度和垂直擴散速度等一系列復雜的因子,取樣位點外源花粉在空中滯留或下降,不易帶出,致使花粉易雜交。
轉基因作物與其親緣種之間的雜交是基因漂移的主要形式,也是二者之間基因流動的證據。Dale認為只有親緣關系較近,即僅包含在同一個屬的種之間才可能是親和的[48]。本試驗研究結果表明,外源基因轉移至SGK321非轉基因親本石遠321的基因漂移頻率顯著地高于轉移至亞洲棉中棉35和海島棉吉扎1號。沈法富等研究表明,在6m內陸地棉品種之間發生的基因流高于陸地棉和海島棉品種間的基因流[39],與本試驗結果基本一致。這表明基因漂移的發生,不僅需要轉基因棉花的花粉漂流,而且存在著不同花粉的競爭和授粉選擇。原因可能與花粉活性及其識別、接受、雜交過程有關。與中棉35相比, 石遠321雖同為陸地棉, 但其為轉基因棉花SGK321的非轉轉基因親本,即SGK321是由石遠321轉入雙價抗蟲基因所得,二者親緣關系最近,花粉親和性和接受程度最高,因而轉移至石遠321的基因漂移頻率顯著高于其他品種。SGK321屬于栽培陸地棉,是具有A、D兩染色體組的異源四倍體, 而海島棉吉扎1號亦是分為A、D兩組的異源四倍體,與SGK321的F1代形成配子時減數分裂正常,因此是可育的,但其后代會出現一些不孕植株,其原因是配對的染色體之間存在結構上的細微差異,或由于不同種間基因系統的不協調,即基因不育。由此推測,轉基因棉花的外源基因漂移至親緣關系較近的非轉基因親本的可能性較大。
通過花粉傳播產生的基因漂移是自然界客觀存在的事實。影響轉基因棉花基因漂移的因素非常復雜,風向只是其中的一個重要因素。為了進一步明確風媒因素對基因漂移的影響,風速是不可忽視的因子。在未來的研究中,可利用相對封閉的條件設置不同的風速探索其對基因漂移的影響。另外,本試驗選擇Bt基因作為標記基因,CpTI基因的基因漂移效率及其與Bt基因在漂移時的互作效應需要深入研究。
[1] Huang J K, Rozelle S, Pray C, Wang Q F. Plant biotechnology in China. Science, 2002, 295(5555): 674- 677.
[2] Ellstrand N C. When transgenes wander, should we worry?. Plant Physiology, 2001, 125(4):1543- 1545.
[3] Zhang Y J, Wu K M, Peng Y F, Guo Y Y. The ecological risks of genetically engineered plants. Acta Ecologica Sinica, 2002, 22(11): 1951- 1959.
[4] Ellstrand N C. Current knowledge of gene flow in plants: implications for transgene flow. Philosophical Transactions of the Royal Society B: Biological Science, 2003, 358(1434): 1163- 1170.
[5] Messeguer J. Gene flow assessment in transgenic plants. Plant Cell Tissue and Organ Culture, 2003, 73(3): 201- 212.
[6] Lu B R, Snow A A. Gene flow from genetically modified rice and its environmental consequences. BioScience, 2005, 55(8): 669- 678.
[7] Mercer K L, Andow D A, Wyse D L, Shaw R G. Stress and domestication traits increase the relative fitness of crop-wild hybrids in sunflower. Ecology Letters, 2007, 10(5): 383- 393.
[8] Bates S L, Zhao J Z, Roush R T, Shelton A M. Insect resistance management in GM crops: past, present and future. Nature Biotechnology, 2005, 23(1): 57- 62.
[9] Dalecky A, Bourguet D, Ponsard S. Does the European corn borer disperse enough for a sustainable control of resistance toBtmaize via the high dose/refuge strategy?. Cahiers Agricultures, 2007, 16(3): 171- 176.
[10] Li G P, Wu K M, Gould F, Wang J K,Miao J, Gao X W, Guo Y Y. Increasing tolerance toCry1Accotton from cotton bollworm,Helicoverpaarmigera, was confirmed inBtcotton farming area of China. Ecological Entomology, 2007, 32(4): 366- 375.
[11] Wu K M. Monitoring and management strategy forHelicoverpaarmigeraresistance toBtcotton in China. Journal of Invertebrate Pathology, 2007, 95(3): 220- 223.
[12] O′Callaghan M, Glare T R, Burgess E P J, Malone L A. Effects of plants genetically modified for insect resistance on non-target organisms. Annual Review of Entomology, 2005, 50: 271- 292.
[13] Oliverira A R, Castro T R, Capalbo D M F, Delalibera I. Toxicological evaluation of genetically modified cotton (Bollgard?) and Dipel? WP on the non-target soil miteSchieloribatespraeincisus(Acari: Oribatida). Experimental and Applied Acarology, 2007, 41(3): 191- 201.
[14] Wu W X, Ye Q F, Min H, Duan X J, Jin W M.Bt-transgenic rice straw affects the culturable microbiota and dehydrogenase and phosphatase activities in a flooded paddy soil. Soil Biology and Biochemistry, 2003, 36(2): 289- 295.
[15] Giovannetti M, Sbrana C, Turrini A. The impact of genetically modified crops on soil microbial communities. Rivista di Biologia, 2005, 98(3): 393- 417.
[16] Conner A J, Glare T R, Nap J P. The release of genetically modified crops into the environment. PartⅡ. Overview of ecological risk assessment. The Plant Journal, 2003, 33(1): 19- 46.
[17] Ammann K. Effects of biotechnology on biodiversity: herbicide-tolerant and insect-resistant GM crops. Trends in Biotechnology, 2005, 23(8): 388- 394.
[18] Deng X, Zhao Y C, Gao B D, Zhang Y J, Sun F Z. Advance on the biosafety assessment of insect-resistant transgenic cotton. Acta Ecological Sinica, 2006, 26(12): 4244- 4249.
[19] Slatkin M. Gene flow in natural population. Anunual Review of Ecology amp; Systematics, 1985, 16: 393- 430.
[20] Futuyma D. Evolutionary Biology, 3rd edn. Sunderland, MA: Sinauer, 1998.
[21] Andow D A, Zwahlen C. Assessing environmental risks of transgenic plants. Ecology Letter, 2006, 9(2):196- 214.
[22] Lu B R, Xia H, Yang X, Jin X, Liu P, Wang W. Evolutionary theory of hybridization-introgression: its implication in environmental risk assessment and research of transgene escape. Biodiversity Science, 2009, 17(4): 362- 377.
[23] Messeguer J, Fogher C, Guiderdoni E. Field assessment of gene flow from transgenic to cultivated rice (OryzasativaL.) using a herbicide resistance gene as tracer marker. Theoretical and applied genetics, 2001, 103(8): 1151- 1159.
[24] Zhang K, Li Y, Lian L. Pollen-mediated transgene flow in maize grown in the Huang-huai-hai region in China. Journal of Agricultural Science, 2011, 149(2): 205- 216.
[25] Pasquet R S, Peltier A, Hufford M B, Oudin E, Saulnier J, Paul L, Knudsen J T, Herren H R, Gepts P. Long-distance pollen flow assessment through evaluation of pollinator foraging range suggests transgene escape distances. Proceedings of the National Academy of Sciences of the United States of America, 2008, 105(36): 13456- 13461.
[26] Ahmed S, Compton S G, Butlin R K. Wind-borne insects mediated directional pollen transfer between desert fig trees 160 kilometers apart. Proceedings of the National Academy of Sciences of the United States of America, 2009, 106(48): 20342- 20347.
[27] Ma J, Gao B D, Wan F H, Guo J Y. Ecological risk ofBttransgenic cotton and its management strategy. Chinese Journal of Applied Ecology, 2003, 3(14):443- 446.
[28] Llewellyn D, Fitt G. Pollen dispersal from two field trials of transgenic cotton in the Namoi Valley, Australia. Molecular Breeding, 1996, 2(2): 157- 166.
[29] Umbeck P F, Barton K A, Nordheim E V, Mccarty J C, Parrott W L, Jenkins J N. Degree of pollen dispersal by insects from a field-test of genetically engineered cotton. Journal of Economic Entomology, 1991, 84(6):1943- 1950.
[30] Zhang B H, Pan X P, Guo T L, Wang Q L, Anderson T A. Measuring gene flow in the cultivation of transgenic cotton (GossypiumhirsutumL.). Molecular Biotechnology, 2005, 31(1): 11- 20.
[31] Llewellyn D, Tyson C, Constable G. Containment of regulated genetically modified cotton in the field. Agriculture Ecosystems amp; Environment, 2007, 121(4): 419- 429.
[32] Van Deynze A E, Sundstorm F J, Bradford K J. Pollen-mediated gene flow in California cotton depends on pollinator activity. Crop Science, 2005, 45(4): 1565- 1570.
[33] Zhang H Q. A study on comparative advantage in cotton-growing regions of China[D]. Beijing: Chinese Academy of Agricultural Science, 2004.
[34] Ma X, Du X M. Preliminary study on the methods of extracting cotton genomic DNA. Cotton Science, 2004, 16(1):40- 43.
[35] Wang C Y, Liu Y, Zhou J, Chen J Q, Qin P. Monitoring of pollen-mediated gene flow from transgenicBtcotton. Chinese Journal of Applied Ecology, 2007, 18(4): 801- 806.
[36] He S P, Zhao J, Yi G X, Liu X, Deng A X, Nan T G, Li Z H, He Z P, Wang B M. Development of a dipstick assay for detection of transgenicBt-cotton. 2006 Annal Conference and the 7th congress of the China Cotton Association, China, 2006: 189- 220.
[37] Zhang B H, Guo T L. Frequency and distance of pollen dispersal from transgenic cotton. Chinese Journal Applied Environmental Biology, 2000, 6(1):39- 42.
[38] Zhang C Q, Lv Q Y, Wang Z X, Jia S R. Frequency of 2, 4-D resistant gene flow of transgenic cotton. Scientia Agricultura Sinica, 1997, 30(1): 92- 93.
[39] Shen F F, Yu Y J, Zhang X K, Bi J J, Yin C Y.Btgene flow of transgenic cotton. Acta Genetica Sinica, 2001, 28(6): 562- 567.
[40] Wang T Y, Zhao Z H, Yan H B, Li Y, Zhu X H, Shi Y S, Song C Y, Ma Z Y, Darmency H. Gene flow from cultivated herbicide-resistant foxtail millet to its wild relatives: a basis for risk assessment of the release of transgenic millet. Acta Agronomica Sinica, 2001, 27(6): 681- 687.
[41] Scheffer J A, Parkinson R, Dale P J. Evaluating the effectiveness of isolation distances for field plots of oilseed rape (Brassicanapus) using a herbicide-resistance transgene as a selectable marker. Plant Breeding, 1995, 114(4): 317- 321.
[42] Goggi A S, Caragea P, Lopez-Sanchez H. Statistical analysis of outcrossing between adjacement maize grain production fields. Field Crops Research, 2006, 99(2/3): 147- 157.
[43] Ma B L, Subedi K D, Reid L M. Extent of cross-fertilization in maize by pollen from neighbouring transgenic hybrids. Crop Science, 2004, 44(4): 1273- 1282.
[44] Song Z P, Li B, Chen J K. Genetic diversity and conservation of common wild rice (Oryzarufipogon) in China. Plant Species Biology, 2005, 20(2): 83- 92.
[45] Rong J. Pollen-mediated gene flow and its modeling in rice (OryzasativaL.)[D]. Shanghai: Fudan University, 2006.
[46] He J, Zhu J L, Liu X X, Zhang Q W. Comparison between two methods for detecting gene flow and analysis on the influencing factors. Agricultural Science amp; Technology, 2011, 12(6): 840- 841, 861.
[47] Rieger M A, Micheal L, Christopher P. Pollen-mediated movement of herbicide resistance between commercial fields. Science, 2002, 296(5577): 2386- 2388.
[48] Dale P J. Spread of engineered genes to wild relatives. Plant Physiology, 1992, 100(1): 13- 15.
參考文獻:
[3] 張永軍, 吳孔明, 彭于發, 郭予元. 轉基因植物的生態風險. 生態學報, 2002, 22(11): 1951- 1959.
[18] 鄧欣, 趙延昌, 高必達, 張永軍, 孫福在. 轉基因抗蟲棉生物安全評價研究進展. 生態學報, 2006, 26(12): 4244- 4249.
[22] 盧寶榮, 夏輝, 楊簫, 金鑫, 劉蘋, 汪魏. 雜交- 漸滲進化理論在轉基因逃逸及其環境風險評價和研究中的意義. 生物多樣性, 2009, 17(4):362- 377.
[27] 馬駿, 高必達, 萬方浩, 郭建英. 轉Bt基因抗蟲棉的生態風險及治理對策. 應用生態學報, 2003, 3(14): 443- 446.
[28] 劉謙, 朱鑫泉. 生物安全. 北京: 科學出版社, 2001.
[33] 張海清. 中國棉花產區比較優勢研究[D]. 北京:中國農業科學院, 2004.
[34] 馬軒, 杜雄明. 提取棉花基因組DNA的一點探討. 棉花學報, 2004, 16(1):40- 43.
[35] 王長永, 劉燕, 周駿, 陳建群, 欽佩. 花粉介導的轉Bt基因棉花田間基因流監測. 應用生態學報, 2007, 18(4): 801- 806.
[36] 何素平, 趙靜, 羿國香, 劉威, 鄧艾興, 南鐵貴, 李召虎, 何鐘佩, 王保民. Bt毒蛋白免疫金標試紙條的制備與應用. 中國棉花學會2006年年會暨第七次代表大會, 中國, 2006: 189- 220.
[37] 張寶紅, 郭騰龍. 轉基因棉花基因花粉散布頻率及距離的研究. 應用與環境生物學報, 2000, 6(1):39- 42.
[38] 張長青, 呂群燕, 王志興, 賈士榮. 抗2, 4- D轉基因棉花基因漂流頻率的研究. 中國農業科學, 1997, 30(1): 92- 93.
[39] 沈法富, 于元杰, 張學坤, 畢建杰, 尹承脩. 轉基因棉花的Bt基因流. 遺傳學報, 2001, 28(6): 562- 567.
[40] 王天宇, 趙治海, 閆洪波, 黎裕, 朱學海, 石云素, 宋燕春, 馬峙英, H.Darmency. 谷子抗除草劑基因從栽培種向其近緣野生種漂移的研究. 作物學報, 2001, 27(6): 681- 687.
[45] 戎俊. 花粉介導的水稻(OryzasativaL.)基因漂移及其模型的研究[D]. 上海: 復旦大學, 2006.
Theinfluenceofwinddirectiononpollen-mediatedgeneflowintransgenicinsect-resistantcotton
ZHU Jialin, HE Juan, NIU Jianqun, ZHANG Qingwen, LIU Xiaoxia*
DepartmentofEntomology,ChinaAgriculturalUniversity,Beijing100193,China
With the extensive environment release and large scale cultivation of genetically modified (GM) crops, the ecological environment security problems, caused by transgene escape from GM crop to its non-GM counterpart and wild relatives, have appeared gradually. During the early years of breeding and seed increase, there are some uncertainty among regulators about containment measures needed to prevent the movement of regulated GM traits into adjacent fields and possibly into the human or animal food chain. Cotton, one of the wind-pollinated and highly outcrossing cash crops, is planted on millions of hectares annually and is the third most abundant GM crop.
This study investigated the occurrence of gene flow from transgenic cotton to non-transgenic cottons under closed conditions using an insect-resistant gene as a tracer marker. The selectable marker used in this study was the syntheticGFMCryⅠA(C)gene which encoded Bt insecticidal protein with resistance to cotton bollworm. In order to determine the impacts of wind direction on pollen-mediated gene flow (PGF), two greenhouse experiments under different wind conditions (directed and indirected) were establishied. The growth area for each treatment was 480m2, and the row and plant spacing was 80cm×30cm. To observe the frequency and distance of gene flow, the transgenic cotton, SGK321 (GossypiumhirsutumL.), was used as a pollen donor, and conventional varieties Shiyuan321 (GossypiumhirsutumL.), Zhongmian35 (Gossypiumhirsutum( L.) and Jizha1 (GossypiumbarbadenseL.) were applied as pollen recipients, separately. Following natural pollination under the wind conditions, the seeds were collected from each conventional varieties at varying distances and sown in pot culture. DNAs were extracted by the CTAB procedure during cotyledon period, then screened forCryⅠA(C) gene by PCR assays. The positive samples in the PCR assays were identified for Bt insecticidal protein by dipstick assay. The results showed that there was a variation of PGF with increasing distances from the donor plots. The highest frequencies of gene flow were (33.33±12.02)% at 6.4m in thg directed-wind treatment and (36.67±6.67)% at 12.8 m in the indirected-wind treatment. The maximal gene flow distance observed were 25.6m in the directed-wind treatment and 36m in the indirected-wind treatment. PGF to the island cotton (Jizha1) was higher under the indirected-wind treatment than that under the directed-wind treatment, associated with the possibility that the gargantuan island cotton could hinder the wind, leading to cross pollination. PGF to non-transgenic counterpart (Shiyuan321) was obviously higher than those to the island cotton (Jizha1) and the upland cotton (Zhongmian35), indicating that genetic relationship played an important role in cross-pollination with non-GM crops.
Such control by individual factor (wind direction) under closed conditions in affecting the occurrence of gene flow rather than open-field trial designs could also be proved very useful in studying the influence of a certain factor on the gene flow. Our research may provide some reference value for the ecological safety assessment of transgenic cotton with the aim of establishing strategies to prevent pollen dispersal.
transgenic cotton;Btgene; transgene escape; qualitative PCR; biosafety
轉基因生物新品種培育科技重大專項(2011ZX0811-002)
2012- 07- 04;
2012- 10- 26
*通訊作者Corresponding author.E-mail: liuxiaoxia611@cau.edu.cn
10.5846/stxb201207040932
朱家林,賀娟,牛建群,張青文,劉小俠.風向因素對轉基因抗蟲棉花基因漂移效率的影響.生態學報,2013,33(21):6803- 6812.
Zhu J L, He J, Niu J Q, Zhang Q W, Liu X X.The influence of wind direction on pollen-mediated gene flow in transgenic insect-resistant cotton.Acta Ecologica Sinica,2013,33(21):6803- 6812.

