4個縊蟶群體遺傳多樣性和系統發生關系的微衛星分析
劉 博, 邵艷卿, 王 侃, 滕爽爽, 柴雪良, 方 軍, 張炯明, 肖國強
(浙江省海洋水產養殖研究所 浙江省近岸水域生物資源開發與保護重點實驗室, 浙江 溫州325001)
縊蟶(Sinonovacula ConstrictaLamarck)俗稱蟶子, 屬軟體動物門(Mollusca), 瓣鰓綱(Lamellibranchia)竹蟶科(solenidae)縊蟶屬(sinonovacula)貝類。縊蟶為廣溫廣鹽性海產雙殼類, 具有重要的經濟價值, 是我國四大養殖貝類之一。縊蟶養殖歷史悠久,過去主要集中在福建和浙江一帶, 隨著縊蟶養殖業的不斷擴展, 山東、天津沿海地區開始從福建、浙江等地區引進苗種。浙江沿海為了能提早出苗, 每年9月份也大量從廣東湛江和福建云霄等地購買親貝催產育苗。無序的養殖、盲目引種、移養等使溢蟶種質資源混淆不清。因此, 分析不同地理群體遺傳結構及種群間關系, 對縊蟶種質資源合理保護、開發和利用具有重要指導意義。
微衛星標記(SSR)與其他分子標記相比, 具有多態性高、多等位性、共顯性、可重復性高、數量豐富和對基因組有很好的覆蓋性等特點[1], 被廣泛的用于種質資源及品種(系)鑒定、遺傳結構分析、分子標記輔助選擇等多方面的研究[2]。微衛星標記技術在太平洋牡蠣(Crassosttea Gigas)[3]、蝦夷扇貝[4-5]、文蛤[6-8]、皺紋盤鮑(Haliotis discushanna)[9]等已有報道, 關于縊蟶在遺傳多樣性和遺傳分化的研究已有線粒體COⅠ標記法, RAPD和同工酶標記等[10-12]; 目前, 縊蟶微衛星方面的研究僅局限在標記開發[13,14]和單一養殖群體的遺傳多樣性分析[15], 而關于用微衛星標記分析不同地理居群縊蟶遺傳多樣的報道至今還沒有,本文首次利用SSR標記分析了4個縊蟶居群的遺傳結構, 在分子水平上探討了處于不同地理區域的縊蟶群體遺傳多樣性差異和遺傳分化水平, 為其種質資源管理和遺傳育種工作提供科學的理論依據。
1 材料與方法
1.1 實驗材料
實驗用4個縊蟶養殖群體, 于2011年9月分別采自福建云霄、廣東湛江、天津塘沽及浙江樂清灣, 各群體隨機取樣30顆, 活體解剖取其水管、斧足放入無水乙醇固定保存, 帶回實驗室備用。
1.2 DNA提取
基因組 DNA的提取采用常規的蛋白酶K消化,苯酚、氯仿/異戊醇抽提, 無水乙醇沉淀的方法, 提取縊蟶肌肉組織的基因組 DNA, 用 TE緩沖液稀釋至終濃度100 ng/μL用于實驗分析。
1.3 PCR擴增及產物檢測
微衛星位點的引物來自己發表的12對縊蟶引物序列(見表 1)[15]。PCR 反應體系為 15 μL, 包括 10×PCR buffer 1.5 μL, 25 mmol/L Mg2+1.5 μL, 2.5 mmol/L dNTP 1.2 μL, 10 μmol/L 上下游引物各 1.5 μL, Taq 酶(5U)0.075 μL, DNA 模板1.5 μL, 加滅菌雙蒸水至15 μL。
PCR反應程序為: 94℃條件下變性5 min后, 進入30個PCR循環: 在94℃下變性30 s, 復性(溫度見表1)30 s, 72 ℃下延伸45 s, 再最后72 ℃下延伸7 min,并將產物于4℃下保存。擴增產物用1.5%瓊脂糖凝膠檢測, 鑒定有擴增產物后再用8%的變性聚丙烯酰胺凝膠電泳檢測, 銀染。

表1 12個多態微衛星引物序列Tab. 1 Primers sequences for the 12 polymorphic loci
1.4 數據分析
根據電泳圖譜, 并參照引物設計時預期片段的大小統計條帶, 應用 POPGENE32軟件計算微衛星引物的期望雜合度(Expected heterozygosity,He)、觀測雜合度(Ho)、哈代-溫伯格平衡值(Hardy-Weinberg equilibrium,HWE)、FST、FIS及FIT等參數。根據遺傳距離利用 MEGA4.0軟件構建 UPGMA系統進化樹。多態信息含量(Polymorphic information content,PIC)的計算公式如下:
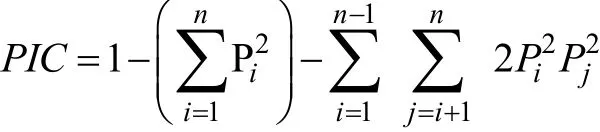
其中,Pi,Pj是某個位點第i、j個等位基因的基因頻率,n為某個位點上的等位基因數。
2 結果
2.1 四個縊蟶群體的微衛星等位基因及多樣性分析
12對引物共檢測出 46個等位基因, 其中YC30位點等位基因數目最多為12個, 表現為高度多態; 每個位點等位基因數從2~12個不等, 平均每個位點觀測等位基因數目為 3.83; 有效等位基因數從位點 YC8的 1.65到位點 YC30的 9.75, 平均有效等位基因數為3.03; 擴增片段長度在130~180 bp之間。
2.2 四個縊蟶群體內遺傳變異
4個縊蟶群體的等位基因數(Ao)、有效等位基因數(Ae)、觀測雜合度(Ho)、期望雜合度(He)、多態信息含量(PIC)及 Hardy-Weinberg檢驗結果見表 2。4個縊蟶群體的遺傳差異較小, 平均等位基因數Ao在3.25~3.50, 有效等位基因數Ae在 2.57~2.69, 等位基因數最低的是天津縊蟶群體, 最高的是福建縊蟶群體, 平均值為 2.69。平均多態信息含量(PIC)在0.46~0.50之間, 以福建群體最高為0.50。群體觀測雜合度Ho在 0.59~0.74之間, 最高的為天津群體,最低的為福建群體; 而4個群體的期望雜合度(He)差異不大, 在 0.55~0.58之間, 最高的是福建群體為0.58。Hardy- Weinberg平衡檢驗表明, 除福建云霄群體有4個位點偏離平衡外, 廣東湛江群體、天津塘沽群體和浙江樂清灣群體中的大部分微衛星位點都偏離了Hardy- Weinberg平衡(表2)。
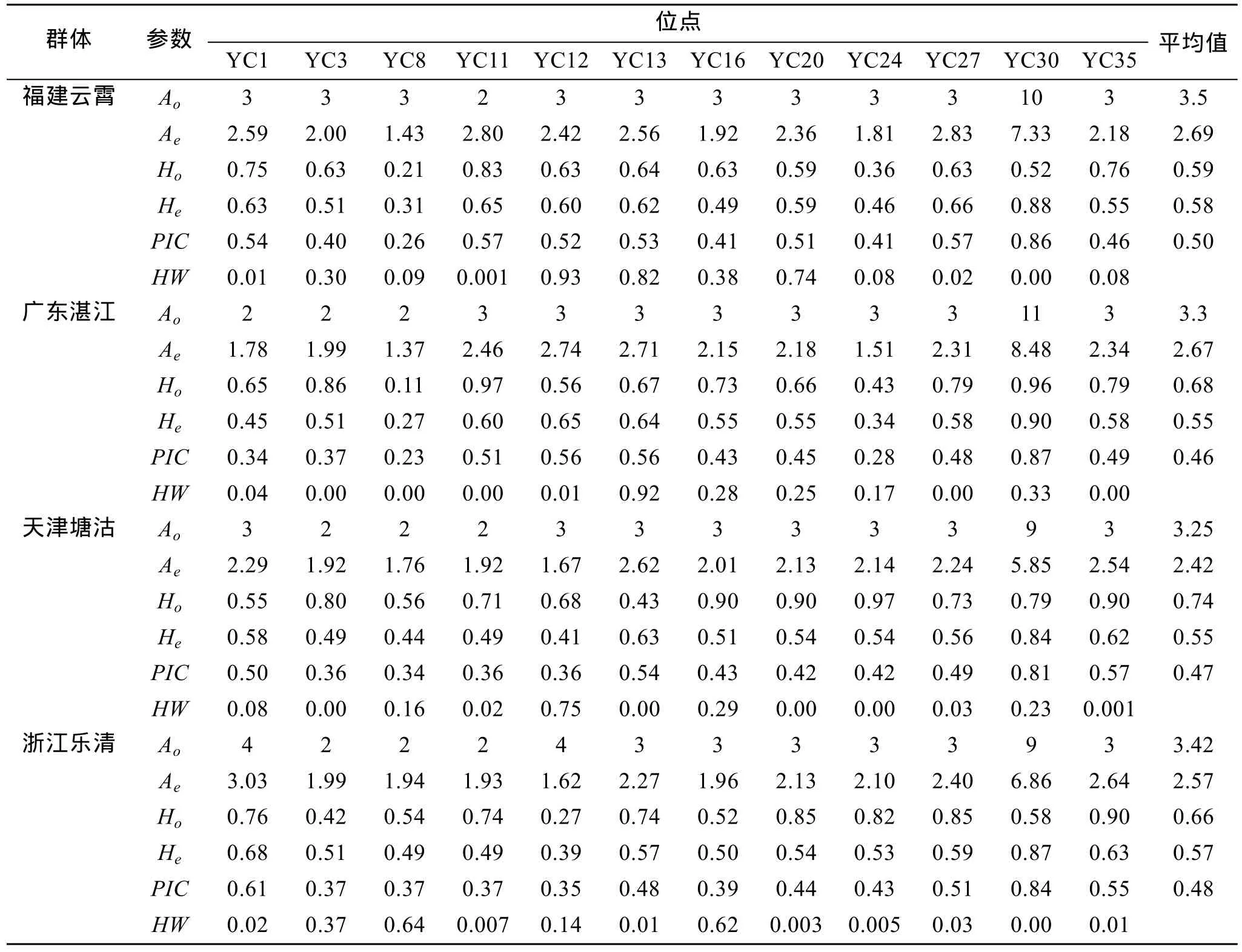
表2 12個微衛星位點在4個縊蟶群體中的遺傳多樣性及Hardy-Weinberg平衡檢驗Tab. 2 Genetic diversity and Hardy-Weinberg in four strains of S. constricta for the 12 polymorphic loci
2.3 不同縊蟶群體間遺傳分化
應用Popgene32軟件對4個縊蟶群體進行了F-統計量及基因流分析, 結果見表3。由表3可知, 4個縊蟶群體僅有 3個位點 YC8、YC12和 YC30的F-統計量為正值, 其余9個位點均為負值,Fis平均值為-0.21,Fit平均值為-0.31; 度量群體間遺傳差異程度的Fst平均值為0.07。遺傳分化系數(Fst)和基因流(Nm)分析不同群體的遺傳分化情況, 發現縊蟶群體除了YC11、YC20、YC27與YC30等4個基因座固定指數小于0.05, 其余均大于種間無遺傳分化的標準(Fst=0~0.05)[16], 基因流Nm平均值為3.33。
利用 MEGA4分析軟件, 采用非加權的組平均法(UPGMA)對 4個群體進行聚類分析, 獲得聚類樹系圖(圖1), 4個縊蟶群體之間的Nei’s遺傳距離和遺傳相似度見表 4。4個縊蟶群體共分為 2大支系:浙江樂清灣群體和天津塘沽群體之間的遺傳距離最小為 0.0344, 聚為第一支; 福建云霄群體和廣東湛江群體之間的遺傳距離為0.103, 聚為第二支。
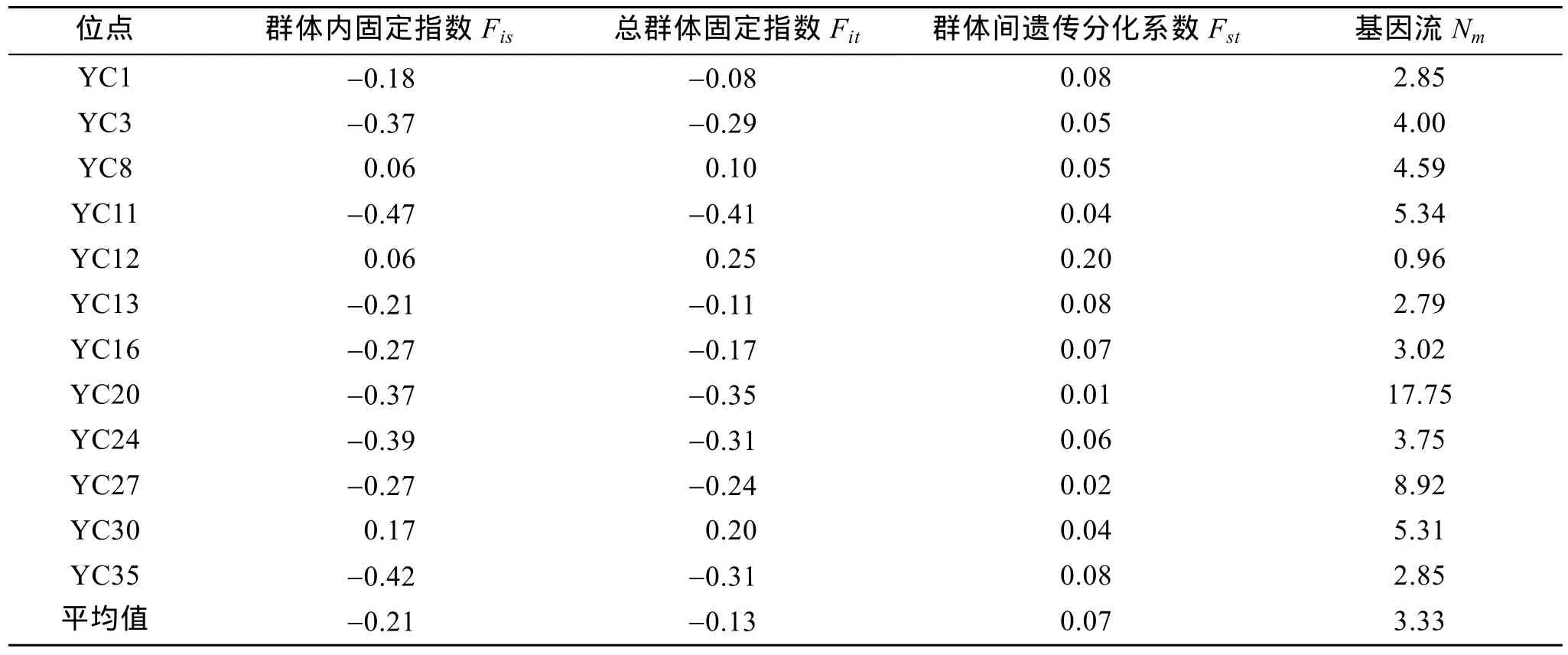
表3 12個微衛星位點的固定指數及基因流Tab. 3 Fixation index and gene flow of 12 polymorphic loci
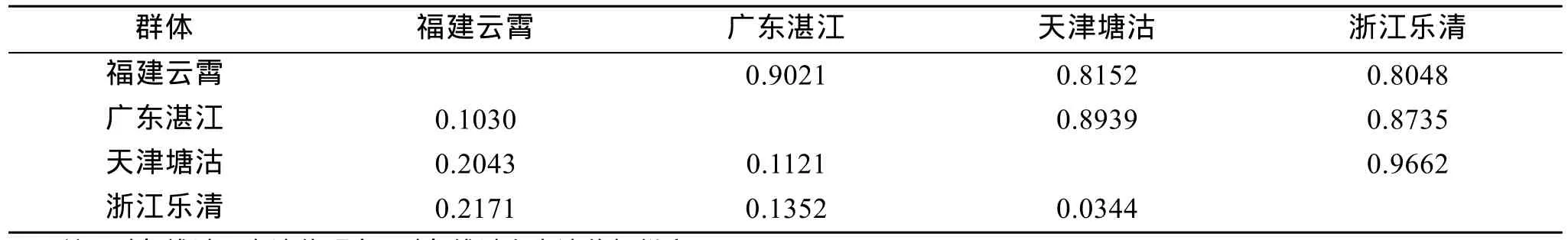
表4 Nei遺傳相似度和遺傳距離Tab. 4 Nei genetic similarity and genetic distance

圖1 根據群體間遺傳距離用UPGMA法構建了4個泥蚶群體的系統進化樹Fig. 1 Phylogenetic tree based on genetic distance of 4 four strains of S. constricta by UPGMA assay
3 討論
3.1 縊蟶群體內遺傳多樣性
依據PIC判斷標準:PIC<0.25為低度多態, 0.25≤PIC<0.5為中度多態,PIC≥0.5為高度多態[17], 可以看出本研究的 4個縊蟶群體的平均多態信息含量(PIC)基本一致在 0.46~0.50之間, 相差無幾且接近于0.5, 表明4個縊蟶群體的遺傳多樣性處于中等偏上水平, 4個群體未出現明顯的種質退化。
李成華等[18]和王冬群等[12]分別利用RAPD技術和同工酶技術分析了不同地理群體的野生縊蟶群體和養殖群體的遺傳多樣性, 野生群體的遺傳多樣性略高于養殖群體, 但與本實驗相比, 以上各群體的遺傳多樣性明顯低于本研究中 4個群體的, 分析原因主要與各實驗采用的標記類型不同有關。顯性標記RAPD與共顯性標記SSR相比, 其無法區分顯性純合子和雜合子, 其多態信息含量僅是 SSR的 1/2;而 England等[19]通過對同工酶和微衛星標記多態性的比較發現, 同一群體的微衛星位點等位基因數為5.2個, 同工酶為 3.0個, 因此不同類型標記影響了對群體遺傳多樣性的分析。本研究中以天津群體的觀測雜合度最高, 但從 4個縊蟶群體的平均等觀測位基因數Ao來看, 天津群體的平均值最低為 3.25,等位基因數目最少, 基因型單一, 原因可能是這些基因型對環境的適應度較高, 經歷了長時間的遺傳漂變和適應性選擇逐漸呈現出了高的基因頻率, 進而產生了與其他地區群體不同的遺傳結構[20]。
3.2 縊蟶群體間的遺傳分化
群體Fst值在 0~0.05之間, 則表明其各亞群間不存在分化; 若Fst值在 0.05~0.15之間, 為中度分化; 若Fst值在 0.15~0.25之間, 則為高度分化。本研究結果群體間遺傳差異程度的Fst=0.07, 表明4個縊蟶群體間的遺傳變異為 7.0%, 而 93.0%為群體內的遺傳變異, 兩者比例懸殊, 說明所選4個縊蟶群體間的變異絕大部分來自群體內, 群體間的遺傳分化程度不高, 而且這種差別也未影響到遺傳水平。
固定指數Fis是檢驗群體雜合子缺失或過剩的指標,群體內固定指數Fis>0時, 表示品系內存在較嚴重的近交現象, 導致雜合子缺失;Fis<0時, 存在遠交現象, 有雜合子過剩。本研究中發現12個位點中有9個位點存在雜合子過剩現象, 并且平均Fis=-0.21, 表示群體內并無明顯的近交現象。一般認為雜合子過剩情況多的出現在研究對象為相對小的群體或者封閉群體, 如養殖群體中的子代群體往往由于親本數量所限, 奠基者效應(Founder effect)會導致連鎖不平衡現象, 繼而造成雜合子過剩。分析本實驗所出現的雜合子過剩的現象,可能與實驗的樣本量較小有關系。
當雜合子嚴重過剩即Fis明顯偏離0時, 往往會引起群體顯著地偏離Hardy-Weinberg平衡。本次研究中福建云霄群體、廣東湛江群體、天津塘沽群體和浙江樂清灣群體的12個微衛星位點中分別有4、7、7和8個位點偏離了Hardy-Weinberg平衡。這種現象在其他海水貝類, 如類海灣扇貝[21]、櫛孔扇貝[22]中也普遍存在。本研究中的4個縊蟶群體均處于不平衡狀態, 這可能與群體間的無序養殖、遷移有關。浙江樂清灣是一個相對封閉的海灣, 但近年來人工育苗中, 為了提早培育出苗, 每年9至10月份都會從廣東、福建等地大量采購親貝進行人工繁殖, 而培育出的苗種除部分在本地中培和養殖外, 其余的又重新銷往福建、廣東、天津、遼寧等地, 因此遷移造成了對基因型的影響, 可能是導致這種不平衡的主要原因。
3.3 縊蟶群體間遺傳距離與系統發生關系
遺傳距離是研究物種遺傳多樣性的基礎, Crawford等[23]指出由微衛星得出的遺傳距離更能反映分化時間的長短, 能客觀地反映品種間的遺傳變異和分化。本研究中, 浙江樂清灣群體和天津塘沽群體之間的遺傳距離最小為 0.0344, 聚為第一支; 福建云霄群體和廣東湛江群體之間的遺傳距離為 0.103, 聚為第二支。聚類結果與牛東紅[24]利用ISSR標記對6個野生縊蟶群體的聚類結果差異較大, 并未表現出遺傳距離和地理距離之間的正相關。出現這種情況可能與近年來縊蟶無序養殖有關, 福建、廣東、浙江等沿海存在相互間盲目引種和移養的現象, 造成縊蟶資源混亂并使本土自然資源混淆不清, 目前已經很難找到未受人為因素干擾的野生縊蟶群體了。另外, 聚類結果的差異, 也可能與采用的分子標記類型不同有關。
3.4 縊蟶種質資源的保護與開發策略
由于本研究中的 4個群體均來源自當地的養殖群體, 可能已經受到了外來縊蟶群體的污染, 不能充分代表當地野生縊蟶種質資源狀況, 因此建議在做好對當地原始野生縊蟶資源收集的情況下, 利用不同分子標記技術分析各地野生縊蟶原種間及野生原種和人工養殖群體的遺傳差異, 根據主要遺傳差異來源的不同(群體間還是群體內), 制定合理的保種策略。
針對本研究 4個縊蟶群體的變異絕大部分來自群體內, 群體間的遺傳分化程度不高的情況, 筆者認為可以采取如下保種和開發的措施: (1)采取原位保存的措施, 通過建立原種場和自然保護區等方法加強野生原種的保護, 避免外來縊蟶群體對原種的污染。(2)由于4個群體的遺傳變異主要來自群體內,說明合理有序的引種不會帶來不利的遺傳效應[25],因此, 在正確收集和保護好野生原種的基礎上, 可以適當引進其他地理群體的優質縊蟶種質來提高當地種質資源的生產率。
[1]Powell W, Machray G C, Provan J. Polymorphism revealed by simple sequence repeats[J]. Trends Plant Science, 1996, 1: 215-222.
[2]Zane L, Bargelloni L, Patamello T. Strategies for microsatellites isolation: a review [J]. Molecular Ecology, 2002, 11: 1-16.
[3]李琪, 木島明博.長牡蠣(Crassostrea gigas)微衛星克隆快速分離及特性分析[J].海洋與湖沼, 2004, 35(2):364-370.
[4]趙瑩瑩, 朱曉琛, 孫效文, 等.磁珠富集法篩選蝦夷扇貝微衛星序列[J].中國水產科學, 2006, 13(5):749-755.
[5]李春艷, 丁君, 常亞青, 等. 蝦夷扇貝微衛星標記的分離及其養殖群體的遺傳結構分析[J]. 中國水產科學, 2009, 16( 1): 39-46.
[6]Lu X, Wang H X, Liu B Z, et al. Microsatellite-based genetic and growth analysis for a diallel mating design of two stocks of the clam,Meretrix meretrix[J]. Aquaculture Research, 2011: 15-18.
[7]朱東麗, 董迎輝, 林志華, 等. 利用微衛星標記對文蛤 4個殼色花紋品系的遺傳分析.水產學報, 2012,36(2): 202-209.
[8]陳淑吟, 吉紅九, 許廣平, 等. 文蛤微衛星 DNA 的篩選及其特性分析[J]. 生物技術通報, 2009, 12:134-138.
[9]李莉, 孫振興, 楊樹德, 等. 用微衛星標記分析皺紋盤鮑群體的遺傳變異[J]. 遺傳, 2006, 28(12):1549-1554.
[10]牛東紅, 陳慧, 王樹亮, 等. 我國沿海縊蟶居群遺傳結構的mtDNA-COⅠ分析[J]. 動物學雜, 2010, 45(2): 11-18.
[11]于穎, 孟祥盈, 王秀利, 等. 縊蟶遺傳多樣性的RAPD分析[J]. 生物技術通報, 2007, 6:138-143.
[12]王冬群, 李太武, 蘇秀榕, 等. 象山縊蟶養殖居群和野生居群遺傳多樣性的比較[J]. 中國水產科學, 2005,12(2):138-143.
[13]Niu D H, Li J L, Liu B D. Polymorphic microsatellite loci for population studies of the razor clamSinonovacula constricta[J].Convser Genet, 2008, 9: 1393-1394.
[14]劉 博, 滕爽爽、邵艷卿, 等. 縊蟶(Sinonovacula constricta)EST-SSR分布特征及引物開發利用[J].海洋與湖沼, 2012, 43(1):132-137.
[15]劉博, 邵艷卿, 滕爽爽, 等. 樂清灣養殖縊蟶群體遺傳結構的微衛星標記分析[J].中國農學通報,2012,28(2):69-73.
[16]Strauss W M. Preparation of genomic DNA from mammalian tissues. (In): Ausubel F.M, Brent R, Kingston R E, eds. Current protocol in molecular biology[M]. New York, John Wiley and Sons, 1989, 221-222.
[17]Botstein D, White R L, Skolnick M, et al. Construction of a genetic linkage map in man using restriction fragment length polymorphisms[J]. Am J Hum Genet,1980, 32:314-331.
[18]李成華, 李太武, 宋林生, 等. 4個縊蟶群體遺傳結構的RAPD分析[J].水產科學, 2004,23(12):26-28.
[19]England P R, Briscoe D A, Frankham R. Microsatellite polymorphisms in a wild population of Drosophila melanogaster[J]. Genetical Research, 1996(67): 285-290.
[20]游翠紅, 童金茍, 俞小牧. 大鱗副泥鰍 7個群體遺傳變異的微衛星分析[J].水生態學雜志, 2012, 33 (1):84-91.
[21]Zhan A B, Hu J J, Wang X L, et al. A panel of polymorphic EST-derived microsatellite loci for the bay scallop (Argopecten irradians)[J]. Journal of Molluscan Studies, 2006, 72(4): 436-439.
[22]戰愛斌, 胡景杰, 胡曉麗, 等. 富集文庫-菌落原位雜交法篩選櫛孔扇貝的微衛星標記[ J].水產學報, 2008,32(3): 353-361.
[23]Crawford A M, Littlepohn R P. The use of DNA marker in deciding conservation priorities in sheep and other livestock[J]. Animal Genetic Resources Information,1998, 23(1): 21-26.
[24]牛東紅, 李家樂, 馮冰冰, 等. 縊蟶 6個群體遺傳結構的ISSR分析[J]. 應用與環境生物學報, 2009, 15(3):332-336.
[25]Rossetto M, Weaver P K, Dixon K W. Use of RAPD analysis in devising conservation strategies for the rare and endangeredGrevillea scapigera(Proteaceae)[J].Molecular Ecology, 1995, 4: 321-329

