甜菜EST-SSR引物的開發與應用
史樹德,魏 磊,張子義,邵金旺,田自華
(1.內蒙古農業大學農學院,呼和浩特 010019;2.內蒙古農業大學生命科學院,呼和浩特 010018;3.內蒙古農業大學甜菜生理研究所,呼和浩特 010018)
甜菜是我國主要經濟作物之一,其不僅是主要的制糖工業原料,也是發展畜牧業的優良飼料,更是新興的能源作物,在農業產業結構調整和發展地方經濟中占有重要地位。但由于中國不是甜菜起源國,缺少種質資源,再加之相關研究積淀較薄,使我國的甜菜研究水平整體落后,甜菜育種水平落后于發達國家。而隨著基因組學研究的飛速發展,分子設計育種成為作物遺傳改良的重要途徑之一,新的分子標記不斷被開發和廣泛應用,從而加速了作物基因組輔助育種和設計育種的進程[1]。近幾年,國內外在甜菜分子生物學領域的研究正在興起,但可利用分子標記的匱乏嚴重制約其發展。目前,在甜菜基因組輔助育種研究中應用到的分子標記十分有限,僅有關于 RAPD[2-6]、AFLP 和 SNP[7]以及少數 SSR[8-11]、ISSR[12]標記的初步報道,其中可利用的甜菜SSR標記很少。
簡單序列重復(simple sequence repeats,SSRs)或微衛星(microsatellites)是以 1~6個堿基為基元的串聯重復序列,它們普遍存在和均勻分布于大多數真核生物的基因組中,重復數高度變異[13-15]。SSR標記具有多態性高、容易用PCR檢測、重復性高等特點,作為理想的分子標記已廣泛應用到許多植物的遺傳學研究中[16-18]。SSR標記的開發有幾種[19-22]。EST-SSR是近年發展起來的新型分子標記,作為表達基因部分序列的EST和cDNA數據近年來高速增加。截至2011年3月1日,GenBank(http://www.ncbi.nlm.nih.gov/dbEST)中的甜菜EST數據已有29830條。EST-SSR反映了基因的編碼部分,可以直接獲得基因表達的信息,為功能基因提供“絕對”的標記,使得有可能對決定重要表型或農藝性狀的等位基因進行直接鑒定,省去了SSR引物開發過程中的克隆和測序步驟,充分利用了現有測序數據,降低了開發成本,EST-SSR的開發在多種作物中已有報道[23-27]。
為此,本研究目的是利用GenBank中已有的EST序列,開發基于EST序列的SSR標記,并用其進行甜菜種質資源的遺傳多樣性驗證,以篩選多態性高、重復性好的EST-SSR標記;評價收集的甜菜種質資源遺傳多樣性,為建立甜菜高密度遺傳圖譜、功能性分子標記發掘、重要農藝性狀QTLs克隆等研究奠定基礎。
1 材料與方法
1.1 供試材料
本試驗所用甜菜品種為甜研309和KWS9412,KWS9419,KWS5416,農大甜研4號和包育302,種植于溫室,采用CTAB法[28]提取甜菜苗期新鮮葉片基因組DNA。
1.2 EST-SSR的引物開發
首先計算機安裝PC版perl程序,然后從GenBank/dbEST(http://www.ncbi.nlm.nih/entrez)中以FASTA格式下載sugar beet和Beta vulgaris L.中所有的EST序列,共29830條。 利用Blastclust(v.2.2.10;http://www.ncbi.nih.gov/web/newsltr/spring04/blastlab.htm)軟件,對這些EST序列進行冗余性查找,利用CD_HIT(http://www.bioinformatics.org/cd-hit/)快速批量去冗余序列;利用 est_timmer(http://pgrc.ipk-gatersleben.de/misa/)去除EST 序列中過短的序列(<100bp)和過長的序列(>700bp)以及 mRNA 的“帽子”和“尾巴”,利用 misa.pl(http://pgrc.ipk-gatersleben.de/misa/)識別和定位SSR,其中配置文件misa.ini用來設置識別SSR標記的標準;利用primer3模塊批量設計SSR引物,引物設計的主要參數是,引物長度20~26bp;退火溫度Tm值48℃~65℃;(G+C)含量30%~70%;PCR擴增產物長度大于150bp;利用MacVector7.0軟件對所設計的引物進行驗證,引物由上海生工生物技術有限公司合成。
1.3 EST-SSR的擴增與檢測
PCR反應在25μL的體積中進行,含PCR緩沖液、50~80 ng的模板DNA、1.5 U Taq聚合酶,上、下游引物各0.5μmol/L、200μmol/L dNTP和2.0 mmol/L MgCl2。整個反應過程在PE 9700上進行,循環條件是:94℃預變性3min;然后35個循環,每個循環包括94℃變性45s,復性的合適溫度通過溫度梯度實驗確定,并盡可能提高復性溫度以增加特異性,復性時間60s,72℃延伸1min 20s;最后于72℃延伸8min。復性溫度低于50℃仍無產物出現,則認為該引物不能擴增。PCR產物經2.5%瓊脂糖凝膠電泳檢測。
1.4 多態信息含量和遺傳相似系數分析
多態信息含量常被用來評估所設計引物的潛在利用率。本實驗的每個EST-SSR引物的多態信息含量按Park等[29]提供的公式計算,即其中Pi為i位點的基因頻率,n為等位基因數。遺傳相似系數分析利用NTSYS-pc軟件[30]的SIMQUAL模塊計算6個甜菜品種間的遺傳相似系數。然后基于遺傳相似系數矩陣,依據UPGMA算法并利用NTSYS-pc軟件的SHAN聚類程序對6個材料進行聚類分析。
2 結果和分析
2.1 甜菜EST-SSR的特征
從NCBI網站下載的29830條主要來源于sugar beet和Beta vulgaris L.的EST序列(截止2011年3月1日)。經過除去ploy A或T“尾巴”和載體等序列后共獲得20109條無冗余EST序列,序列總長度為11287687bp。按照上述查找標準,共發現6951條至少含有1個SSR的EST序列,占無冗余EST總數的34.6%,表明甜菜EST-SSR含量比較豐富。按照SSR具體實驗引物設計要求,從4338個SSR重復中設計得到2845對EST-SSR引物。這些引物對中有1052個含有1個SSR重復,其余1793對含有2個或2個以上的SSR重復。在鑒定的SSR中A/T單堿基重復最多,其最少重復9次,最大重復28次;AAG/CTT三核苷酸重復次之,最少重復5次;AG/CT二核苷酸重復居第三,最少重復6次;四、五和六核苷酸重復數量最少,其最少重復5次(見表1);在同一條EST序列中所含2個SSR位點之間最小相距2個核苷酸,最大相距100個核苷酸。其中除單核苷酸重復外,AAG/CTT三核苷酸重復基元是出現頻率最高的EST-SSR類型,占總SSR的12.7%,AG/CT次之,占10.4%,ACCTCC/AGGTGG等六核苷酸重復最少,約占1%。

表1 在20109條甜菜EST序列中發掘的不同基元的SSR數量及百分率統計
2.2 EST-SSR引物的擴增效率
在所得的2845對SSR引物中隨機挑選并合成了100個SSR引物對,在前述給定的條件下對6個品種甜菜基因組DNA進行PCR擴增,有47對(47%)引物擴增出了SSR特征條帶,共產生621個位點,平均每對引物產生13.2個位點。表2為47個具有功能性引物對的相關信息。在47個功能性引物對中,有14個引物對(29.7%)擴增出1條產物帶,有25個引物對(31.9%)擴增出2條條帶,8個引物對(17.2%)擴增出2條以上條帶;有38個引物對的擴增產物在預期片段大小范圍之內,占功能性引物對的80.8%,9個引物對的擴增產物小于預期片段大小;此外,在33個擴增出2條或2條以上條帶的引物對中,有9個引物對擴增出比預期片段大的條帶,在47個功能性引物對中,有25個引物對在6個甜菜品種間擴增出多態性,多態性引物對占53.2%。25個引物對在6個品種中共擴增出138個多態性標記。
2.3 EST-SSR引物的多態性分析
每對引物的多態性信息含量用PIC值表示,并用量估計等位基因的變異情況。依據47對引物在6個甜菜品種中擴增得到的等位基因的變異信息來計算各引物的PIC值。它們的PIC值變化在0.13~0.71之間,平均為0.47,引物S9的PIC值最大,達0.71;而引物S10的最小,只有0.13。
2.4 遺傳相似系數分析
基于這 25個多態性EST-SSR標記,依據UPGMA算法利用NTSYS-pc2.1軟件將本研究所用6個甜菜品種進行聚類分析,可將6個品種劃分為不同的兩組(圖1):第一組為國內品種,其中甜研309與農大甜研4號和包育302親緣關系較遠,第二組為親緣關系較近的德國KWS系列3個品種,系統發育分析表明國內外兩組材料親緣關系較遠,遺傳相似度較低,相似系數為0.31。

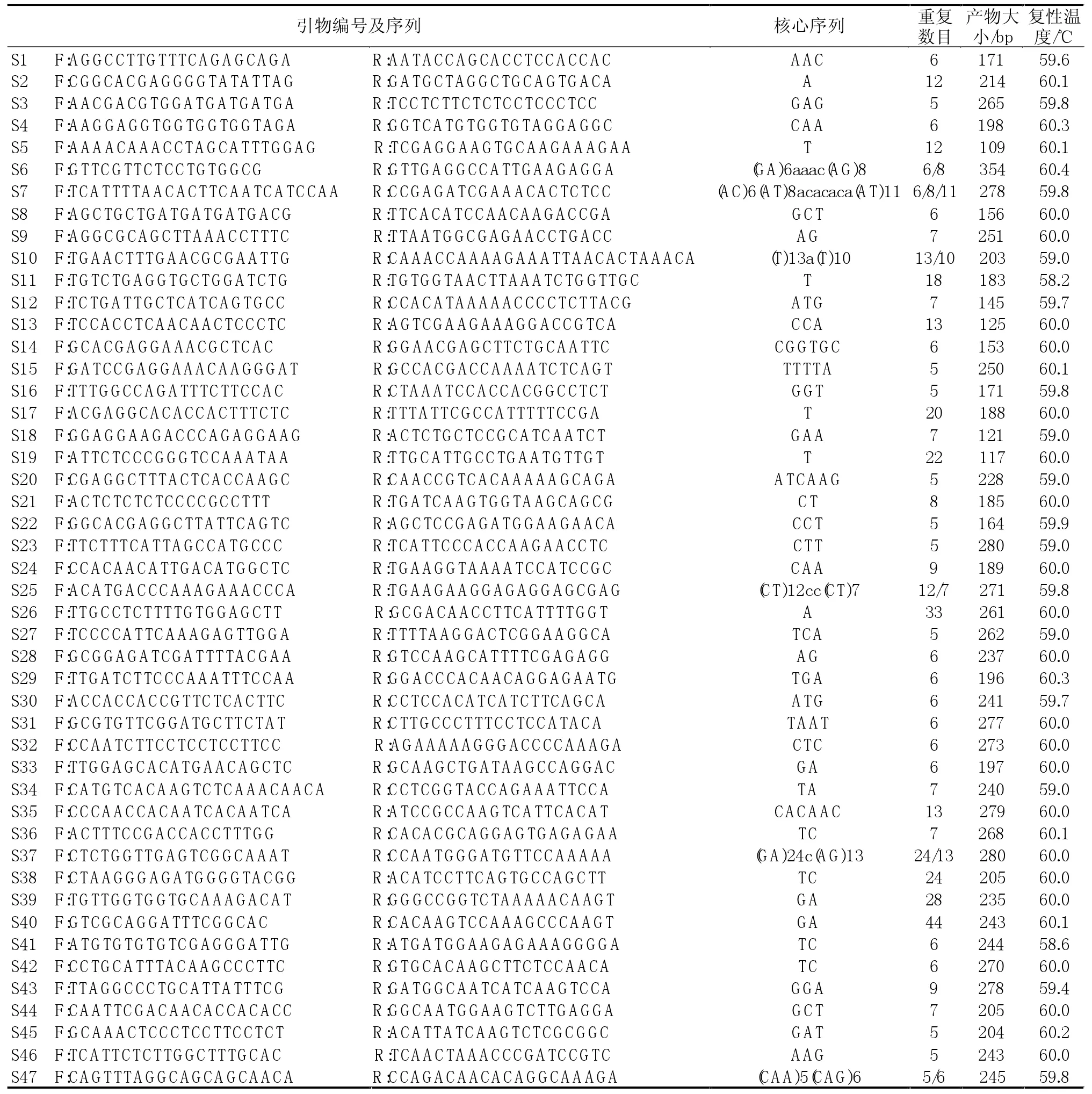
表2 甜菜ESTs中分離到的部分微衛星引物
3 討論
與其他分子標記相比,EST-SSR標記來自表達基因的全部或部分序列,其多態性可能直接與該基因功能相聯系。研究發現,水稻的蠟質基因5'端非編碼區中的SSR序列(CT)n長度變化不僅與直鏈淀粉的含量有關[31],而且還決定了糯稻淀粉的理化特性[32]。因此根據EST包含的SSR位點開發的分子標記,是直接與功能相關的功能標記[33],EST-SSR標記的這一特點決定了其在功能基因標記方面應用前景廣闊。
本研究中,多態信息位點PIC值變化在0.13~0.71之間,較已知甜菜SSR標記PIC值低[34],可能是由于EST標記來源于基因編碼區,具有很高的保守性,因此它們在品種之間的多態性要低于來源其它基因組區域分子標記[35],但是通過根據EST 5'端或3'端的UTR區開發的SSR標記多態性要稍好些[33]。因此,在設計引物時,可設定軟件條件,應盡量使引物靠近3'或5'端非編翻譯區。本研究中較低的PIC值與張艷欣和Taliercio等人的研究結果一致[36,18],表明基于表達基因的EST-SSR標記的多態性確實低于基因組SSR[37]。在100對引物中有47對成功擴增出了SSR特征條帶,其它未獲得擴增產物的引物可能的原因是正向引物或反向引物或二者都恰好跨過了mRNA的剪切位點,也可能是由于在相應的基因組DNA中存在較大的內含子,這兩個原因都會導致引物無法與模板DNA結合而不能擴增,從而導致擴增效率較低。基于遺傳相似系數的聚類分析表明,國內和國外品種間遺傳差異度較大,可能是其親本來源不同所致。本研究中,三核苷酸重復數量高于二核苷酸重復,此結果與EST中三核苷酸比二核苷酸更為豐富的結論相符[32]。
綜上,EST-SSR標記易于開發、成本較低,其功能可以通過序列同源性比對獲得。而且由于來自于轉錄譜,能夠反映出轉錄區的差異,使得EST-SSR標記在遺傳多樣性分析、比較作圖和分子標記輔助選擇育種的研究中具有更高的應用價值。
[1]Peleman J D,van der Voort J R.Breeding by design[J].Trends Plant Sci.,2003,8(7):330-334.
[2]張福順.葉用甜菜種質資源遺傳多樣性及親緣關系的研究[D].哈爾濱:中國農業科學院甜菜研究所,1999.
[3]UphoffH,WrickeG.Ageneticmapofsugarbeet(BetaVulgarisL.) basedonRAPDmarkers[J].PlantBreeding,1995,114(4):355-357.
[4]路運才.甜菜多倍體品種和親本的生物學特性及其系統發育關系的研究[D].哈爾濱:中國農業科學院甜菜研究所,2001.
[5]Askar,N.A.,Reza,A.,Ali,H.N.,Jalal,S.,Mahmood,M.Use of Molecular Marker for Assay Gene Dosage Resistant Gene to Rhizomania Disease (Rz1) in Sugar beet(Beta vulgaris L.) [J].Asian J.of Biotech., 2009,1(1):7-41.
[6]Amiri R.,Mesbah M.,Moghaddam M.,Bihamta M.R.,Mohammadi S.A.and Norouzi P.A new RAPD marker for beet necrotic yellow vein virus resistance gene in Beta vulgaris[J].Biologia Plantarum,2009,53(1):112-119.
[7]Grimmer M.K.,Trybush S.,Hanley S.,Francis S.A.,Karp A.and Asher M.J.C.An anchored linkage map for sugar beet based on AFLP,SNP and RAPD markers and QTL mapping of a new source of resistance to Beet necrotic yellow vein virus[J].Theoretical and Applied Genetics.,2007,114(7):1151-1160.
[8]Annika Hjerdin Panagopoulos.Application of Molecular Markers in Sugar Beet Breeding[D].Lund:Lund University,2003.
[9]Schneider K,Sch?fer-Pregl R,Borchardt C and Salamini F.Mapping QTLs for sucrose content,yield and quality in a sugar beet population fingerprinted by EST-related markers[J].Theor.Appl.Genet.,2002,104(6-7):1107-1113.
[10]趙尚敏,白晨,張惠忠,等.適于甜菜遺傳多樣性分析的SSR引物篩選[J].中國糖料,2009(4):6-8.
[11]王華忠,吳則東,王曉武,等.利用SRAP與SSR標記分析不同類型甜菜的遺傳多樣性[J].作物學報,2008,34(1):37-46.
[12]付增娟,史樹德,張少英,等.甜菜ISSR-PCR反應體系的優化[J].中國糖料,2008(4):7-9.
[13]Powell W,Machray G C,Provan J.Polymorphism revealed by simple sequence repeats[J].Trends Plant Sci.,1996,1(7):215-222.
[14]TóthG,GáspáriZ,JurkaJ.Microsatellitesindifferenteukaryoticgenomes:surveyandanalysis[J].GenomeResearch,2000,10(7):967-981.
[15]Tautz D,Renz M.Simple sequences are ubiquitous repetitive components of eukaryotic genomes[J].Nucleic Acids Research,1984,12(10):4127-4138.
[16]朱振東,賈繼增.小麥 SSR 標記的發展及應用[J].遺傳,2003,25(3):355-360.
[17]Blair M W,Giraldo M C,Buendía H F,et al.Microsatellite marker diversity in common bean (Phaseolus vulgaris L)[J].Theor.Appl.Genet.,2006,113(1):100-109.
[18]Rongwen J,Akkaya M S,Bhagwat A A,et al.The use of microsatellite DNA markers for soybean genotype identification[J].Theor.Appl.Genet.,1995,90(1):43-48.
[19]張亞平,王文,宿兵,等.大熊貓微衛星DNA的篩選及其應用[J].動物學研究,1995,16(4):301-306.
[20]Rassmann K,Sehlotterer C,Tautz D.Isolation of simple sequence loci for use in Polymerase chain Reaction-based DNA finger printing[J].Electrophoresis,1991,12(2-3):113-118.
[21]Karagyozov L,Kalcheva I D,Chapman V M.Construction of random small-insert genomic libraries highly enriched for simple sequence repeats[J].Nucleic Acids Research,1993,21(16):3911-3912.
[22]Lian C L,Zhou Z H,Hogetsu T.A simple method for developing microsatellite markers using amplified fragments of intersimple sequence repeat[J].Journal of Plant Research,2001,114(3):381-385.
[23]Taliercio E,Allen R D,Essenberg M,et al.Analysis of ESTs from multiple Gossypium hirsutum tissues and identification of SSRs[J].Genome,2006,49(4):306-319.
[24]Scott K D,Eggler P,Seaton G,et al.Analysis of SSRs derived from grapes EST[J].Theor.Appl.Genet.,2000,100(5):723-726.
[25]Eujayl I,Sorrells M E,Wolters P,et al.Isolation of EST-derived microsatellite markers for genotyping the A and B genomes of wheat[J].Theor.Appl.Genet.,2002,104(2-3):399-407.
[26]Rungis D,Berube Y,Zhang J,et al.Robust simple sequences repeat markers for spruce (Picea spp.) from expressed sequence tags[J].Theor.Appl.Genet.,2004,109:1283-1294.
[27]魏利斌,張海洋,鄭永戰,等.芝麻EST-SSR標記的開發和初步研究[J].作物學報,2008,34(12):2077-2084.
[28]Doyle J J,Doyle J L.A rapid DNA isolation procedure for small quantities of fresh leaf tissue.Phytochemical Bulletin,1987,19:11-15.
[29]Park Y H,Alabady M S,Ulloa M,et al.Genetic mapping of new cotton fiber loci using EST-derived microsatellites in an interspecific recombinant inbred (RIL) cotton population[J].Mol Genet Genom,2005,274(4):428-441.
[30]Rohlf F J.NTSYS-pc:Numerical Taxonomy and Multivariate Analysis System,Version 2.1,User Guide[CP].Exeter Software,New York,2000.
[31]Ayers,N.M,McClung,A.M,Larkin,P.D,et al.Microsatellites and a single nucleotide polymorphism differentiate apparent amylose classes in an extended pedigree of US rice germplasm[J].Theor.Appl.Genet.,1997,94:773-781.
[32]Bao J S,Corke H,Sun M.Microsatellites in starch synthesizing genes in relation to starch Physiochemical Properties in waxy rice(Oryza sativa L.).Theor.Appl.Genet.,2002,105(6-7):898-905.
[33]Thiel T,Michalek W,Varshney R K,et al.Exploiting EST databases for the development and characterization of gene-derived SSR-markers in barley (Hordeum vulgare L.) [J].Theor.Appl.Genet.,2003,106:411-422.
[34]Christopher M.R,Mary B,Sharon e.M,et al.Polymorphic microsatellite markers for inferring diversity in wild and domesticated sugar beet(Beta vulgaris L.) [J].Molecular Ecology Notes,2004,4:243-245.
[35]Eujayl l,Sorrells M E,Baum M,wolters et al.Assessment of genotypic variation among cultivated durum wheat based on ESTSSRs and genomic SSRs[J].EuPhytica,2001,119(1):39-43
[36]張艷欣,林忠旭,李武,等.海島棉EST-SSR引物的開發與應用研究 [J].2007,52(15):1779-1787.
[37]Zhang L Y,Bemard M,Uroy P,Feuillet C.High transferability of bread wheat EST-derived SSRs to other cereals[J].Theor.Appl.Genet.,2005,111:677-687.

