長牡蠣(Crassostrea gigas)EST串聯重復序列的組成和分布
張琳琳,李 莉,張國范
(1.中國科學院 海洋研究所,山東 青島 266071;2.中國科學院 研究生院,北京 100049)
長牡蠣(Crassostrea gigas)也稱太平洋牡蠣,具有體型大、生長快、產量高、適應性強等優點,在我國北部沿海大面積養殖,是我國雙殼貝類養殖中規模大、產量高的養殖品種之一。長牡蠣作為冠輪動物超門的模式種,在大片段文庫和遺傳圖譜的構建,表達譜差異分析,雜種優勢探討等方面進行了較詳細的研究[1-4],但基于大規模數據的重復序列方面的研究相對較少[5-6]。本文主要對長牡蠣EST進行串聯重復序列結構類型,分布,豐度等的比較分析。
串聯重復序列是指 1~200個堿基左右的核心重復單位,以頭尾相串聯的方式重復多次所組成的重復序列。它們在基因組中有著基因表達調節,群體遺傳多樣性分析等重要作用,與多種疾病相關[7]。 而簡單序列重復,即微衛星(Simple sequence repeat,SSR),更是廣泛地應用于遺傳連鎖圖譜構建[8-9]和物種基因組結構的分析[10]。雖然長牡蠣大規模系統的基因組測序工作還沒有完成,但NCBI上公布了大量的長牡蠣EST(Expressed sequence tags,表達序列標簽)數據。所謂EST是指通過對cDNA文庫隨機挑取克隆進行大規模測序所獲得的cDNA的5’或3’端序列,長度一般為150~500bp。研究表明長牡蠣EST中存在大量重復序列,可用于SSR標記的開發[6],這為從EST中尋找并分析串聯重復序列提供了依據。通過物種間和物種內串聯重復序列的比較,研究轉錄本的結構特征,分析其串聯重復序列特別是 SSR的分布特征和可能的功能,將有助于了解基因組的起源和進化,同時更好地發揮這些序列在串聯重復序列標記方面的應用。
截至2009年11月1日,在NCBI數據庫中已登錄了57 139條長牡蠣ESTs,但未有對上述57 139條EST全面的串聯重復序列的報道。本研究旨在對現有長牡蠣EST中的串聯重復序列信息進行結構類型,分布和豐度比較分析,以明確長牡蠣串聯重復序列的發生頻率和特點。同時分析了SSR在全長cDNA中的分布特點,以探討長牡蠣轉錄本的結構和進化壓力。本研究有助于促進串聯重復序列特別是 SSR標記在基因組結構進化和長牡蠣遺傳育種中的應用。
1 材料與方法
1.1 長牡蠣EST序列的下載和預處理
從NCBI庫中下載57 139條長牡蠣ESTs(2009-11-01),過濾長度小于 100 bp的序列并與 UniVec(http://www.ncbi.nlm.nih.gov/VecScreen/UniVec.html)比對去除載體序列,在去掉3’末端的PolyA后,得到56 968條序列。利用 Sequence Assembly Program,CAP3[11]對上述序列進行初步聚類,采用的參數為重疊長度閾值N>30,重疊的一致性百分比N>90。
1.2 長牡蠣EST串聯重復序列的分析
利用Tandem Repeat Finder (TRF)[12]對預處理的EST進行串聯重復序列尋找,比對參數(匹配,不匹配,插入缺失)為 2,7,7,最小比對分值 30,重復單位最大長度500。過濾掉重復序列長度不足15bp的重復序列。如果同一位置出現的不同重復序列預報,本研究取重復序列長度最大的類型。長牡蠣的HindIII衛星序列的多序列比對采用 DNAMAN5.2.2(Lynnon Biosoft Company)。
1.3 長牡蠣 EST 5’UTR,3’UTR 和編碼區CDS SSR分析
從NCBI庫中下載644條長牡蠣蛋白質序列對應的EST序列,手工篩選出含有編碼區全長和5’UTR,3’UTR的序列,共 80條。分別使用 TRF分析其5’UTR,3’UTR和CDS中SSR的分布情況。
2 結果
2.1 長牡蠣EST衛星重復序列的分析
在處理后的長牡蠣EST中共有10 997條串聯重復序列(397 019 bp),其中小衛星重復序列(7~436 bp)有8 392條,共335 207 bp,占分析EST序列的1.58%(圖1a,b,c)。重復序列單元總數目和重復類型間有一定規律性。重復序列單元總數目較多集中到7~12 bp,其中9 bp重復單元數目最多,為3 067個重復單元,其次是8 bp,10 bp,11 bp,12 bp,7 bp。從13 bp重復類型開始,重復單元數目降至1 000以下。隨著重復單元長度的不斷增加,重復單元數目大致上不斷減少。在24~50 bp重復之間,重復單元數目波動相對較大。重復單元長度大于55 bp的區域中,在63 bp時出現一個峰,重復單元數目為 32.4,其他的重復單元類型相應的重復單元數目均小于25 bp。重復單元長度大于300 bp的只有3個重復類型,相應的重復單元總數目為 6.6。另一方面,串聯重復序列平均拷貝數與重復類型并沒有表現出線性關系,而是呈現不規律性的波動(圖1 d)。
在長串聯重復序列的分析中(本文中指串聯重復序列的長度大于 100bp的重復類型),162~167bp 重復單元呈現一個明顯的峰(圖1 c)。將此部分序列提出,分析發現與長牡蠣的HindIII satellite DNA具有保守性(圖2)。
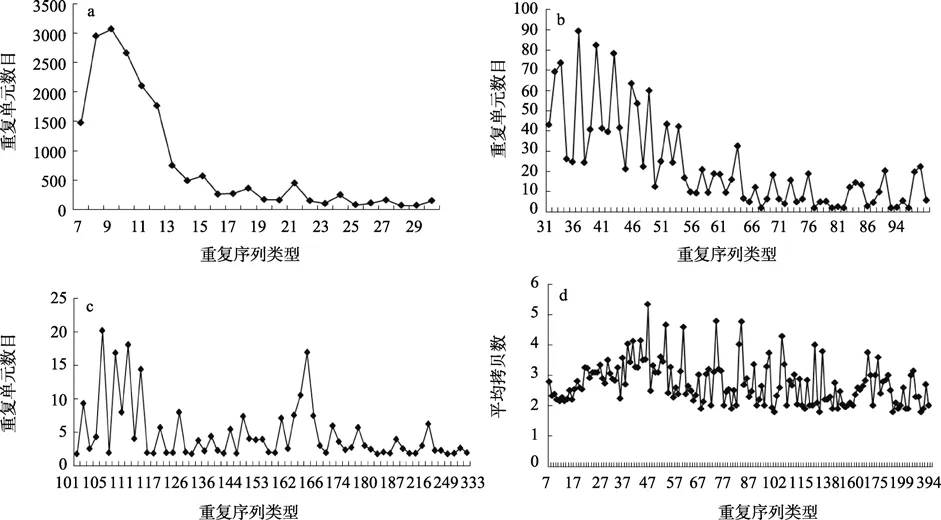
圖1 不同串聯重復序列類型在長牡蠣中的拷貝數特征Fig.1 The copy number of tandem repeats in the pacific oyster ESTs
2.2 長牡蠣EST簡單串聯重復序列的分析
長牡蠣 EST中含有豐富的 SSR,共 2 602個,61 744 bp,占分析序列總堿基的0.29%(表1)。重復序列數目表現為六堿基重復序列>單堿基>二堿基>三堿基>五堿基>四堿基,分別為851,805,307,258,240和141。重復序列長度、簡單重復序列類型與拷貝數的研究過程中,發現重復序列單元長度與平均拷貝數成反比。另一方面,相同重復單元長度不同重復類型的重復序列數目、重復序列長度和平均拷貝數有很大的差別。每種重復單元類型代表其互補或順序不同的所有重復單元,如 ATC代表ATG/TGA/GAT/CAT/ATC/TCA 6種重復類型。由于四堿基、五堿基、六堿基重復序列的重復類型較多,我們用 AT的百分比代替分析 SSR的分布特征和結構[9]。研究發現,A串聯重復遠遠大于T。對于G串聯重復最大拷貝數為 974,是因為 NCBI號為 FP000596的序列低質量測序,在除去此序列的影響后,G串聯重復序列的最大拷貝數為 26。不同的重復序列重復類型重復序列的拷貝數目不同,如二堿基重復中,AG的重復序列數目遠遠大于AT、AC和GC。相同重復單元長度不同重復類型的平均拷貝數也有很大差別,并且與該重復類型的重復序列數目無關,如 ATC重復類型的重復序列數目約為ACT的30倍,但ACT重復類型的平均拷貝數大于ATC重復類型。
EST-SSR在標記應用時,多是以PCR為基礎的,對SSR兩側的側翼序列有一定長度的要求。因此,本研究統計了簡單重復序列兩側的側翼序列不低于30bp的微衛星位點,統計表明長牡蠣有1 954個簡單重復序列位點符合要求,這些位點是微衛星標記開發的候選。

圖2 長牡蠣HindIII 衛星序列的多序列比對Fig.2 Alignment of multiple HindIII satellites in Pacific oyster
2.3 長牡蠣cDNA 5’UTR,3’UTR和編碼區CDS SSR分析
對挑選的含有5’UTR,3’UTR以及完整的編碼區的80條長牡蠣序列分析發現UTR區域SSR長度所占的比例(0.005和 0.0026)遠遠大于 CDS區域 SSR所占的比例(0.0011)(表2)。因為5’UTR序列總長度相對較少,SSR 重復單元數目的關系為:5’UTR <CDS < 3’UTR,分別為 19,32.9 和 64.3。此外,cDNA的位置對簡單串聯重復序列的重復類型具有選擇性。5’UTR區域只含有單堿基重復單元,CDS區域只含有三堿基倍數重復單元(三堿基/六堿基),3’UTR所含的重復單元類型較為豐富,含有單堿基,二堿基和五堿基重復單元。
3 討論
3.1 長牡蠣EST中串聯重復序列類型豐富,162~167bp類型含量高
從NCBI上下載的長牡蠣的EST序列中含有豐富的串聯重復序列類型。覆蓋從 1~436bp重復類型的 152種。對長牡蠣 100bp的重復類型中 162~167范圍的峰值的分析表明,14個重復序列中有5個與長牡蠣的HindIII衛星序列具有高的相似度。南極貝(Adamussium colbecki)中曾報道了一個170bp重復單元的衛星序列,占基因組序列的 0.2%[13]。該衛星序列之后又被證明在牡蠣中具有中間的保守性,與哺乳動物的CENP-B box具有保守性,并被用來做牡蠣物種分類的標記[14]。
3.2 長牡蠣SSR分布廣泛,1 594個候選位點
在簡單重復序列中,從單堿基重復到六堿基重復均覆蓋大多數重復序列類型。不同的簡單重復序列類型的拷貝數目有很大差異。在二堿基重復中,AG的重復序列數目高達221,AT和AC均不超過50,GC最少為0,這與前人的報道相一致[8,15-16]。在三堿基重復序列中,ATC重復序列數目最多為73次,其次為AAC,AAT,AAG,AGG,其他的類型重復次數均小于15次,這與之前在櫛孔扇貝中的報道類似[15]。在四、五、六堿基重復序列中,我們發現第二高AT百分比的重復序列類型擁有更高的重復序列數目,這與家蠶中的報道相一致[9]。從引物設計的角度考慮,
有1 594個位點為微衛星標記開發的候選位點,該結果為進一步開發長牡蠣EST-SSR標記奠定了基礎。

表1 長牡蠣EST微衛星重復序列的數目、長度和拷貝數特征Tab.1 The number,length,and copy number of SSR in the Pacific Oyster EST
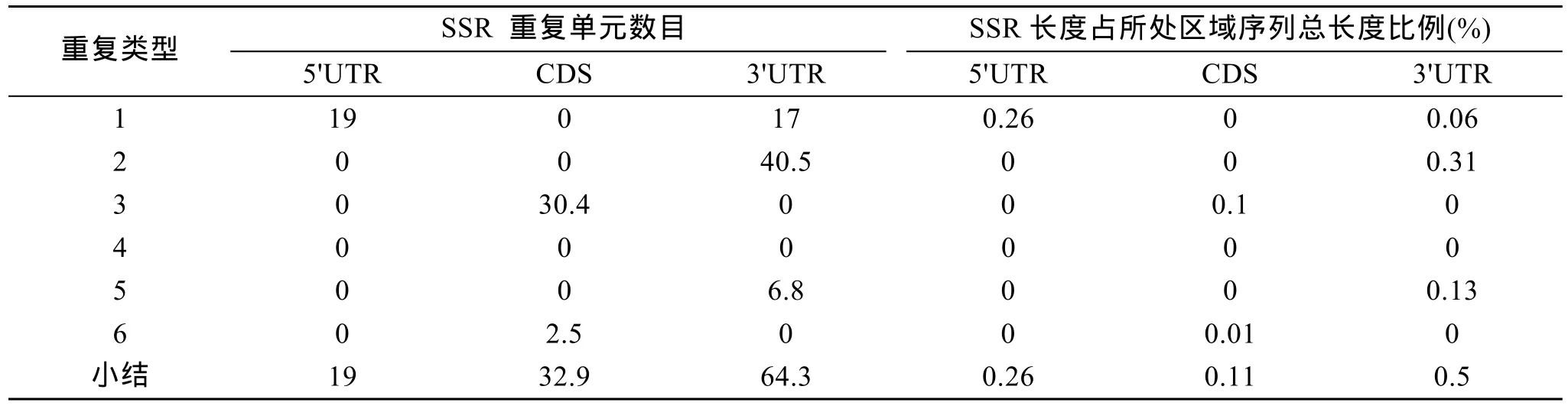
表2 長牡蠣EST簡單重復序列5’UTR,3’UTR和CDS特征Tab.2 The distributions of 5’UTR,3’UTR and CDS of SSR in the Pacific Oyster EST
3.3 長牡蠣EST的CDS區承受更大的選擇壓力
CDS區域簡單串聯重復序列相對較少,這與編碼區受到的選擇壓力大于UTR區域有關,而編碼區的重復序列類型為三堿基和六堿基,這兩種堿基類型均為編碼氨基酸的密碼子數目3的倍數,這更說明了非3倍數的簡單重復序列對編碼區具有破壞作用,而自然選擇將這部分破壞的簡單重復序列淘汰了,這與水稻中的報道相一致[17]。在本研究中,編碼區三堿基重復序列的類型為ACA,GAA和GAT重復,推測該三種重復類型可能與串聯重復數目具有一定聯系,其進一步研究可能需要使用更多的全長cDNA才能得出更明確的結論。
[1]Cunningham C,Hikima J,Jenny M J,et al.New resources for marine genomics:bacterial artificial chromosome libraries for the Eastern and Pacific oysters (Crassostrea virginicaandC.gigas)[J].Mar Biotechnol (NY),2006, 8(5):521-533.
[2]Hubert S,Hedgecock D.Linkage maps of microsatellite DNA markers for the Pacific oysterCrassostrea gigas[J].Genetics,2004, 168(1):351-362.
[3]Fleury E,Huvet A,Lelong C,et al.Generation and analysis of a 29,745 unique Expressed Sequence Tags from the Pacific oyster (Crassostrea gigas) assembled into a publicly accessible database:the Gigas Database[J].Bmc Genomics,2009, 10:341.
[4]Hedgecock D,Lin J Z,DeCola S,et al.Transcriptomic analysis of growth heterosis in larval Pacific oysters(Crassostrea gigas)[J].Proc Natl Acad Sci U S A,2007,104(7):2313-2318.
[5]Wang Y,Guo X.Development and characterization of EST-SSR markers in the eastern oysterCrassostrea virginica[J].Mar Biotechnol (NY),2007, 9(4):500-511.
[6]Wang Y,Ren R,Yu Z.Bioinformatic mining of EST-SSR loci in the Pacific oyster,Crassostrea gigas[J].Anim Genet,2008, 39(3):287-289.
[7]Richard G F,Kerrest A,Dujon B.Comparative genomics and molecular dynamics of DNA repeats in eukaryotes[J].Microbiol Mol Biol Rev,2008, 72(4):686-727.
[8]Toth G,Gaspari Z,Jurka J.Microsatellites in different eukaryotic genomes:survey and analysis[J].Genome Res,2000, 10(7):967-981.
[9]Prasad M D,Muthulakshmi M,Madhu M, et al.Survey and analysis of microsatellites in the silkworm,Bombyx mori:frequency,distribution,mutations,marker potential and their conservation in heterologous species[J].Genetics,2005, 169(1):197-214.
[10]Subramanian S,Mishra R K, Singh L.Genome-wide analysis of microsatellite repeats in humans:their abundance and density in specific genomic regions[J].Genome Biol,2003, 4(2):R13.
[11]Huang X Q,Madan A.CAP3:A DNA sequence assembly program[J].Genome Research,1999, 9(9):868-877.
[12]Benson G.Tandem repeats finder:a program to analyze DNA sequences[J].Nucleic Acids Res,1999, 27(2):573-580.
[13]Canapa A,Barucca M,Cerioni P N,et al.A satellite DNA containing CENP-B box-like motifs is present in the antarctic scallopAdamussium colbecki[J].Gene,2000, 247(1-2):175-180.
[14]Lopez-Flores I,de la Herran R,Garrido-Ramos M A,et al.The molecular phylogeny of oysters based on a satellite DNA related to transposons[J].Gene,2004,339:181-188.
[15]Zhang L,Chen C,Cheng J,et al.Initial analysis of tandemly repetitive sequences in the genome of Zhikong scallop (Chlamys farreriJones et Preston)[J].DNA Seq,2008, 19(3):195-205.
[16]Li Y C,Korol A B,Fahima T,et al.Microsatellites:genomic distribution,putative functions and mutational mechanisms:a review[J].Mol Ecol,2002, 11(12):2453-2465.
[17]Zhang Z and Xue Q.Tri-nucleotide repeats and their association with genes in rice genome[J].Biosystems,2005, 82(3):248-256.

