兩種體色三疣梭子蟹線粒體DNA部分片段序列的比較分析
遲大利, 高 煥 沈頌東, 閻斌倫
(1. 淮海工學院江蘇省海洋生物技術重點建設實驗室, 江蘇 連云港 222005; 2. 蘇州大學生命科學院, 江蘇蘇州 215123)
兩種體色三疣梭子蟹線粒體DNA部分片段序列的比較分析
遲大利1,2, 高 煥1, 沈頌東2, 閻斌倫1
(1. 淮海工學院江蘇省海洋生物技術重點建設實驗室, 江蘇 連云港 222005; 2. 蘇州大學生命科學院, 江蘇蘇州 215123)
三疣梭子蟹(Portunus trituberculatus)作為重要的海洋經濟動物, 其常見體色(頭胸甲)為茶綠色。近年來在我國沿海海域海捕三疣梭子蟹中開始出現體色為紫色的一類梭子蟹, 除體色不同外, 二者表型特征并無顯著差異。對兩種體色三疣梭子蟹不同個體的線粒體COI和16S rRNA基因進行了序列比對分析以判定二者之間的親緣關系。結果發現COI和16S rRNA基因在兩種體色三疣梭子蟹群體中的核苷酸序列一致性分別為99.87%和99.88%, 表明紫色三疣梭子蟹并未發生亞種的分化, 即紫色和茶綠色群體屬于同一個種。并利用GenBank數據庫檢索了梭子蟹科其他15種海產蟹的16S rRNA序列, 進行了序列同源性比對分析和分子系統學方面的探討, 為梭子蟹種屬分類學研究提供分子依據。
三疣梭子蟹; 體色; 線粒體DNA; 16S rRNA; COI
三疣梭子蟹(Portunus trituberculatus)隸屬于甲殼綱(Crustacea), 十足目(Decapoda), 梭子蟹科(Portunidae), 梭子蟹屬(Portunus), 又稱梭子蟹、槍蟹、海螃蟹、海蟹、門蟹等, 是一種大型海洋經濟動物, 廣泛分布于中國、日本、朝鮮等西太平洋沿岸海域[1], 具有肉質好、食物鏈短、生長快、產量高等優點[2], 是我國重要的海洋漁業資源。鑒于其較高的經濟價值及其在生態環境中的重要作用, 自20世紀50年代起, 國內外學者相繼對三疣梭子蟹生活習性、生理生化、胚胎發育、養殖模式、群體遺傳結構、超微結構觀察和核型分析等方面做了深入的研究, 而對三疣梭子蟹分子生物學展開的研究工作尚處于起步階段, 且主要集中在分子標記研究領域[3]。
常見三疣梭子蟹體色(頭胸甲)為茶綠色, 近年來, 在我國從渤海到東海廣大海域的海捕野生群體中發現一類體色為紫色的梭子蟹。從形態上觀察, 除了身體顏色存在差異外, 兩種體色梭子蟹其他表型特征相似。對于此類紫色梭子蟹的來源有不同的說法: 有的認為雌性梭子蟹為紫色, 雄性蟹為茶綠色[4];有的認為它們屬于兩個不同的種[5]。因此, 有必要了解不同體色梭子蟹之間的關系及其與其他蟹類的系統發生學關系。
由于母性遺傳、進化率低以及缺乏分子間遺傳重組等特點, 線粒體DNA已經廣泛應用于群體遺傳結構分析和不同分類水平物種間親緣關系的研究[6]。迄今為止, 線粒體兩個保守序列基因COI(細胞色素c氧化酶亞基I基因)和16S rRNA基因已經成功應用于甲殼類動物的生物學親緣關系鑒定[7]。通過應用長PCR技術, Yamauchi等[7]首次對三疣梭子蟹線粒體基因組進行全序列分析研究。郭天慧等[2]對三疣梭子蟹線粒體DNA COI和16S rRNA基因片段序列進行比較研究發現, 16S rRNA基因較 COI基因更為保守,表明 COI基因適于種內遺傳多態性研究, 而 16S rRNA基因適于種屬間遺傳多態性分析。
Place等[8]對可口美青蟹(Callinectes sapidus)整個線粒體基因組進行了測序和基因定位, 發現可口美青蟹和三疣梭子蟹線粒體基因組長度(分別為16 263 bp 和16 026 bp)十分接近, 且二者線粒體基因組內基因序列及相關功能基因的定位均十分相似,表明三疣梭子蟹和可口美青蟹的親緣關系相近, 但是, 三疣梭子蟹和可口美青蟹分別生活在太平洋和大西洋沿岸[8], 處于地理隔離狀態, 分類學上分別隸屬于梭子蟹屬和青蟹屬(Callinectes), 且二者體色亦有不同, 因此有必要了解二者在分子系統分類學中的關系。
本文期望通過線粒體DNA COI和16S rRNA部分基因片段在茶綠色和紫色梭子蟹間的序列比對分析, 以闡釋清楚兩種體色梭子蟹的親緣關系, 即二者是否達到亞種的分化水平; 并對三疣梭子蟹與梭子蟹屬其他 14種梭子蟹以及青蟹屬可口美青蟹的16S rRNA基因進行序列同源性比對分析, 以探討三疣梭子蟹與其他海產蟹類的分子系統發育關系。
1 材料與方法
1.1 材料
實驗所用紫色和茶綠色三疣梭子蟹購自連云港水產品市場, 時間為2007年10月至2008年3月。
1.2 樣品DNA的提取
取三疣梭子蟹附肢肌肉, 使用上海賽百盛基因技術有限公司生產的樹脂型TM基因組 DNA提取試劑盒提取基因組DNA, 經Gene QuantPro核酸定量儀(通用公司, 美國)測定基因組DNA濃度, -20 ℃保存備用。
1.3 PCR擴增及目的基因序列測定分析
三疣梭子蟹線粒體DNA COI和16S rRNA基因片段擴增所用特異性引物由上海生工生物工程技術服務有限公司合成。引物序列參見郭天慧等[2]的文獻,分別為 COI-H/COI-L(正向: 5’-taaacttcagggtgaccaaaaaatca-3’, 反 向 : 5’-ggtcaacaaatcataaagatattgg-3’)和16S-AR/16S-BR(正向: 5’-cgcctgtttatcaaaaacat-3’, 反向: 5’-ccggtctgaactcagatcacgt-3’)。
PCR 反應體系(25.0 μL)為: ddH2O 12.3 μL, 10×PCR 緩沖液 2.5 μL, 25 mmol/L MgCl22.0 μL, 10 μmol/L dNTPs 2.0 μL, 5 U/μLTaqDNA 聚合酶 0.2 μL,10 μmol/L 上下游引物各 2.0 μL, 500 μg/L DNA 模板2.0 μL。PCR擴增條件為: 94 ℃預變性5 min; 94 ℃變性45 s, 48 ℃退火1 min, 72 ℃延伸1 min, 循環30次; 最后72 ℃延伸5 min。PCR試劑均購自上海賽百盛基因技術有限公司。
PCR擴增所得目的DNA片段直接送至上海生工生物工程技術服務有限公司進行序列測定, 再通過DNAMAN(Version 6.0.40)軟件進行紫色和茶綠色梭子蟹線粒體DNA COI和16S rRNA基因片段序列比對分析。
1.4 16種海產蟹16S rRNA基因片段的序列分析及分子系統學探討
根據三疣梭子蟹的種屬特異性及序列分析結果,在GenBank數據庫中有選擇性地檢索出同屬于梭子蟹科(Portunidae)的、梭子蟹屬其他 14種梭子蟹(P.anceps;P. bahamensis;P. binoculus;P. depressifrons;P.floridanus;P. gibbesii;P. ordwayi;P. rufiremus;P. sayi;P. sebae;P. spinicarpus;P. spinimanus;P. ventralis;P.vossi)及青蟹屬可口美青蟹(C. sapidus)的 16S rRNA基因片段, 共15種海產蟹類型。利用DNAMAN軟件對三疣梭子蟹與這15種海產蟹的線粒體DNA 16S rRNA基因片段核苷酸序列進行結構分析, 并以可口美青蟹作為外類群進行分子系統分析。鄰接法(Neighbor-joining, NJ)分析使用Kimura 2-paramater,系統樹分支的置信度采用重復抽樣分析(Booststrap analysis)的方法, 重復抽樣的次數為1000次。
2 結果
2.1 線粒體DNA COI和16S rRNA基因片段PCR擴增結果
本實驗采用三疣梭子蟹基因組DNA為模板對其線粒體 COI和 16S rRNA基因部分片段分別進行PCR擴增及電泳檢測。結果表明, 兩個基因片段的PCR擴增產物均產生了清晰可辨的擴增條帶, 且二者擴增條帶分子量大小與參考文獻[2]相一致。
2.2 線粒體DNA COI和16S rRNA基因的序列分析
從COI和16S rRNA基因PCR擴增樣品中分別隨機選取4個紫色和4個茶綠色個體的擴增樣品進行測序分析(圖1和圖2)。經測定分析后得到了長度為658 bp的COI基因序列和長度為524 bp的16S rRNA基因序列。相關序列分析結果為: 紫色和茶綠色梭子蟹個體中COI基因(G+C)摩爾分數(DNA堿基比)分別為46.8%和46.7%; 兩類個體中16S rRNA基因(G+C)摩爾分數均為53.15%; 經DNAMAN軟件分析合并的COI和16S rRNA基因中(A+T)含量分別為63%(A=36.6%; C=16.4%; G=20.5%; T=26.4%)和69.7%(A=34.5%; C=12.4%; G=18.0%; T=35.2%), 這與三疣梭子蟹整個線粒體DNA序列中70.2%的(A+T)含量相近[7], 而且, COI和16S rRNA基因在紫色和茶綠色群體間的核苷酸序列一致性(sequence identity)分別達到了99.87%和99.88%。
2.3 三疣梭子蟹 16S rRNA基因與其他海產蟹的同源性比較
經 GenBank數據庫選擇性地檢索, 得到用于同源性比對分析的梭子蟹科共15種海產蟹16S rRNA基因片段, 相應分類和GenBank登錄號見表1。

圖1 紫色(P)和茶綠色(G)梭子蟹個體COI基因片段的序列比對Fig. 1 Multiple sequence alignment of COI gene fragments of tea-green (G) and purple (P) crab samples
由于選取的 16種海產蟹屬于同一科(梭子蟹科)內不同屬間的類型, 其中 15種為同一屬(梭子蟹屬)內不同種間的類別, 本文通過常用于系統發生比較的線粒體DNA 16S rRNA基因對16種海產蟹的核苷酸序列同源性進行分析比較。DNAMAN軟件進行序列同源性比對分析所得結果見表2。

圖2 紫色(P)和茶綠色(G)梭子蟹個體16S rRNA基因片段的序列比對Fig. 2 Multiple sequence alignment of 16S rRNA gene fragments of tea-green (G) and purple (P) crab samples

表1 用于16S rRNA基因比對分析的梭子蟹科15種海產蟹的分類學位置及GenBank登錄號Tab. 1 Classification of 15 species of Portunidae and the GenBank accession numbers for 16S rRNA analysis
16種海產蟹之間的16S rRNA基因核苷酸序列一致性(sequence identity)為 88.06%, 序列同源性(sequence homology)范圍為 76.2%~100%。梭子蟹P.anceps和P. rufiremus之間的16S rRNA基因核苷酸序列同源性最低, 為 76.2%; 梭子蟹P. bahamensis和P.depressifrons以及P. spinimanus和P. vossi之間的16S rRNA基因核苷酸序列同源性最高, 均達到100%。與其他15種海產蟹16S rRNA基因同源性比對分析結果表明, 三疣梭子蟹和其他不同種屬海產蟹間的核苷酸序列的同源性為81.7%~91.7%, 其中, 與賽氏梭子蟹(P.sayi)同源性最高, 達到91.7%; 與P. rufiremus的同源性最低, 為81.7%; 而與可口美青蟹(C. sapidus)的同源性僅次于賽氏梭子蟹, 達到87.7%。

表2 16種海產蟹線粒體DNA 16S rRNA基因核苷酸部分序列同源性比較Tab. 2 Comparison of sequence homology (%) among 16S rRNA genes from 16 species of Portunidae
2.4 16S rRNA基因在系統學分析中的應用
基于線粒體 DNA 16S rRNA基因核苷酸序列,以可口美青蟹為外群對16種海產蟹進行系統發育分析。通過應用鄰接法(NJ法)構建的的分子系統發育關系樹(圖 3), 枝長表示分歧度, 枝上的數值是 1000次重復抽樣檢驗的置信度值。聚類分析結果表明, 三疣梭子蟹首先與賽氏梭子蟹(P. sayi)聚為一類, 再與可口美青蟹聚在一起。另外, 梭子蟹P. bahamensis和P. depressifrons以及P. spinimanus和P. vossi之間的 16S rRNA基因核苷酸序列的高同源性在分子系統樹的聚類結果中也反映了這一點。由于針對梭子蟹種屬內的研究主要集中在三疣梭子蟹等重要經濟種類, 且天然地理分布阻隔也造成對梭子蟹屬內各種之間的分類學研究較為匱乏, 該聚類分析結果可為梭子蟹種屬分類學研究提供分子依據。
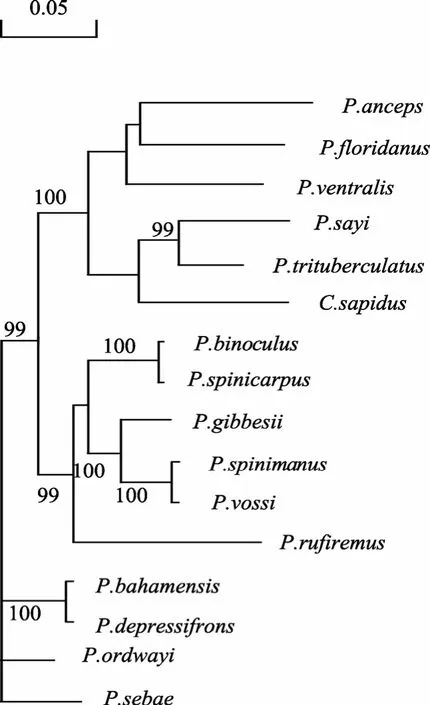
圖3 基于16種海產蟹線粒體DNA 16S rRNA基因核苷酸序列通過鄰接法構建的分子系統樹(NJ樹)Fig. 3 Molecular phylogenetic tree on the nucleotide sequence of 16S rRNA gene from 16 species of Portuni dae constructed by Neighbour-Joining method (NJ tree)
3 討論
3.1 線粒體保守序列基因分析在系統分類學中的應用探討
當前, 動物分子分類學研究成果多建立在線粒體DNA相關基因序列分析的基礎之上。線粒體中核糖體 RNA基因(rRNAs)和細胞色素 c氧化酶亞基 I基因(COI)的核苷酸序列比對, 是分子系統親緣分析行之有效的方法[9]。陳麗梅等[10]對 3種蟶類 16S rRNA和 COI基因片段序列進行了比較并對其系統學進行了初步研究, 發現COI基因的進化速率與16S rRNA基因相比要快, 16S rRNA基因則相對保守一些。徐琰等[11]通過運用16S rRNA基因序列初步探討了部分真蝦類的系統發育關系, 結果表明, 16S rRNA基因片段的序列很適合研究真蝦類屬間系統發育關系, 而在研究屬以上高級階元或種以下階元間的系統發生關系時較不敏感, 因其變異對于種級水平顯得過于保守, 而對于科級以上階元又顯得過快。蔣欽楊等[12]在 3個不同群體羅氏沼蝦(Macrobrachium rosenbergii)線粒體16S rRNA基因序列分析及遺傳差異比較中也指出, 線粒體16S rRNA基因是動物種內個體間較為保守的序列, 主要用于種以上水平的變異分析。在細菌分類學鑒定方面,16S rRNA基因序列分析是當前細菌分類中最精確的一種技術[13]。劉佳文等[14]指出通過分子生物學技術分析16S rRNA基因, 可將分枝桿菌(Mycobacterium)鑒定到種的水平。由于具有較強的中間解析和鑒別能力, 線粒體16S rRNA基因正廣泛應用于脊椎動物和無脊椎動物的系統與進化學及種類鑒別中[15]。
目的基因序列測定及比對分析可以直觀反映兩個群體之間的親緣關系。以細菌鑒定為例, 當兩DNA間同源性為 100%, 即可認為是源于同一個體;同源性為80%~90%則被認源于為同一種內或同一亞種; 同源性為 60%~70%被認為源于同一種內的不同亞種, 同源性為 20%~60%則被認為是源于同一屬的不同種(http://space.biox.cn/3776/viewspace-4586.html)。對不同個體或群體間DNA堿基比(G+C)摩爾分數的差異比較的分析方法已經廣泛應用于微生物的分類學的親緣關系鑒定的研究中。一般認為, (G+C) 摩爾分數相差超過5%就不可能屬于同一個種, 相差超過10%就可考慮是不同屬[16]。本實驗選取的線粒體DNA兩個保守基因的序列比對分析結果顯示了COI和16S rRNA在兩個群體中均接近100%的核苷酸序列一致性, 且紫色和茶綠色梭子蟹群體中 COI基因(G+C)摩爾分數差別為 0.1%; 16S rRNA基因(G+C)摩爾分數則完全相同, 說明紫色和茶綠色梭子蟹群體間親緣關系十分相近, 二者同屬于一個種, 紫色三疣梭子蟹并未發生亞種分化。
在 COI基因序列比對分析中, 紫色梭子蟹個體間僅發現單堿基差異, 序列歧異為轉換(G-A); 而茶綠色梭子蟹不同個體間有較多的(6個)堿基突變, 序列歧異基本上是轉換(T-C, G-A)多于顛換(G-C), 且其中有一處單堿基序列差異(G-A)與紫色梭子蟹個體相同, 這說明紫色梭子蟹群體的線粒體DNA序列在進化關系上較為保守(圖1); 在16S rRNA基因序列比對分析中, 紫色和茶綠色梭子蟹群體內不同個體間均只出現單堿基突變, 而且僅為單堿基 T的重復序列數目不同(圖2), 反映了16S rRNA基因高度保守的特性, 說明16S rRNA基因更適用于親緣關系較遠物種之間的分類學研究鑒定。
在利用線粒體DNA 16S rRNA基因對16種海產蟹的核苷酸序列同源性進行分析中, 三疣梭子蟹與可口美青蟹的同源性僅次于賽氏梭子蟹(91.7%), 達到87.7%, 且DNAMAN軟件分析表明三疣梭子蟹與可口美青蟹間的 16S rRNA基因核苷酸序列一致性達到 83.00%, 表明二者相近的親緣關系。以上結果表明, 海產蟹線粒體DNA 16S rRNA基因是相對保守的, 適合用于梭子蟹科內不同種間同源性比較及遺傳多樣性的分析。
鑒于本實驗所用線粒體DNA COI和16S rRNA基因序列均未檢測到可用于鑒定兩種體色三疣梭子蟹間的差異, 可進一步對線粒體DNA中核苷酸序列變化較大的區域(如, 線粒體 DNA 控制區[17,18]等)進行序列比較分析, 以得到基于堿基序列差異的不同體色三疣梭子蟹間的具體親緣關系。
3.2 三疣梭子蟹體色成因分析
體色在海洋蟹類的生活史中扮演重要的角色。首先它可以作為一種保護色, 與周圍生活環境融為一體, 有利于規避天敵和捕食獵物; 其次, 體色可以作為一種交流信號, 與其特定的生活生理特征相聯系; 再次, 體色的改變可能與其生活環境的變化有關, 如招潮蟹(Uca pugilator)便具有因環境溫度高低和光強的強弱而改變身體顏色的特性[19]。
自然界中, 同一物種內具有不同體色個體或群體的現象十分普遍, 針對體色遺傳變異等方面的研究也比較深入。鄭懷平等[20]根據貝殼顏色的差異對海灣扇貝(Argopecten irradians)家系的建立及其成長發育過程進行了觀察研究, 發現體色(殼色)作為一個可遺傳的質量性狀, 它與表型性狀的聯系可能與貝類的遺傳、生理特性以及生態環境等有關。在本研究所觀察到的三疣梭子蟹既然同屬于一個種, 那么其體色的變化很可能是由環境因素或者遺傳因素引起的。同時在實際觀察中了解到, 雌蟹和雄蟹中都有紫色和茶綠色個體的存在, 因此否定了雌蟹為紫色, 雄蟹為茶綠色的說法[4]。另外, 在養殖中雖然有個別紫色個體體色會褪色為接近茶綠色個體體色的現象, 但其與茶綠色體色仍存在著明顯的差異, 且同等條件下絕大多數紫色個體顏色不會發生明顯變化。因此, 基于以上分析, 可以認為三疣梭子蟹產生紫色和茶綠色兩種明顯體色差異的原因是遺傳和環境因素共同作用的結果, 產生體色差異的具體原因有待進一步地深入研究。
[1] Hamasaki K, Fukunaga K, Kitada S. Batch fecundity of the swimming crab Portunus trituberculatus (Brachyura: Portunidae)[J]. Aquaculture, 2006, 253: 59-365.
[2] 郭天慧, 孔曉瑜, 陳四清, 等. 三疣梭子蟹線粒體DNA 16S rRNA和COI基因片段序列的比較研究[J].中國海洋大學學報, 2004, 34(1): 22-28.
[3] 遲大利, 閻斌倫, 高煥, 等. 三疣梭子蟹分子生物學研究進展[J]. 水產養殖, 2008, 29(4):11-15.
[4] 蘇秀榕, 李太武, 歐陽芬, 等. 三疣梭子蟹營養成分的研究[J]. 營養學報, 1996, 18(3): 342-346.
[5] 孫先交. 三疣梭子蟹越冬、育苗及養殖[J]. 齊魯漁業,2001, 18(4): 18-20.
[6] Avise J C. Phylogeography[M]. London: Harvard Univesity Press, 2000. 447.
[7] Yamauchi M M, Miya M U, Nishida M. Complete mitochondrial DNA sequence of the swimming crab,Portunus trituberculatus (Crustacea: Decapoda:Brachyura)[J]. Gene, 2003, 311: 129-135.
[8] Place A R, Feng X J, Steven C R, et al. Genetic markers in blue crabs (Callinectes sapidus) II: Complete mitochondrial genome sequence and characterization of genetic variation[J]. Journal of Experimental Marine Biology and Ecology, 2005, 319: 15-27.
[9] Bandyopadhyay P K, Stevenson B J, Cady M T, et al.Complete mitochondrial DNA sequence of a Conoidean gastropod, Lophiotoma (Xenuroturris) cerithiformis:Gene order and gastropod phylogeny[J]. Toxicon, 2006,48: 29-43.
[10] 陳麗梅, 孔曉瑜, 喻子牛, 等. 3種蟶類線粒體 16S rRNA和COI基因片段的序列比較及其系統學初步研究[J]. 海洋科學, 2005, 29(8): 27-32.
[11] 徐琰, 宋林生, 李新正. 用 16S rDNA序列初步探討部分真蝦類的系統發育關系[J]. 海洋科學, 2005,29(9): 36-41.
[12] 蔣欽楊, 楊學明, 郭亞芬, 等. 3個不同群體羅氏沼蝦線粒體16S rRNA基因序列分析及遺傳差異[J]. 水產科學, 2005, 24(10): 28-31.
[13] 郭亞輝, 郭堅華, 李斌. 根據 16S rRNA序列對假單胞菌屬分類學的研究進展[J]. 微生物學雜志, 2004,24(2): 38-41.
[14] 劉佳文, 呂紅艷, 吳雪瓊, 等. 16S rRNA基因序列在分枝桿菌分類鑒定的研究進展[J]. 國外醫學臨床生物化學與檢驗學分冊, 2001, 22(5): 265-268.
[15] 孔曉瑜, 姜艷艷, 相建海, 等. 魁蚶線粒體16S rRNA和 COI基因片段序列測定及其應用前景[J]. 海洋科學, 2001, 25(12): 46-48.
[16] 張文治. 微生物學[M]. 北京: 高等教育出版社, 2005:p240.
[17] 朱世華, 鄭文娟, 鄒記興, 等. 科魚類線粒體DNA控制區結構及系統發育關系[J]. 動物學研究, 2007,28(6): 606-614.
[18] 郭學光, 郭喬楠. 線粒體 DNA 控制區研究進展[J].腫瘤防治雜志, 2005, 12(22): 1747-1750.
[19] Silbiger N, Munguia P. Carapace color change in Uca pugilator as a response to temperature[J]. Journal of Experimental Marine Biology and Ecology, 2008,335: 41-46.
[20] 鄭懷平, 張國范, 劉曉, 等. 不同貝殼顏色海灣扇貝(Argopecten irradians)家系的建立及生長發育研究[J].海洋與湖沼, 2003, 34(6): 632-639.
Received: Sep., 10, 2009
Key words:Portunus trituberculatus; boby-color, mitochondrial DNA; 16S rRNA; COI
Abstract:The blue swimming crabs, an important commercial species, with different body-colors were discovered along the coast of East China in recent years. Both purple and tea-green crabs have similar, even the same physical characteristics except for the carapace color. In order to identify the relationships between them,we amplified and compared two conserved mitochondrial gene fragments of color-different crabs through DNA sequencing analyses. The sequence alignment analysis of color-different crabs presented 99.87% and 99.88% nucleotide sequence identity for the COI and 16S rRNA genes, respectively, indicating that there was no species or subspecies differentiation between them. Based on the alignment of nucleotide sequences, a phylogenetic tree of 16S rRNA genes from 16 species of Portunidae was constructed. It may serve as a basis for the taxonomy of portunid crabs.
(本文編輯:張培新)
Comparison analysis between purple and tea-green individuals of Portunus trituberculatus using mitochondrial partial genes
CHI Da-li1,2, GAO Huan1, SHEN Song-dong2, YAN Bin-lun1
(1. Jiangsu Key Laboratory of Marine Biotechnology, Huaihai Institute of Technology, Lianyungang 222005,China; 2. Life Sciences College, Suzhou University, Suzhou 215123, China)
Q178.532
A
1000-3096(2010)11-0027-08
2009-09-10;
2009-11-12
“十一五”國家科技支撐計劃重大項目(2006BAD09A01)作者簡介: 遲大利(1983-), 男, 山東龍口人, 碩士研究生, 從事三疣梭子蟹體色相關分子標記研究, E-mail: charliedl@163.com; 通信作者: 閻斌倫, 教授, 從事蝦蟹類養殖工作, 電話: 0518-85895252, E-mail:
yanbinlun@yahoo.com.cn

