黑龍江省馬鈴薯晚疫病菌與采集年份和地理來源相關性分析
王佳楠 呂文河* 金光輝 白雅梅 李文霞
(1東北農業大學農學院,黑龍江哈爾濱 150030;2黑龍江八一農墾大學農學院,黑龍江大慶163319;3東北農業大學資源與環境學院,黑龍江哈爾濱 150030)
馬鈴薯(SolanuMtuberosuML.)是世界第四大糧食作物,同時也是蔬菜、飼料、加工及工業原料作物(蔣敏華 等,2006)。晚疫病是危害馬鈴薯生產最為嚴重的病害之一。晚疫病菌(Phytophthora infestans)通常以無性生殖方式繁殖,當存在A1和A2兩種交配型時,亦可通過有性的卵孢子進行繁殖(李汝剛 等,1997)。P. infestans具有8~10條染色體(Sansome &Brasier,1973),基因組全長約237 Mb(Tooley & Therrien,1987),GC含量約52 %。隨著生物信息學的快速發展,EST-SSR標記以其開發成本較低,高效迅速等優點在植物病原菌遺傳多樣性研究中具有巨大的潛力。本試驗在供試馬鈴薯晚疫病菌株均為 A1交配型的條件下,利用 21對EST-SSR引物分別對菌株與采集年份、菌株與地理來源的相關性進行了分析,以期為馬鈴薯抗晚疫病育種等工作提供科學的參考依據。
1 材料與方法
1.1 試驗材料
在晚疫病菌株與采集年份相關性分析研究中,選取 2005、2006、2008年同為采自黑龍江省哈爾濱市的17份菌株(均為A1交配型)作為試驗材料(表1);在晚疫病菌株與地理來源相關性分析研究中,選取同為 2006年采集,但分別來自黑龍江省加格達奇、哈爾濱、黑河、海倫、克山、青岡、訥河、綏化8個地點共計46份菌株(均為A1交配型)作為試驗材料(表2)。EST-SSR引物采用已發表的21對EST-SSR引物(王佳楠,2009)。本試驗于2009年在東北農業大學農學院馬鈴薯研究室完成。
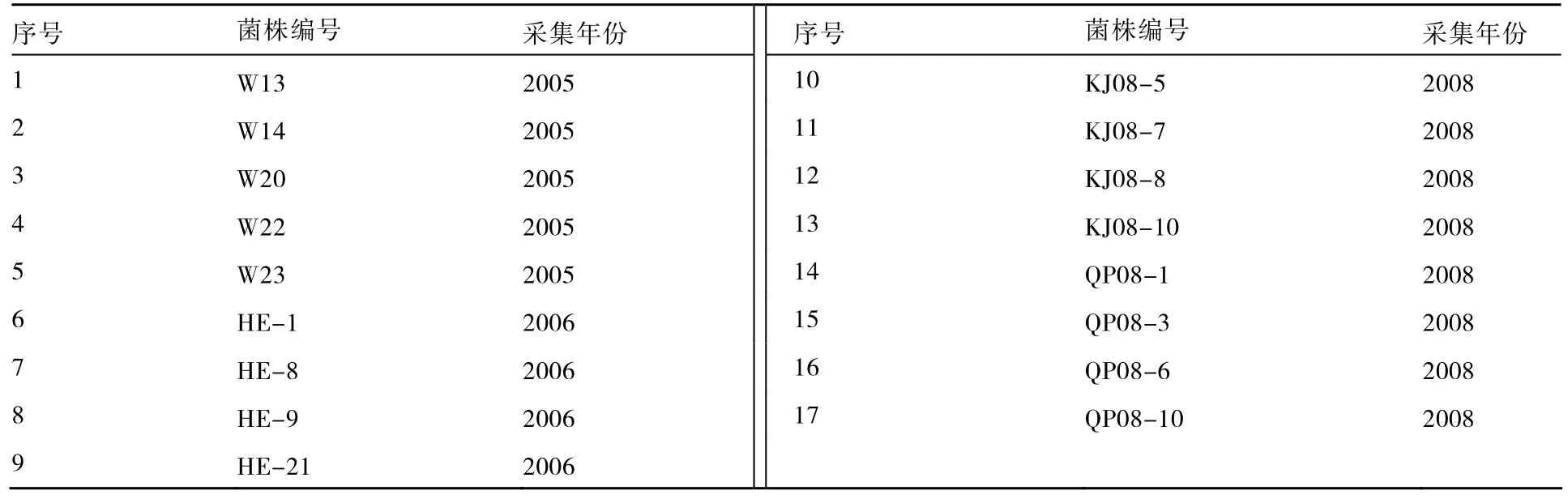
表1 2005、2006、2008年采自黑龍江省哈爾濱市的17份菌株
1.2 試驗方法
1.2.1 EST-SSR分析 采用Pipe等(2000)的方法提取致病疫霉基因組DNA。PCR反應體系:25 ng模板 DNA,0.5 mmol·L-1dNTPs,2 μL 10×Buffer(Mg2+free),1.75 mmol·L-1MgCl2,15 pmol引物,1.2 UTaqDNA聚合酶,加ddH2O至20 μL。PCR擴增程序:94 ℃預變性2 min;94 ℃變性30 s,63 ℃退火30 s,72 ℃延伸30 s,共35個循環;72 ℃延伸7 min;4 ℃保存。PCR產物用6 %變性聚丙烯酰胺凝膠電泳分離,在恒定60 W功率下電泳約1.5 h,銀染法染色。
1.2.2 數據處理 根據EST-SSR擴增產物,將有條帶的個體記為1,沒有的記為0,從而生成由1和0組成的原始矩陣。使用NTSYS-pc2.11a軟件(Rohlf,2000),對原始矩陣用SimQual程序求簡單匹配系數(simple matching coefficient),并獲得遺傳相似系數矩陣(similarity coefficient matrix),用SAHN程序中的UPGMA方法進行聚類分析,并通過Tree plot模塊生成聚類樹狀圖。將聚類結果轉換為協表征矩陣(cophenetic matrix),用Mxcomp程序對聚類結果和相似系數矩陣之間的相關性進行Mantel檢驗。

表2 2006年采自黑龍江省8個地點的46份菌株
2 結果與分析
2.1 晚疫病菌株與采集年份相關性分析
利用已發表的 29對引物進行多態性篩選(王佳楠,2009),引物全部能夠擴增出產物。選取能產生多態性且條帶清晰的21對引物(占72.4 %)分別對菌株與采集年份、菌株與地理來源的相關性進行分析。
采用相似系數(Sm)以UPGMA法生成聚類樹狀圖(圖 1)。此外,將聚類結果轉換為協表征矩陣,對協表征矩陣和相似系數矩陣的相關性進行Mantel檢驗,結果表明兩種矩陣顯著相關(r=0.746 05)。由圖 1可知,在相似系數為 0.87處聚類結果分為3組。其中,采于2005年的 3份菌株聚為第一類;而采于2005年的另2份菌株與采于 2006年的 4份菌株聚為第二類;采于2008年的8份菌株全部聚為第三類。據此,在相同地點采集的晚疫病菌株的聚類結果與采集年份之間具有一定的相關性。
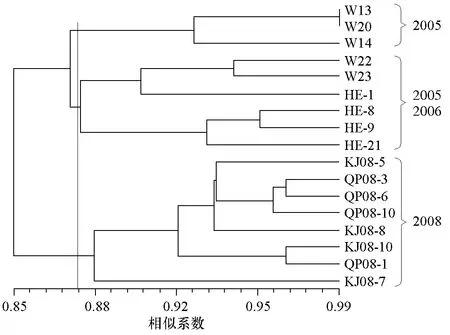
圖1 2005、2006、2008年采自黑龍江省哈爾濱市的17份菌株聚類樹狀圖
2.2 晚疫病菌株與地理來源相關性分析
采用相似系數(Sm)以UPGMA法生成聚類樹狀圖(圖2)。此外,將聚類結果轉換為協表征矩陣,對協表征矩陣和相似系數矩陣的相關性進行 Mantel檢驗,結果表明兩種矩陣顯著相關(r=0.859 66)。由圖2可知,在相似系數為0.89處,分類趨勢比較明顯。采自加格達奇、綏化、訥河的菌株在聚類圖上親緣關系較近,采自青崗除 QG06-5(1)外的菌株、采自哈爾濱除 HE-1外的菌株、采自海倫除HL06-113外的菌株在聚類圖上親緣關系也較近,但采自克山和黑河的菌株分散于各個類群,沒有表現出聚類趨勢,可知采自克山和黑河的菌株遺傳背景較為復雜。據此,在相同年份采集的晚疫病菌株的聚類結果與地理來源之間具有一定的相關性。
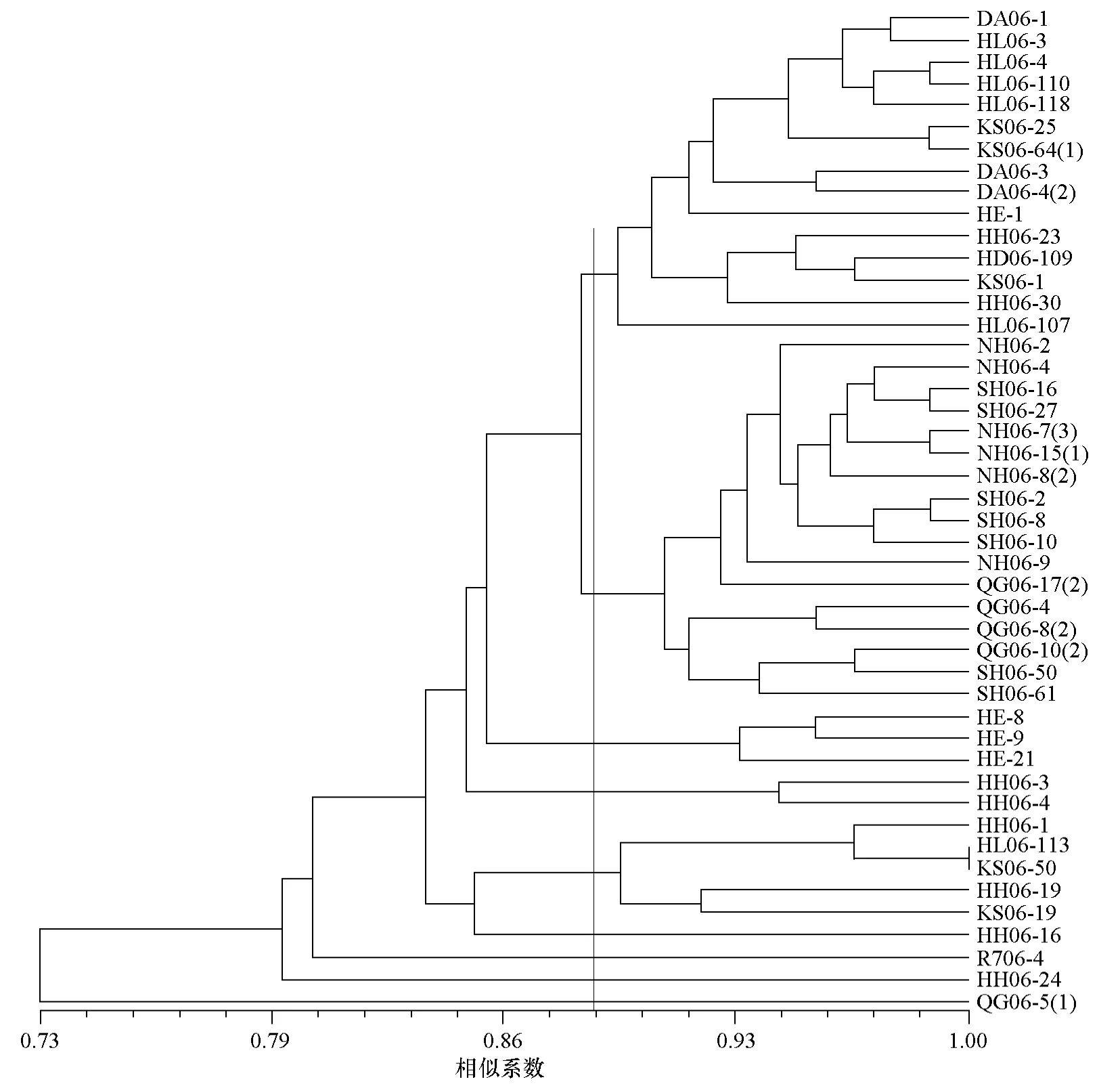
圖2 2006年采自黑龍江省8個地點46份菌株聚類樹狀圖
3 結論與討論
分子標記技術在生物多樣性分析研究中具有很大的優勢,Knapova和 Gisi(2002)的研究表明SSR標記適合針對晚疫病菌基因型多樣性的研究。同AFLP相比,SSR技術具有位點特異的優點,這樣更有利于分析馬鈴薯晚疫病菌的群體結構。本試驗是在本實驗室自行開發9對具有多態性產物的EST-SSR引物基礎上,結合國外12對具有多態性產物的SSR引物,首次報道了黑龍江省馬鈴薯晚疫病菌與采集年份和地理來源之間的關系。但客觀地講,由于受到SSR引物數量,特別是用于開發 EST-SSR引物所需的高質量晚疫病菌EST序列數量的限制,引物數量尚不充足,需要進一步拓展SSR引物的開發策略,以期進一步豐富SSR引物的數量。
在晚疫病菌遺傳多樣性與采集年份相關性研究方面,本試驗聚類結果表明,在相同地點采集的晚疫病菌株的聚類結果與采集年份有一定相關性。由分析結果可知,黑龍江省哈爾濱市 2008年的菌株群體結構與2005、2006年的菌株群體結構存在明顯不同,即2008年菌株的群體結構發生了顯著改變。其原因可能是2007年黑龍江省內罕見的干旱導致初侵染源發病情況發生變化,但具體原因尚須結合生理小種鑒定和抗藥性鑒定等群體表現型研究結果綜合考慮,作出更為全面的解釋。
在晚疫病菌遺傳多樣性與菌株地理來源相關性研究方面,采自克山和黑河的菌株分散于各個類群,沒有表現出聚類趨勢,這種情況可能與以下兩個原因有關:第一,與克山地區菌株采樣的具體地點有關。來自克山地區的菌株有一少部分采自黑龍江省農業科學院克山分院的馬鈴薯育種田中,由于育種田中種植了部分引種材料和無性系材料,這些材料可能對晚疫病菌株存在篩選作用。第二,來自克山的另一部分菌株與來自黑河的菌株采自當地的商品薯田,由于兩地種薯、商品薯的調運比較頻繁,也會對當地晚疫病菌群體的遺傳結構造成影響。因此,有理由認為,來自克山和黑河的菌株遺傳背景比其他6個地點的菌株遺傳背景復雜。本試驗結果與瑞士的Knapova和Gisi(2002)的報道有所不同,Knapova和Gisi(2002)的研究結果認為用AFLP、SSR等分子標記檢測的DNA多樣性與菌源的地理位置等無相關性,但Knapova和Gisi同時承認,雖然SSR分子標記能檢測出豐富的病菌群體結構多樣性,但是只有少數的SSR基因型占統治地位,這可能與病害流行時以某些生理小種為主有關,不同地區的氣候條件、主栽品種、耕作制度不同,病原菌與寄主在長期適應的過程中,適者生存,必然以某些類群為主,因此在試驗結果上可能表現不出地理來源相關性。
在今后進一步的研究中,隨著引物數量的不斷增加,可以進一步采集來源更廣泛,采集年份更多的菌株,通過分子標記技術進行聚類分析,從而更加準確地分析馬鈴薯晚疫病菌EST-SSR遺傳多樣性是否與菌株的采集年份和地理來源具有相關性。
蔣敏華,楊清,李麗,黃先群.2006.馬鈴薯抗晚疫病轉基因研究進展.種子,25(12):46-50.
李汝剛,伍寧豐,范云六.1997.馬鈴薯抗晚疫病研究進展.馬鈴薯雜志,11(4):243-250.
王佳楠.2009.黑龍江省馬鈴薯晚疫病菌群體遺傳多樣性EST-SSR分析〔碩士論文〕.哈爾濱:東北農業大學.
Knapova G,Gisi U.2002.Phenotypic and genotypic structure ofPhytophthora infestanspopulations on potato and tomato in France and Switzerland.Plant Pathology,51:641-653.
Pipe N D,Azcoitia V,Shaw D S.2000.Self-fertility inPhytophthora infestans:heterokaryons segregate several phenotypes.Mycological Research,104:676-680.
Rohlf P J.2000.NTSYSpc,Numerical Taxonomy and Multivariate Analysis System,Version 2.1.Exeter Software,Setauket,NY,USA.
Sansome E,Brasier C M.1973.Diploidy and chromosomal structural hybridity inPhytophthora infestans.Nature,241:344-345.
Tooley P W,Therrien C D.1987.Cytophotometric determination of the nuclear DNA content of 23 Mexican and 18 non-Mexican isolates ofPhytophthora infestans.Exp Mycol,11:19-26.

