冠菌素誘導橡膠樹次生乳管分化的形成層細胞CUT&Tag文庫構建和初步分析



關鍵詞:巴西橡膠樹;次生乳管分化;維管形成層;原生質體;組蛋白乙酰化;CUTamp;Tag
中圖分類號:S576;S59 文獻標志碼:A
2019年4月29日,美國西雅圖HENIKOFF博士在《Nature Communications》上首次發布Cleavage Under Targets and Tagmentation(CUTamp;Tag)技術[1]。CUTamp;Tag 技術是利用超高活性的新型pG-Tn5 轉座酶,在特異抗體引導下精準靶向切割目的蛋白附近的DNA 序列。在進行CUTamp;Tag 實驗時,首先進行靶蛋白特異性抗體(一抗)孵育,使抗體進入細胞與靶蛋白結合,為了放大信號,同理接著進行二抗孵育,最后孵育pAG-Tn5 轉座體,使得轉座體進入細胞并與抗體結合,這樣就把轉座體間接地固定在靶蛋白上,隨后加入Mg2+,激活Tn5 酶的切割活性,切斷靶蛋白結合的DNA 區域。由于Tn5 連有測序接頭,在打斷的同時直接在片段化的DNA 上加接頭,然后提取DNA,進行PCR 擴增構建文庫。在傳統的染色質免疫共沉淀(ChIP-Seq)試驗中,使用超聲波隨機打斷染色質,經過抗體免疫沉淀DNA,通常得到DNA 長度均一性差。然而,在CUTamp;Tag 中,轉座子只在接近蛋白質結合位點的附近切割染色質,從而使DNA 序列長度縮短[2]。因此即使較低的測序深度(3~5 M reads)也能得到高質量的數據。與傳統的ChIP-Seq 相比,CUTamp;Tag 技術具有細胞投入量低、信噪比高、可重復性好等優勢,尤其適用于早期胚胎發育、干細胞、腫瘤以及表觀遺傳學等研究領域[1-3]。目前,CUTamp;Tag 在組蛋白修飾方面的研究己經在動物細胞和植物中都有成功應用[3-4],甚至利用CUTamp;Tag 進行組蛋白修飾的研究己經發展到了單細胞水平[1, 3, 5]。然而,植物細胞存在細胞壁、大液泡和復雜的次生代謝產物,對抗體和pG-Tn5 轉座酶進入到植物細胞內有阻礙作用,CUTamp;Tag 技術應用于植物蛋白質修飾的研究較成熟,而針對植物的轉錄因子與染色質結合研究的CUTamp;Tag 試驗仍然是個挑戰,試驗成功案例還很少[6]。
天然橡膠與石油、煤炭、鋼鐵并稱四大工業原料,是國防和經濟建設的重要戰略物資。世界所需的天然橡膠,98%以上是來自巴西橡膠樹(Hevea brasiliensis Muell. Arg.)[7]。橡膠樹樹干樹皮中的次生乳管是天然橡膠合成和貯存的場所,由維管形成層紡錘狀原始細胞分化而來[8-9]。割膠生產上,通過切斷樹皮中的乳管,收集從乳管排出的膠乳,加工成天然橡膠。維管形成層細胞分化次生乳管后,會不斷向樹皮外側推移,乳管細胞要經過發生-幼嫩-成熟-衰老-死亡的過程。為維持橡膠樹樹皮產膠部位的乳管數量,需要維管形成層不斷分化出新的次生乳管進行補充。而這些次生乳管的數量與天然橡膠產量直接相關,次生乳管數量取決于維管形成層分化成次生乳管的頻率,即次生乳管分化能力,這是橡膠樹產量育種的主要指標[7]。前期研究中,我們團隊最早發現外施茉莉酸(jasmonic, JA)及其前體亞麻酸(LA)能誘導維管形成層細胞分化成次生乳管[9-11]。后續我們又發現使用活性形式的茉莉酸(JA-Ile)結構類似物冠菌素(coronatine, COR)[12],在誘導橡膠樹次生乳管分化的效應方面比茉莉酸甲酯(MeJA)更好[13],并建立了一種穩定的COR 誘導橡膠樹萌條次生乳管分化的實驗系統[14-19]。最近,我們發現組蛋白去乙酰化酶(HDACs)的抑制劑曲古抑菌素A(trichostatin A, TSA)也能夠高效誘導橡膠樹次生乳管分化[16]。使用COR 處理,能顯著提高維管形成層區的組蛋白乙酰化程度,而且HDA 活性和含量也受到影響。基于這些研究基礎,以及組蛋白乙酰化修飾作為基因轉錄調控的重要方式,推測JA 誘導橡膠樹次生乳管分化,可能是通過組蛋白乙酰化修飾調控橡膠樹次生乳管分化相關基因轉錄而實現。
本研究利用COR誘導橡膠樹萌條次生乳管分化的實驗系統,通過分離COR 誘導橡膠樹次生乳管分化的樹皮樣品,使用酶解法分離形成層區細胞的原生質體。采用CUTamp;Tag 技術,通過組蛋白H3 乙酰化抗體引導下精準靶向切割組蛋白乙酰化修飾區域附近的DNA 序列,進行CUTamp;Tag 試驗,構建cDNA 文庫并質檢和測序。研究結果為使用CUTamp;Tag 技術構建植物組織cDNA文庫提供操作方法,為解析組蛋白乙酰化修飾調控橡膠樹乳管分化的分子機制提供理論基礎。
1 材料與方法
1.1 材料
植物材料為巴西橡膠樹無性系熱研7-33-97的1 年生萌條,種植在中國熱帶農業科學院橡膠研究所的五隊增殖苗圃,這些萌條每年都經過鋸桿,并通過基部的潛伏芽重新生長新的萌條,并在1 年內生長5~6 個伸長單位(extension unit,EU)[9, 13]。
1.2 方法
1.2.1 冠菌素(COR)處理橡膠樹萌條 選取一年生橡膠樹萌條的第2 伸長單位(EU2),節間長且健壯的樹干作為實驗材料,在EU2 樹干中部,使用單面刀片刮去面積為2 cm× 4 cm 的莖表皮及部分皮層,用面積略大的滅菌無塵紙包裹處理部位,施加含有20 μmol/L COR 溶液,然后用塑料封口膜纏繞并密封包裹。處理時間為1 d。然后拆去塑料封口膜和無塵紙,剝取處理部位的樹皮(不帶部分木質部),并立即置于冰水中,帶回實驗室。COR處理橡膠樹萌條,取3 組生物學重復,每組生物學重復取自5 株橡膠樹萌條樹皮進行混合。選取與COR 處理相同狀態的橡膠樹萌條,不做任何處理。在COR 處理剝取樹皮樣品的同時,剝取未處理的萌條樹皮(不帶部分木質部),即為對照處理(CK)膠樹萌條。對CK 的樹皮樣品,同樣取3 組生物學重復,每組生物學重復取自5株橡膠樹萌條樹皮進行混合。
1.2.2 橡膠樹萌條樹皮形成層細胞的原生質體制備 將取回的COR 和對照處理橡膠樹萌條的樹皮樣品,立即置于一次性塑料培養皿內,分割成約0.5 cm×2cm的小塊,并立即置于冰水中,充分洗滌,洗掉膠乳和細小的雜質。再通過酶解法從新鮮樹皮中制備原生質體,參考擬南芥和楊樹[20-21]的實驗方案加以改進,分離橡膠樹萌條樹皮形成層區域的原生質體。針對橡膠樹樹皮中存在大量膠乳,相應地增加洗滌步驟和樹皮材料酶解時間。將得到的原生質體懸浮液添加到含有0.01 mg/mL雙醋酸熒光素(fluorescein diacrtate, FDA)和2.5 μg/mL 碘化錠(propidium iodide, PI)緩沖液中,使用FDA 和PI 雙重染色法測定原生質質體活力,并使用血細胞計數板進行計數。
1.2.3 CUTamp;Tag 實驗 使用南京諾唯贊公司的Hyperactive Universal CUTamp;Tag Assay Kit for Illumina試劑盒進行cDNA 文庫構建,針對本研究的植物細胞原生質體,對實驗步驟進行優化,詳情如下:
(1)緩沖液(Buffer)配制。①Binding Buffer:取30 μL 10×Binding Buffer,加無酶水至300 μL,混勻。②Wash Buffer:取150 μL 10×Wash Buffer,加入30 μL 50×蛋白酶抑制劑,加入1320 μL 無酶水,混勻。③50×蛋白酶抑制劑:取1 片蛋白酶抑制劑混合片劑(Roche, EDTA-free Protease InhibitorCocktai, 11873580001)溶于1 mL 無酶水中,–20 ℃保存。④Antibody Buffer:取50 μLAntibody Buffer(-),加入0.5 μL 5% Digitonin,混勻后置于冰上預冷。⑤Dig-wash Buffer:取792 μL步驟②中配制的Wash Buffer,加入8 μL 5%Digitonin,混勻。⑥Dig-300 Buffer:取100 μL10×Dig-300 Buffer,加2 μL 5% Digitonin 和20 μL50×蛋白酶抑制劑,加入878 μL 無酶水,混勻。⑦試劑盒中的Buffer WA 和Buffer WB,在首次使用時,加入試劑盒說明書要求的無水乙醇。
(2)ConA Beads 處理。取1 支200 μL 的8連管,每個樣本加入100 μL Binding Buffer。使用移液槍吹打充分重懸ConA Beads,取出10 μLConA Beads 加到Binding Buffer 中,混合均勻,置于磁力架上,待溶液澄清后(約2 min),用移液槍吸棄上清。將8 連管從磁力架上取下,加入100 μL Binding Buffer,用移液槍輕輕吹打混勻(請勿振蕩混勻)。將8 連管置于磁力架上,待液體澄清后(約2 min),用移液槍吸盡上清。再加入10 μL Binding Buffer 重懸ConA Beads。
(3)細胞原生質體收集。在細胞原生質體通透之前的所有步驟都在室溫下進行,使細胞受到的應力最小化。操作過程中,避免劇烈的渦旋振蕩,造成細胞原生質體破裂。在室溫條件下收集原生質體,并用血細胞計數板計數。重懸原生質體,取所需數量的原生質體(約150 000個)于1.5 mL EP 管中,室溫下2500 r/min(600×g)低速離心5 min,用移液槍吸盡上清。在室溫條件下,加入500 μL Wash Buffer 重懸細胞,2500 r/min(600×g)低速離心5 min,用移液槍吸盡上清。在每個樣本中,加入100 μL Wash Buffer 重懸原生質體。
(4)一抗孵育。在每個樣本中,加入50 μL預冷的Antibody Buffer 重懸細胞(細胞核)-磁珠復合物。使用Merck 公司的組蛋白H3 乙酰化修飾抗體( Anti-acetyl-Histone H3 Antibody,06-599)作為一抗,進行COR 誘導橡膠樹萌條次生乳管分化形成層區樣品和對照的CUTamp;Tag 文庫構建。CUTamp;Tag 文庫需要設置陽性對照和陰性對照, 其中陽性對照使用細胞內高表達的H3K27me3 抗體,陰性對照使用結合能力強的IgG抗體。參照抗體說明書推薦的免疫濃度,按照1∶50 的濃度,將抗體加入8 連管中,上下顛倒混勻。瞬時離心,收集液體于管底(切忌因離心時間過長,導致磁珠聚集在管底),將8 連管置于冰箱4 ℃,靜置孵育過夜。
( 5 ) 二抗孵育。二抗為羊抗兔CI , 用Dig-wash Buffer 按照一定的比例,預先稀釋好二抗(常規推薦使用1∶100 比例稀釋,二抗濃度為1∶50),每個樣本的二抗工作液用量為50 μL。從冰箱取出孵育過夜的8 連管,瞬時離心收集反應液,將8 連管置于磁力架上,待溶液澄清后(2 min),用移液槍吸棄上清。加入稀釋好的二抗工作液,上下顛倒數次,使抗體與細胞(細胞核)-磁珠復合物混合均勻,室溫下旋轉孵育1 h,由于溶液較少,旋轉孵育期間每15 min 取下八連管,輕彈混勻1 次。二抗孵育完成后,將8 連管置于磁力架上,待溶液澄清后(2 min),用移液槍吸盡上清。向8 連管中加入200 μL Dig-washBuffer,上下顛倒數次,確保Buffer 與細胞(細胞核) - 磁珠復合物充分混合。重復上述的Dig-wash Buffer 洗滌步驟2 次。
( 6 ) pA/G-Tnp 轉座子孵育。取2 μLpA/G-Tnp 加入98 μL Dig-300 Buffer 混合,預先稀釋好pA/G-Tnp,使其終濃度為0.04 μmol/L,每個樣本pA/G-Tnp 轉座子工作液用量為100 μL。取Dig-wash Buffer 洗滌的8 連管,瞬時離心,將8 連管置于磁力架上,待溶液澄清后(2 min),用移液槍吸盡上清。每個樣本加入100 μL 稀釋的pA/G-Tnp 轉座子工作液,上下顛倒數次,使轉座子與細胞(細胞核)-磁珠復合物混合均勻。室溫下旋轉孵育1 h,由于溶液較少,旋轉孵育期間每10 min 取下8 連管,輕彈混勻1 次。pA/G-Tnp 轉座子孵育完成后,瞬時離心,將8 連管置于磁力架上,待液體澄清后(30~120 s),用移液槍吸盡上清。向8 連管中加入200 μL Dig-300 Buffer,上下顛倒數次,確保Buffer 與原生質體-磁珠復合物充分混合均勻。重復上述的Dig-wash Buffer 洗滌步驟3 次。
(7)DNA 片段化。取40 μL Dig-300 Buffer,加入10 μL 5×TTBL,混合均勻,預先稀釋TTBL,每個樣本TTBL 工作液用量為50 μL。取下pA/G-Tnp 轉座子孵育的8 連管,瞬時離心,將8連管置于磁力架上,待液體澄清后(2 min),用移液槍吸盡上清。向每個樣本中加入50 μL 稀釋的TTBL 工作液,混合均勻。將8 連管置于PCR儀中,37 ℃孵育1 h(可不設置熱蓋,使PCR 儀保持開蓋狀態進行PCR 反應,期間每20 min 取下8 連管,輕彈混勻1 次)。
(8)DNA 提取。向37 ℃孵育完成的每個DNA 片段化樣本中,加入5 μL Proteinase K、100 μL Buffer L/B 和20 μL DNA Extract Beads,充分渦旋混勻。將8 連管置于PCR 儀中,55 ℃孵育10 min,其間顛倒混勻2~3 次。瞬時離心,將8 連管置于磁力架上,靜置約2~3 min,小心移除上清(切勿吸走磁珠,用10 μL 槍頭盡量將上清吸除干凈)。將上述樣本取下磁力架,每個樣本中加入200 μL Buffer WA(使用前要加入無水乙醇),充分渦旋混勻。瞬時離心收集反應液,將8 連管置于磁力架上,靜置2 min,用移液槍吸盡上清。重復上述的Buffer WB 洗滌步驟2 次。開蓋室溫晾置約10 min,直至管內無液體殘留,磁珠表面無反光(為保證DNA 純度,Buffer WB 要充分晾干揮發乙醇,但不宜過度干燥而導致磁珠龜裂)。將上述樣本取下磁力架,加入22 μL 滅菌超純水,用移液槍吹打混勻,室溫洗脫5 min,其間輕輕振蕩2~3 次。將8 連管置于磁力架上,待溶液澄清后(約1 min),吸取20 μL 上清至新的8 連管中,得到提取的DNA 樣本。該DNA 樣本即為CUTamp;Tag 文庫模版,可置于–20 ℃長期保存,避免反復凍融。
(9)文庫PCR 擴增。在PCR 管中配制文庫PCR 擴增的反應體系:純化后的片段化DNA15 μL,2×CAM 25 μL,N5XX 5 μL,N7XX 5 μL,總體積50 μL,使用移液槍輕輕吹打混勻。在PCR儀中,進行如下反應:72 ℃鏈置換反應3 min;95 ℃預變性3 min;98 ℃變性10 s,60 ℃退火延伸7 s,共12 個循環;72 ℃ DNA 合成1 min;4 ℃終止反應。
(10)PCR 產物純化。使用磁珠法對PCR 產物進行純化,將VAHTS DNA Clean Beads(Vazyme# N411)回溫并渦旋振蕩混勻,緩慢吸取100 μL加到文庫的PCR 反應產物中,使用移液槍輕輕吹打10 次,保證整個體系均勻,室溫孵育5 min。將反應管瞬時離心并置于磁力架上分離磁珠和液體,待溶液澄清后(約5 min),小心用移液槍吸盡上清,注意不要擾動到磁珠。保持PCR 管始終在磁力架上,加入400 μL 新鮮配制的80%乙醇漂洗磁珠,室溫孵育30 s,用移液槍吸盡上清。重復乙醇漂洗步驟2 次。保持PCR 管始終處于磁力架上,開蓋空氣干燥5 min。磁珠晾干后,將PCR管從磁力架上取出,加入22 μL 滅菌超純水洗脫,使用移液槍輕輕吹打10 次充分混勻磁珠,室溫孵育5 min。將PCR 管瞬時離心,置于磁力架上分離磁珠和液體,待溶液澄清后(約5 min),小心吸取20 μL 上清轉移到新的EP 管中,該純化的PCR 產物樣本即為CUTamp;Tag 文庫,置于–20 ℃長期保存,避免反復凍融。
(11)文庫質量檢測和長度分布檢測。對構建好的CUTamp;Tag 文庫,通過濃度檢測和長度分布檢測進行質量評價。使用Agilent FragmentAnalyzer 5400 全自動毛細管電泳系統進行文庫質量和長度分布檢測。
1.2.4 CUTamp;Tag 文庫測序和分析 使用Illminanovaseq 6000 對構建的CUTamp;Tag 文庫進行測序。首先對Illmina 測序獲得的CUTamp;Tag 文庫原始數據,采用剪切方式截去測序數據的測序接頭和低質量片段,去除低質量的數據和保證clean reads的質量,具體步驟如下:①刪掉質量值小于15 的堿基超過該reads 堿基數的40%的reads;②截去堿基為N 的超過6 個的reads,③去除帶接頭的reads;④舍棄修剪后短于50 bp 的reads。得到的clean reads 將通過以下的生物信息學軟件進行分析,分別使用BWA 軟件進行Mapping 分析,使用DeepTools 軟件進行樣本間相關性分析,使用MACS2 軟件進行peak calling 分析,使用HomerfindMotifsGenome.pl 進行motif 識別,使用Goseq、TopGO 和Bioconductor(2.13)軟件進行GO 富集分析,使用KOBAS 軟件進行KEGG 富集分析。
2 結果與分析
2.1 COR 誘導橡膠樹萌條次生乳管分化和樹皮形成層細胞的原生質體分離
使用COR 誘導橡膠樹萌條次生乳管分化實驗系統,自然狀態下橡膠樹EU2樹皮的橫切面見圖1A。在20 μmol/L COR誘導處理7 d 的橡膠樹樹皮中可觀察COR誘導產生的次生乳管(圖1B)。采集COR 誘導處理1 d 的橡膠樹萌條樹皮樣品,使用酶解法分離橡膠樹萌條樹皮形成層細胞的原生質體。使用徒手切片的方法對酶解前后的樹皮進行橫切,可以通過光學顯微鏡快速地觀察到樹皮的結構變化。結果發現酶解前的橡膠樹樹皮從外到內分別為表皮、皮層、初生韌皮纖維、初生韌皮部、次生韌皮部和形成層區,各部分組織都很完整,且形成層區約有7~11 層形成層細胞(黑色三角所示),無其他雜質(圖1C);而酶解后的橡膠樹樹皮,表皮、皮層、初生韌皮纖維、初生韌皮部和次生韌皮部等組織都很完整,而形成層區細胞已經完全酶解(空心三角所示)(圖1D)。對酶解法獲得的樹皮形成層區細胞原生質體(圖1E)進行FDA-PI 染色和熒光顯微鏡鏡檢,發現在原生質體懸濁液中,被PI 染色呈紅色的死細胞和雜質較少(圖1F),而被FDA 染色發綠色熒光的具有活性的原生質體數量較多(圖1G);再使用血細胞計數板計數法對具有活性的原生質體進行計數,發現COR 處理得到具有活性原生質體數量約為3750 個/μL,總數約為3.75×105 個,CK樣品中具有活性原生質體數量約為3850個/μL,總數約為3.85×105 個。可見使用酶解法獲得橡膠樹樹皮形成層區的有活性的原生質體,質量較好、數量較多,能滿足后續CUTamp;Tag 文庫構建的要求。
2.2 CUTamp;Tag文庫構建和質量檢測
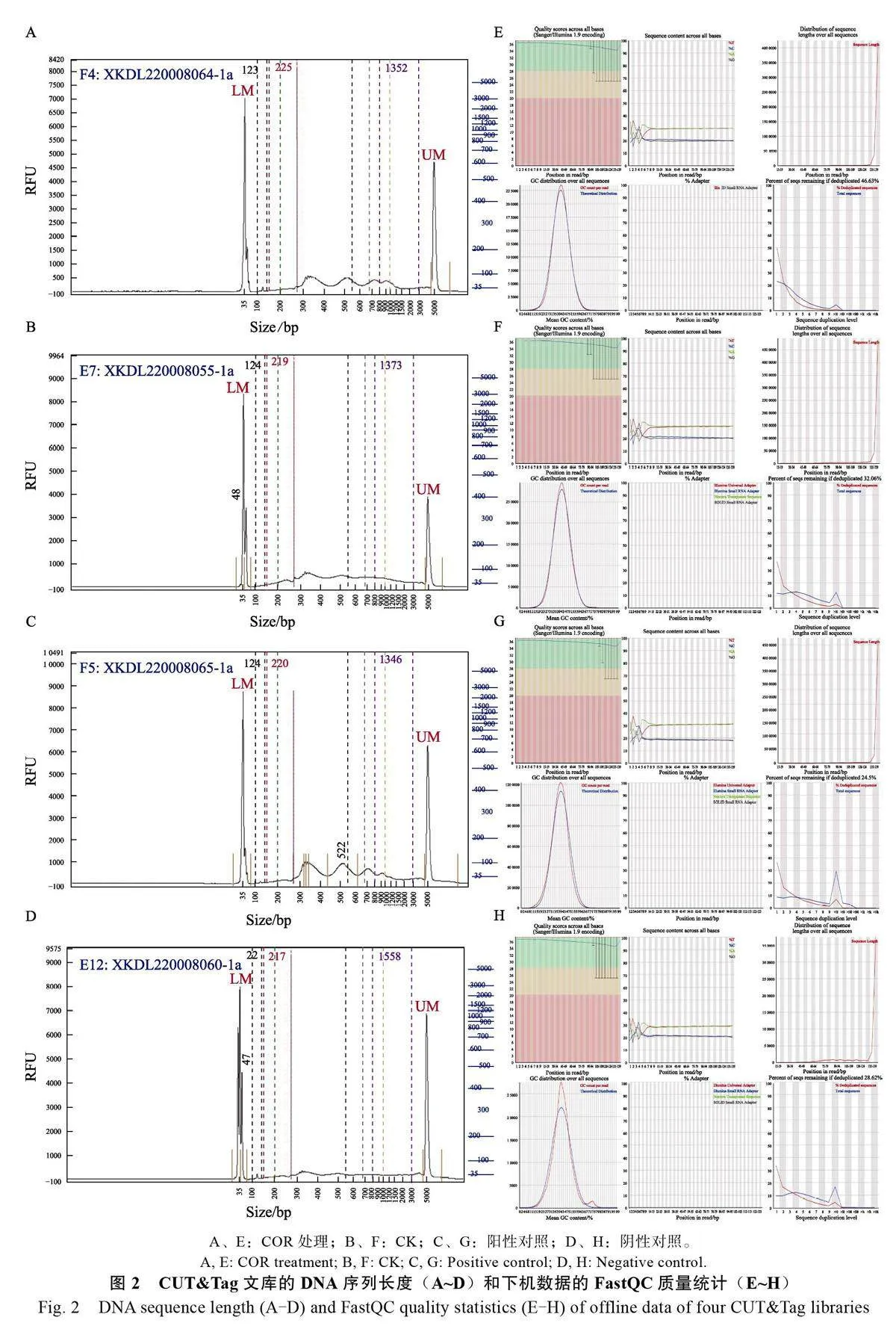
對酶解法獲得的原生質體進行重懸,按照原生質體的濃度吸取約150 000 個原生質體,分別使用組蛋白H3 乙酰化修飾抗體(COR 處理和CK)、H3K27me3 抗體(陽性對照)和IgG 抗體(陰性對照),構建COR 處理組、CK、陽性對照和陰性對照的CUTamp;Tag 文庫。通過文庫質量檢測,發現獲得的各CUTamp;Tag 文庫的DNA 片段長度大致在250~1000 bp(圖2A~圖2D),符合CUTamp;Tag 文庫要求。通過Illumina 測序獲得CUTamp;Tag 文庫的數據信息,使用FastQC 軟件對測序數據的質量進行評估,數據質量較好(圖2E~圖2H)。
2.3 CUTamp;Tag 文庫的數據質控和參考基因組比對
通過對Illumina 測序獲得的CUTamp;Tag 文庫的數據進行過濾,采用剪切方式截去測序數據的測序接頭和低質量片段,去除低質量的數據,保證clean reads 的質量。修剪之后剩余reads 長度足夠長,依然可以用于后續生物信息學分析,從而較高效地利用測序數據。
獲得的COR 處理、CK、陽性對照和陰性對照4 個CUTamp;Tag 文庫:clean reads 數量分別為4 782 119、5 926 919、24 499 257 和512 401,除了陰性對照的數據量較少以外,其他3 個文庫的clean reads 數據量均較好。堿基G 和C 的數量總和占總的堿基數量的百分比分別為40.72%、41.17%、37.86%和42.68%,均在正常水平。判斷文庫質量的關鍵指標, 即Q20 clean 分別為96.13%、96.20%、96.57%和96.39%,Q30 clean分別為89.73%、89.95%、90.56%和90.44%,均屬于同類測序數據的較高水平(表1)。
利用Burrows Wheeler Aligner (BWA)對4 個CUTamp;Tag 數據與巴西橡膠樹高產無性系RY8-79參考基因組[22]的比對分析。COR 處理、CK、陽性對照和陰性對照4 個CUTamp;Tag 文庫的序列比對上RY8-79 參考基因組的reads 數量和百分比,分別為4 645 819(97.15%)、5 698 271(96.14%)、23 073 516(94.18%)和340 394(66.43%),除了陰性對照比對上數據量較少以外,其他3 個文庫的比對上的reads 數量和百分比均較高。在CUTamp;Tag 的分析中,唯一比對且非重復比對的reads 數量是關注的重點,4 個文庫數據uniquemapped dedup 分別為2 224 053(46.51%)、1 686302(28.45%)、6707253(27.38%)和84 869(16.56%),此項指標屬于較好水平(表2)。
2.4 reads 在基因組上的分布統計
對4 個CUTamp;Tag 文庫的Total mapped reads數據比對到巴西橡膠樹高產無性系RY8-79 參考基因組的各個染色體的密度進行統計。正常情況下,整個染色體長度越長,該染色體內部定位的reads 總數越多。從定位到染色體上的reads 數與染色體長度的關系圖中,可以更加直觀看出染色體長度和reads 總數的關系。如圖3 所示,陰性對照比對上基因組各染色體的reads 數量較少(圖3D),COR 處理、CK 和陽性對照3 個文庫數據比對上基因組各染色體的reads 數量較多,且三者之間的豐富程度相差不大(圖3A~圖3C)。可見本次構建的COR 處理和CK 的CUTamp;Tag 的數據質量較好。
利用deepTools 軟件的computeMatrix 模塊對轉錄起始位點(TSS)上下游3 kb 區域的CUTamp;Tag信號進行統計,是將整個區域按照50 bp 的窗口大小劃分成bin,然后計算每個bin 中的平均信號強度,可以獲得每個樣本的TSS 上游和下游3 kb區域上的平均信號分布(圖4A~圖4D),COR 處理、CK 和陽性對照的結果在TSS 處均有顯著的高峰,而且TSS 上游和下游的曲線很均勻;雖然陰性對照在TSS 處也有顯著的高峰,但TSS 上游和下游的曲線比較雜亂。同理,利用deepTools軟件的plotProfile 模塊對gene body 上下游3 kb區域的CUTamp;Tag 信號進行統計,得到每個樣本的gene body 上下游3 kb 區域上的平均信號分布(圖4E~圖4H),COR 處理、CK 和陽性對照的結果在TSS和TES處均有顯著的高峰,TES-TSS連接的曲線很均勻,顯著高于TSS 和TES 之外的區域;雖然陰性對照在TSS 和TES 處也有顯著的高峰,但TES-TSS 連接的曲線比較雜亂,也是顯著高于TSS 和TES 之外的區域曲線。同理,利用deepTools 軟件的computeMatrix 模塊對peak 峰頂上下游3 kb 區域的CUTamp;Tag 信號進行統計,得到每個樣本CUTamp;Tag 信號貫穿基因組的TSS 兩側的分布圖(圖4I~圖4L),橫坐標表示reads 相對于peak summit 的位置信息,縱坐標表示每一個區域的reads 信號的聚類結果,可以看出COR處理、CK 和陽性對照的3 個文庫的reads 集中在peak summit 的位置,但漂移出peak summit 的也較多,且上下游的富集reads 信號較少。從以上結果可知,引起陰性對照曲線雜亂的原因可能是陰性對照的數據量少、比對的結果少造成的。
2.5 差異基因的GO和KEGG分析
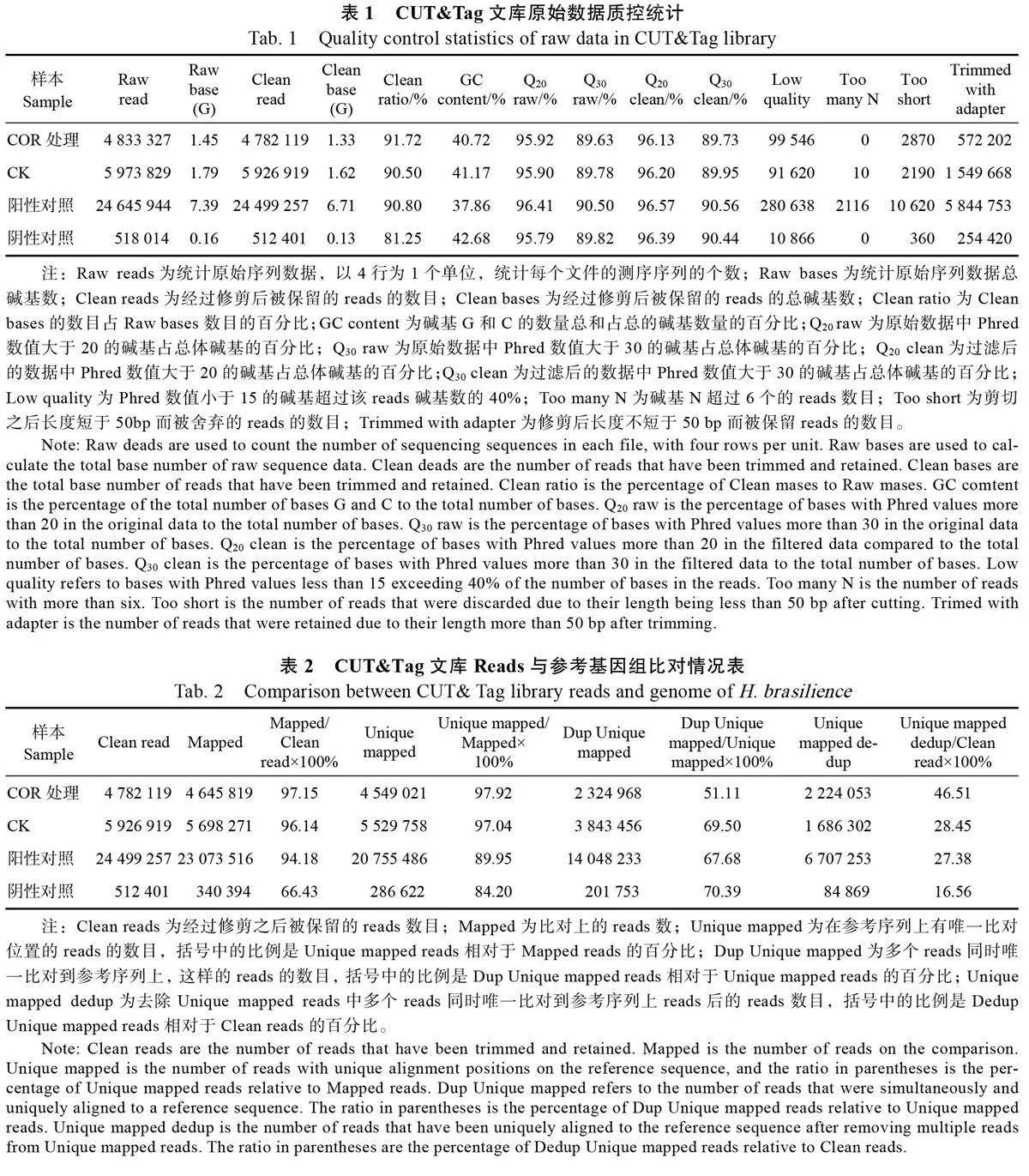
峰最鄰近的TSS 所對應的基因被認為是峰相關基因。使用Goseq 軟件對各CUTamp;Tag 文庫的峰相關基因進行GO 富集分析,根據GO 富集的生物過程(biological process)、細胞組分(cellularcomponent)和分子功能(molecular function)3個類別,對過度呈現P 值(over represented Pvalue)最小的30 項中具有峰值項的基因數量(gene with peak item)最多的幾項進行篩選。如圖5A~圖5D 所示:(1)COR 處理的CUTamp;Tag文庫差異峰對應基因,在GO 富集的3 個類別中,分別是生物過程6 項,其中DNA 整合相關基因220 個;細胞組分12 項,其中泛素化連接酶復合體相關基因130 個、泛素化-蛋白轉移酶相關基因128 個;分子功能12 項,其中泛素化連接酶復合體相關基因178 個、泛素化-蛋白轉移酶相關基因178 個和ADP 結合相關基因166 個等。(2)CK的CUTamp;Tag 文庫差異峰對應基因,在GO 富集的3 個類別中,分別是生物過程2 項,其中對生長素的反應相關基因33個;細胞組分12 項,其中泛素化連接酶復合體相關基因117 個和核泛素連接酶復合物相關基因115個;分子功能16 項,其中ADP 結合相關基因170 個、泛素蛋白轉移酶活性相關基因165個和泛素樣蛋白轉移酶活性相關基因165 個等。(3)組蛋白H3K27m3 抗體對應的陽性對照的CUTamp;Tag 文庫差異峰對應基因,在GO 富集的3 個類別中,分別是生物過程10 項,其中對蛋白質磷酸化相關基因338 個和DNA 整合相關基因144 個;細胞組分2 項,其中細胞外基質相關基因18 個;分子功能15 項,其中氧化還原酶活性相關基因165 個和鐵離子結合相關基因155 個等。(4)陰性對照的CUTamp;Tag 文庫差異峰對應基因,在GO 富集的3 個類別中,分別是生物過程14 項,其中光合作用相關基因13 個;細胞組分7 項,其中含蛋白質復合物相關基因25個和膜蛋白復合體相關基因13 個;分子功能9項,其中葉綠素結合相關基因3 個和脫氧核糖核酸酶活性相關基因3 個等。可見COR 處理、CK 與陽性和陰性對照文庫的GO 富集結果之間存在巨大差異。
使用KOBAS 軟件進行峰差異基因KEGG 富集分析,對P 值和校正后的P 值的最小20 項中,具有輸入的基因數量(input gene number)較多的幾項進行篩選。如圖5E~圖5H 所示,(1)COR處理的CUTamp;Tag 文庫的差異峰對應的基因校正P 值最低且富集數量較多的KEGG 通路,依次為植物病原體相互作用(P=0.1288,110 個基因),類黃酮生物合成(P=0.1631,35 個基因)、光合作用(P=0.0553,36 個基因)、氨基糖和核苷酸糖代謝(P=0.6543,71 個基因)、核糖體(P=0.6543,156 個基因)等;(2)COR 處理對照的CUTamp;Tag文庫的差異峰對應的基因校正P 值最低且富集數量較多的KEGG 通路,依次為植物病原體相互作用(P=0.0553,107 個基因),類黃酮生物合成(P=0.1631,36 個基因)、光合作用(P=0.12,44個基因)、晝夜節律-植物(P=0.3826,28 個基因)、核糖體(P=0.3826,156 個基因)等;(3)組蛋白H3K27m3 抗體對應的陽性對照的CUTamp;Tag 文庫的差異峰對應的基因校正P 值最低且富集數量較多的KEGG 通路, 依次為類黃酮生物合成(P=7.12E-06,33 個基因),植物病原體相互作用(P=0.0067,66 個基因)、次生代謝產物的生物合成(P=0.0067,297 個基因)、倍半萜和三萜生物合成(P=0.0069,16 個基因)、光合作用(P=0.0126,29 個基因)等;(4)陰性對照的CUTamp;Tag 文庫的差異峰對應的基因校正P 值最低且富集數量較多的KEGG 通路,依次為光合作用(P=1.68E-14,15 個基因),氧化磷酸化(P=2.8E-06,11 個基因)、萜、哌啶和吡啶生物堿的生物合成(P=0.0219,3個基因)、RNA 聚合酶(P=0.0459,3 個基因)、代謝途徑(P=0.1366,31個基因)等。可見COR處理、CK 與陽性對和陰性對照文庫的KEGG 富集結果之間也存在較大差異。
3討論
3.1 CUTamp;Tag 技術為解析橡膠樹維管形成層分化成次生乳管的分子機制提供了契機
橡膠樹樹干樹皮中的次生乳管是天然橡膠合成和貯存的場所,是由維管形成層紡錘狀原始細胞分化而來的[7, 9]。形成層細胞分化次生乳管后,會不斷向樹皮外側推移,乳管細胞要經過發生-幼嫩-成熟-衰老-死亡的過程。按照橡膠樹樹皮從里向外依次分布形成層區(乳管發生)、水囊皮(幼嫩乳管)、黃皮和砂皮內層(成熟乳管)以及砂皮外層和粗皮(衰老和死亡的乳管)[7]。由于天然橡膠生產中的割膠部位主要是形成層和水囊皮以外的組織——黃皮和砂皮內層,其中分布有大量的成熟乳管,故稱之為有效產膠部位。只有形成層不斷分化新的次生乳管,才能保障有效產膠部位中成熟乳管的數量[7]。橡膠樹樹干樹皮中的次生乳管細胞的命運決定發生在形成層區。最適合觀察此過程的時間為每年的春季新梢生長期(3—4 月),此時樹干的形成層區開始分裂分化活動,形成層的細胞層數最少(約4 層)[13, 23],可以在原位跟蹤到形成層分化成次生乳管的過程。所以研究COR 誘導橡膠樹維管形成層分化次生乳管的分子機制,其植物材料僅限于COR 誘導處理的橡膠樹樹皮的形成層區,形成層細胞且細胞位于木質部和韌皮部之間,難以分離到具有活性且數量較多的形成層細胞,也難以獲得足夠量的原生質體來滿足ChIP-Seq 實驗要求,故我們選擇起始量更低且效率更高的CUTamp;Tag 技術來研究COR 處理的橡膠樹樹皮形成層區的組蛋白乙酰化修飾對次生乳管分化的調控。目前,CUTamp;Tag技術已經廣泛應用于組蛋白修飾/染色質結合蛋白分析、開放染色質分析、單細胞組學、DNA 和蛋白質修飾分析、空間生物學和染色質構象捕獲(3C)等多種研究方向,并且適用于植物和動物的多種模式物種。橡膠樹維管形成層在分化成次生乳管細胞的特點,結合CUTamp;Tag 技術能夠達到單細胞級別的分析,能夠充分還原形成層細胞發生的特異性修飾和基因表達,為解析橡膠樹形成層分化次生乳管細胞的機制提供了良好的契機。
3.2 橡膠樹維管形成層分化次生乳管過程,可能受到JA 信號、組蛋白乙酰化和泛素化協同調控
通過對COR 處理橡膠樹樹皮CUTamp;Tag 文庫的峰相關基因進行GO 和KEGG 富集分析,發現很多生長素、類黃酮代謝相關的基因,COR 和對照處理的文庫還發現了大量的蛋白質泛素化修飾相關的基因,如泛素化連接酶復合體、泛素化-蛋白轉移酶。這說明COR 誘導橡膠樹次生乳管分化過程中,形成層發生了蛋白質乙酰化修飾,而且有蛋白質泛素化修飾參與其調控過程。
蛋白質乙酰化是在乙酰基轉移酶的作用下,在蛋白質賴氨酸殘基上添加乙酰基的過程,是細胞控制基因表達,蛋白質活性或生理過程的一種機制。組蛋白乙酰化多發生在核小體的組蛋白N端堿性氨基酸集中區的特定賴氨酸殘基上。組蛋白乙酰化水平是由組蛋白乙酰基轉移酶(HATs)和組蛋白去乙酰化酶(HDAs)共同決定。在細胞核內,組蛋白乙酰化與組蛋白去乙酰化過程處于動態平衡,進而調控基因的轉錄和表達。蛋白質泛素化,是蛋白質的賴氨酸殘基位點與泛素分子的羧基端相互結合的過程,可改變底物蛋白的穩定性、定位、活性和復合物形成,參與蛋白質降解過程。泛素化調節途徑共有3 類酶催化:泛素激活酶(E1)、泛素接合酶(E2)、泛素-蛋白質連接酶(E3)。與組蛋白乙酰化類似,組蛋白泛素化也是可逆轉的調控,組蛋白泛素化的動態平衡過程由2 個因素決定:分別是細胞內可以利用的游離泛素,以及組蛋白泛素化或去泛素化酶的活性。同樣是發生在蛋白質賴氨酸殘基位點上的蛋白質修飾,2 種修飾方式之間必然存在一些聯系。目前,關于乙酰化和泛素化二者的相互作用的研究還很少。最新的研究證明,泛素化和乙酰化能夠共同調控植物中蛋白質穩定性變化[24]。研究發現依賴于NatA 的NTA 損傷會導致擬南芥蛋白質組的整體不穩定,并發現了一個新的降解決定子(degron),該degron 標記了大多數非乙酰化的胞質蛋白并可通過泛素系統降解[24]。在人類疾病研究中,發現組蛋白泛素化與乙酰化協同作用可以實現染色質酶活性的精細調控[25]。甲基轉移酶Dot1 的活性可由H4K16(H4K16ac)特異性激活,H4K16ac 直接激活Dot1 的活性,H2B 的泛素化(H2BUb)進一步增強了這種效果,從而獲得了Dot1 的最佳催化速率。并通過冷凍電鏡結構對比分析和定點突變結合核小體的酶分析, 證實了H4K16ac 乙酰化和H2BUb 泛素化對Dot1 甲基化的變構刺激在H3K79 甲基化反應中起關鍵作用[25]。
而在JA信號途徑起主導作用的COR 誘導橡膠樹次生乳管分化過程中,不僅有組蛋白乙酰化修飾參與,也有E3 泛素連接酶參與。經典的JA信號途徑:茉莉酸異亮氨酸(JA-Ile)是植物激素茉莉酸的活性形式,植物對茉莉酸的應答依賴SCFCOI1 復合物[26-27],該復合物具有E3 泛素化連接酶活性[28]。關鍵的調控環節為COI-JAZ-MYC,其中JAZ 蛋白是茉莉酸信號途徑的負調控因子,通過抑制MYC 類轉錄因子對下游茉莉酸應答基因的轉錄激活活性,阻斷JA 信號傳導。當JA-Ile與其受體COI1 結合后,SCFCOI1 復合物與JAZ 蛋白結合,使JAZ 蛋白發生泛素化而被26 s 蛋白酶體降解,從而解除JAZ 對MYC 轉錄因子的抑制作用,啟動JA 下游響應基因的轉錄[29-30]。據報道,植物轉錄共激活因子復合體Mediator 的一個亞基MED25,可將COI1 引導至MYC2 靶啟動子上,并促進JAZ 蛋白通過泛素化,被26 s 蛋白酶體降解;此外,MED25 可與組蛋白乙酰基轉移酶1(HAC1)相互作用,此互作可選擇性調節MYC2靶啟動子H3K9 乙酰化,從而在JA 信號轉導中發揮重要作用[31-34]。HAC1 在MYC2 靶啟動子上的富集與活性依賴于COI1 和MED25。因此,MED25將COI1 與HAC1 依賴的H3K9 乙酰化聯系起來,以激活MYC2 對JA 響應基因的轉錄調控[31-34]。由此可見,組蛋白乙酰化修飾和泛素化修飾在JA信號途徑中起很重要的調控作用。近期,我們發現HDA 的抑制劑TSA 也能誘導次生乳管分化[16],且COR 處理顯著提高形成層區的組蛋白乙酰化程度,且HDA 活性, 尤其是HDA6 的含量和HDA/HAT 基因的表達均受到JA 的影響(未發表資料)。據此,我們推測橡膠樹維管形成層分化次生乳管過程,可能是通過JA 信號途徑、組蛋白乙酰化修飾和蛋白質泛素化修飾來共同調控乳管分化相關基因的轉錄而實現。

